CT影像肝脏转移瘤分割与检测深度网络的研究
2021-01-22李佳昇郭树旭张惠茅邱云海李雪妍
李佳昇,郭树旭,张 磊,郑 爽,张惠茅,邱云海,李雪妍
1.吉林大学 电子科学与工程学院,长春130012
2.吉林大学第一医院 放射科,长春130021
约50%的结直肠癌患者会发生肝转移。治愈性肝切除术为患者的治愈和延长生存期提供了机会。采用保守治疗的患者5 年生存率约为5%。相比之下,那些进行治愈性肝切除的患者的5 年生存率约为40%[1-3]。是否进行治愈性肝切除,需要一个多学科决策系统来评估。临床上根据《实体瘤反应评估标准指南(RECIST 1.1)》来量化转移性肿瘤的程度[4]。评估内容包括肿瘤的数目、大小和在肝脏中的分布等。在肿瘤切除术之前或之后都要对患者进行胸部、腹部和骨盆CT检查[5]。随着精准医学综合治疗技术的发展,以往放射科医生对影像主观评价的局限性越来越凸显。机器学习技术在医学影像分析领域中的应用越来越广泛,特别是卷积神经网络(CNN)在医学影像分割、分类和检测等任务中的应用[6-9]。
Kim等人[10]提出了一种改进的Cycle GAN模型,其中多相位的U-Net作为生成器,用于分割肝脏和肝脏肿瘤。Sun 等人[11]设计了一种多通道全卷积神经网络(MC-FCN)模型,通过独立训练不同时期的对比增强CT 影像,并将高维特征组合在一起来分割肝脏肿瘤。Han[12]提出了一种32层的CNN模型,并利用长程连接操作和ResNet短程残差连接,将五张相邻的切片(尺寸为320×320)组成2.5 维的结构并作为输入,生成与中心切片相对应的分割图像。在文献[13]中,提出一种基于2 维CNN 的全自动肝脏CT 影像分割方法,该方法将ResNet模块作为短程连接,并进行基于形态学的后处理从而减少肿瘤的分类错误率。Vorontsov 等人[14]训练的深度网络由两个全卷积神经网络(FCN)网络模型级联而成,用于分割肝脏和肝脏肿瘤。Christ等人[15-16]应用级联U-Net 和三维条件随机场(3DCRF)作为后处理完成肝脏和肿瘤的分割。
本文提出了一种改进的级联网络框架,用于精准分割和检测腹部CT影像中的肝脏肿瘤。第一级U-Net完成肝脏轮廓分割,通过3D CRF完成精准分割后输入到形态学活动轮廓模块,根据肝脏范围完成固定大小框图的捕获。形态学活动轮廓获得较小尺寸的图像,输入由U-Net[17]和Mask R-CNN[18]组成的第二级联网络完成肝脏肿瘤的精准分割和检测。
1 改进级联网络模型方法设计
1.1 数据来源
本文实验数据是97名患者的腹部增强CT影像,来自吉林大学第一医院放射科。医院伦理委员会批准了这项研究。患者均经病理证实患有结直肠癌,影像数据均来自于治疗前。为了保护隐私,个人信息被清洗。CT 影像是以512×512 像素矩阵重建的DICOM 格式存储,体素的大小为0.775 mm×0.775 mm×5 mm。每个患者都有完整的平扫影像、增强动脉和静脉CT影像。
数据的金标准是由两名放射科医生在飞利浦的ISD(IntelliSpace Discovery)平台上手动标记的肝脏轮廓和肝脏肿瘤轮廓,如图1所示。一名放射科主任医生负责检查标注结果。利用Receiver Operating Characteristic Curve(ROC)来评估两位放射科医生勾画的一致性,如果ROC重合率高于80%,则认为勾画正确。

图1 金标准:医生勾画肝脏和肝脏肿瘤轮廓
1.2 数据扩充
数据扩充是提高深度学习网络鲁棒性的有效方法之一。通过数据扩充,可以有效地缓解过拟合问题,提高分割精度。实验数据随机被分成训练集、验证集和测试集(按7∶2∶1 的比例)。所有测试数据均未参与网络参数调整,只做测试评估。对所有数据集进行数据扩充。数据扩充方法以及数据量详见表1,表中所有的数据首先通过平移n 个像素,然后以图片中心为旋转点旋转m 度来扩充。通过仿射变换扩充数据集后,送入深度学习网络训练的一个批次内的切片,依然具有空间相似性。所以在网络训练之前需要随机调整数据的顺序。
1.3 级联网络模型设计
CNN 包括卷积层、池化层、激活函数层和全连接层。FCN通过将CNN的最后一个全连接层更改为卷积层来实现像素级的分类,即为每个像素分配一个标签。FCN 对输入图像进行端到端的预测,即通过一系列卷积、上采样和下采样的方法对输入图像进行处理,特征提取、组合和输出特征。U-Net 是一种改进的FCN 模型,通过对图像进行卷积来实现对每一层的特征进行提取;通过下采样特征图来减少特征参数,从而减少网络模型参数量;通过反卷积上采样特征图,以实现局部信息的融合,并将特征图的大小恢复为原始图像;通过将不同卷积层之间的特征融合在一起,以增强特征的多样性和鲁棒性,使得分割结果更加准确。U-Net在医学影像分割方面表现最佳。Mask R-CNN 在目标检测领域应用广泛。检测网络先生成并筛选位置候选区域,再进行目标检测及实例分割,与分割网络相比能够获取目标更多的位置信息。检测网络相同类别的单一检测的目标区域通过重叠度(IOU)合并在一起,不同的目标即使相邻也会拥有明确的分割边界。如,两个贴近的结直肠癌肝转移肿瘤即使贴合在一起,也会有明确的区分边界。

表1 数据扩充方法
肿瘤在一张切片上所占的像素总数不到整个图像的像素总数的5%。需要分类的像素占总像素的比例太小。实验中结果显示采用U-Net 直接分割肿瘤的Dice系数约为43%,分割精度非常低。因此,由形态学活动轮廓将肝脏区域提取出来,图像大小为512×512剪裁为320×320,在肝脏区域内,病变占整体像素的15%左右。通过级联网络的方法分割肝脏肿瘤,不仅弥补了深度学习对小目标分割的不准确性,使分割结果更加精确,还减少了计算机内存和训练时间的浪费。本文中,肝脏肿瘤的分割是通过级联网络实现的。级联网络的第一层是U-Net,完成肝脏分割;级联网络第二层是U-Net,完成肝脏肿瘤精准分割,Mask R-CNN 用于检测肝脏肿瘤。级联网络模型如图2所示。本文采用的U-Net模型结构如图3所示,模型参数详见表2。
网络由输入块、卷积块、反卷积块和输出块组成。表2描述Kernel列中,“3×3×2”表示2层、卷积核大小为3×3的网络层,Output size(1)、Output size(2)分别是级联的U-Net 模型的第一层和第二层的输出大小。网络层 中 的Up1、Up2、Up3 和Up4 分 别 为Conv_layer4 和Conv_layer5 的第二个卷积层的融合并进行反卷积、Conv_layer3 以及Deconv_layer1 的第二个卷积层的融合并进行反卷积、Conv_layer2 以及Deconv_layer2 的第二个卷积层的融合并进行反卷积、Conv_layer1 和Deconv_layer3的第二个卷积层的融合并进行反卷积。
1.3.1 形态学活动轮廓

图2 级联网络模型示意图

图3 U-Net网络结构示意图
级联网络第一层目的是获得肝脏轮廓。本节提出在两级网络间使用形态活动轮廓的方法。形态学活动轮廓设计如图4所示,伪代码如下:
算法Morphological activity framework
Input:for example:(a),(b),(c);
Output:for example:(d),(e)
1.FOR traverse all Fig(a)do
2.find the centroid of connected region in (a),named m
3.m extends 160 pixels to the upper,lower,left and right directions to form a block diagram and crop(a).
4.IF the edge of the cropped image without any pixel whose value is 1 THEN
5.(b)and(c)crop the image at the same location,then save(d)and(e)
6.ELSE
7.the parallel lines of the upper, lower, left and right directions called yl,yr,xu,xd,are translated in turn
8.FOR i<4 do
9.IF parallel scan line hits a pixel value of 1 THEN
10.get the distance from the parallel line to the coordinate axis
11.Continue;
12.END IF
13.END FOR
12.obtain the distance from the coordinate axis is ylm,yrm,xum,xdm。
13.ym=1/2(ylm+yrm);
14.xm=1/2(xum+xdm);
15.m=(xm,ym)
16.m extends 160 pixels to the upper,lower,left and right directions to form a block diagram and crop(a).
17.(b)and(c)crop the image at the same location,then save(d)and(e)
18.END IF
19.END FOR

表2 级联网络模型参数设置

图4 形态学活动轮廓
图4 (a)、(b)和(c)大小均为512×512,(a)是第一层级联网络分割输出,经3D CRF处理后所生成的肝脏二值图像。(b)为原始的腹部CT 影像,(c)是原始腹部CT影像肝脏肿瘤所对应的标签。(d)和(e)大小均为320×320,分别为形态活动轮廓处理的腹部CT影像和相应原始腹部CT影像的肝脏肿瘤标签。
设计形态活动轮廓,其目的是消除肝脏和肿瘤以外的冗余信息,从而降低训练速度并提高模型的准确性。实验中,腹部CT 影像肝脏区域至少需要300×300 像素才能涵盖。320×320是被U-Net网络层的卷积核整除的最小尺寸。为保证肝脏全覆盖以及编解码特征的正确融合,因此选择大小为320×320作为活动轮廓裁剪尺寸。
肝脏在一个患者的一组腹部CT影像中形状并不规则。在形态活动轮廓的伪代码设计过程中,直接选择中心的方法更适合于具有凸边的肝脏,而扫描平行线的方法则更适合肝脏边缘凹形。针对肝脏轮廓的不同情况,本文针对采用两种方法适应图像特点选择中心点。
1.3.2 Mask R-CNN模型设计
Mask R-CNN 可以被应用于肝脏肿瘤的检测和分割,以及区分肿瘤和背景的分类任务。图5 表示Mask R-CNN的网络结构图,由三部分组成,第一部分共享卷积层(backbone),实现特征的提取,第二部分是候选区域生长网络(RPN)。RPN模块类似于树状分支结构,可以完成两个任务。模块主干是一个3×3的卷积层,树枝由两个1×1的卷积层组成,分别解决了前后景的输出和边框的修正。第三部分对候选网络进行分类的网络,蓝色框图中数字表示分辨率和通道数,箭头表示卷积、反卷积或全连接层(可以通过上下文推断,卷积减小维度,反卷积增加维度)。所有的卷积都是3×3 的,除了输出层,是1×1 的。反卷积是2×2 的,步长为2。“×4”表示堆叠的4 个连续的卷积。添加卷积的目的通过扩大输出维度,使得预测更加准确。Mask R-CNN可以通过这个模块完成分类,检测框图,以及目标分割的任务。

图5 Mask R-CNN的网络结构示意图
Mask R-CNN相比于Faster-R-CNN的改进有两点:将最后的预测层面上,集成了原有的框图检测、类别分类以及新增的语义分割为将原来ROI Pooling Layer改进为ROI Align,剔除了Faster-R-CNN 在下采样过程中以及将特征转化为固定维度时取整的操作,利用双线性差值的方法来更加精确地找到每个框图对应的特征。这样相对于原来的目标识别可以排除一部分误差,更加精确地进行像素级别的细化,实现分割要求。
本实验中使用迁移学习完成了Mask R-CNN 网络的训练。共享了ResNet50 模型在COCO 数据集训练的卷积层参数。这样可以解决数据量小的问题选择FPN[19]网络作为输出层的“head”。迁移学习的优势在于保持原有的训练网络模型以及效力的同时,可以大量减少深度学习对大量数据集的要求,以及减少大量的训练时间。
2 实验结果
2.1 实验环境
实验是在装有Windows 10 64位操作系统,有Intel i9-7920X 2.90 GHz,128 GB RAM 和2 块NVIDIA GeForce GTX 1080Ti(11G)显卡的计算机上进行的。采用Python 3.5 编写代码。深度学习和迁移学习的训练和测试均以TensorFlow 作为后端框架,Keras 作为前端模块完成。
2.2 参数设置与结果
本文提出一种改进的级联网络。U-Net作为级联网络的第一层,该网络负责分割肝脏。之后,通过形态活动轮廓获得级联网络第二层的输入数据和对应肿瘤标签。根据肝脏分割轮廓,将大小为512×512的图像裁剪为320×320。选择迁移学习网络的Mask R-CNN 和深度学习网络的U-Net作为级联网络的第二层。Mask RCNN 可以完成肿瘤的检测和实例分割,并获得被判定为肿瘤的置信度。
选用Dice系数来评价本文的网络模型,Dice系数定义如下:

其中,X 是医生手工勾画的金标准在图片中所占的区域,Y 是通过网络自动生成ROI 区域,分子代表X、Y具有相同像素值的像素数,分母表示X、Y 的总像素。
级联网络的第一层学习率设置为0.000 1;batch_size设置为10,迭代次数设置为60。经过30次迭代后,模型开始稳定,并测试了批量大小设置为10的验证数据集。验证数据集的Dice 系数为97%。将3D CRF 作为后处理模块以获得更好的分割效果,Dice系数增加到98%。
级联网络的第二级,U-Net 网络参数设置为学习率0.000 01;batch_size 设置为10,迭代次数设置为100 次,70次迭代以后模型开始稳定,对验证集数据进行测试,batch_size 也设置为10。因为肿瘤是小目标,为防止模型过拟合,选择更小的学习率。Mask R-CNN是迁移学习模型,不需要大量数据集。每个GPU 上的batch_size设置为2。为了防止过拟合,选择学习率逐渐衰减的学习方法。学习率设置为0.001,学习率初始动量LEARNING_MOMENTUM=0.9,设置权重衰减正则化系数WEIGHT_DECAY=0.000 1,置信度设置为0.9。得到动静脉时期肿瘤影像训练迭代50 次,在30 次左右迭代达到稳定;动静脉集合数据训练迭代次数70 次,在50 次左右迭代达到稳定。
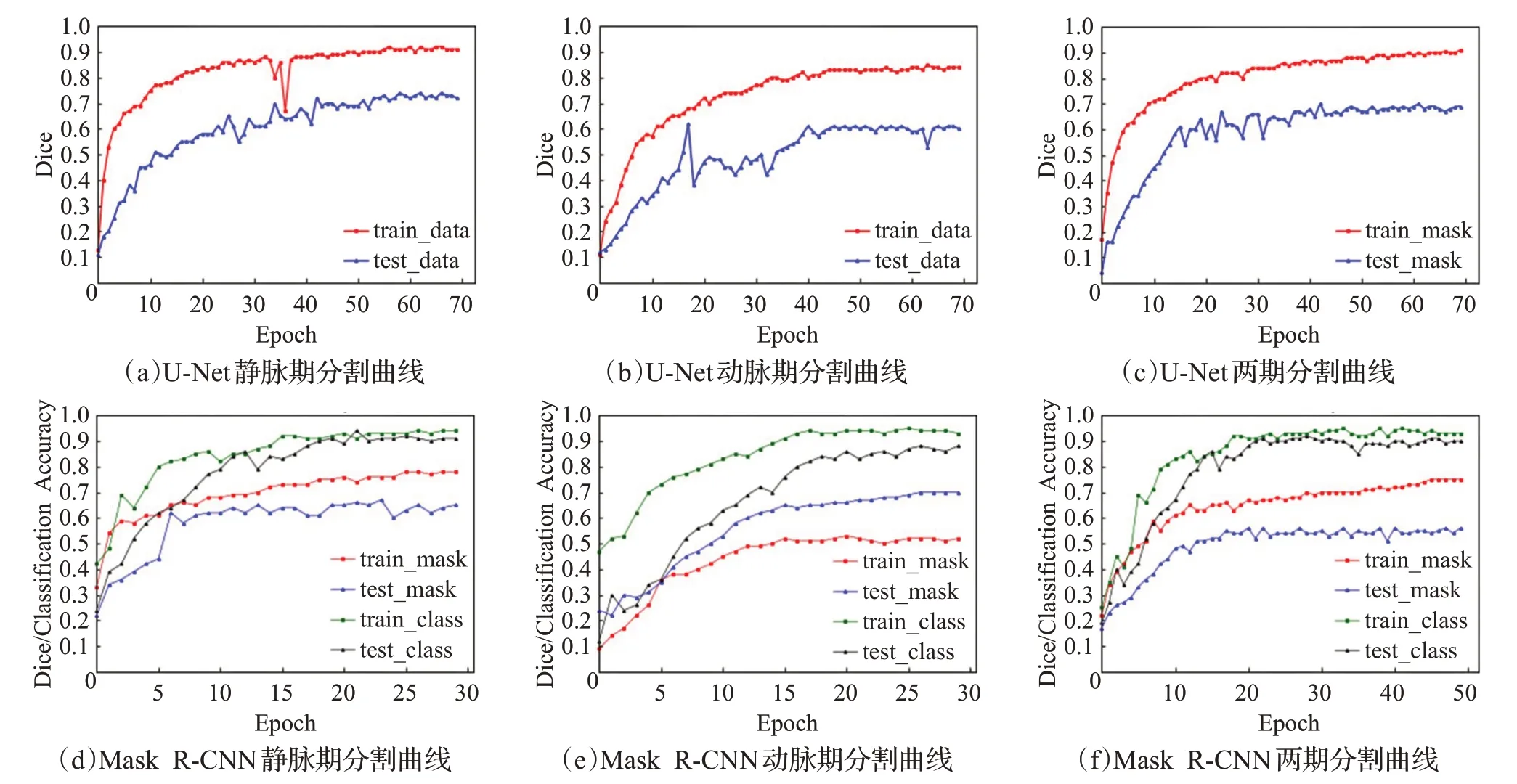
图6 级联网络第二层分割肿瘤训练曲线图
图6 是级联网络第二层U-Net 或Mask R-CNN 肝脏肿瘤分割检测网络训练曲线图。“train_class”和“train_mask”分别表示训练数据集的肿瘤的分类精度,和训练数据集肿瘤分割的Dice系数。当Dice系数和分类精度均稳定时,则认为该模型已完成训练。当模型稳定时,Dice 系数和分类精度上下波动不超过2%。通过对测试集的验证,对肝脏肿瘤直接用U-Net网络进行分割,得到Dice系数仅为43%。应用本文的级联网络分割肝脏肿瘤Dice系数为74%。实验结果表明,改进的级联网络可以有效地提高分割精度Mask R-CNN 不仅可以获取肿瘤分割结果,还可以在其他非肿瘤背景下获得分类准确性。Mask R-CNN 分割并以3D CRF 作为后处理检测肿瘤和U-Net 分割肿瘤的结果示例如图7 所示。Mask R-CNN在检测方面,均值平均精度(mAP)为88%。
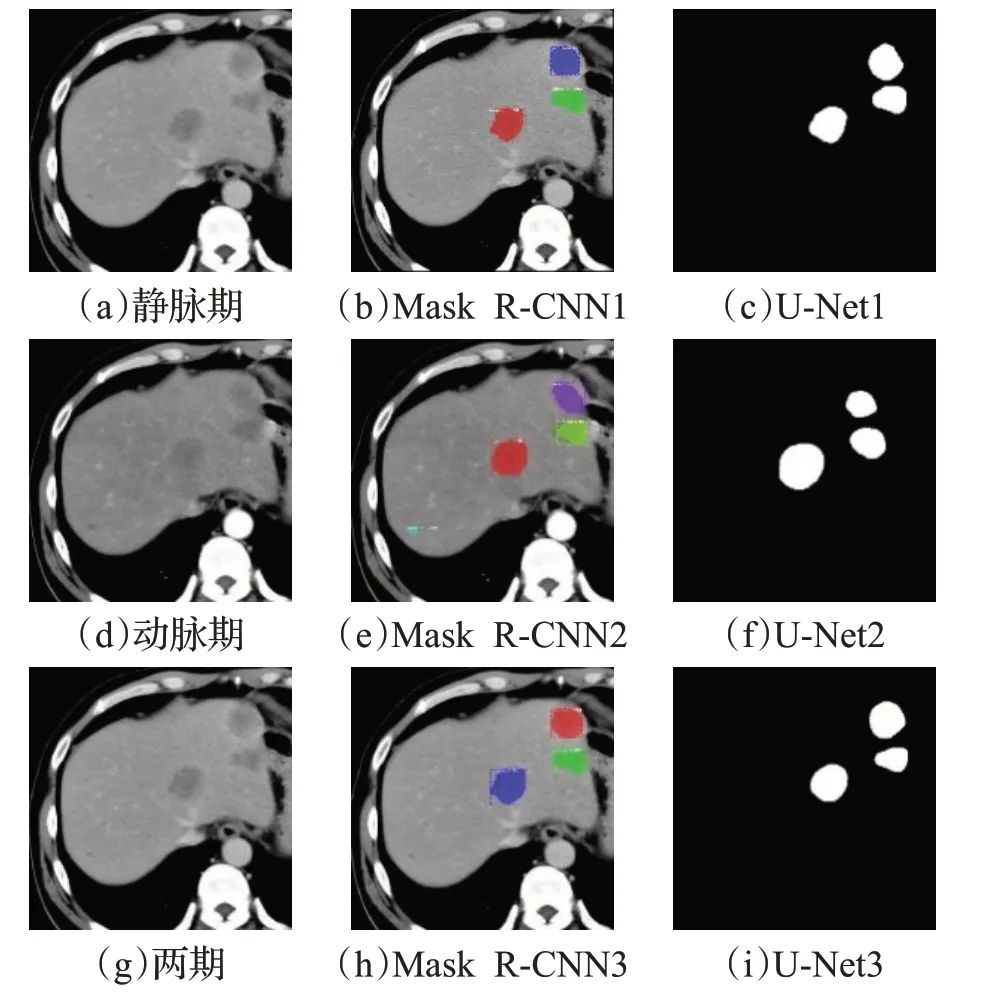
图7 级联网络第二层分割肿瘤结果
图7 中从上至下分别代表静脉期、动脉期和两期的组合影像。每行图像分别代表原始CT影像、MASK RCNN分割的肿瘤结果图和U-Net分割并用3DCRF做后处理优化的肿瘤分割结果图。(a)代表静脉期的原始CT影像,(b)和(c)代表(a)分别通过MASK R-CNN 和UNet 并以3DCRF 作为后处理的肿瘤分割结果。(d)代表动脉期的原始CT 影像;(e)和(f)代表(d)分别通过MASK R-CNN 和U-Net 并以3D CRF 作为后处理的肿瘤分割结果。(g)代表两个时期的原始CT 影像,(h)和(i)代表(g)分别通过MASK R-CNN 和U-Net 并以3D CRF作为后处理的肿瘤分割结果。
U-Net 分割肿瘤以及Mask R-CNN 分割和检测肿瘤的结果的详细信息如表3 和表4 所示。在表3 和表4中,Mask R-CNN 因为肿瘤检出率较高,在区分肿瘤和非肿瘤背景具有很高的准确性。实验结果表明,Mask R-CNN能够准确识别肿瘤和肿瘤位置。

表3 U-Net+3D CRF肝脏肿瘤分割结果(Dice/检出率) %

表4 Mask R-CNN肝脏肿瘤分割分类结果(Dice/检出率/置信度) %
分割结果是否精确的评估标准是Dice系数、关注边缘分割是否准确。与Mask R-CNN相比,U-Net具有更精确的边缘分割效果。针对本文实验数据,与其他分割模型相比,Dice系数具有更高的精度,见表5。
3 结语
通过改进的级联网络模型的设计,针对测试集中的肝脏CT 影像,肝脏分割Dice 系数为97.5%。若级联网络第二层选择U-Net,针对所有测试集中的静脉肝脏肿瘤CT影像,肝脏肿瘤分割Dice系数为74%;若级联网络第二层选择Mask R-CNN去检测肿瘤,针对相同的肝脏肿瘤测试集平均Dice 系数为67%,mAP 为88%。本文改进的级联网络通过分割网络与分割网络的级联,和分割网络与检测网络的级联完成了肝脏肿瘤的细化分割和快速检测。

表5 肝脏肿瘤静脉分割对比结果
Mask R-CNN属于目标检测集成框架,它可以输出检测到目标类别,目标区域置信度以及目标的实例分割边界。将Mask R-CNN应用于患者肝脏是否包含肿瘤,肿瘤位置在哪里,具有明显优势。检测网络可以更精确地定位位置信息和给出肿瘤分类的正确率。如果没有肿瘤,它也可以迅速告知医生没有检测到感兴趣的区域。以Mask R-CNN 为模型的检测框架可以帮助医生诊断感兴趣区域是肿瘤还是非肿瘤的背景区域,帮助医生实现快速筛查和定位肝脏肿瘤。
