多组学分析在炎症性肠病中的应用与研究进展
2021-01-22张立兴王丽娜康广博黄鹤
张立兴 王丽娜 康广博 黄鹤
(天津大学化工学院 系统生物工程教育部重点实验室,天津 300350)
炎症性肠病(Inflammatory bowel disease,IBD)是一种累及末端小肠至直肠的慢性肠道炎症性疾病,主要包括溃疡性结肠炎(Ulcerative colitis,UC)和克罗恩病(Crohn’s disease,CD)。患者多伴有复杂的临床症状,如腹痛、直肠出血和体重减轻等[1]。IBD的发病率和流行率在过去几十年里急剧增加,影响已经超过350万人,逐渐成为一种全球流行性疾病[2]。尽管近年来得到了医学研究团体的广泛关注,但个体遗传变异、饮食、环境因素、肠道生态失调等多因素相互作用共同导致了疾病的发生,如图1所示。因此仅从易感基因座位、肠道微生态对宿主的影响等单一维度无法揭示IBD的发病机制。
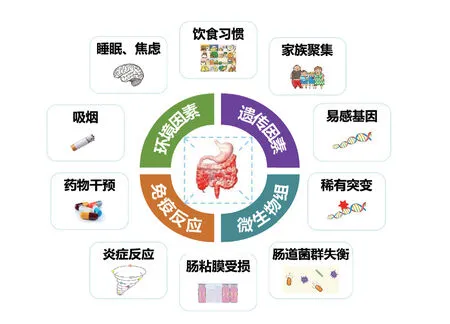
图1 IBD发病因素之间的复杂相互作用
得益于测序和质谱技术的不断进步和完善,涌现出的宏基因组测序、转录组学和蛋白质组学等技术手段能够在不同维度上反映生物体形貌。如图2所示,自2007年以来,每年涉及炎症性肠病和高通量组学数据的研究文章和摘要的数量(log值)正在逐年上升。单一组学研究在疾病的诊断和治疗中起着不可替代的作用,但是单组学的研究方式通常存在局限性,仅能从单一维度去描述多系统、多层次的IBD病理变化。此外富集分析结果范围较大,其有效性有待考证,例如转录组学研究中尽管上游基因突变或丧失功能,但如果激活其他途径,mRNA表达可能保持不变甚至增加,这显然已无法满足病因学研究的需要。相较于传统的单组学技术手段,多组学联合分析具有系统性与整体性的特点且组学间交叉验证,其结果在IBD的发病机理,诊断和治疗方面更具说服力。IBD表型异质性是由基因-环境相互作用所驱动,并且被肠道菌群失调介导。多组学联合分析能够更好地反映宿主免疫反应、宿主环境因素和肠道微生物群之间复杂的相互作用特征。
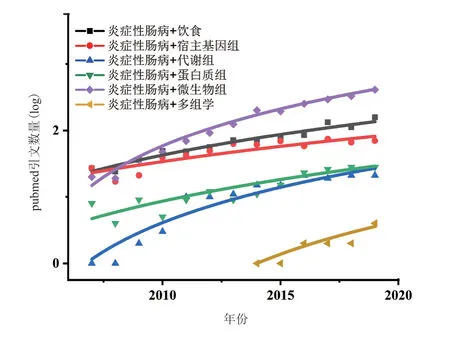
图2 组学在IBD相关研究文章中的历史趋势
由美国国立卫生研究院(USA National Institutes of health,NIH)资助的人类微生物组项目联盟召集了一批科学家来探索微生物群落及其与人类宿主的关系。人类微生物组计划的第一阶段(HMP)[3-4]通过确定包括胃肠道、鼻、皮肤、泌尿生殖道等在内的人类微生物组的多样性和组成以评估与健康相关的微生物多样性的共同模式;这些努力揭示了与人类有关的巨大微生物多样性,并为宿主与微生物组间的相互作用关系提供了新的见解。人类微生物组计划的第二阶段(iHMP)[5]通过与微生物有关的3种人类疾病模型的研究,利用包括16S扩增子、宏基因组和宏转录组、代谢组和蛋白组数据、单细胞测序、病毒组测序以及血清学在内的多组学数据,建立了完整的分子互作网络,并且系统揭示了肠道菌群的组成、多样性以及稳定性与人体正常的生理功能以及疾病的发生密切相关。这3项纵向研究集中在(1)妊娠和早产;(2)肠道疾病发病,以炎症性肠病为模型;(3)呼吸道病毒感染和2型糖尿病发病。作为iHMP研究的3个领域之一,研究人员深入剖析了炎症性肠病宿主以及微生物活动的内在和外部关联,同时也为应用多组学关联分析技术进一步探究IBD的发病机制、寻找IBD的治疗新靶点指明方向。此外,世界范围内不同国家和地区也进行了多项IBD患者队列的多组学研究,产生了海量IBD特征数据,这将有利医学研究团队深层次探究病因的分子机制。这些研究项目寻找出的有效靶点将成为未来IBD干预治疗策略研究的新方向、新思路。目前根据临床特征将IBD分为多个亚型,如果要开发一种更有针对性的治疗方法,需要发现IBD的分子亚型,这些亚型可以作为新的药物靶点。为了实现这一目标,需要从相同的IBD患者中获得多层分子数据。本文探讨了多组学关联分析在IBD研究中的重要潜在应用,包括发病机理分析、疾病异质性特征分析、治疗反应预测、新型靶向药物开发、精准医疗等,为IBD的诊疗提供理论参考和借鉴。
1 宿主基因组学
基因分型(Genotyping),是通过使用生物学分析方法检查个体的DNA序列并将其与其他个体的序列或参考序列进行比较来确定个体的遗传组成(基因型)差异过程的技术。免疫芯片、全局筛选芯片和人外显子组芯片的应用使得大规模检测IBD易感基因座,探究人类遗传因素与疾病表型之间的关系成为可能,也为宿主基因型和肠型之间的相互作用提供了强有力的证据。由英国、德国、瑞典等多个国家联合成立的国际炎症性肠病遗传学联盟(UK IBD Genetics Consortium,IIBDGC)致力于建立IBD患者和健康人群的高质量基因组DNA和表型信息,目前拥有来自IBD患者和健康对照者的大约40000个DNA样品,现已发现了99个明确的IBD易感基因座。同时应用Immuno Chip定制芯片,为大约15000名CD患者,12000名UC患者和21000名健康对照患者进行分型,联合基因型-表型分析技术挖掘全基因组关联研究(Genome-Wide Association Studies,GWAS)数据,以鉴定更多的新型药物靶点、改进现有诊疗方法为目标,为个性化治疗提供数据支持。此外,我国炎症性肠病学组证实了IBD遗传易感性在东西方人群中存在显著差异,这为我国IBD发病机制研究提供了新的研究方向[6]。
传统芯片筛选技术仅能检测出疾病已知关联位点,针对潜在位点对疾病的遗传影响不能很好地阐明,因此需要通过其他测序手段了解疾病的遗传学机理。对特定细胞类型或组织中IBD全外显子组测序和易感性基因座靶点重测序研究有望了解IBD发病机制中基因位点的功能作用,通过测序平台的高特异性、灵敏度以及覆盖度,深度发掘与IBD发病相关的基因信息。核苷酸结合寡聚化结构域蛋白2(NOD2)在CD的发病风险中最显著,也是第一个被发现与IBD相关的基因[7-9]。与全基因组重测序相比,外显子组测序和IBD易感基因座靶向重测序具有低成本、高效快捷的优势,且能针对潜在变异位点反复测序,测序深度可达100x-150x,检测到更多低频和罕见变异,有利于更好地解释所得变异与疾病的关系。宿主基因型和肠道型都与疾病表型有关,这为宿主基因型和肠道型之间的相互作用提供了强有力的证据。Weersma等[10]通过宿主基因组外显子测序和全基因组测序,鉴定到12个微生物数量性状基因座(microbial Quantitative Trait Locus,mbQTLs),包 括IBD相 关 基 因IL17REL、MYRF、SEC16A和WDR78。产短链脂肪酸途径中乙酰辅酶A生物合成减少与基因MYRF变异相关,同时基因CYP2D6、GPR151和CD160的一些罕见变异也会导致代谢相关功能通路的变化,此外相互作用分析证实了TNFSF15基因中IBD疾病特异性mbQTLs。
通过测序技术找到的基因组易感位点范围往往很大,其中许多变异位点仅仅具有统计意义上的关联性,而不具有因果性,产生了很多和疾病无关的候选基因列表。黄海亮等[11]设计出新的精细化定位算法,精确定位出和疾病具有因果关系的易感位点,可以更多地了解与IBD发病机制相关的基因信息,并可拓展应用到其他复杂遗传疾病上。尽管近十年以来,GWAS已经确定了许多IBD疾病易感位点,至少涉及300个基因[12],但与IBD发生机制的关系还有待进一步阐释。
2 环境组学和表观基因组学
宿主环境包括饮食、种族、性别、抗生素用药史等,在IBD患者发病病因中起到重要作用,并且能影响患者肠道微生物表型。例如,饮食被认为是长期和短期时间尺度上微生物结构和功能的重要决定因素[13-14],涉及肠道菌群的IBD研究应在研究设计阶段尽可能控制饮食。抽烟、阑尾切除术与UC呈负相关,但它们却能加重CD;高脂饮食、精细碳水化合物与IBD的发生密切相关,相反富含水果、蔬菜和不饱和脂肪酸的饮食可以预防这类疾病;此外,维生素D供给不足、空气污染、睡眠障碍、心理因素等也能影响IBD发生及发展[15]。这些结果表明在IBD的研究设计和分析中都应该包含这些影响因素。
许多证据表明,DNA甲基化、组蛋白修饰和miRNAs表达改变能解释IBD发病过程中基因与环境因素的关系。“表观遗传学”的概念由Waddington等提出,包括DNA甲基化、组蛋白修饰和非编码RNA[16]。表观基因组描述了不同时期、不同细胞类型中的表观遗传修饰的位置,并找到它们的功能相关性,可以为炎症性肠病的发病机理提供新的研究思路。Nimmo等[17]针对IBD患者外周血样品绘制了CD不同阶段的全局甲基化谱,与对照组相比,CD患者的甲基化水平显著改变,包括一些免疫激活改 变 的 基 因:MAPKI3、FASLG、PRF1、S100A13、RIPK3和IL-21R等,证实相关基因的甲基化状态是疾病状态的预测指标。Kelly等[18]研究分析了CD患儿回肠上皮细胞的组蛋白甲基化特征,鉴定出组蛋白H3K4me3表观遗传修饰发生显著变化的基因,主要集中在免疫调节、细胞生存和信号转导等代谢通路,与患者的肠道炎症程度密切相关。Kim等[19]通过甲基化特异性PCR和亚硫酸氢盐测序技术对207例CD患者进行了功能验证,确定了脆性组胺酸三联体基因(FHIT)在CD患者中以疾病特异性的方式高度甲基化,并且发现其蛋白表达水平下调,确认甲基化FHIT是一种很有前景的CD生物标志物。涉及IBD发病机制的基因调控网络研究不再是“单线程”(如基因组学或表观基因组学)的研究方式,而应结合两者共同解释克罗恩病或溃疡性结肠炎的基因分子调控机制,为IBD的诊断与治疗提供新的研究方向。
3 宿主转录组学
IBD病因的复杂性超出了基因组和表观遗传学的范畴,并涉及基因调控下游的其它组件,包括转录组、蛋白质组和代谢组。转录组是某一生理条件下,细胞内全部RNA转录本的集合,能够从整体上揭示细胞中基因的表达情况及调控规律。转录失调与包括IBD在内的多种疾病相关,分析组织中RNA的表达可以提供疾病活动、病程和治疗效果等信息。Clark等[20]通过整合微阵列与miRNA数据集发现IBD患者中上调最多的基因与糖尿病发生(REG1A,REG1B)、细菌信号(TLRs、NLRs)、先天免疫(DEFA6、IDO1、EXOSC1)、炎症(CXCLs)和基质降解(MMPs)相关;下调紧密连接蛋白(CLDN8)、溶质转运蛋白(SLCs)和粘附蛋白基因表达,将重新定位现有IBD治疗方法的药物途径和治疗靶标。Momozawa等[21]通过大型转录组数据集鉴定了23650个顺式-表达数量性状基因座,确定了驱动63个IBD相关基因座的疾病相关性的调控模块。此外,转录组还被应用于发现诊断和治疗UC和CD患者的生物标志物。Lin等[22]识别出炎症性肠病中的新型miRNA标志物包括miR-31、miR-206、miR-424和miR-146a,这将有助于炎症性肠病的诊断。转录组学的主要挑战之一是不同宿主细胞类型的表达谱差异性。随着高通量测序技术的发展,这些局限性会被逐步优化和改进,从黏膜活检标本中分离单个宿主细胞进行单细胞测序可以缓解这一问题。
4 蛋白质组学
蛋白质组学针对某一生物、组织或细胞在特定生理或病理条件,系统性研究所有蛋白质的特征、数量和功能,能够获得包括功能多样性肠道菌群在内的环境样本,揭示正在进行的代谢过程以及受环境变化如疾病状况的影响,更有可能反映宿主与微生物生态系统的实际情况以及与IBD的内在关联。Washington等[23]利用支持向量机算法和机器学习技术研究蛋白质组特征的预测效力,在区分CD和UC分类上获得了较好的预测结果。Manfredi等[24]评估了UC与CD的血清蛋白谱,发现UC与CD患者相比对照组均有不同程度的蛋白表达差异,其中有些蛋白如KAIN、PRCC和GELS仅在CD中发现差异,还有些蛋白如LPPRC,SURF4和CHADL仅在UC中发现差异。Di等[25]发现与健康组相比,IBD患者中上调的蛋白在趋化因子和细胞因子活性、免疫性疾病生物标志物和自身免疫性疾病GWAS信号中显著富集;相反,下调的蛋白在营养和代谢方面富集。蛋白质组学的应用有助于挖掘新的IBD标志物,发现的IBD相关生物标志物主要有血小板因子4[26]、结合珠蛋白[27]、波形蛋白[28]、人中性粒细胞防御素组分[29]等,这种蛋白质组学诊断模式为IBD的鉴别诊断提供具有临床价值的参考标准。蛋白质组学可以有效地加速互补生物标志物的发现,从而能够持续监测患者的治疗反应;还可以进一步改善治疗方法,并最终促进针对IBD的个性化药物治疗[30]。
5 代谢组学
代谢组是指细胞、组织或器官中所有代谢组分的集合,尤其是指分子质量在1000以下的小分子物质[31]。与细胞的主要功能单位蛋白质不同,代谢物是指代谢过程中生化活动产生的小分子,基因与蛋白质的微小调控最终反映在代谢物表型的变化[32]。近年来,研究人员开始利用代谢组学技术探讨IBD病因机制,旨在改善疾病诊断和治疗效果。Schicho等[33]采用核磁共振氢谱(NMR)分析IBD患者的血清和血浆样本,发现IBD患者中的甲醇、甲酸盐、甘露糖、氨基酸含量明显增加,而尿素和柠檬酸盐含量明显降低。Wilson等[34]研究人员从血浆、血清、尿液、粪便、粪便挥发物、呼气中的挥发性有机化合物等不同生物样本对IBD人群进行定性定量分析发现,与非IBD人群相比,IBD患者的氧化三甲胺TMAO水平有所下降。这些数据表明,TMAO可能有潜力作为一种生物标志物,以支持IBD诊断以及评估UC的疾病活动。Marchesi等[35]采用无创代谢组学方法识别不同类型IBD粪便标本的代谢物特征,观察到CD患者的粪便代谢谱相比UC患者存在显著差异,表明CD引起的炎症更广泛,涉及整个肠道。这些研究说明了代谢组学在识别潜在生物标志物方面应用前景广阔。此外,代谢组学作为其他组学技术的补充,能够揭示关键生物学机制,具有明显的应用价值。
6 微生物组学
尽管IBD病因尚不清楚,但越来越多证据表明IBD与肠道生态失衡有关[36]。目前IBD患者肠道微生物群落结构研究主要是通过粪便标本检测,结果与肠道活检标本检测有明显差异[37],并不能很好地解释IBD与肠道微生态失衡的强相关性。但仍然有其他证据,如服用抗生素后的新生儿在未来患有IBD的风险更高[38],以及IBD风险位点与模式识别受体(PRRs)、免疫细胞因子相关[39]。PRRs能够对病原菌产生应答,如与CD患者相关的NOD2基因能编码胞内PRR,若该基因上的功能位点缺失,会导致肠道微生态中的有害菌清除功能障碍[40]。这些证据都能表明IBD发病与肠道微生物群落失衡有关,为进一步对IBD病因机制研究提供了方向。对肠道微生物群落的深入分析,以及对人体肠道和宿主微生物群落之间的相互作用分析,能够更好地发现微生物相互作用(如合作、竞争、利他、侵略)对IBD发病机制的新见解。
6.1 16S rRNA扩增子测序
16S rRNA扩增子测序是研究微生物群落结构的常用手段。16S rDNA是细菌编码核糖体16S亚基的基因,存在于原核微生物基因组中[41],具备高度保守性(保守区)和特异性(可变区),是系统发育研究和分类学研究的理想目标。在一项中西方IBD患者队列中,利用16S rRNA测序技术发现不同人种患者粪便标本中的肠道菌群变化模式相似,并且开发出一套用于IBD诊断的预测模型,CD与UC的预测准确率分别为87.5%和79.1%。另外发现对英夫利昔单抗治疗应答的患者治疗后肠道菌群多样性恢复,梭菌目(Clostridiales)相对丰度显著增加,这使得英夫利昔单抗更有针对性地治疗IBD,从而达到降低成本和药物不良反应发生率的目的[42]。在IBD患者肠道微生态中寻找丰度特异性变化的生物标志物成为临床个性化治疗IBD的重要基础,发现是否有跨种族通用或特定的肠道微生物标志物可以显示和预测疾病进展和药物治疗反应,为不同IBD患者菌群模式提供更多精准治疗方案。
6.2 宏基因组学
宏基因组学是生境中全部微小生物遗传物质的总和,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的[43]。通过宏基因组测序(Metagenomic Sequencing,MGS)得到的不同层面特征信息可用于给IBD疾病状态和治疗反应分类及预测。Douglas等[44]在肠检样本中鉴定出可作为标志物的肠道微生物,并研究了肠道菌群功能,鉴定出的嗜粘蛋白-艾克曼菌属(Akkermansia muciniphila)在CD患者中丰度较低,MGS鉴定出的功能相关代谢通路如ko00633、KO7793等可较准确地预测治疗反应。在一项西方IBD患者队列研究中发现,IBD患者的粪便微生物多样性相比健康组物种多样性显著减少[45],这种变化与疾病发展紧密相关。更进一步研究表明,厚壁菌门丰度特异性减少,变形菌门丰度特异性增加,这种变化与复杂微生物群落介导宿主炎症反应的环境应力相一致。
肠道菌群的纵向数据收集成本较高,但在某些情况下信息解释度并不够,因为肠道微生物具有一定的可塑性和稳定性。微生物群落对人类健康风险的影响在很大程度上取决于微生物之间合作交流、竞争侵略等相互作用[46]。因此,系统阐明微生物相互作用对宿主疾病的调控机制研究很有意义。姜立波等[47]将博弈论、行为生态学等学科理论整合到微生物相互作用关系的网络架构中,并开发出一套基于物种丰度依赖性的互作网络算法。微生物相互作用经验法则的开发设计对解释IBD遗传-环境相互作用网络具有重大意义。
7 多组学联合分析
尽管基因组、转录组、蛋白质组、代谢组、微生物组等多个组学在探索IBD发病机制研究和提供更优的治疗策略方面提供了极大的帮助。然而多数研究仅通过单一的组学从单一的因素入手,而没有考虑到它们在健康或疾病中的复杂相互作用。基于IBD疾病的复发性,从单一角度可能无法很好的解释疾病的发展过程,所以整合组学对于IBD的解读拥有无可比拟的优势。IBD系统生物学通过各种组学数据集结合多层次分析,能全面描述IBD分子特征,是IBD发生发展研究的理想方式。利用基因组学、转录组学和免疫组学可以观察宿主遗传结构和免疫应答;通过宏基因组测序和宏转录组可以测量肠道微生物;蛋白质组学、宏蛋白质组学和代谢组学可以测量宿主信号和微生物共代谢;通过饮食日记、饮食频率问卷和标准化临床记录来测量环境因素。但多组学整合分析的工作还较为欠缺,样本量、测序深度及数据库注释度是限制IBD多组学研究的主要因素,这会导致不同研究中得到的结果相互矛盾,表明标准化的必要性。如表1-2所示,世界各地的多个研究小组和协会正在生成大型数据集,包括广泛的临床数据与高通量组学数据。
多组学技术应用于肠道微生物研究,为遗传、代谢和生化过程的变化提供了一个全局视角。如图3所示,这种方法现在已被应用于IBD患者的肠道菌群,能帮助我们更好地理解IBD患者微生物组致病作用的具体机制,以及宿主-微生物相互作用的功能阐释。IBDMDB项目作为iHMP的一部分,研究涉及宏基因组、宿主和宏转录组、蛋白质组、代谢组、病毒组、甲基化分析和16S测序,将微生物组成与基因关联分析,在直肠和回肠分别获得了106对和31对基因-OTU对,这与区分不同部位的不同基因表达模式一致;最后整合多组学构建出大规模交叉测量型关联网络,其中普拉氏梭杆菌(Fecalibacterium prausnitzii)相关性最强,能解释在“失调”时相关基因、转录及蛋白水平表达下调。酰基肉碱与许多失调相关的物种有关,包括肺炎克雷伯菌(Klebsiella pneumoniae),副流感嗜血杆菌(Haemophilus parainfluenzae)和鲍氏梭菌(Clostridium bolteae)等,这意味着包括长期生长和短期转录在内的多种调控尺度都参与其中[64]。在一项Alenghat等[65]主导的研究中,他们通过包括全基因组表达谱、16S rRNA扩增子测序和染色质免疫沉淀测序(ChIP seq)在内的多组学联合分析发现,宿主遗传和环境因素包括共生细菌信号,能影响IBD的发展和严重程度。他们认为HDAC3是协调肠上皮细胞内在转录途径网络的关键作用机制,HDAC3可调节IBD患者中多种细胞过程和共生菌信号。对肠道微生物群的反应改变是疾病风险的主要决定因素和疾病的可能机制,许多与保护肠道菌群相关的宿主生物功能易受组成基因中有害基因突变的影响。我们可以整合微生物组学和GWAS数据,研究宿主遗传变异对微生物组的影响。在一项包括10000多名IBD患者和5000多名非IBD患者的多中心研究中,证明一种特定的错义变体(rs13107325)与CD和结肠粘膜微生物组分的变化有关,所有样本均在同一位置进行基因分型,验证队列最终证明了特定的IBD风险变异导致肠道微生物群的致病性变化[66]。

献文[67][5][64][48][49][50][51][52][53][54][55][56]本本本本本本本本标标标标标标标标便检便便便便便便///粪/活/粪//粪/粪//粪/粪/粪序序本序序序序本序序序序序序测测标测测测测标测测测测测测法子检法子法子检子子子本法子法枪增活枪增枪增活增增增标枪增枪学鸟鸟组组本本组组肠活组组物因标标因因和膜因因生基便便基基粪——16S rRNA扩和鸟16S rRNA扩鸟16S rRNA扩道便粘基基微宏粪粪宏宏16S rRNA扩16S rRNA扩16S rRNA扩检肠宏16S rRNA扩鸟宏————便/粪/粪/结学组质本组组质组组质白质白白标蛋本蛋本质蛋织白白主标主标主组蛋本宿便宿便蛋——宏标————————————宿肠——组(SCAFs)/粪浆本本本人)标匀标标便便便便列(/粪/粪/粪/粪组队组组究谢谢组谢/粪谢代学代代代谢研学组向向向向代本谢靶靶靶靶向标组代——非非——————————非非靶便————多病本检本本检本本肠标活标标活标标性便肠便检道本织血症/粪/粪标组/全录RNA-Seq/结录RNA-Seq/活序序RNA-Seq/肠RNA-Seq/胞细检肠片1 炎测测维芯学组组纤达成组RNA-Seq/活RNA-Seq/结表表录转主本转主主本主肠因转——宏宿标宏宿宿标宿人————————基/位析本本/分标标感本析/化液液分片基血化芯DNA甲/血血和IBD易标疫/序检和周基免析测组/活序/外和测序细片标组本子析组测DNA甲胞维芯本胞学因标显分子重组纤型子标GWAS分本细组基血外化显向因成分显血化白因主周主基外靶基肠因外周基血基——宿外宿甲全点全人基人外————————甲全量数12 90 1321 215 61821 561 63 183934——UC/CD 0/10 36/3638/67495/6150/367/115 0/1561 17/1982/509/015/9——The GEM Project Inflammation at IBDMDB称HMP iHMP目名Med into Grad 1000 IBD Project IBDGC——Interfaces——————项

献文[57][58][59][60][61][62][63]物容内肠/盲序测子增学组物微————16S rRNA扩——生——————本标本本本肠标标标结膜本膜本肠本标结标肠标粘脏肠脏肠肠和鼠)和粘/肝/结/肝/结/结/肝/结组组究(组组组组组研学质质质质质质质组白白白白白白白学质蛋蛋蛋蛋蛋蛋蛋组白主主主主主主主宿宿多宿宿蛋宿宿宿的型本标模脏本内肝标体和浆病浆血本肠血和标脏脏性冻症/冷/肝/肝组2 炎组组谢谢谢学代代代组向向向表谢靶非————靶非————靶代非本本本标本标标肠标肠肠结膜结本本结本和粘和标标和标脏肠脏肠肠脏肠/肝/结/肝/结/结/肝/结片片片片片片片芯芯芯芯芯芯芯学达达达达达达达组表表表表表表表录因因因因因因因转基基基基基基基型型鼠模模小炎炎陷肠炎鼠性肠肠肠炎炎大肠缺结因移结-/-)转导导导导结结DSS诱模DSS诱型DSS诱IL-10基胞T细DSS诱Mdr1a(
多组学联合分析还能用于研究IBD患者中肠道菌群与免疫系统的相互作用。适应性免疫通过抗原特异性应答来保护微生物,免疫应答受损会改变肠道菌群结构,进而导致肠道菌群紊乱或失衡;反过来肠道菌群能够促进粘膜免疫系统和全身免疫系统的正常发育与成熟,其生态失调则会破坏免疫系统。Erickson等[67]在6对健康或受CD影响的双胞胎的粪便样本中,通过宏基因组学和宏蛋白质组学技术,旨在明确与CD相关的基因和蛋白质水平的功能差异。最后发现在回肠克罗恩病患者的蛋白谱中,负责机体炎症反应的蛋白质明显增多,并且伴随着细菌碳水化合物代谢和细菌-宿主相互作用的改变。多组学技术被越来越广泛地用于阐析IBD患者中肠道微生物、免疫系统和宿主遗传结构三者之间的相互作用关系,组学数据之间能互相补充和证明,确保研究结果的深入性和可靠性。在一项IBD宏基因组研究中,发现IBD患者和健康组之间有12%的代谢功能途径存在显著差异。另一项宏基因组学和宏蛋白质组学研究证实,iCD中丁酸盐和丙酸盐代谢基因减少,丁酸盐和其他短链脂肪酸的总体水平降低,这与16S研究发现的产短链脂肪酸厚壁菌减少一致[68]。
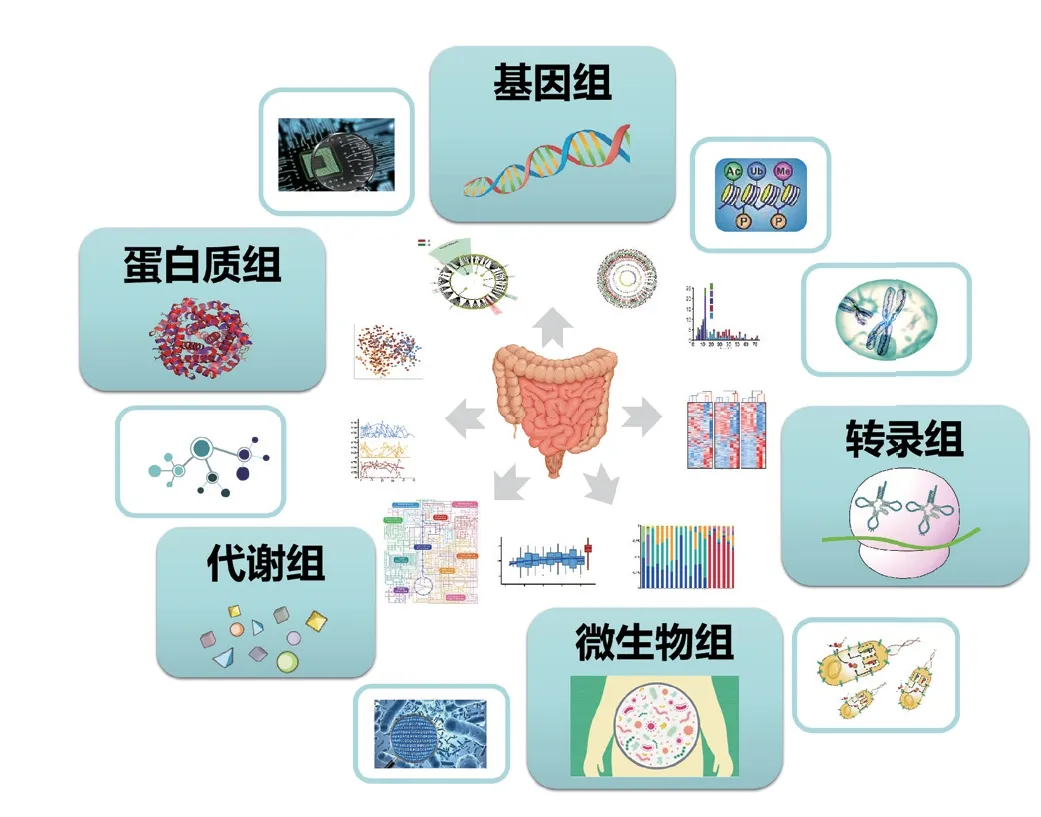
图3 IBD多组学联合分析
此外,还能利用在IBD多组学产生的大规模数据指导早期靶向药物开发和肠道微生物组干预治疗策略[69]。Mills等[70]收集40名UC患者患者粪便样品进行16S rRNA、基因组、代谢组学、蛋白质组学,同时对血清样品进行蛋白质组测序,整合测序数据集分析与UC疾病严重程度相关的微生物因素,所有组学分析结果均显示出与疾病严重性相关的大规模变化,其中代谢组和蛋白质组学最能预测UC疾病的严重性,最后确定拟杆菌属(Bacteroides)蛋白酶是反映UC疾病严重程度的一个明显特征,而这主要是由于蛋白酶蛋白表达的变化所引起,体内和体外实验的证据均显示细菌蛋白酶抑制剂可能是未来治疗UC的一种新型治疗方法。Jia等[62]通过使用葡聚糖硫酸钠(DSS)诱导的结肠炎小鼠模型进行多组学评估,发现欧芹通过改善肠道菌群失调进而干预IBD。更好地了解营养物质和肠道微生物群之间的协同相互作用,将有助于制定更有效的IBD干预手段。
8 总结与展望
多种高通量测序技术可用于识别和定量分子表型,包括基因和miRNA表达水平、表观遗传特征和肠道菌群组成等,可鉴定出一系列与临床表型相关的IBD亚型。更好地将IBD划分为不同的表型不仅有助于更好地了解疾病,还有可能有助于确定特定的患者亚群,这些患者将从特定的干预措施中受益。尽管多组学数据的整合与建模仍具有挑战性,但多组学IBD分子表型分析,有助于了解IBD病因及开发个性化疾病管理和疗法。当常规抗炎药物失效时,在分子途径水平上识别IBD亚型将有助于确定更适配的治疗方案。为了合理使用这些治疗资源,发现能够可靠地识别哪些病人会从某种药物中受益或受到伤害的生物标志物将是至关重要的。例如,美国PRISM队列采用16S rRNA扩增子和宏基因组测序技术发现微生物谱可以预测维多株单抗(Vedolizumab,一种肠道选择性生物制剂)疗效[71]。Rueedi等[72]对两组不同队列CD患者的尿液进行了代谢组和全基因组关联分析,结果表明,一种特异性SNP(rs492602)与岩藻糖基转移酶FUT2之间存在联系,FUT2与CD相关,得出FUT2可能是一个有用的CD预后生物标志物这一结论。这项研究表明遗传风险位点是IBD蛋白表达变化的驱动因素,值得进一步研究以证实其因果性和有效性。
虽然新型生物标志物(表3)在临床研究中表现出较好的预测效能,但是仍存在一定的局限性。生物标志物的结果可靠性有赖于研究对象的基因背景、治疗方案、微生态组成、随访时间等多个影响因素[78],其临床价值不具备普适性。因此,我们需要进行多中心、大规模、标准化的临床研究,提高生物标志物对IBD的诊断与鉴别水平,有利于扩大IBD人群的适用范围。确定IBD个性化治疗方案不仅需要临床特征信息的输入,还需要联合复杂的多组学数据。因此,在大数据研究中整合这些多维数据源对IBD精准医疗的发展显得十分重要。可以乐观地预见,多组学联合分析技术将为IBD患者提供特异性的药物治疗方案。

献文参[73]考[73][73][73][73][74][74][73][75-76]、醇PUS10,、肌miR-98,TNFSF8,IGNG,盐酸ERAP2,IL10,CREM,、乳酸氨LRRK2、缬PIM3,IL/R,NDFIP1,TAGAP,IL2R,PRDM1,酸SLC22A4,氨、亮↓)TNFRSF9,氨TNFSF8,IL12B,IL23,酸GPC4,GPX1,亮、异miR-362-3p,miR-532-3p,ICAM3(CCL8,CCR6,胺IL8RB,氨酰盐miRs-103-2*,物CCL2,CCL7,酸IL8RA,ADO,志标BACH2,TNFSF4,VMP1,、谷CUL2,MST1,物miR-98,miR125b-1*,let-7e*,IBD生、谷盐氨酸、甲CCL11,CDH1,ERRFI1,碱的现CPEB4,HNF4A,WDR8,ITGB2,CARD9,CDH1(发↑);DOK2,、胆究MUC19,酸氨研ICOSLG,TNFSF*15,PRDX5,学GNA12,IL27,SBNO2,FASLG,THAA,组多)酸);IFITM1(↓过LRRK2,DAP,DLD JAK2,TYK2,著(↑);丙显3 通为氨碱HSPA6,、赖PARK7,表胆,CXCL1最酰IL23R,miRplusF1159,miR20b,磷CDH11等GBGT1,因↑)达PEX13、甘表分IBD vs. controlsCARD 9,RER,XBP1,基DENND1B,异FGR2a*/B,IL21,糖萄物FANCC,差:19个志miR-484(、葡油miRplus-E1035,酸标IGFBP4,FAM10A4(↑物:CYLD、IL23,IL1R1,IL1R2,SERNC3,ORMDL3,UTS2,肠膜SLC11A1,LNPEP,CBGT1,: 精的NOD2,ITLN1,ATG16L1,STAT3,IRGM,结粘状肠氨生肠在miR340*,结潜乙结CD vs. controls UC vs. controls组IBD vs. controlsTHRAP2,CD vs. controls IBD vs. controls CD vs. controls IBD vs. controlsmiRs-3180-3p,UC vs. controls)态组状因组基化组组学因观基录谢组基甲表(转代[75-76][77][77])[77]4、[77][75][75](↓、视白盐蛋白合酸菌菌蛋二应结杆球醇黄拟胃、丁)C-反弱瘤、视脆泼(↓Fusobacterium、醇白肌酸菌杆盐氨蛋色应梭、乳酸、氏-3、CXCL16、拉素普)酸酸(↓氨氨组集-3、C-反Clostridium difficil、活凝素缬)盐、菌、酸(↓酸胺糖)集乳(↓凝↑)梭Fusobacterium varium、性)氨胺马氨半糖难菌样艰杆尿酰酸多氨(↓IL-8、IL-18、、三盐、乳物生Faecalibacterium)、变肠、TNF-RI、TNF-RII、IL-8、IL-18、CXCL16、半微属亮甲、谷antichitin、antilaminarin、PAB、IL-18、CXCL16(亮甲柠酸、酸酸甲色菌三、Campylobacter concisus、梭赖酸异、、形)、酸基氨氨胺檬氨、盐盐);丙杆氨酸酸(↑粪酰丁硫(↑氨、酚酸尿、TNF-RII、盐氨白);马胺酸组蛋丁、TNF-RI、Dialister invisus、);4-甲二丁、卫曲Klebsiella pneumoniae、氨盐酸);乙菌酸钙菌谷盐(↑谷杆特斯氨酰、酸酸、弯菌Roseburia hominis和酸谷利明伯(↑(↑二酸基白阿组简雷菌戴、酸氨羟氨氨甲、卫盐氨组、:S100A12、蛋氏浊酸赖基酸钙炎膜瑞浑肺、甲索)、拜基谷胺甲粘酸、胡斯(↓酯罗酰、酸冬碱氨酯延白Enterotoxigenic Bacteroides fragilis人胺乙甲基);天胆缬酸、)S100A12、蛋、)卫Escherichia coli、克大Bacteroides vulgatus、菌如杆、乙酸((↑PAB、(↓);结酸酸基氨氨氨胍脯素(↑杆:atypical pANCA、GAB、S100A12、钙菌白)、、(↑拟盐胺盐酸ACCA、gASCA、anti-OmpC、AMCA、肠);脂蛋菌三胍anti-OmpC、弱酸甲酸索(↑酸酸氨连白脆、盐酸铁(↑);丙亮、、蛋↑)性丁(↑乙延乳白铁菌素产Mycobacterium avium paratuberculosis、、)醇胡碱亮乙、氨醇蛋胆(↑Bifidobacterium adolescentis、菌卫酸酸酸拟肠异、酸(↓钙、S100A12、连、S100A12、蛋杆通产菌枝素:atypical pANCA、IL-8()普酰、酸氨乳gASCA、素杆胰歧:S100A12、)氨氨氨铁Phylum proteobacteria、油甘甘、磷油氨甘、、、甘核杆毒)4、双白结(↓赖甘甘、、、酸);脂白(↑春蛋酸盐氨盐盐盐酸酸酸酸氨蛋青白:副Phylum Firmicutes、岛:ALCA、白蛋:天:谷:葡:丙:甲丁(↑卫:柠:天结:钙萄变Bacteroides fragilis、素:钙膜合:乳病门粘:ALCA、菌门恩菌二氨檬冬便液清肠便液清清醇便糖氨酸冬清岛便肠清清便形罗壁粪尿血结粪尿血血黄粪血胰粪结血血粪Ruminococcus gnavus、分克 厚prausnitzii、)蛋卫CD vs. controls CD vs. controls UC vs. controls CD vs. UC UC vs. CD IBD vs. controls)调调上IBD vs. controls下((组组质物白生蛋微
