基于Faster R-CNN的疟疾血涂片检测改进算法
2021-01-19刘乾宇
刘乾宇
(1.北京邮电大学 软件学院,北京 100876;2.北京邮电大学可信分布式计算与服务教育部重点实验室,北京 100876)
0 引 言
疟疾是由疟原虫属寄生虫所致,自2000年以来,世界在减少疟疾病例和死亡总数方面取得了显著进展。但疟疾患病及死亡人数仍然居高不下,每两分钟就有一名儿童死于这种可防治的疾病。每年报告的新发病例超过2亿。据世界卫生组织颁发的《2018年世界疟疾报告》,2017年,估计在87个国家有2.19亿疟疾病例。2015年至2017年期间没有在减少疟疾病例方面取得明显进步。全球疟疾应对工作面临众多挑战,及时诊断和治疗是防止疟疾轻症发展为严重疾病和死亡的最有效手段。寄生于人体的疟原虫有四种,即间日疟原虫、三日疟原虫、恶性疟原虫和卵形疟原虫。其中恶性疟原虫和间日疟原虫危害最大。疟原虫的生命周期很复杂,包括环状体(rings)、滋养体(trophozoites)、裂殖体(schizonts)和配子体(gametocytes)。疟疾血涂片镜检法是检测疟原虫和疟疾病例诊断依据的最直接和最重要的方法,是由世界卫生组织推荐用于疟疾检测的金标准[1]。由于近些年国内疟疾的防治和减少,基层医院与疾病控制机构不再重视疟疾血涂片的镜检,从而导致疟疾血涂片镜检水平不断下降[2-4],产生疟疾的漏诊和误诊[5]。因此使用计算机辅助诊断方法自动识别细胞并识别其疾病种类,进而实现自动细胞分类和计数,由于其相较人工更快更准确的定量结果,将对疟疾疾病的检测带来重要意义。
针对疟疾血涂片细胞分类以及计数问题存在的不足与经验,该文提出了一种基于Faster R-CNN模型的疟疾细胞检测算法,对网络结构做出优化和修改,增加卷积滤波器层,并对特征提取网络进行优化从而提高疟疾图像的检测效果。
1 相关工作
随着计算机视觉技术的发展,深度学习在学术界受到了广泛的关注,也对医疗影像检测领域产生了巨大的影响。基于机器学习的SVM等算法解决大规模训练样本的图像分类尤其是多分类问题较为困难。基于深度学习的算法,尤其以卷积神经网络(convolutional neural network,CNN)[6]为基础的算法在物体检测任务中被广泛应用。Girshick等[7]提出R-CNN解决了图像目标检测问题,该算法首先对图片中的物体画框,再判断框出的物体类别。R-CNN采用选择性搜索(selective search)方法生成大约2 000个方框,用已训练好的CNN对每一个方框内的图片提取出特征,再放入SVM进行分类,同时将特征放入回归器中得到更精确的候选框。由于R-CNN太多的候选框存在重叠的问题,需要大量重复计算,随后Fast R-CNN[8]合并了R-CNN中用CNN提取特征、SVM分类、回归这三个过程,形成端到端整体的模型,并且引入感兴趣区域池化层(ROI pooling)共享特征,速度和准确率都得到了提升。但Fast R-CNN采用的选择性搜索方法进行特征提取仍然非常耗时,而Faster R-CNN[9]在Fast R-CNN基础上引入了RPN(region proposal network)改善了这一问题,进一步提高了检测速度,并在检测精度和速度上达到了较好的平衡,实现了端到端网络结构[10]。除了上述以R-CNN为代表的基于区域提案(region proposal)的目标检测算法,还有一类基于回归的目标检测算法,如YOLO[11]把目标检测问题转化为回归,采用一个完整的端到端网络,由输入的原始图像直接输出目标位置和分类的检测结果,网络更轻训练速度更快。但YOLO对小样本群体检测效果不好,泛化能力偏弱。为了解决这些缺点,SSD[12]整合了YOLO中的回归思想和Faster R-CNN中的anchor思想,在不同尺度的特征图进行目标位置和类别的训练和预测,从而达到多尺度检测的目的,提高了模型的泛化能力,同时保证了检测速度。
近年来,人工智能技术与医疗健康领域的融合不断加深,人工智能逐渐成为影响医疗行业发展,提升医疗服务水平的重要因素。医疗影像辅助诊断是一个热点内容。过去已经进行了许多关于使用自动化方法进行疟疾血涂片检测的研究。常用的方法是数字图像处理技术。一些研究者开发了辅助诊断工具,例如使用粒度分析法来估计细胞大小[13],或使用基于形态学算子的自动阈值检测来分析感染的血细胞图像[14],但是由于使用了体外培养标本而不是实际的外周血,未从根本解决疟疾检测的任务。Raviraja等人[15]提出了一种从感染的血液图像中自动分离出寄生虫(滋养体,裂殖体和配子体细胞)的方法。使用颜色、形状和大小的信息,然后通过缩放,整形以重建图像,将图像与受感染的图像进行比较。对返回的图像进行统计分析并进行比较,此外还评估了寄生虫细胞核的大小和形状。Savkare等人[16]开发了一种系统,该系统可根据感染者血液中的疟原虫颜色和形态特征快速准确地诊断人类血液介质中的疟疾红细胞。Snehal Suryawanshi等人[17]提出了用于疟疾寄生虫检测的增强技术,其中细胞分割包括多个步骤,例如使用基于泊松分布的最小误差阈值对图像进行二值化,然后在形态学上进行细化,最后使用支持向量机(SVM)作为分类器进行分类,与基于欧氏距离分类器的精度相比更高。虽然研究者们使用图像处理的技术对疟疾检测进行了广泛研究,但是受疟疾感染血液的显微镜图像在显微镜照明强度、细胞形状、密度和颜色由于样品制作的过程的细微差异而不同,并且其中个别类别即使是专家也很难识别。另外有学者尝试自动化识别并量化疟疾[18-20]的过程使用一组预定的测量(强度、形状和纹理)中的要素,使用复杂的工作流进行图像处理和机器学习分类。然而,由于缺乏通用性,这些方法都没有获得很大反响。因此,该文尝试针对图像预处理进行改进,增强疟原虫的可检测性,提高算法模型的可靠性。
2 整体模型介绍
2.1 Faster R-CNN目标检测原理
Faster R-CNN在Fast R-CNN的基础上引入区域候选网络,用RPN来提取预测框,使得RPN和Fast R-CNN模型能够共享卷积后的特征。这样特征图同时作为RPN和Fast R-CNN的输入,提高了性能。Faster R-CNN模型结构如图1所示。首先Faster R-CNN使用卷积层提取特征,该特征图被共享用于后续RPN层和全连接层,RPN用于生成区域候选框,并且通过对锚点(anchors)分类以及边界框回归(bounding box regression)修正anchors以获得精确的区域候选框。随后传入感兴趣区域池化层(regions of interest pooling,RoI Pooling),进一步提取特征,进入全连接层判定目标类别,同时再次通过边界框回归以获得检测框最终的精确位置。
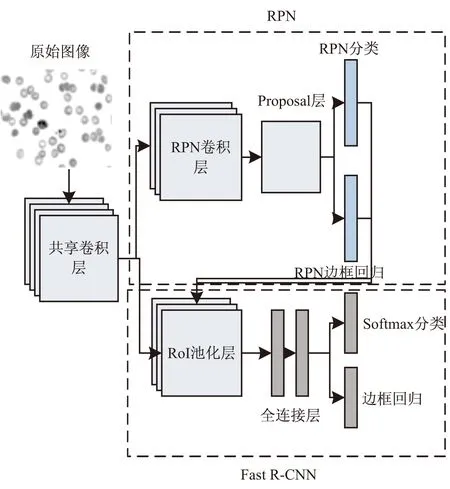
图1 Faster R-CNN基本结构
相对于传统的滑动窗口和选择性搜索算法,Faster R-CNN采用RPN生成目标区域候选框提高了候选框的生成速度,用于对目标识别、分类以及边界框回归。回归层用于估计目标原始的位置,分类层用于对目标区域进行分类,得出目标属于某类的概率。每一个滑动窗口,同时预测9个候选框,一般由3种面积128×128、256×256、512×512,3种比例1∶1、1∶2、2∶1组成,最终输出为多个候选框和该候选框被判断为目标的得分。
感兴趣区域池化层是对空间金字塔池化层(spatial pyramid pooling,SPP)的改进。由于全连接层中图像维度的约束,SPP层使用金字塔结构处理图像尺寸不同的问题,RoI Pooling通过下采样、上采样的方法,将非均匀尺寸的输入,即不同候选区域的特征图执行最大池化,转换成固定大小的特征图。此处固定大小的选择是由该网络的超参数预定义的。主要目的是为了加快训练与测试时间,优化性能,采用端到端联合的方式训练整个系统。
2.2 文中模型
2.2.1 卷积滤波器
滤波是对于图像的每一个像素点,计算它的邻域像素和滤波器矩阵的对应元素的乘积,然后加起来,作为该像素位置的值。而基于核的滤波器被称为卷积滤波器,核也成为卷积矩阵,其有奇数行、奇数列,中心的元素对应于感兴趣的像素,其他的元素对应于这个像素周围的邻近像素,每个元素都有一个整数或浮点数的值,这些值是应用在像素上的权重。
针对疟疾血涂片细胞的特点,细胞数量多且细胞边缘与内部细节不够清晰,故在Faster R-CNN共享卷积层之前加入卷积滤波器层,该层的网络结构为自定义的一个3×3和一个5×5的核,分别为H1、H2。首先使用H1对图像进行平滑处理,再使用H2增强图像的细节,得到较原图更为清晰的图片。
如图2所示,经卷积滤波器层处理过的图像输入到Fast R-CNN网络中进行进一步的特征提取。
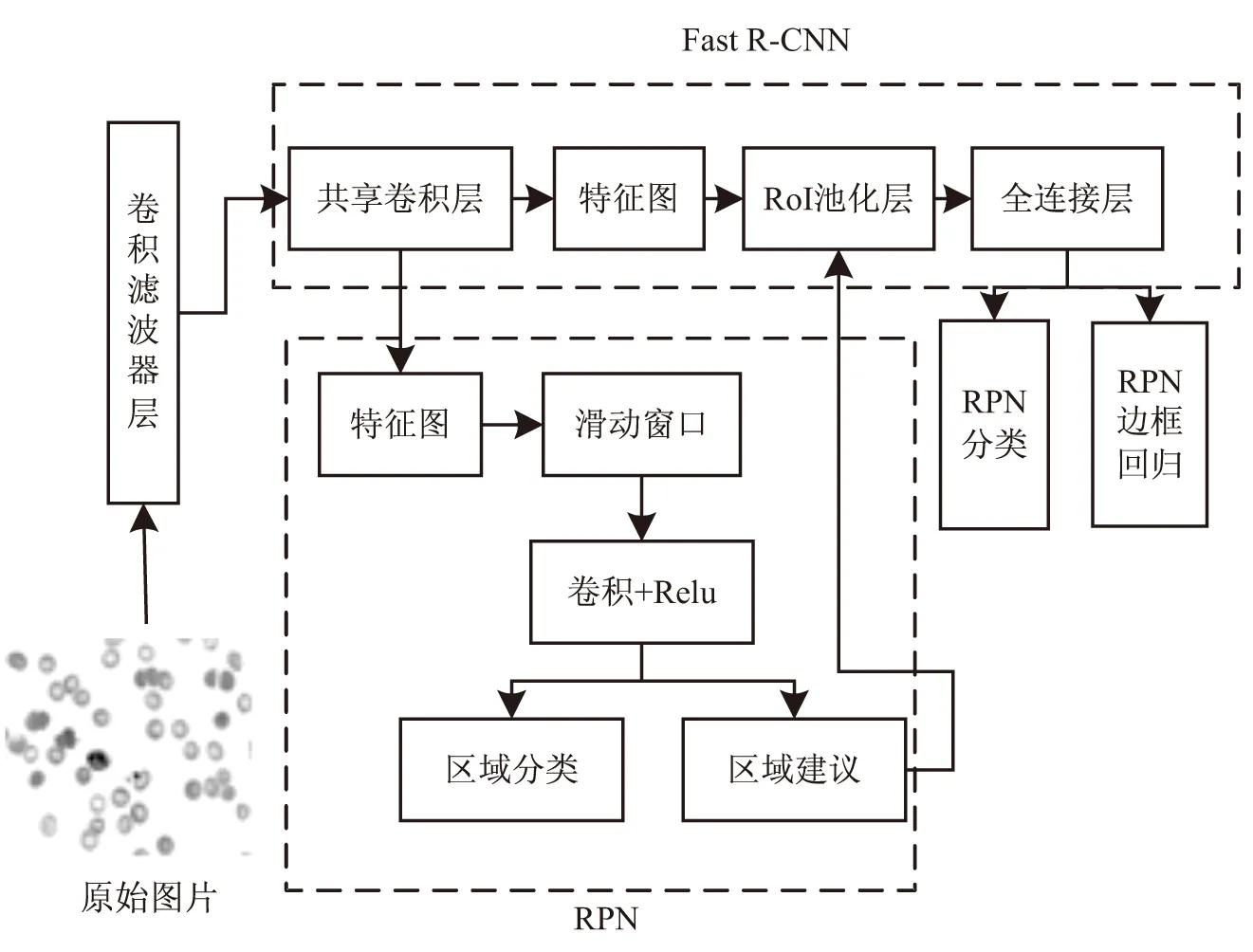
图2 文中模型基本结构
2.2.2 残差网络
原始的Faster R-CNN采用了VGG16[21]作为特征提取网络。VGG16采用堆积的小卷积核,其网络架构如图3所示。
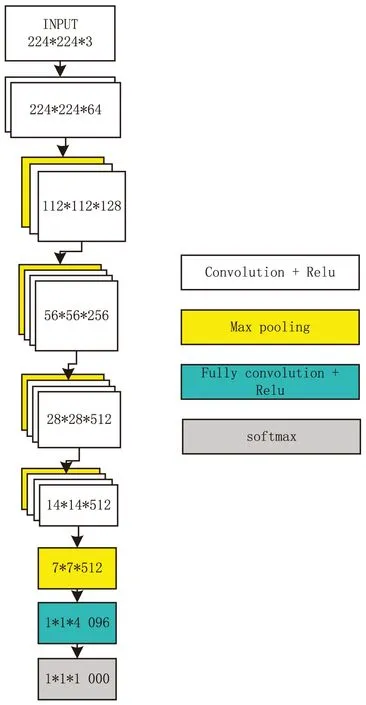
图3 VGG16网络结构
VGG16前几层使用3×3卷积核,通过最大池化(max pooling)层减少神经元数量,最后增加2个全连接层和1个归一化指数函数(softmax)层。而随着网络深度的增加,网络的准确度应该同步增加,但增加网络深度后,梯度反向传播过程中会逐渐减小,造成梯度消失,导致网络退化。
该文引入残差网络(ResNet)[22]以优化上述问题。ResNet通过引入残差学习单元,允许前面输入的信息直接传到后面的层中,使新添加的层只需要学习上一个网络输出的残差,可以使网络在深度增加的情况下不退化,训练效果更好。
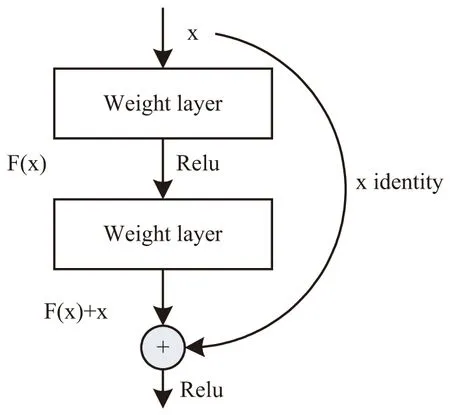
图4 ResNet中残差学习单元
如图4所示,残差学习单元除了正常的卷积层输出外,还有一个分支把输入直接连到输出上,该输出和卷积的输出做算术相加得到最终的输出,用公式表达就是H(x)=F(x)+x,其中x是输入,F(x)是卷积分支的输出,H(x)是整个结构的输出。对于一个堆积层结构,当输入为x时其学习到的特征记为H(x),若想学习到残差F(x)=H(x)-x,则原始的学习特征是F(x)+x。相比原始特征,残差学习更加容易。当残差为0时,此时堆积层仅仅做了恒等映射(identity mapping),网络性能至少和浅层网络性能一样,且残差不会为0,使得堆积层在输入特征基础上学习到新的特征,从而拥有更好的性能,有效缓解网络退化问题。
3 实验过程
3.1 数据集
该文采用的数据集为间日疟原虫(疟疾)感染人血凃片数据集[23],图像为PNG和JPG格式,由1 364张图像组成,总计约80 000个细胞,用Giemsa试剂染色。该数据集由两类未感染细胞即红细胞(red blood cells)和白细胞(leukocytes)和四类感染细胞即配子体(gametocytes)、环状体(rings)、滋养体(trophozoites)和裂殖体(schizonts)组成。数据集的标注信息包括以上全部类别,其中一些识别困难的细胞被标注为困难(trouble)类型。每张图片分辨率为1 600×1 200,在人工标注的细胞中,Ground Truth框的面积(分辨率)大小多为100×100左右。
3.2 实验环境
实验基于TensorFlow框架,采用的系统为Linux,GPU为Tesla P100。数据集中,训练验证集占80%,测试集占20%。训练验证集中训练集占80%,验证集占20%。其中标注为困难的类型不参与训练。
3.3 评价指标
在疟疾血涂片细胞数据中,针对单个类别,使用平均精度(average precision,AP)作为评价指标,对于各个类别,分别计算AP,取所有类别的AP平均值为mAP(mean average precision),用来衡量文中模型在所有类别上识别效果的好坏。
3.4 预处理
深度卷积神经网络的参数数量巨大,需要数量足够多的数据才能满足训练要求,而文中采用的数据集相对来说非常有限。为了能够增加训练的数据量,以提高神经网络模型的泛化能力,文中采用对训练集图像进行垂直翻转、水平翻转以及逆时针旋转90度的方法,对现有的数据集进行扩充。
3.5 实验结果
实验部分采用三组对比实验,实验一仅加入卷积滤波器层,而不采用ResNet特征提取网络和修改的锚点框尺度比;同理,实验二仅采用ResNet进行特征提取,实验三仅采用修改的锚点框尺度比;最后综合实验一、二、三的改进,得出一个综合的输出结果。
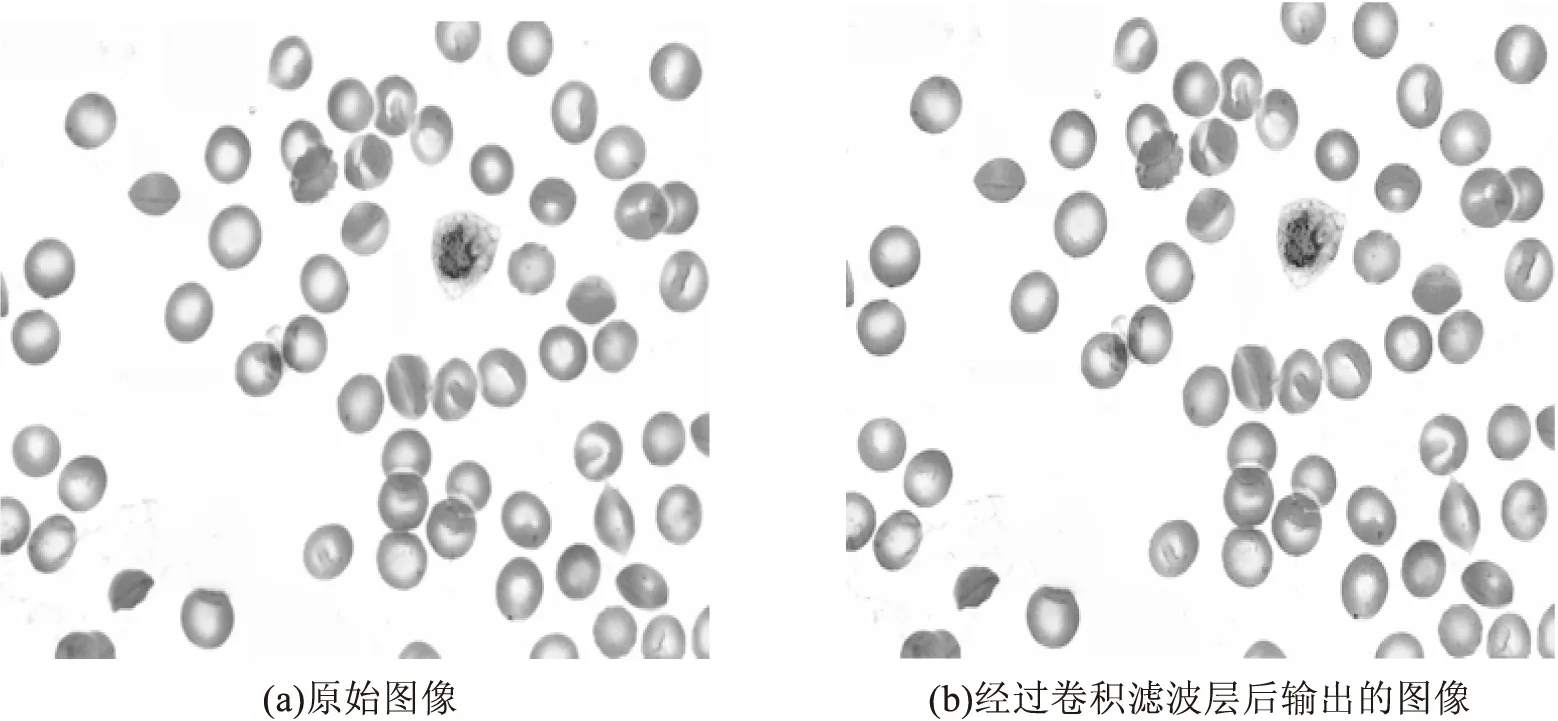
图5 卷积滤波器层输出特征图
图5为疟疾血涂片数据集中的图像经过卷积滤波器层的输出结果。对比图5(a)与图5(b)不难看出图5(b)中细胞的结构与边缘更加明显,图像更清晰,该层的输出特征图作为Faster R-CNN的输入数据。
实验结果表明,经过数据扩充的训练集在添加了卷积滤波器层并且优化过的Faster R-CNN模型训练后,测试集的mAP为79.56%,能够达到辅助检测的效果,而对于原始Faster R-CNN模型的测试结果,测试集的mAP为70.72%,实验一只加入卷积滤波器层提高了1.64%,实验二采用ResNet进行特征提取mAP提高了4.57%,实验三通过修改锚点框尺寸使mAP提高了2.72%,综合所有改进的模型实验结果使mAP提高了8.84%(见表1)。文中综合方法部分测试图像效果如图6所示,其中图6(a)、图6(c)为在原始Faster R-CNN模型下的测试结果,图6(b)、图6(d)为在文中方法下的测试结果,相同血涂片检测结果中图6(b)相对于图6(a)多检测出了滋养体类型的细胞,图6(d)相对于图6(c)在检验细胞数量和准确率均得到了提高,文中方法在疟疾血涂片细胞检测中的效果更好。

表1 实验结果mAP对比

图6 原始Faster R-CNN与文中模型实验结果对比
4 结束语
将深度学习应用于生物医学领域有广阔的前景,在原始Faster R-CNN的基础上提出了一种改进的Faster R-CNN疟疾血涂片细胞的检测与识别算法。通过试验,相比于原始Faster R-CNN模型,文中方法结合ResNet残差网络解决了网络退化问题,并通过添加卷积滤波器层等方式在间日疟原虫(疟疾)感染人血凃片数据集的检测中提升了检测效果。采用文中方法,在检测人员的镜检水平参差不齐的环境下,可以大大提高识别率,同时降低了对原始图像的质量要求,以及训练样本数量的要求,在医学领域尤其是医疗诊断方面具有重要意义。在未来的研究中,将继续优化模型,用来帮助研究人员对视野图像中的细胞进行自动分类和分阶段,并识别和区分受感染阶段的特征。另外需要测试不同来源的疟疾血涂片样本来测试算法,以拓展此算法的应用场景。
