SSllla基因在云南高抗性淀粉特种稻的遗传变异
2020-12-21李霞杜娟杨晓梦普晓英杨加珍曾亚文
李霞 杜娟 杨晓梦 普晓英 杨加珍 曾亚文


摘要:对云南省高抗性淀粉特种稻品系及功米系列品种进行SSHla基因已报道剪接位点SNP检测,结果显示,云南省高抗性淀粉材料未出现已报道的剪接位点SNP突变。进一步对云南高抗性淀粉特种稻SSllla基因的序列进行遗传变异分析,共发现SNP遗传变异位点59个,插入缺失变异位点6个。其中,分布于基因编码区域的SNP位点18个,非同义变异12个。分析这些非同义变异位点在6份高抗性淀粉特种稻材料与云南省地方对照品种的差异,结果显示6份高抗性淀粉特种稻材料与对照品种的差异,主要为籼粳稻之间的差异,只有功米2号存在SNP特异性变异。
关键词:SSIlla基因;抗性淀粉;水稻;遗传变异
中图分类号:S511
文献标识码:A
文章编号:0439-8114( 2020) 20-0035-04
D01:10.1408 8/j .cnki.issn0439-8114.2020.20.008
随着饮食习惯和生活方式的改变,Ⅱ一型糖尿病的发病人数逐年增加[1]。研究表明,合理增加抗性淀粉的摄取量,可有效控制体重,具有潜在预防慢性病的功效[2,3]。水稻是全球1/3以上人口的主食,水稻品种间血糖指数GI值变幅为48-93,是开发预防糖尿病及降低餐后血糖主食的重要来源[4,5]。目前,国内已培育出一些高抗性淀粉含量水稻品种,如云南省农业科学院的功米3号[6],上海市农业科学院的降糖稻1号[7],浙江大学培育的突变体RSlll[8]。虽然高抗性淀粉水稻已有一定规模的应用,但对其遗传机制的研究却较少。有研究表明,SSIlla基因剪接位点的SNP突变可以导致水稻子粒抗性淀粉含量的升高[9]。同时,SSllla基因在不同水稻材料基因组中较其他淀粉合成相关基因表现出较高的多态性,是淀粉合成酶类中最多样化的基因[10]。因此,本研究对云南省高抗性淀粉特种稻品种功米系列及2份高抗性淀粉品系进行SSllla基因的同源克隆,并对其基因序列进行比较分析,以期通过上述检测,明确SSllla基因在云南省高抗性淀粉特种稻的遗传变异,为下一步云南省高抗性淀粉特种稻的育种工作提供技术支持。
1 材料与方法
1.1 材料
供试材料为云南省农业科学院生物技术与种质资源研究所功能食品作物团队选育高抗性淀粉水稻功米系列品种及品系,共计6份(抗性淀粉含量>3%),分别为功米2号、功米3号、功米4号、功米5号、17SM36、17SM37,对照为云南粳稻品种合系35、籼稻品种滇屯502,二者为抗性淀粉含量偏低材料。
1.2方法
1.2.1 DNA提取选取各材料的幼嫩叶片,利用天根生化科技(北京)有限公司植物基因组提取试剂盒(DP320)提取试验材料的基因组DNA,具体操作参照试剂盒说明书进行。提取后的DNA样品置于-20℃冰箱保存备用。
1.2.2 引物设计及PCR扩增 利用参考基因组SSllla基因序列设计特异引物(表1)。以8份材料的基因组DNA为模板,高保真酶I-5TM 2x High-FidelityMaster Mix(TPOOI,北京擎科生物技术有限公司)进行PCR扩增。扩增程序为98℃预变性2 min;98℃变性10 s,56℃退火15 s,72℃延伸60 s,35個循环;72℃延伸10 min,25℃保存。在1.0%琼脂糖凝胶中进行电泳产物检测,紫外凝胶成像系统拍照。
1.2.3 目的片段回收及克隆利用DNA凝胶回收试剂盒(CE0101-50型)对目的片段进行回收,将回收片段连接到pClone007载体上(007VS型),转化感受态TreliefrM 5a Chemically Competent Cell( TSC01型),进行阳性克隆筛选并进行PCR鉴定。以上试剂均购于北京擎科生物技术有限公司,试验操作均按照上述试剂盒说明书进行。
1.2.4
SSllla序列分析对鉴定的阳性克隆送样测序,每个样品送3个阳性克隆,测序由北京擎科生物技术有限公司实施。序列分析利用DNAstar软件完成。Blast分析通过NCBI( http://www. ncbi.nlm. nih.gov/)在线进行。
2 结果与分析
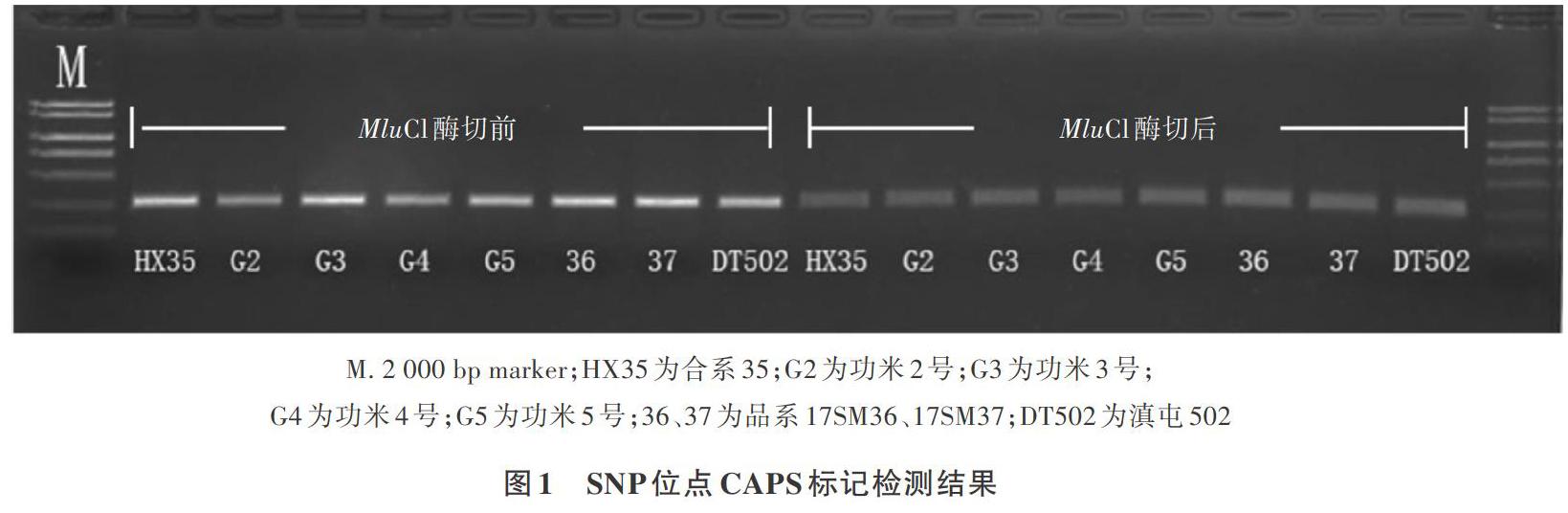
2.1 已报道SSHla基因剪接位点SNP突变检测
利用已报道的CAPS标记,对云南省高抗性淀粉特种稻进行SSllla基因第六外显子剪接位点SNP突变检测。通过MluCl内切酶对合系35、功米2号、功米3号、功米4号、功米5号、17SM36、17SM37、滇屯502进行酶切反应检测。结果如图1所示,6份高抗性淀粉特种稻材料不能被MluCl内切酶切开,说明这6份材料并未发生相应的SNP变异,也未产生新的MluCl酶切位点。
2.2 SSIHa基因在高抗性淀粉特种稻的遗传变异
结合参考基因组序列信息,通过对6份云南省高抗性淀粉特种稻材料及对照品种的SSllla基因序列的遗传变异分析,共计发现了66个变异位点,其中SNP变异位点59个,41个位于内含子及基因上游区域,18个SNP突变位点位于外显子区域(表2)。同时,位于外显子区域的SNP位点中有同义突变6个,非同义突变12个。除了上述的SNP的变异外,还发现了6个插入/缺失突变,1个在基因上游序列出现,其他均位于内含子区域(表2)。
2.3 SSHla基因在高抗性淀粉特种稻与对照品种的差异分析
通过对6份云南省高抗性淀粉特种稻材料及对照品种的SSllla基因全长序列与参考基因组序列的遗传变异分析,共计发现12个非同义突变位于外显子区域。由于编码区的非同义突变可能会引起蛋白质功能的改变,因此重点分析了上述12个SNP变异位点在2个对照品种与高抗性淀粉特种稻之间的差异。结果(表3)显示,功米3号及17SM36与对照籼稻品种滇屯502基因型在这12个SNP位点上表现出了完全相同的基因型。功米4号、功米5号及17SM37基因型与粳稻品种合系35在这12个位点表现出很高的相似性,只在5353483、5353812、5355663、5357570这4个位点上与滇屯502有一致的基因型,推测这些差异可能来源于籼粳杂交的结果。功米2号在5353162、5353784、5353826位点上表现出与2个对照品种不同的基因型差异。同时,6份高抗性淀粉特种稻材料在5353319、5353685、5354337、5354415这4个位点表现出了籼粳稻之间的差异。
3 讨论
随着中国大健康产业的不断发展及人民生活水平的提高,人们的营养保健意识不断增强。抗性淀粉作为安全健康的新型功能性食品,越来越受到消费者的青睐。水稻作为中国人口的主粮,开展高抗性稻米的基础研究和种质资源的选育意义重大[11]。目前,已克隆的水稻子粒中影响抗性淀粉含量的基因包括SSllla和SEB[9,12],二者都属于淀粉合成酶基因。本研究对云南省抗性淀粉含量较高的功能稻米品种及品系SSfffa基因的序列分析显示,功米3号、功米4号、功米5号及17SM36品系与对照品种SSllla基因序列基本一致,编码区域的12个非同义突变也只在籼粳稻之间存在差异,基本确定上述5份高抗性淀粉材料的抗性淀粉升高与SSllla基因的变异无关。功米2号在5353162、5353784、5353826位点上表现出与其他7份材料不同的基因型差异。由于其他5份高抗性淀粉材料的基因型完全一致,并且以上3个位点与2个对照表现出相同的基因型,因此推测这些位点可能并不影响抗性淀粉的含量。此外,由于功米2号是其中惟一的有色米材料,以上3个差异也可能与其特殊的种皮颜色有关。基于以上结果推测,云南省高抗性淀粉特种稻抗性淀粉含量的升高,可能与SSllla基因的遗传变异没有直接的相关性。研究表明,作物抗性淀粉含量与直链淀粉的比例呈正相关[13.14],而GBSSI编码的Wx蛋白质对水稻直链淀粉含量有积极影响[15]。由此说明,除了淀粉合成基因SSllla和SEB对抗性淀粉含量有影响外,其他淀粉合成基因也可能影响抗性淀粉含量。目前,参与淀粉合成的酶类主要有6大类,分为可溶性淀粉合成酶(Soluble starch syn-thase)、淀粉分支酶(Starch branching enzyme)、淀粉脱支酶( Debranching enzyme)、颗粒结合淀粉合酶( Granulebound starch synthase)、ADP葡萄糖焦磷酸化酶( AGPase)和质体淀粉磷酸化酶(Pho)[16.17]。已有报道显示,淀粉合成相关酶类的遗传变异对水稻淀粉的物理特性有不同程度的影响[18],因此,后续试验将对云南省高抗性淀粉特种稻在其他淀粉酶合成基因的遗传变异进行深入分析,探究其与云南省高抗性淀粉特种稻之间的关系,以期为云南省高抗性淀粉特种稻形成的遗传基础研究提供线索。
参考文献:
[1] CHEN L, MAGLIADO J, ZIMMET P Z.The worldwide epidemiolo-gy of type 2 diabetes mellitus-present and future perspectives[Jl.Nat Rev Endocrinol. 2011.8: 228-236.
[2] RAICOND P, EZEKIEL R, RAIGOND B. Resistant starch in food:A review[l].J Sci Food Agric .2015, 95( 10): 1968-1978.
[3] YAMADA Y. HOSOYA S,NISHIMURA S,et al.Effect of breadcontaining resistant starch on postprandial blood glucose levels inhumans [J]. Bioscience. biotechnology and biochemistry. 2005,69:559-566.
[4] BOERS H M. SEIJEN T H J.MELA D J.A systematic review of theinfluence of rice characteristics and processing methods on post-prandial glycaemic and insulinaemic responses[J] British journalof nutrition. 2015. 114: 1035-1045.
[5] KAUR B,RANAWANA V.HENRY J. The glycemic index of riceand rice products:A review, and table of GI values[J]. Critical re-views in food science and nutrition, 2016. 56: 215-236
[6]羅曦,黄锦峰,朱永生,等.水稻功米3号高抗性淀粉性状的遗传分析[J]农业生物技术学报,2014. 22(1):10-16.
[7]孙志敏,白建江,朱辉明,等混配比例及加工方式对“降糖稻1号”稻米产品抗性淀粉含量的影响[J].核农学报,2012, 26(2):318-323.
[8]杨朝柱,李春寿,舒小丽,等.富含抗性淀粉水稻突变体的淀粉特性[J].中国水稻科学,2005( 19):516-520.
[9] ZHOU H, WANG L, LIU G,et al.Critical roles of soluble starchsynthase SSIHa and granule-bound starch synthase Waxy in synthe-sizing resistant starch in rice[J]. Proc Natl Acad Sci. 2016. 113:12844-12849.
[10] KHARABIAN-MASOULEH A.WATERS D L.REINKE R F.et al.Discovery of polymorphisms in starch-related genes in rice germ-plasm by amplification of pooled DNA and deeply parallel sequenc-ing[J]. Plant Biotechnol J, 2011. 9(9): 1074-1085.
[11]李颜方,王高鸿,杜艳伟,等.作物抗性淀粉研究进展[J].中国农业科技导报,2019(21):56-62.
[12] YANG R F,SUN C L,BAI J J,et al.A putative gene sbe3-rs forresistant starch mutated from SBE3 for starch branching enzyme inrice( Oryza sativa L.)[J]. PLoS One. 2012.7(8):e43026.
[13] CAI J, MAN J,HUANG J, et al.Relationship between structureand functional properlies of normal rice starches with different am-ylose contents[Jl. Carbohydrate polymers, 2015, 125: 35-44.
[14] LIN L, GUO D, HUANG J, et al. Molecular structure and enzymat-ic hydrolysis properties of starches from high-amylose maize in-bred lines and their hybrids [Jl. Food hydrocolloids. 2016. 58:246-254
[15] CROFTS N.ABE K, AIHARA S, et al. Lack of starch svnthase Illaand high expression of granule-bound starch sVnthase I synergisti-cally increase the apparent amylose coment in rice endosperm[J].Plant science, 2012, 193-194: 62-69.
[16]王自布,黃燕芬,吴坤,等.籽粒淀粉合成酶与淀粉合成关系的研究进展[J]生物技术进展,2013(3):336-341
[17] JEON J S,RYOO N, HAHN T R. et al. Starch biosynthesis in ce-real endosperm[Jl Plant physiology and biochemistry, 2010, 48:383-392
[18] KHARABIAN-MASOULEH A. WATERS D L E,REINKE R F,et al SNP in starch biosynthesis genes associated with nutritionaland functional properties of rice[J]. Scientific reports.2012,2: 557.
作者简介:李霞(1981-),女,河南获嘉人,助理研究员,博士,主要从事功能作物分子育种研究,(电话)15398480628(电子信箱)lixia_napus@163.com;通信作者,曾亚文,男,二级研究员,博士,主要从事功能作物育种研究,(电子信箱)zengyw1967@ 126.com,
