基于枯草芽孢杆菌多组学数据全基因组规模代谢模型与蛋白质降解模拟
2020-12-17江明锋官久强安添午张翔飞罗晓林
袁 萍,江明锋,官久强,安添午,张翔飞,罗晓林*
(1.西南民族大学青藏高原动物遗传资源保护与利用,四川省教育部重点实验室,成都 610041;2.四川省草原科学研究院,成都 611731)
枯草芽孢杆菌(Bacillus subtilis)是一种典型革兰氏阳性细菌,分布广泛,易分离培养,具有抑制病原菌生长、增强免疫应答、促进养殖动物生长、净化水质等特点,在饲料、食品行业、基因工程和医药等领域应用广泛[1]。关于枯草芽孢杆菌培养的微生物学、遗传学和分子生物学方法非常成熟,已测定多株枯草芽孢杆菌基因组序列[2]。枯草芽孢杆菌可利用低成本碳氮源大规模培养,因其可分泌大量胞外酶,如碱性蛋白酶(Extracellular alkaline protease,aprE)等,可将环境中不溶性蛋白质降解为多种寡肽和氨基酸,并从中衍生出其他含氮化合物供菌体生长[3]。基于此特性,有较强蛋白质降解能力的枯草芽孢杆菌被筛选用于降低水体蛋白质含量。
基因组规模代谢网络模型(Genome-scale metabolic models,GEMs)通过基于约束的流量平衡算法,利用摄取和分泌通量有限数据预测代谢表型[4]。随组学技术发展,GEMs提出与应用已近20年。基于其对基因型和表型关系综合性描述能力,GEMs已广泛应用于设计代谢工程策略和方面[5]。目前GEMs重构在模式微生物如大肠杆菌等发展和应用趋于成熟,非模式生物中应用待进一步研究[5-6]。枯草芽孢杆菌代谢模型已有相关报道,但利用GEMs通过分子与蛋白水平预测枯草芽孢杆菌对环境中蛋白质降解能力研究较少。
随水产养殖规模不断扩大,养殖水体富营养化成为水产养殖主要问题。已有联合利用枯草芽孢杆菌、光合细菌、硝化菌及反硝化菌等降解水体有机氮,改良水体富营养化相关研究。目前主要集中在调整不同细菌比例,根据有机氮或氨氮降解水平表型数据评估和筛选高效有机氮降解菌株,却未见利用枯草芽孢杆菌基因组和代谢数据系统研究并基于计算推测其蛋白质降解能力的相关报道。本试验首次利用全基因组规模代谢模型作为整合调控途径与枯草芽孢杆菌碳氮循环代谢模拟不同碳氮条件下枯草芽孢杆菌降解蛋白质,使用菌株试验数据和细胞内通量数据评估模型准确性,为系统筛选并最大化枯草芽孢杆菌蛋白质降解能力提供参考。
1 材料与方法
1.1 试剂与培养基
本研究DNA操作所需酶和试剂盒均购自Takara Biotechnology(日本)和Parkson基因技术有限公司(中国上海)。其他试剂均购自国药控股化学试剂有限公司(中国上海),具有最高分析级。
Luria-Bertani(LB)培养基和基本培养基用于细菌培养。LB培养基由NaCl 10 g·L-1,胰蛋白10 g·L-1,酵母提取物5 g·L-1和1 L H2O组成。根据需要使用1.5%琼脂固化。
鱼粉蛋白培养基用于蛋白降解菌分离筛选。鱼粉蛋白培养基由含 NaCl 0.5 g·L-1,MgSO4·7H2O 0.5 g·L-1,NH4Cl 2 g·L-1,KH2PO40.2 g·L-1,K2HPO41 g·L-1,H2O 1 L和0.5 mL微量培养基(CoCl2·6H2O 0.1 g·L-1,MnCl2·4H2O 0.425 g·L-1,ZnCl20.05 g·L-1,NiCl2·6H2O 0.01 g·L-1,CuSO4·5H2O 0.015 g·L-1,Na2MoO4·2H2O 0.01 g·L-1,Na2SeO4·2H2O 0.01 g·L-1,pH 7.0)组成无机盐培养基加入10 g·L-1鱼粉蛋白作为唯一碳氮源。
改良葡萄糖蛋白胨培养基用于测定不同碳氮源条件下菌株生长速率和蛋白质降解速率,葡萄糖10 g·L-1,NaCl 5 g·L-1,不同浓度蛋白胨(40、30、20、10、5、3.3和2.5 g·L-1)。碳氮源比分别为1∶1,1∶2,1∶3,1∶4,2∶1,3∶1,4∶1。
1.2 菌株分离鉴定
本研究所用枯草芽孢杆菌菌株采自四川省成都二龙桥鱼塘底部2~7 cm处泥层,用淤泥采样器取50 g左右泥样。采用文献[7-8]方法从养殖水体中分离高效蛋白质降解菌株。将10 g底泥接种到500 mL鱼粉蛋白培养基中,37℃180 r·min-1培养72 h,提取10 mL培养基接种于500 mL鱼粉蛋白培养基中,37℃180 r·min-1培养24 h。将少量培养基涂布在固体LB培养基上,37℃培养24 h。挑取单克隆菌斑,接种于LB培养基扩大培养。使用通用引物27F(5'AGAGTTTGATCCTGGCTCAG 3')和1492R(5'TACGGYTACCTTGTTACGACTT 3')PCR扩增筛选得到菌株,获得其16S rDNA。通过NCBI BLAST(https://blast.ncbi.nlm.nih.gov/)对该 16S rDNA序列线上比对,利用MEGA X构建其系统发育树[9]。
1.3 菌株生长速率和菌株蛋白质降解速率鉴定
本研究通过测定培养基中菌体干重变化计算不同培养条件下菌株生长速率。首先,将接种菌株的培养基在37℃180 r·min-1分别培养0、12和24 h后摇匀抽取50 mL,离心取沉淀加纯水50 mL混匀冲洗,然后离心取沉淀,重复3次。最后将沉淀烘干称重。
使用凯氏定氮法测定培养基中蛋白含量。将接种菌株LB培养基或鱼粉蛋白培养基在37℃180 r·min-1培养0和12 h后摇匀抽取50 mL,离心取上清液10 mL加入Viskase公司MD44透析袋(8 000~12 000 D)透析去除无机氮源、氨基酸和小分子寡肽。定量透析产物并使用凯氏定氮仪(上海赫冠仪器有限公司)测定蛋白质含量。
1.4 枯草芽孢杆菌HD-2菌株基因组DNA提取建库测序及生物信息学分析
HD-2基因组DNA提取使用TaKaRa公司生产细菌基因组提取试剂盒。完成基因组DNA抽提后,利用1%琼脂糖凝胶电泳检测收集基因组DNA。使用Covaris M220将基因组DNA打断为长度300~500 bp片段。使用TruSeqTMDNA Sample Prep Kit建库试剂盒构建测序文库并使用TruSeq PE Cluster Kit v3-cBot-HS试剂盒扩增。最终文库通过Illumina Hiseq 2000测序平台完成扫描测序。测序产生的DNA序列数据以FastQ格式记录,对原始测序数据质控并去除测序错误和低质量测序片段。使用SOAPdenovo v2.04拼接软件对测序片段组装。使用Glimmer 3.02软件预测细菌基因。
1.5 基因组规模代谢模型构建和生长模拟校验
使用 RAST软件(http://rast.nmpdr.org/)注释HD-2基因组预测得到的基因序列并通过MODEL SEED软件生成包含有完整Gene-Protein-Reaction(GPR)列表初始代谢草图。根据KEGG和BioCyc代谢数据完善并设定反应方向。使用MEMOTE软件评估模型完整性[10],填补模型缺口,最终构建菌株HD-2基因组规模代谢模型。
本试验使用Cytoscape2.8.2软件作网络可视化,并使用其Network Analyzer插件分析网络拓扑结构,查找核心功能所在子网络[11]。此后,使用COBRA2.0分析流平衡[12]。分别使用生长速率最大和单一产物量最大作为目标函数,函数如下:
maximize:biomass/target product
subject:S*v=0
0.01 ≤biomass≤0.02
通过模拟菌株在不同碳源和氮源条件下菌株最大生长速率,将模拟最大生长速率下碳氮比与试验中结果比较,评估模型准确性。
1.6 枯草芽孢杆菌不同碳氮源降解蛋白质预测
利用上述构建的GEMs模型,通过改变碳源氮源输入浓度,模拟aprE基因不同碳氮比营养条件下表达变化。将文献报道不同碳氮营养条件下枯草芽孢杆菌生长数据[3]与试验测量结果和模拟结果比较。将模拟结果中蛋白酶合成速率最大碳氮源比例等同于试验中测定蛋白质降解速率最大时碳氮源比例,总蛋白酶合成量作为目标函数,最大葡萄糖流量设定为0.0050 g·DW(dry weight,干重)-1·h-1,即包含0.0020 g碳元素碳源设置为碳源输入量,将包含0.0004 g氮元素 0.0025 g·DW-1·h-1总蛋白作为细胞外初始氮源输入量(初始条件:碳氮比为5∶1)。此后,以0.0005 g·DW-1·h-1梯度逐步提高细胞外氮源输入量至0.0125 g·DW-1·h-1作为细胞外最终氮源输入量,模拟碳氮比1∶1营养条件。分别模拟蛋白质降解速率最大时碳氮比和蛋白酶合成量最大时碳氮比。调整模型中各反应对应流量变化范围,使模拟结果与试验结果数据变化趋势一致。最后,利用调整后模型模拟分析连续梯度变化碳氮比条件下枯草芽孢杆菌蛋白质降解,确定枯草芽孢杆菌蛋白质降解最适碳氮源比例。
2 结果与分析
2.1 菌株选择与鉴定
本研究分离鉴定得到1株不溶性蛋白质高效降解菌株HD-2,12 h培养基中不溶性蛋白降解率达89.76%。表明HD-2具有高不溶性蛋白质降解能力。以枯草芽孢杆菌HD-2基因组DNA为模板,利用细菌通用引物扩增16S rDNA,1.5 kb处有1条特异性条带。纯化16S rDNA的PCR扩增产物,并作序列测定。结果表明,枯草芽孢杆菌HD-2的16S rDNA长度为1 531 bp。在GenBank中BLAST比对分析,结果表明与菌株HD-2的16S rDNA序列相似性最高的是枯草芽孢杆菌BS49和BS34A菌株,达100%。利用其亲缘关系标注系统发育树见图1,结果表明菌株HD-2属于芽孢杆菌属枯草芽孢杆菌。
2.2 枯草芽孢杆菌HD-2菌株基因组测序组装和基因预测结果
枯草芽孢杆菌HD-2菌株基因组扫描测序共产生读段(reads)15 412 018对,包含1 557 602 908个碱基,其中Q20以上reads占比92.77%。经质控保留有效reads共15 049 172对,1 415 852 922个碱基。有效reads组装得到206个长序列片段(scaffolds),包含5 753 251个碱基,测序reads覆盖深度234x,详细测序和组装结果见表1。
枯草芽孢杆菌HD-2的因预测结果包含5 585个可能基因序列,共4 953 741 bp个碱基。基因平均长度886 bp,基因座平均分布密度为每千碱基分布0.97个基因。基因中平均GC含量为68.1%。全部基因序列储存为ffn格式文件。基因组测序数据和组装结果已提交至NCBI Genome数据库,编号为SUB8287272。

图1 利用HD-2菌株16S rDNA序列构建系统发育树Fig.1 Phylogenetic analysis of the 16S rDNA of strain HD-2

表1 枯草芽孢杆菌HD-2菌株基因组组装结果Table 1 Results of B.subtilis strain HD-2 genome assembly
2.3 枯草芽孢杆菌HD-2菌株基因组注释
通过RAST在线快速注释软件对枯草芽孢杆菌HD-2菌株作基因组注释。注释结果中包含已知功能基因1 968个,分属30个主要子系统,包括各类生物大分子合成和代谢、细胞结构合成、化合物转运合成和生物防御运动等细胞过程。其中与蛋白酶合成直接相关氮代谢相关基因20个,蛋白质合成相关基因129个和蛋白质降解相关基因25个(见图2)。
HD-2编码外分泌蛋白酶是6个包含编码多个开放阅读框基因簇。这6种蛋白酶分别为胞外中性金属蛋白酶(EC 3.4.24.28)、解封氨肽酶(EC 3.4.11.-)、YpdF氨肽酶、D-丙氨酰-D-丙氨酸羧肽酶(EC 3.4.16.4)、耐高温羧肽酶1(EC 3.4.17.19)和氨肽酶PepA(EC 3.4.11.1)。
2.4 枯草芽孢杆菌HD-2基因组规模代谢模型iBac2019构建
本试验构建HD-2菌株基因组尺度代谢模型iBac2019。该模型分为细胞外和细胞质两个区域,共包含846个基因,1 394个反应和1 490种化合物,其中包含82个转运反应模拟大分子跨膜运输(见表2)。iBac2019还包含17个虚拟反应,模拟转录因子调控下游基因表达和被碳氮源供应量限制的表达水平。这17个虚拟反应包含存在于枯草芽孢杆菌中蛋白酶表达调控通路(见图3),将转录调控过程转化为虚拟生化反应,以便定量流平衡分析。
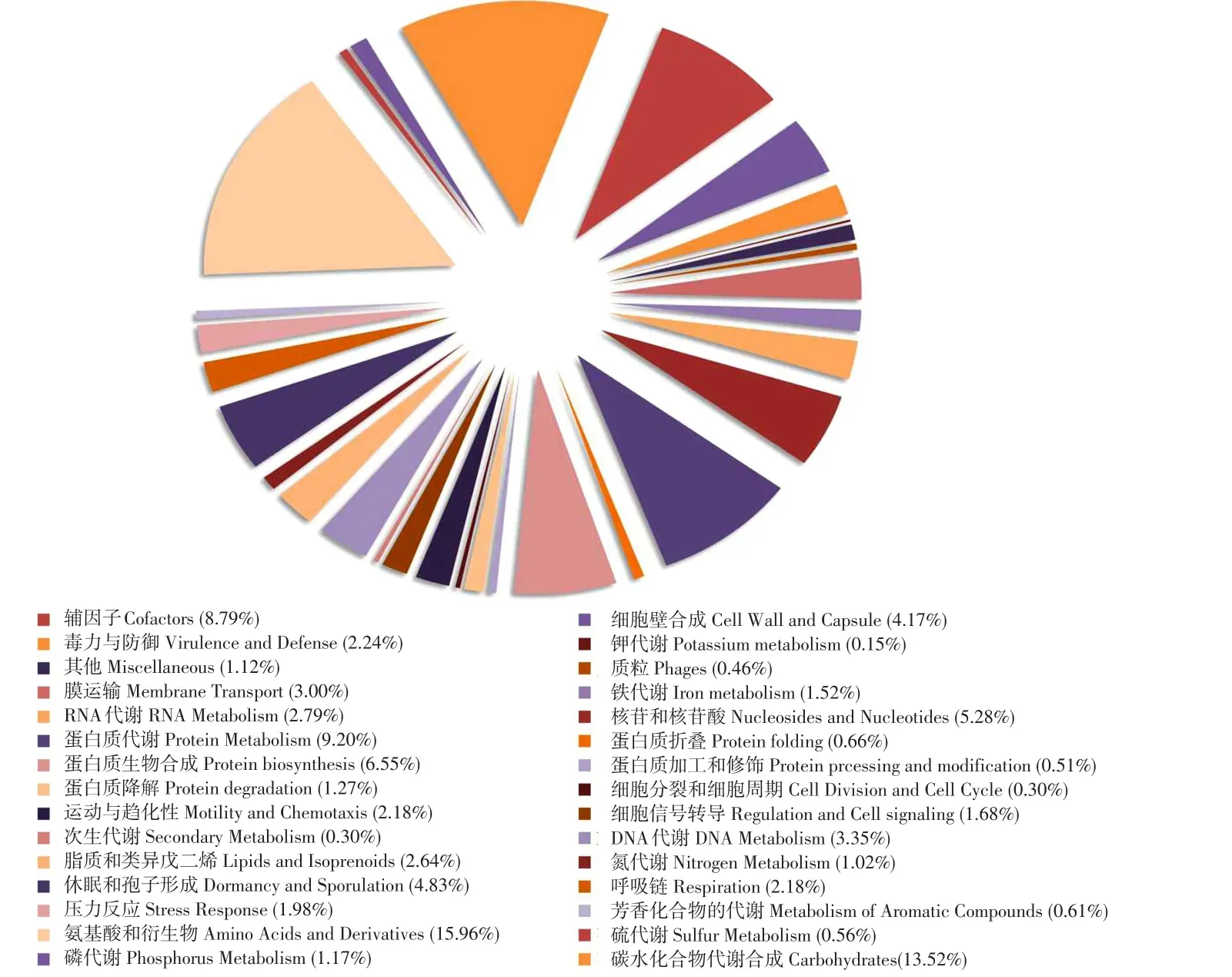
图2 枯草芽孢杆菌HD-2注释基因功能分类Fig.2 Gene annotation and classification in strain HD-2
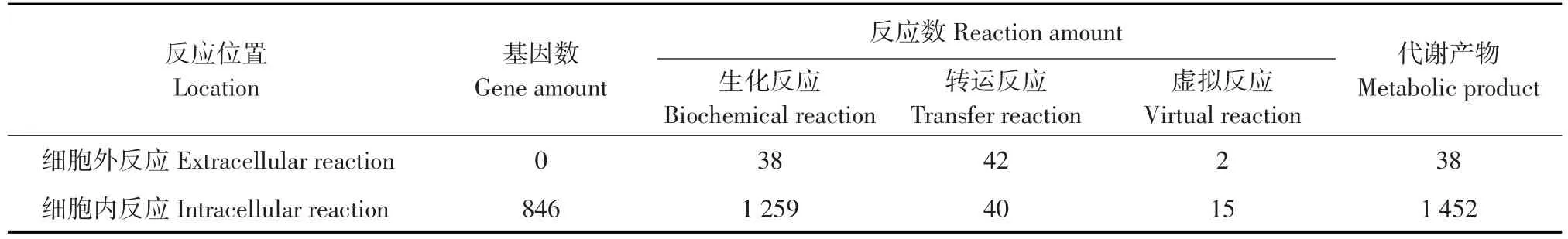
表2 iBac2019模型基本参数Table 2 A summary of GEMs model iBac2019

图3 枯草芽孢杆菌外泌蛋白酶表达调控通路Fig.3 Regulation pathways of exosprotease encoding genes in B.subtilis
2.5 iBac2019不同碳氮比下最大生长速率模拟
实验室分别测定培养基碳氮比为1∶1、1∶2、1∶4、2∶1、4∶1条件下培养12 h菌体干重。根据试验结果,所得不同碳氮条件下相应生长速率如下:碳氮比为1∶1时0.0124 g·DW-1·h-1;碳氮比为1∶2时0.0093g·DW-1·h-1;碳氮比为1∶4时0.0051g·DW-1·h-1;碳氮比为2∶1时0.0212 g·DW-1·h-1;碳氮比为4∶1时0.0083 g·DW-1·h-1(见表3)。使用基于约束的重构、分析和其软件中内置通量变异性分析,确定每个反应通量边界。在此基础上构建iBac2019模型Biomass方程,见附件2。
将目标条件设置为生长速率最大,模拟碳氮比为1∶3和3∶1条件下枯草芽孢杆菌最大生长速率分别为0.0072和0.0128 g·DW-1·h-1,与该条件下试验测量值相差8.3%(0.0066 g·DW-1·h-1)和18.7%(0.0104 g·DW-1·h-1),模拟值与测量值变化趋势吻合,表明模型仿真度较高。
2.6 通过目标产物最大化实验模拟蛋白质降解最适碳氮比
通过MCODE插件寻找模型中碱性蛋白酶合成和转运子网络。子网络拓扑分析表明蛋白酶合成通路和碳代谢通路关系紧密。通过与注释信息比较,选择模型中共有糖酵解、糖异生(30个反应),TCA循环(18个反应),丙氨酸、天冬氨酸代谢(15个反应)以及胞内、胞外蛋白水解(22个反应)4个代谢通路共85个反应组成蛋白酶分泌及其相关代谢反应生物功能模块。蛋白水解相关酶类共有胞内蛋白水解酶16个和胞外蛋白水解酶6个(见表4)。

表3 不同碳氮源比例条件下枯草芽孢杆菌HD-2生长速率和蛋白质浓度变化Table 3 Growth rate of B.subtilis HD-2 and the changes in protein concentration under different carbon-nitrogen ratio

表4 iBac2019模型中与蛋白降解相关的蛋白(酶)Table 4 Proteins(enzymes)related to protein degradation in iBac2019 model

续表
将该模块内总蛋白酶合成量作为目标函数,将最大葡萄糖流量设定为0.0050 g·DW-1·h-1,即包含0.0020 g碳元素设置为碳源输入量,将包含0.0004 g氮元素0.0025 g·DW-1·h-1总蛋白作为细胞外初始氮源输入量,初始条件下碳氮比5∶1。在此条件上,以g·DW-1·h-1梯度逐步提高细胞外氮源输入量至0.0125 g·DW-1·h-1总蛋白作为细胞外最终氮源输入量,模拟碳氮比达1∶1。将胞外总蛋白上限设置为输入蛋白量10倍,添加蛋白酶催化并转运蛋白进入细胞质虚拟反应。模拟菌株分泌蛋白酶,降解蛋白为可利用肽链并增加氮源输入流量。设置上述条件,利用流平衡分析模拟该模块,并记录蛋白酶合成量和菌株最大生长量(见图4)。
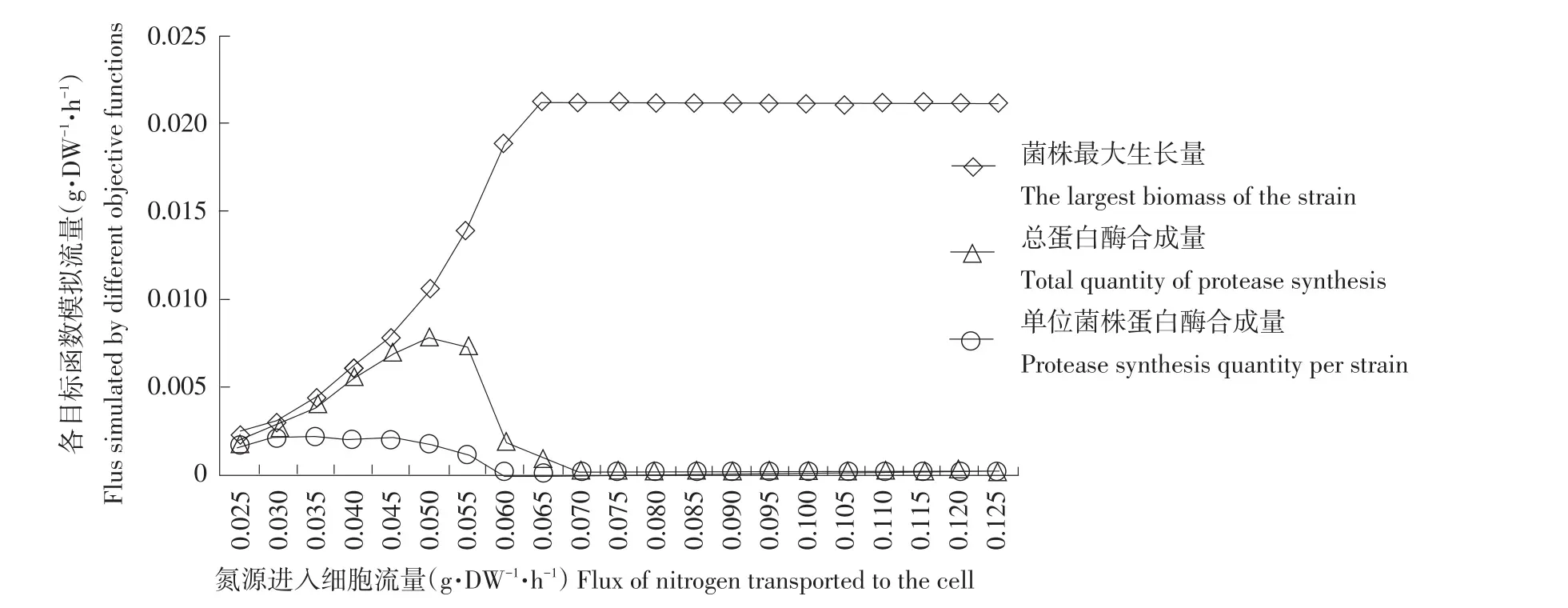
图4 不同氮源细胞流量下枯草芽孢杆菌代谢模型生长速率和蛋白酶合成速率模拟结果Fig.4 Simulation results of growth rate and protein synthesis in B.subtilis under different quantity of nitrogen source
结果表明,枯草芽孢杆菌在氮源进入细胞流量达到0.0650 g·DW-1·h-1时生长量达到顶峰,之后生长速率停止增加。即在碳氮比约为2∶1时,生长速率进一步增加开始受碳源供应量约束,单纯增加氮源供应无法提高菌株生长速度。氮源进入细胞流量增加至0.0350 g·DW-1·h-1时达到最大单位蛋白酶合成速率,氮源进入细胞流量因氮源供应不足而限制菌株蛋白酶合成。单位蛋白酶合成速率达到最大时进一步供应氮源虽增加菌株生长速率,但抑制蛋白酶合成分泌。最终,在考虑菌株生长速度和单位蛋白酶合成速率两个影响因素后,总蛋白酶合成量如图4所示,氮源进入细胞流量达0.05000 g·DW-1·h-1时达到最大,此时碳氮比约为2.8∶1。进一步试验验证,在碳氮比达2.6∶1时,枯草芽孢杆菌HD-2菌株溶液蛋白质降解能力最高,达2.0000 mg·L-1·h-1。
3 讨论
近年来,基因组规模代谢网络模型广泛应用于真核及原核生物。由于原核单细胞细菌有较为简单的基因组结构及代谢通路,其代谢模型研究最为深入有效。目前,GEMs已在药物生产、能源开发和健康监测等多方面发挥重要作用。药物生产方面,游动放线菌基因组规模代谢模型iYLW1028用于发酵模拟,显著提高阿卡波糖含量[13]。能源开发方面,代谢网络模型应用于提高蓝细菌生物乙醇生产效率[14]。在健康监测方面,肠道菌群在机体健康中发挥重要作用,肠道菌群全基因组规模代谢模型已广泛应用于集体营养状况检测等[15]。枯草芽孢杆菌作为一种模式细菌,其代谢网络模型已构建并发展至今[16]。与传统GEMs相比,本模型整合最新组学数据,显著提高预测代谢通量分布和表型能力。
枯草芽孢杆菌降氮能力运用广泛。据报道,枯草芽孢杆菌生物肥料可有效控制农业氨氮排放,保持较高农作物产量并减轻环境干扰[17]。此外,将枯草芽孢杆菌直接饲喂可降低犬粪便中氮发酵产物浓度和气味[18]。在农业及环境领域,筛选具有环境蛋白质降解能力枯草芽孢杆菌菌株意义重大。本研究在水产养殖环境中分离鉴定得到1株高效降解蛋白质菌株HD-2,利用其基因组扫描测序结果进一步探究其蛋白质降解潜能。
从机制上说,枯草芽孢杆菌可降解环境中不溶性蛋白质,通过分泌胞外酶(如aprE基因产物等)使蛋白质分解为多肽等,可进一步摄取产生自身所需含氮化合物[19]。同时可通过电子传递链将氨转化为氮气释放到空气中,起到对水环境等脱氮效果[20]。大量分泌的蛋白酶和其基因表达调控因子表明这些胞外蛋白酶对宿主枯草芽孢杆菌的重要性,但这也对枯草芽孢杆菌生长代谢造成负担,因此需根据其细胞所处营养状况和外界环境严格控制枯草芽孢杆菌胞外蛋白酶表达[4]。本研究利用系统计算方法,调整HD-2所处营养条件,预测其蛋白质降解能力,探究枯草芽孢杆菌HD-2最适蛋白质降解条件。与现阶段通过大规模筛选试验寻找蛋白质降解能力强菌株相比,本研究提出一种更高效便捷获得环境氮降解菌株的方法。
4 结论
本研究成功分离得到枯草芽孢杆菌菌株HD-2,通过序列测定确定其亲缘关系。基因组注释分析得到20个与蛋白酶合成直接相关氮代谢相关基因、129个与蛋白质合成相关基因、25个与蛋白质降解相关基因以及6个分泌胞外蛋白酶。此外,利用枯草芽孢杆菌HD-2多组学数据重构基因组规模代谢网络模型iBac2019。利用此模型,预测不同碳氮条件下蛋白水解相关胞内蛋白水解酶16个,胞外蛋白水解酶6个,获得蛋白质高效降解最优生长状态为碳氮比2.8∶1。通过试验验证预测结果准确性,发现碳氮比达2.6∶1时,枯草芽孢杆菌HD-2菌株溶液蛋白质降解能力最高。本研究成功将该模型应用于枯草芽孢杆菌对蛋白质降解能力预测,为从系统水平研究并筛选具蛋白质降解能力枯草芽孢杆菌提供理论基础。
