linc00619通过AKT/ERK信号抑制BEAS-2B细胞的增殖和迁移
2020-12-11吕业超凤羽龄周萍萍湛孝东唐小牛姜玉新
孙 甜,吕业超,凤羽龄,周萍萍,湛孝东,唐小牛,姜玉新,2
(1.皖南医学院 基础医学院,安徽 芜湖 241002;2.嘉兴学院 医学院,浙江 嘉兴 314000)
过敏性哮喘是一种长期的慢性气道炎性疾病[1]。慢性气道炎症、气道高反应性和气道重塑等是其主要临床病理特征[2-3]。气道上皮细胞增生引起的气道阻力增加是导致过敏性哮喘病程加重的主要因素[4]。作为常见的气传变应原,屋尘螨(house dust mite,HDM)可刺激螨性过敏患者的气道上皮细胞产生炎性应答、气道重塑、黏液过度增多,进而加重哮喘[5]。
长链非编码RNAs(lncRNAs)是一类长度>200 nt的非编码RNAs,参与包括细胞增殖和分化、细胞周期等诸多生理和病理生理过程的调控[6-9]。研究证实,lncRNAs在气道平滑肌细胞(airway smooth muscle cells,ASMCs)中发挥关键作用,进而调节哮喘的发病进程[10-11]。例如,浆细胞瘤变异易位1(plasmacytoma variant translocation 1,PVT1)在糖皮质激素不敏感的重度哮喘患者中的表达显著高于糖皮质激素敏感的轻度哮喘患者;抑制PVT1的表达可促进ASMCs增殖,同时抑制IL-6的释放[12]。RNA00882在血小板源性生长因子处理的胚胎ASMCs中显著高表达,抑制其表达导致胚胎ASMCs增殖下降[13]。BCYRN1通过上调瞬时受体电位1(transient receptor potential canonical 1,TRPC1)的表达来促进哮喘大鼠ASMCs的增殖和迁移[14]。GAS5通过控制miR-10a/BDNF信号促进哮喘ASMCs增殖[15]。此外,TCF7通过靶向TIMMDC1并活化Akt信号促进ASMCs增殖[16]。但目前大多数lncRNAs的功能未知。因此,探讨哮喘中lncRNAs对肺上皮细胞增殖分化的调控机制,不仅有利于深入认识哮喘的发病机制,也有助于为哮喘的免疫治疗提供新的实验证据。
本研究首先检测长链非编码RNA 00619(linc00619)的亚细胞定位;随后探讨了其对BEAS-2B生物学功能的影响;最后通过基因芯片分析linc00619对BEAS-2B影响的潜在分子机理,从而为过敏性哮喘的免疫治疗提供实验基础。
1 材料与方法
1.1 材料 细胞:293T和人肺上皮细胞BEAS-2B购自中科院上海细胞库。主要试剂:DMEM培养基、胎牛血清(FBS)购自上海Gibco公司。慢病毒载体pLV6购自上海吉玛制药技术有限公司。嘌呤霉素购自美国Merck公司。Genomic DNA Extraction kit购自日本Takara公司;Max DNA聚合酶购自南京维诺赞公司。CCK8检测试剂盒、RIPA裂解液购自上海生工生物。RNeasy mini Kit、RNase-free DNase set购自德国QIAGEN公司。SBC Human(4×180K) lncRNA芯片及配套试剂盒购自美国Agilent公司。FISH TagTMRNA Red Kit购自美国ThermoFisher公司。其他试剂为国产分析纯。
1.2 方法
1.2.1 细胞培养 293T或BEAS-2B细胞用DEME培养基(含10%FBS、1.5 mg/L谷氨酰胺、100 U/mL青霉素、100 μg/mL链霉素)中,于37℃、5%CO2饱和湿度下常规培养。
1.2.2 linc00619的亚细胞定位 按FISH TagTMRNA Red Kit说明书标记linc00619探针,并对BEAS-2B细胞进行RNA原位杂交和DAPI复染。
1.2.3 linc00619双突变载体的制备和细胞转染 根据linc00619(Gene ID:414260)核苷酸序列,设计3对双导引RNAs(dgRNA),即dgRNA1:CAT TAG AGG ACC GGA CAT GGT GG(+)和AAT AAA TCT GGA CCT ACA CAT GG(-);dgRNA2:CTT GCT TCT GAC GAA AGC TCA GG(+)和AGC CTA CAA TTC TAA GTA TTT GG(-);dgRNA3:CTC CAT TCC TGT GTT TAG GCA GG(+)和ATC TCG ATT CAA GTT TGT TGA GG(-),分别产生791、700和300 bp突变体。将它们分别插入慢病毒pLV6中。
将BEAS-2B细胞(2.5×105细胞/孔)铺于24孔板,加入0.5 mL含10% FBS的DMEM于37℃、5%CO2过夜;弃去培养液,加入0.5 mL稀释后的不同病毒液37℃、5%CO2培养12~24 h;弃去培养液并加入0.5 mL DMEM+10% FBS,37℃、5%CO2继续培养24~48 h;荧光显微镜观察细胞。
1.2.4 linc00619突变体的鉴定 用Genomic DNA Extraction kit提取BEAS-2B基因组DNA,PCR扩增用下列引物:正向5′-ACA GCT TGC TTG GCA CTG AAA TG-3′和反向5′-AAC CAT CCC CTA CCA TCA TCC AT-3′。PCR体系(50 μL):25 μL 2× Max buffer、1 μL dNTP Mix(10 mmol/L)、各2 μL正/反向引物(10 μmol/L)、200 ng基因组DNA、2.5 U Max DNA聚合酶、17 μL ddH2O。PCR反应程序:95℃变性3 min, 95℃ 5s、60℃ 15s、72℃ 40s循环42次,72℃ 5s,终延伸16℃ 1 min。
1.2.5 荧光定量PCR 采用Trizol一步法提取细胞总RNA,并进行逆转录。逆转录反应体系(10 μL):RNA模板1 μg,引物(50 pmol/L)1 μL,dNTP(10 mmol/L)1 μL,补充ddH2O至10 μL。65℃ 5 min,反应产物迅速置于冰上,加入2×逆转录缓冲液4 μL,逆转录酶100 U,ddH2O 5.5 μL。混匀后42℃ 60 min,75℃ 15 min,4℃保持。PCR反应(20 μL):2×PCR Master Mix 10 μL,正/反向引物(10 μmol/L)各0.4 μL,cDNA模板1 μL,补充ddH2O至20 μL。定量PCR反应程序:95℃预变性30 s,95℃ 10 s,60℃ 30 s,共40个循环。
1.2.6 细胞增殖试验 将100 μL linc00619敲除的BEAS-2B细胞(1×105细胞/孔)铺于96孔板,37℃、5%CO2培养24 h;弃去培养液,根据CCK8试剂盒说明书加入CCK8试剂,避光37℃、5%CO2孵育2 h;用酶标仪在450 nm处检测光密度值。
1.2.7 细胞迁移试验 向Transwell小室上层无血清DMEM培养液(含0.2% BSA)中加入linc00619敲除的BEAS-2B细胞(1×105细胞/孔),下层培养液为含10% FBS的DMEM,细胞培养48 h后,用DAPI染色,荧光显微镜下细胞计数。
1.2.8 Western blot 用PBS洗涤linc00619敲除的BEAS-2B细胞,加入100 μL/孔的RIPA细胞裂解液,4℃ 30 min,12 000 r/min 10 min收集上清,12.5% SDS-PAGE分离并转到PVDF膜,膜用1%的BSA 37℃封闭1 h后,加入鼠抗人p-AKT、AKT、p-MEK、MEK、p-ERK、ERK及β-actin一抗(1∶1000稀释)37℃孵育1 h,1×TBST缓冲液洗膜3次,5分钟/次。加HRP标记的驴抗鼠IgG二抗(1∶1000稀释),37℃孵育1 h,1×TBST缓冲液洗膜3次,5分钟/次。加化学发光液进行曝光。
1.2.9 芯片杂交、数据提取和分析 用Trizol一步法提取细胞总RNA,用NucleoSpin RNA Clean-up XS kit和RNase-Free DNase Set纯化。按照Agilent表达谱芯片套装试剂盒操作说明扩增总RNA和荧光标记,并用RNeasy mini kit纯化。将1.65 μg标记的cRNA用Gene Expression Hybridization Kit与芯片在65°C杂交17 h。Gene Expression Wash Buffer Kit清洗芯片后,用Agilent Microarray Scanner扫描芯片,Feature Extraction software 12.0读取数据,limma软件包对数据进行分析。
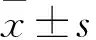
2 结果
2.1 linc00619定位于细胞质 用荧光标记的linc00619探针对BEAS-2B细胞进行linc00619 RNA原位杂交及DAPI复染结果显示,linc00619 RNA分布于细胞质(图1)。
2.2 linc00619敲除的鉴定 基因组PCR结果显示,dgRNA1和dgRNA3均产生预期大小的突变体,并用于后续研究。定量PCR结果表明,与对照组相比(1.006 3±0.116 6),dgRNA1~3均可抑制linc00619的表达,抑制效率分别0.001 1±0.000 2、0.000 3±0.000 1和0.004 6±0.000 7(P<0.01)(图2B)。但dgRNA2未检测到突变体(图2A),且其抑制linc00619表达的机制有待阐明。
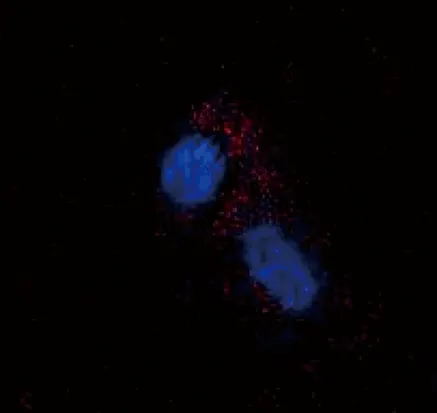
图1 linc00619定位于BEAS-2B细胞的细胞质(400×)
A.PCR扩增基因组DNA中linc00619突变体;B.荧光定量PCR分析linc00619敲除BEAS-2B细胞中linc00619表达(n=3,F=222.556,P=0.000;多组间两两比较,**P<0.01)。
2.3 linc00619敲除促进了BEAS-2B增殖和细胞迁移 CCK8结果表明,与对照组相比,dgRNA1介导linc00619敲除24 h促进了BEAS-2B的增殖(P<0.01),但dgRNA3对BEAS-2B的增殖影响无统计学意义(P>0.05);dgRNA1和dgRNA3介导linc00619敲除48 h均促进了BEAS-2B的增殖(P<0.05),但和dgRNA1相比,dgRNA1的促进更明显且差异有统计学意义(P<0.01)。dgRNA1介导linc00619敲除72 h促进了BEAS-2B的增殖(P<0.01),而dgRNA3抑制了BEAS-2B的增殖(P<0.01)。见表1。迁移结果显示,相比于对照组(215±8),dgRNA1(376±17)和dgRNA3(317±12)介导linc00619敲除均促进BEAS-2B的迁移(P<0.01)(图3)。
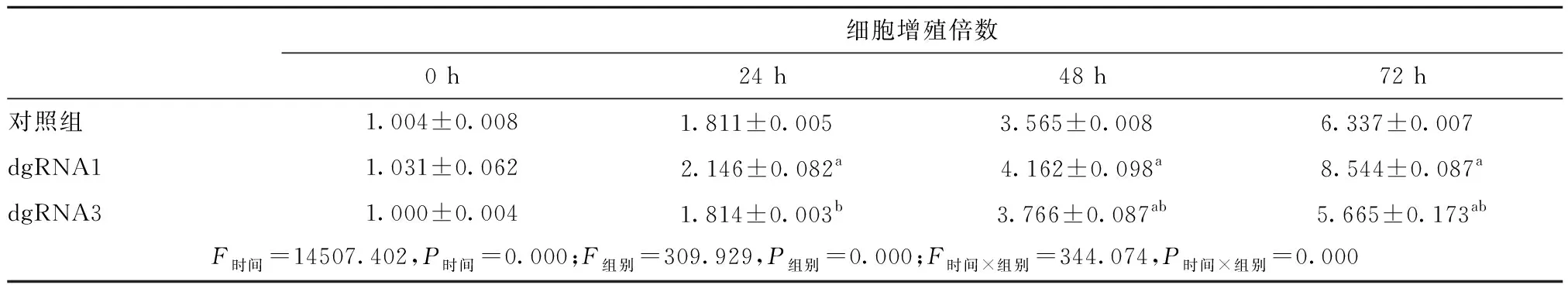
表1 linc00619敲除对BEAS-2B细胞增殖的影响
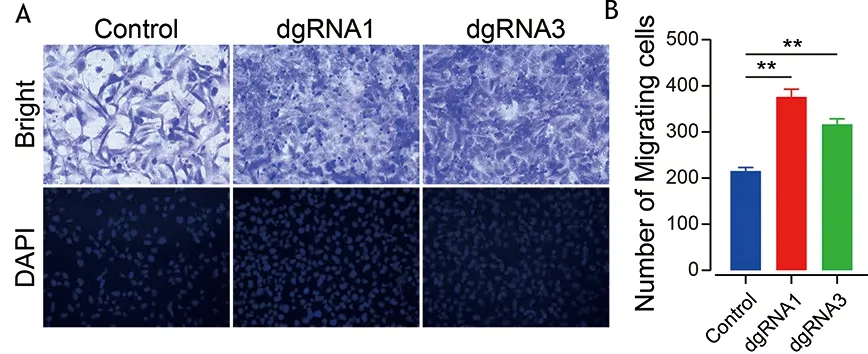
A.BEAS-2B细胞迁移的代表性图片(200×);B.计数并比较A中迁移的BEAS-2B细胞(n=3,F=120.139,P=0.000;多组间两两比较,**P<0.01)。
2.4 linc00619敲除对mRNA表达谱的影响 为探讨linc00619影响BEAS-2B细胞的潜在分子机理,我们用lncRNA芯片分析linc00619敲除的差异表达mRNAs,并进行KEGG信号富集。结果显示,共获得3 461个差异mRNAs(│差异倍数│>2),其中1 298个上调,2 163个下调(图4A)。有922个基因在KEGG信号通路被富集,图4B列出前30个信号通路,包括MAPK信号。本研究检测了增殖相关分子AKT、MEK和ERK的表达。结果表明,linc00619敲除活化了AKT和ERK [AKT:与对照组(0.57±0.11)相比,dgRNA1(1.29±0.05)和dgRNA3(1.18±0.14)升高(P<0.01);ERK:与对照组(0.82±0.13)相比,dgRNA1(2.44±0.05)和dgRNA3(1.38±0.03)升高(P<0.01)]。
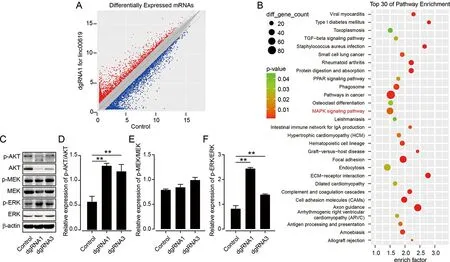
A.差异表达mRNAs的散点图;B.差异表达mRNAs的KEGG信号通路富集的排名前30的信号通路;C.Western blot分析AKT、MEK和ERK蛋白表达;D~F.对C中AKT、MEK和ERK蛋白的灰度分析(n=3,FAKT=39.588,PAKT=0.000;FMEK=2.389,PMEK=0.173;FERK=300.118,PERK=0.000。多组间两两比较,** P<0.01)。
3 讨论
本研究首先检测到linc00619位于细胞质。随后发现linc00619敲除促进了BEAS-2B细胞的增殖和迁移。芯片结果表明linc00619敲除导致MAPK信号显著富集。Western blot结果显示AKT和ERK被显著活化。这些结果表明,linc00619可能通过AKT/ERK信号参与了BEAS-2B细胞的生物学功能。
lncRNAs的亚细胞定位决定了其功能。细胞核lncRNAs可能参与包括染色质重塑和修饰及核小体等功能[17];而细胞质lncRNAs主要参与包括mRNA周转、mRNA修饰等[18-19]。本研究发现linc00619位于细胞质,提示其可能参与了转录后调节,其详细机理有待阐明。
lncRNAs在哮喘中也发挥重要作用。如Hu等用基因芯片筛选并获得了224个TGFβ处理的BEAS-2B中差异表达lncRNAs[20]。Li等发现,LncRNA-ENST00000501520可促进恶性BEAS-2B细胞的增殖[21]。此外,BEAS-2B中lncRNAuc001.dgp.1可增强PM2.5诱导的肺部炎症[22]。本研究发现,linc00619敲除促进了BEAS-2B细胞的增殖和迁移。Gao等也发现,lncRNA-DQ786227沉默抑制了恶性BEAS-2B细胞的增殖[23]。为清晰linc00619在BEAS-2B中的作用机理,我们用lncRNAs芯片筛选并获得了3 461个linc00619敲除的差异表达mRNAs。其中有922个基因被富集到KEGG信号通路,包括MAPK信号。Western blot证实,linc00619敲除活化了AKT和ERK。但其详细机理有待阐明。
综上所述,本研究表明,linc00619位于细胞质,敲除该分子导致BEAS-2B细胞的增殖和迁移增加,以及AKT和ERK的活化。但其具体的分子机制有待进一步深入研究。
