miRNA-210-3p在大肠癌细胞迁移侵袭中的促进作用及其靶基因的生物信息学分析
2020-10-26李婉明
潘 鑫 李婉明 于 敏 方 瑾
EffectofmiRNA-210-3pinInvasionofColorectalCancerCellsandBioinformaticAnalysisofTargetGenes.PanXin,LiWanming,YuMin,etal.DepartmentofCellBiology,KeyLaboratoryofCellBiology,MinistryofPublicHealth,KeyLaboratoryofMedicalCellBiology,MinistryofEducation,ChinaMedicalUniversity,Liaoning110122,China
AbstractObjectiveTo investigate the effect of miR-210-3p in colorectal cancer (CRC) cells migration and invasion and to analyze the enriched signaling pathways of target genes by bioinformatic methods.MethodsThe expression of miR-210-3p in colorectal cancer was analyzed by dbDEMC and OncomiR database. Real-time quantitative PCR was used to detecte the expression of miR-210-3p in five types of CRC cells (CL187,HCT-8,SW480, HCT-116,LoVo). miRNA mimics or inhibitor was transfected in CRC cells to up or down regulate the expression of miR-210-3p. Transwell assays were used to evaluated migration and invasion of CRC cells after transfection. Target gengs were pridected by miRDB, TargetScan7.2, starBase and miRPathDB. Gene ontology analysis and signal pathway enrichment were performed by DAVID.ResultsdbDEMC and OncomiR database showed that miR-210-3p was highly expressed in colorectal cancer, which was related to tumor metastasis and prognosis. Real-time quantitative PCR tested expression of miR-210-3p in highly invasiive cell lines(HCT-116 and LoVo) was significantly higher than that in moderate invasive cell lines SW480. And low invasive cell lines(CL187, HCT-8) revealed the lowest expression. Overexpression of miR-210-3p significantly enhanced SW480 cell migration and invasion, while LoVo cell migration and invasion was decreased after knockdown miR-210-3p. A total of 3035 target genes were predicted and the functions were mainly enriched in biological processes such as cell migration, adhesion, protein metabolism. The enriched pathways include cancer pathway, HIF signaling pathway and Rap1 signaling pathway. RGMA is significantly down regulated in clinical CRC tissues and may be the target gene of miR-210-3p.ConclusionmiR-210-3p can improve the migration and invasion abilities of colorectal cancer cells, which indicates that it might be a new biological target for colorectal cancer treatment.
KeywordsmiR-210-3p; Colorectal cancer; Invasion; Bioinformatic analysis; Target gene
大肠癌是消化系统常见的一种恶性肿瘤,其发生率和病死率居高不下。引发大肠癌患者死亡的主要原因是早期诊断不及时,确诊时约30%的患者已发生转移[1]。因此寻找肿瘤转移的分子标志物对了解大肠癌转移机制和提高患者预后有重要意义。microRNA(miRNA)是一类长约22个核苷酸的内源性非编码RNA,与靶mRNA 3′UTR结合,促进其降解或抑制其翻译,从而对基因表达进行负调控[2]。大量研究表明,miRNA在多种肿瘤中表达异常,参与肿瘤的发生、发展及转移[3~5]。
miR-210-3p位于人11号染色体短臂,多项研究表明其在多种肿瘤中发挥着重要的作用。研究报道,在三阴性乳腺癌、非小细胞肺癌、胰腺癌等恶性肿瘤中,miR-210-3p的表达水平与患者的临床病理特征相关,可以作为预后及诊断的生物学标志物[6~8]。在肺腺癌中,miR-210-3p通过抑制赖氨酰氧化酶样4(LOXL4)促进癌细胞增殖和迁移,发挥癌基因作用;而在膀胱癌中,miR-210-3p则发挥抑制肿瘤生长和转移的抑癌作用[9,10]。但在大肠癌中miR-210-3p的作用仍不清楚,本研究旨在探讨miR-210-3p对大肠癌细胞迁移和侵袭能力的影响,预测相关靶基因及其富集的GO功能和信号转导通路,为深入开展miR-210-3p对大肠癌的转移机制的研究提供理论依据。
材料与方法
1.细胞系及主要试剂:选取不同转移潜能的大肠癌细胞系,高转移细胞系HCT-116和LoVo,中低转移细胞系SW480,低转移细胞系CL187、HCT-8,以上细胞系购自中国科学院上海细胞库。miR-210-3p模拟物,miR-210-3p抑制剂,miRNA NC和Transfection Kit均购自广州锐博生物科技有限公司;miRNA Isolation Kit购自美国Invitrogen公司;miRNA反转录引物、反转录试剂盒、检测试剂盒(All-in-one miRNA qRT-PCR Detection Kit)购自广州复能公司;胎牛血清、RPMI1640和DEME培养基购自以色列BI公司。
2.细胞培养及转染: HCT-8、SW480、HCT-116和LoVo细胞培养于含10%胎牛血清的RPMI 1640培养基,CL187细胞培养于含10%胎牛血清的DMEM培养基,置于37℃、5% CO2培养箱中培养。将对数生长期的细胞接种于培养皿中,培养至细胞汇合度达到30%~50%。将miR-210-3p 模拟物、miR-210-3p抑制剂、miRNA-NC按照转染试剂盒说明书提供的方法转染至SW480细胞中,继续培养用于后续实验。
3.miRNA的提取及实时荧光定量PCR实验:在无RNA酶条件下严格按照miRNA提取试剂盒说明书提取细胞miRNA。将miRNA反转录合成cDNA后,进行实时荧光定量PCR扩增。以U6为内参,采用2-ΔΔCt法计算miRNA表达。
4.Transwell迁移、侵袭实验:实验分为空白对照组、阴性对照组、miR-210-3p 模拟物组、miR-210-3p 抑制剂组。用无血清培养基制备细胞悬液,调整细胞浓度至2.0×105个/毫升,接种至无基质胶包被或包被Matrigel基质胶的Transwell小室上层,下室加入600μl含10%胎牛血清的培养基,培养48h后,95%乙醇固定30min,4%台盼蓝染色30min,于显微镜下计迁出细胞数。随机选择5个视野计算平均值。
5.生物信息学分析:使用dbDEMC和OncomiR数据库分析miR-210-3p在大肠癌组织中的表达。利用TargetScan7.2、starBase、miRDB和miRPathDB进行miR-210-3p靶基因的预测,利用DAVID数据库对靶基因进行生物学过程富集分析和基于KEGG的Pathway信号通路的富集分析,利用GEPIIA数据库分析关键靶基因在临床病理组织中的表达。

结 果
1.miR-210-3p在大肠癌组织和癌细胞中的表达:dbDEMC数据库分析显示,大肠癌组织中miR-210-3p表达量明显高于癌旁组织(P<0.01);转移组织中miR-210-3p的表达量显著高于原发癌组织(P=0.000);OncomiR数据库分析显示,miR-210-3p高表达患者生存期低于miR-210-3p低表达组(P<0.05,图1)。荧光定量PCR结果显示,miR-210-3p在高转移性细胞LoVo、HCT-116中的表达水平高于中低转移性细胞SW480及低转移性细胞CL187和HCT-8(图2)。
2.miR-210-3p模拟物、抑制剂转染对大肠癌细胞迁移、侵袭能力的影响:Transwell迁移和侵袭实验结果显示,上调中低转移性SW480细胞内miR-210-3p的表达后,迁出和侵入小室的细胞数目明显多于对照组(图3A,P<0.05);下调高转移性LoVo细胞内miR-210-3p的表达后,迁出和侵入小室的细胞数目明显低于对照组(图3B,P<0.05)。
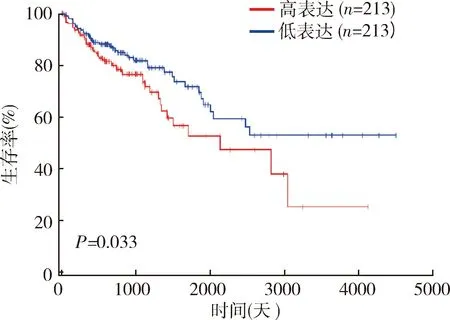
图1 miR-210-3p的表达与大肠癌患者的生存分析

图2 不同转移潜能大肠癌细胞中miR-210-3p的表达
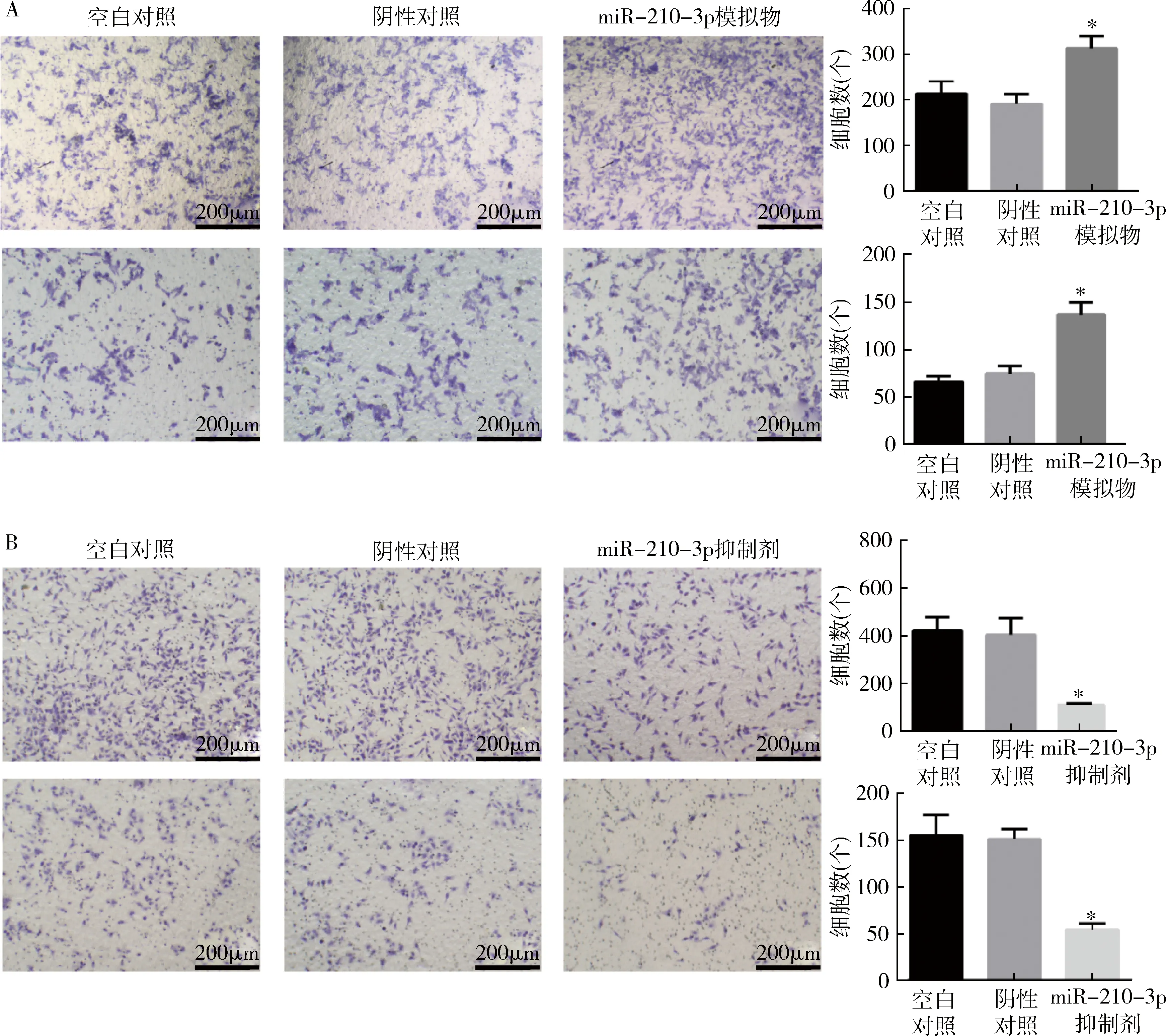
图3 转染miR-210-3p模拟物、抑制剂对大肠癌细胞迁移和侵袭能力的影响A.SW480细胞迁移和侵袭结果;B. LoVo细胞迁移和侵袭结果;与空白对照比较,*P<0.05
3.miR-210-3p靶基因的预测和分析:利用miRDB、TargetScan7.2、starBase和miRPathDB数据库对miR-210-3p的靶基因进行预测,得到3035个靶基因。选取两两数据库的交集进行GO生物学功能注释以及KEGG通路富集分析,在生物功能方面,靶基因功能主要富集在细胞迁移、细胞黏附、血管生成等;在细胞组分方面,主要富集在细胞核、胞质、吞噬小泡中;在分子功能方面,主要富集在转录因子活性、铁锌离子转运等;KEGG信号转导通路主要富集在癌症通路、HIF信号通路、Rap1信号通路等多个迁移相关信号转导通路(图4)。为了更为准确地筛选靶基因,笔者将预测到靶基因数据导入String数据库中分析蛋白间的相互作用,FGFRL1、E2F3、PTEN、BMP2、RGMa等编码蛋白质在相互作用网络图(PPI)中发挥稳定结构的作用(图5)。
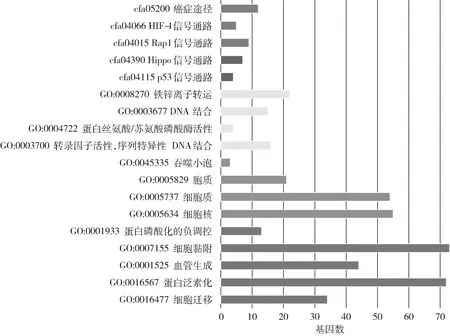
图4 候选基因GO分析以及KEGG分析蓝色.KEGG信号通路;黄色.分子功能;绿色.细胞组分;红色.生物学过程
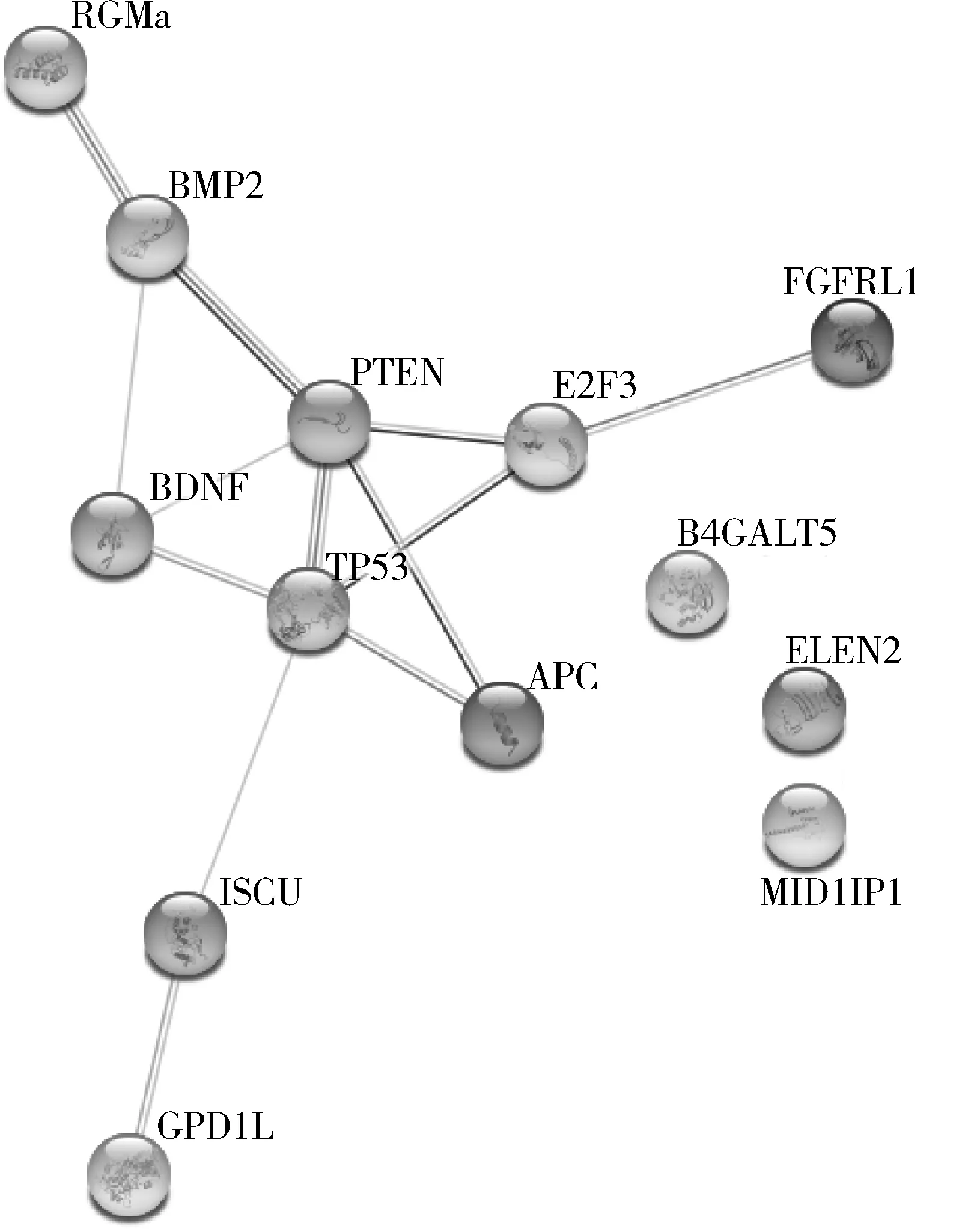
图5 候选基因PPI分析
4.关键靶基因验证:用GEPIA数据库进一步验证分析存在相互作用的靶基因在大肠癌组织(275例)和正常组织(349例)中的表达情况,结果发现,APC、GPD1L、ISCU、RGMa在大肠癌组织中的表达低于正常组织,其中RGMa的表达差异有统计学意义(P<0.05,图6)。
讨 论
miRNAs是一种内源性非编码RNA,与靶基因mRNA的3′UTR结合,在转录或转录后水平抑制mRNA的翻译,影响蛋白表达,从而发挥生物学功能[2]。近年来,大量研究证实miRNA不仅参与调控包括细胞增殖、分化、凋亡以及新陈代谢等各种正常的生理功能,更与多种肿瘤的发生、发展、转移以及耐药相关[11]。在大肠癌中,已报道多种miRNA存在表达异常,通过上调或下调表达抑制或者促进大肠癌细胞的增殖和转移[12~14]。因此,研究miRNA作用对于提高对大肠癌转移机制的认识,开发新的肿瘤转移标志物具有重要的临床意义。
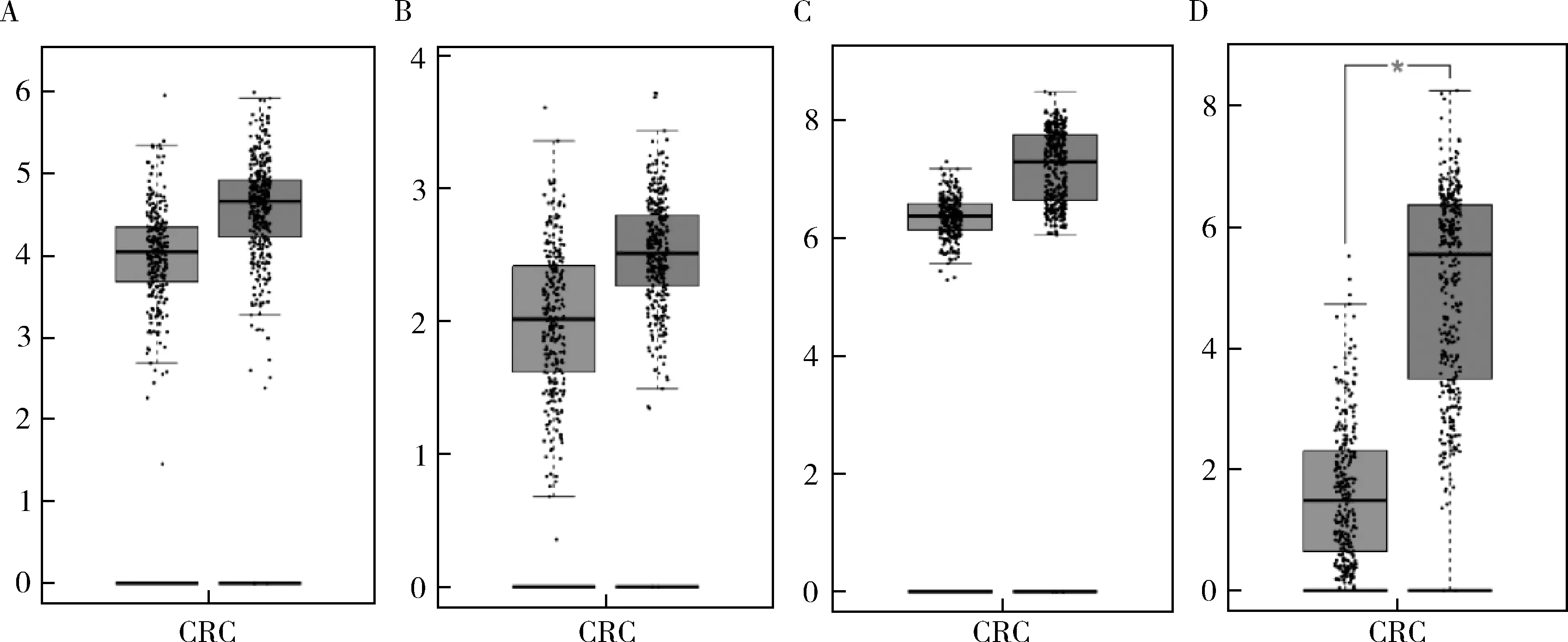
图6 关键基因在大肠癌组织及正常组织中的表达水平(肿瘤组织:n=275;正常组织:n=349)A~D分别为APC、GPD1L、ISCU、RGMa在肿瘤组织和正常组织中的表达情况。*P<0.05
大量研究证明miR-210-3p在多种肿瘤中表达异常,参与调控细胞增殖、凋亡、迁移、侵袭和药物敏感度等过程。Ren等[15]研究发现,miR-210-3p通过靶向SIN3A调节非小细胞肺癌细胞增殖和凋亡,Zhang等[16]研究结果显示,miR-210-3p可促进乳腺癌细胞迁移,Xie等[17]则发现下调miR-210-3p的表达可提高胆管癌的耐药性。然而miR-210-3p在大肠癌转移中的作用机制尚不清楚。信息学分析结果显示,miR-210-3p在大肠癌组织中的表达量高于癌旁组织,以及其在转移组织中的表达量显著高于原发癌组织,并且miR-210-3p高表达的患者生存时间明显低于miR-210-3p低表达组;荧光定量PCR结果进一步发现,miR-210-3p在高转移性大肠癌细胞中的表达量显著高于中低转移性大肠癌细胞,提示miR-210-3p的高表达可能起到促进大肠癌侵袭、转移的作用。在过表达miR-210-3p后,笔者观察到中低转移性SW480细胞的迁移和侵袭能力明显增强,而降低miR-210-3p表达后,高转移性LoVo细胞迁移和侵袭能力又明显减弱,说明miR-210-3p具有促进大肠癌细胞迁移和侵袭能力的作用。
为进一步探究miR-210-3p促进大肠癌迁移和侵袭的分子机制,笔者利用miRDB、TargetScan7.2、starBase和miRPathDB数据库对miR-210-3p的靶基因进行预测,发现在生物功能方面,靶基因功能主要富集在细胞迁移、细胞黏附、血管生成等;细胞组分主要富集在细胞核、胞质、吞噬小泡中;分子功能富集在转录因子活性、铁锌离子转运等;KEGG信号转导通路主要富集在癌症通路、HIF信号通路、Rap1信号通路等多个迁移相关信号转导通路。进一步分析靶基因的编码蛋白间的相互作用,发现FGFRL1、E2F3、PTEN、BMP2、RGMa等编码蛋白质在相互作用网络图中发挥稳定结构的作用,Yang等[10]已鉴定出FGFRL1可作为miR-210-3p的靶基因抑制膀胱癌的生长和转移,表明笔者的分析具有可行性和可信性。在临床大肠癌的肿瘤组织中,笔者发现关键基因的编码蛋白APC、GPD1L、ISCU、RGMa在大肠癌组织中低表达,其中RGMa的表达差异有统计学意义(P<0.05)。
排斥性导向分子a(repulsive guidance molecule,RGMa)是排斥性导向分子家族成员之一,被认为是细胞迁移、分化、铁稳态和凋亡等许多基本过程的关键分子,可通过新生素抑制血管新生,还可通过调控Smad信号抑制前列腺癌的细胞生长和黏附,发挥抑癌作用[18~20]。
已有报道显示其在结肠癌中经常发生遗传和表观失活而导致肿瘤发生恶性转变,在胶质母细胞瘤中还受miR-127-3p的抑制而调控细胞的恶性增殖。笔者通过生物信息学分析,预测出RGMa极有可能也是miR-210-3p的潜在靶基因,可能在大肠癌中起到抑制肿瘤转移的作用,因此后续将通过相关实验验证miR-210-3p与RGMa之间的相关性,以探索大肠癌的转移机制。
综上所述,miR-210-3p在大肠癌组织及转移性大肠癌组织中呈高表达,其过表达可增强大肠癌的细胞迁移和侵袭能力,发挥促癌作用。笔者预测了包括RGMa等在内的靶基因及其富集的信号通路,为miR-210-3p成为大肠癌转移的早期诊断指标和治疗靶点提供依据。
