稻瘟病菌细胞周期控制蛋白的生物信息学分析
2020-10-20方淑梅侯雪邱凯华孔祥森鞠世杰梁喜龙
方淑梅 侯雪 邱凯华 孔祥森 鞠世杰 梁喜龙


摘要:稻瘟病菌是重要的模式致病真菌,该菌引发的稻瘟病也是全球水稻最严重的病害之一,因此对稻瘟病菌的研究具有重要的学术意义和实际价值。细胞周期受多层次、多因子共同调控,其相关控制蛋白在真菌的形态建成、发育分化、逆境适应及致病性等方面发挥重要作用。为明确稻瘟病菌细胞周期控制蛋白的生物信息学特性,利用多种生物信息学软件和网站对获得的3种细胞周期控制蛋白Cwf19、Cwf16和Cwf14的理化性质、亚细胞定位、分子进化、翻译后修饰、空间结构、互作蛋白等进行分析,探讨了其可能的作用机制,为进一步利用反向遗传学手段深入研究其生物学功能奠定基础。
关键词:稻瘟病菌;细胞周期控制蛋白;生物信息学
中图分类号:S435.111.4+1 文献标志码: A
文章编号:1002-1302(2020)18-0060-06
收稿日期:2020-07-08
基金项目:黑龙江省自然科学基金(编号:C2016047);黑龙江省教育厅项目(编号:12531452);黑龙江八一农垦大学研究生创新科研项目(编号:YJSCX2019-Y14)。
作者简介:方淑梅(1977—),女,辽宁东港人,博士,副教授,主要从事植物逆境分子生物学等方面的教学和科研工作。E-mail:fangshumei520@126.com。
通信作者:梁喜龙,博士,教授,主要从事植物化控及生物逆境方面研究。E-mail:xilongliang@126.com。
稻瘟病菌(Pyricularia oryzae or Magnaporthe oryzae)是重要的模式植物致病真菌,在真菌的科学研究中具有重要地位。而且由稻瘟病菌引起的稻瘟病是在世界范围内严重影响水稻产量和品质的最严重的病害之一,调查显示全球受影响国家多达80个,每年受稻瘟病灾害导致的水稻减产率高达全世界水稻总产量的10%~30% [1]。而我国一直以来水稻种植业饱受稻瘟病的困扰,每年的产量损失高达30亿kg以上。另外稻瘟病菌还可侵染小麦、大麦和黍等禾本科作物[2]。因此对稻瘟病菌致病分子机制的研究对于发现防治稻瘟病菌的特异性作用靶点,为高效药物研发提供科学可行的重要理论基础。
细胞周期是受多层次、多因子共同调控的影响细胞分裂、分化和凋亡等所经历的过程。 细胞周期控制蛋白(cell cycle control protein)参与细胞正常运转的蛋白质,是真核生物生命进程中重要的调控因子,可通过调控细胞周期而参与生命体的形态建成、生长发育、细胞分化、环境适应及疾病发生等诸多过程[3-8]。研究显示,许多pre-mRNA剪接因子是重要的细胞周期控制蛋白,通过参与剪接体的形成与RNA或其他蛋白结合而在剪接过程中发挥作用,从而影响基因表达[9-13]。
随着稻瘟病菌基因组数据库的公布以及遗传重组手段的日益成熟,稻瘟病菌致病相关基因挖掘工作取得重要进展[14-15]。然而对稻瘟病菌细胞周期控制蛋白的研究仍然较少。为此,笔者所在实验室利用生物信息学方法分析了稻瘟病菌中的细胞周期控制蛋白的分子进化、理化性质、结构特征、序列特征及互作蛋白等,探讨其可能的作用机制,旨在为进一步利用反向遗传学手段研究其生物学功能奠定基础。
1 材料与方法
1.1 稻瘟病菌细胞周期控制蛋白的获取与确定
利用稻瘟病菌数据库网站(http://fungi.ensembl.org/Magnaporthe_oryzae/Info/Index),输入检索词“cell cycle control protein”搜索,获取所有细胞周期控制蛋白的基因ID和基因在染色体中的位置、外显子数、编码外显子数等信息,并利用MapChart 2.2软件,参照基因和染色体长度按比例进行绘制。
1.2 稻瘟病菌细胞周期控制蛋白的理化特性及修饰位点预测
利用在线工具ProtParam(https://web.expasy.org/protparam)预测稻瘟病菌细胞周期控制蛋白的亲水性和稳定性;利用在线软件SignalP-5.0 Server(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白质信号肽,参数设置为默认值。利用在线软件DISPHOS1.3(http://www.dabi.temple.edu/disphos/)预测蛋白质磷酸化位点,利用软件GPS-SUMO 1.0预测蛋白质的Sumo化位点,Sumoylation threshold和SUMO interaction threshold均设置为中等。
1.3 稻瘟病菌细胞周期控制蛋白的系统进化分析
将稻瘟病菌细胞周期蛋白的氨基酸序列提交到NCBI(http://www.ncbi.nlm.nih.gov),利用序列同源性获取同源蛋白,整理为.fasta格式,然后利用软件ClustalX 1.83进行氨基酸序列的多重匹配分析,参数设为默认值,进一步用MEGA 4.0软件转化为.meg文档后,利用Neighbor-Joining法绘制系统进化树。
1.4 稻瘟病菌细胞周期控制蛋白的空间结构预测
首先利用在线软件NPS@SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl? page=/NPSA/npsa_sopma.html)进行二级结构分析,窗口宽度17,相似性阈值8。利用SWISS-MODEL(https://swissmodel.expasy.org/)在線网站预测蛋白质的三级结构,获取3D模型及相关信息。
1.5 稻瘟病菌细胞周期控制蛋白的互作蛋白分析
互作蛋白预测利用STRING 11.0在线软件(https://string-db.org/cgi/input.pl)进行,minimum required interaction score 设置为高置信度(high confidence)0.700,互作蛋白来源于试验证实(Experiments)。
2 结果与分析
2.1 稻瘟病菌细胞周期控制蛋白及基因信息确定
通过在稻瘟病菌数据库网站搜索,获得注释为细胞周期控制蛋白的基因ID共3个,分别为 MGG_00102、MGG_12308和MGG_06309(表1),其所对应的蛋白分别为Cwf19、Cwf16和Cwf14,来源菌株均为Pyricularia oryzae 70-15。
2.2 稻瘟病菌细胞周期控制蛋白基因的染色体定位
由表1和图1可见,3种稻瘟病菌细胞周期蛋白基因MGG_00102、MGG_12308和MGG_06309分别位于5号、2号和4号染色体上,其中MGG_00102位于染色体末端,而MGG_12308和MGG_06309均分布在靠近染色体末端1/4处。
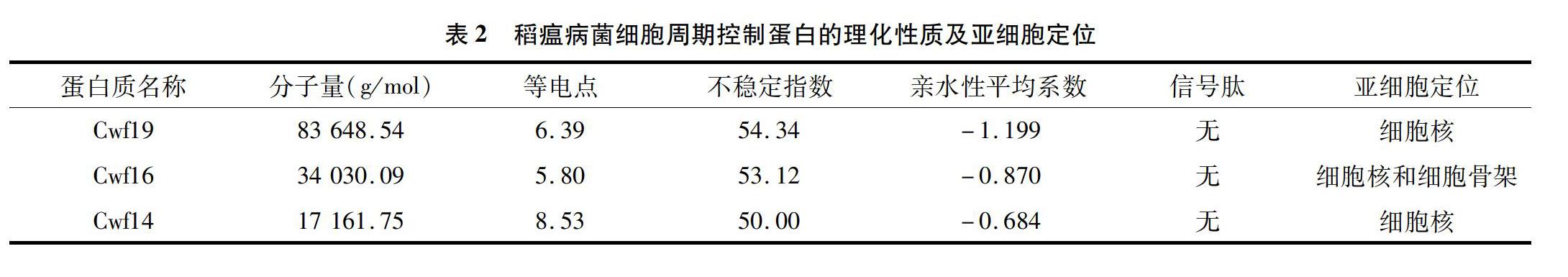
2.3 稻瘟病菌细胞周期控制蛋白的理化特性与亚细胞定位
由表2可以看出,3种蛋白质的分子量差异较大,其中Cwf14分子量最小,为17 161.75 g/mol,Cwf16大约是它的2倍,而Cwf19分子量最大,大约是它的5倍;在等电点方面,Cwf14偏碱性,而Cwf19和Cwf16偏中性;3种蛋白均为不稳定的亲水性蛋白,均无信号肽,亚细胞定位分析显示Cwf19和Cwf14仅存在于细胞核,Cwf16除位于细胞核外,还参与细胞骨架构成。
2.4 稻瘟病菌细胞周期控制蛋白修饰位点预测
利用DISPHOS在线网站预测细胞周期控制蛋白磷酸化修饰位点显示,Cwf19磷酸化位点共20个, 其中Ser 15个、Thr 2个、Tyr 3个;Cwf16磷酸化位点共5个,其中Ser 4个、Thr 1个,无Tyr磷酸化;Cwf14中无磷酸化位点。GPS-SUMO软件预测3种蛋白质的SUMO化位点结果如表3所示,Cwf19的具有1个SUMO化位点(181Lys);Cwf16具有2个SUMO化位点(28Lys和156Lys)和1个SUMO互作基序(185-189Val-Val-Val-Ala-Gln);Cwf14具有1个SUMO化位点(129Lys)。其中,距离Cwf19 SUMO化位点最近的磷酸化位点为173 Ser和190Ser,距离Cwf16 SUMO化位点最近的磷酸化位点为21Thr、36Thr和152Thr。
2.5 稻瘟病菌细胞周期控制蛋白的系统进化分析
为了明确稻瘟病菌细胞周期控制蛋白的进化关系,本研究利用MEGA 4.0软件,采用Neighbor-Joining法绘制了系统进化树。如图2所示,3 种细胞周期控制蛋白均各自形成一组分支,表明他们在真菌的不同种属间具有保守性。其中Cwf19,稻瘟病菌与小麦全蚀病菌(Gaeumannomyces tritici)聚在一起,具有相同节点,亲缘关系最近,而与新型隐球菌(Cryptococcus neoformans)亲缘关系最远;对于Cwf16,稻瘟病菌与Microdochium bolleyi和Phaeoacremonium minimum具有较近的亲缘关系,与二孢白粉菌(Golovinomyces cichoracearum)的亲缘关系最远;而Cwf14与稻瘟病菌亲缘关系最近的是夏季斑枯病菌(Magnaporthiopsis poae),与向日葵间座壳菌(Diaporthe helianthi)的亲缘关系最远。
2.6 稻瘟病菌细胞周期控制蛋白二级结构预测
本研究中细胞周期控制蛋白二级结构预测结果如图3所示。由图3可以看出,3种细胞周期控制蛋白的二级结构均以α螺旋为主,所占比例远高于其他2种形式。另外,Cwf16的α螺旋、β折叠和β转角所占比例均最高于另外2种蛋白,表明其无规卷曲所占比例最小。
2.7 稻瘟病菌细胞周期控制蛋白的三级结构
正确的空间构象是蛋白质具有特定生物学功能的前提。本研究利用在线预测软件SWISS-MODEL预测细胞周期控制蛋白的3D结构,结果如表4和图4所示。Cwf19的预测模板为6id0.1.R,三级结构由2个大小不同的可能的结构域构成;Cwf16的预测模板为6exn.1.F,三级结构由1个结构域和1个αα超二级结构构成;Cwf14的预测模板为3jb9.1.Z,为由1个结构域构成的蛋白质分子。
2.7 稻瘟病菌细胞周期控制蛋白的互作蛋白预测分析
很多蛋白质分子尤其是调控蛋白,在细胞内通常都会与其他调控蛋白相互作用而发挥功能。本研究利用STRING在线软件分别对3种细胞周期控制蛋白的互作蛋白进行预测。所有互作蛋白分子均来源于试验证实,最低互作阈值(minimum required interaction score) 设置为0.700。结果如表5所示,这3个蛋白的互作蛋白均为pre-mRNA剪接/加工因子或剪接体的组分,而且有些为他们共同的互作蛋白,如MGG_08133和MGG_08641同时与Cwf19、Cwf16和Cwf14互作,除此之外,MGG_01426和MGG_03116是Cwf19与Cwf16共同的互作蛋白,MGG_05616是Cwf19与Cwf14共同的互作蛋白,MGG_13500是Cwf16与Cwf14共同的互作蛋白。因此他们可能在pre-mRNA剪接过程中共同发挥作用。
3 结论与讨论
对细胞周期调控的探索一直是真核生物重要的研究内容之一。在真菌,许多细胞周期相关蛋白不断地被发现和解读,如Cln家族、Clb家族等,他们通过参与相关的信号通路而调控细胞的生长、发育以及致病菌株的毒力 [16-17]。除了这些直接作用的蛋白质家族之外,细胞核中存在大量的调控蛋白,如剪接体构成蛋白及剪接因子,他们通过调控基因表达而参与细胞周期的控制[9-13]。本研究中的3种细胞周期控制蛋白均存在于细胞核内,以α螺旋为主要二级结构的不稳定亲水性蛋白,他们的互作蛋白均为pre-mRNA剪接/加工因子或剪接体组分,因此推测这3种蛋白也是pre-mRNA剪接/加工因子或参与剪接体构成的蛋白质,參与 pre-mRNA 剪接或选择性剪接。而且这3种蛋白在发挥作用时具有共同的互作蛋白,并且其中的MGG_01426和MGG_08641都参与细胞周期的控制过程,表明他们可能在共同的调控细胞周期运转的途径中发挥作用。
SUMO(small ubiquitin-like modifier)化修飾通常指SUMO分子(76个氨基酸构成的肽)通过其C端Gly结合于目的蛋白质分子的Lys残基上,也有些SUMO分子通过目的蛋白的SUMO互作基序(SUMO-interacting motifs,SIMs)与之结合,是常见的翻译后修饰过程[18-19]。研究显示SUMO化修饰参与蛋白质的多种功能调节,包括影响蛋白质的稳定性、核酶活性、蛋白质之间的相互作用、蛋白质与核酸结合及亚细胞定位等[20-25]。本研究中的3个细胞周期蛋白均具有结合SUMO分子的位点或互作基序,因此SUMO化修饰可能在其发挥分子定位或参与剪接体组装及作用中发挥作用。然而研究显示并不是所有预测可能的SUMO位点都会真正地被SUMO分子结合,磷酸化修饰也可能在底物的SUMO化中发挥重要作用。Hietakangas等报道Lys-x-Glu-x-x-Ser-Pro是SUMO化位点的一致性序列,其中Ser的磷酸化可影响Lys的SUMO化 [26-27]。虽然本研究并未在这3种细胞周期控制蛋白中发现这种保守的序列,但是磷酸化与SUMO化之间的关系值得深入研究。
参考文献:
[1]Talbot N J. On the trail of a cereal killer:Exploring the biology of Magnaporthe grisea[J]. Annu Rev Microbiol,2003,57:177-202.
[2]Skamnioti P,Gurr S J. Against the grain:safeguarding rice from rice blast disease[J]. Trends Biotechnol,2009,27:141-150.
[3]Dorter I,Momany M. Fungal cell cycle:A unicellular versus multicellular comparison[J]. Microbiol Spectr,2016,4:551-570.
[4]Saunders D G,Aves S J,Talbot N J. Cell cycle-mediated regulation of plant infection by the rice blast fungus[J]. Plant Cell,2010,22:497-507.
[5]Steinberg G,Perez-Martin J. Ustilago maydis,a new fungal model system for cell biology[J]. Trends Cell Biol,2008,18:61-67.
[6]Whiteway M,Bachewich C. Morphogenesis in Candida albicans[J]. Annu Rev Microbiol,2007,61:529-553.
[7]Perezmartin J,Bardetti P,Castanheira S,et al. Virulence-specific cell cycle and morphogenesis connections in pathogenic fungi[J]. Semin Cell Dev Biol,2016,57:93-99.
[8]Susumu K,Zuzana M,Eric V,et al. Cell cycle regulation and hypoxic adaptation in the pathogenic yeast Cryptococcus neoformans[J]. FASEB J,2018,32:533.
[9]Tavanez J P,Caetano R,Branco C,et al. Hepatitis delta virus interacts with splicing factor SF3B155 and alters pre-mRNA splicing of cell cycle control genes[J]. FEBS J,2020,PMID:32352217.
[10]Vijaykrishna N,Melangath G,Kumar R,et al. The Fission yeast pre-mRNA-processing factor 18 (prp18+) has intron-specific splicing functions with links to G1-S cell cycle progression[J]. J Biol Chem,2016,291:27387-27402.
[11]Chawla G,Sapra A K,Surana U,et al. Dependence of pre-mRNA introns on PRP17,a non-essential splicing factor:implications for efficient progression through cell cycle transitions[J]. Nucleic Acids Res,2003,31:2333-2343.
[12]Dahan O,Kupiec M. The Saccharomyces cerevisiae gene CDC40/PRP17 controls cell cycle progression through splicing of the ANC1 gene[J]. Nucleic Acids Res,2004,32:2529-2540.
[13]Saha D,Banerjee S,Bashir S,et al. Context dependent splicing functions of Bud31/Ycr063w define its role in budding and cell cycle progression[J]. Biochem Bioph Res Co,2012,424:579-585.
[14]Dean R A,Talbot N J,Ebbole D J,et al. The genome sequence of the rice blast fungus Magnaporthe grisea[J]. Nature,2005,434:980-986.
[15]董妍涵. 稻瘟病菌的組学分析和致病相关基因的挖掘及功能研究[D]. 南京:南京农业大学,2014.
[16]Dirick L,Bohm T,Nasmyth K. Roles and regulation of Cln-Cdc28 kinases at the start of the cell cycle of Saccharomyces cerevisiae[J]. EMBO J,1995,14:4803-4813.
[17]Linke C,Chasapi A,Gonzalez-Novo A,et al. A Clb/Cdk1-mediated regulation of Fkh2 synchronizes CLB expression in the budding yeast cell cycle[J]. NPJ Syst Biol Appl,2017,3:7.
[18]Kerscher O. SUMO junction-whats your function? New insights through SUMO-interacting motifs[J]. EMBO Rep,2007,8:550-555.
[19]Seeler J S,Dejean A. Nuclear and unclear functions of SUMO[J]. Nat Rev Mol Cell Bio,2003,4:690-699.
[20]Jackson SP,Durocher D. Regulation of DNA damage responses by ubiquitin and SUMO[J]. Mol Cell Biol,2013,49:795-807.
[21]Ulrich H D. Two-way communications between ubiquitin-like modifiers and DNA[J]. Nat Struct Mol Biol,2014,21:317-324.
[22]Jentsch S,Psakhye I. Control of nuclear activities by substrate-selective and protein-group SUMOylation[J]. Annu Rev Genet,2013,47:167-186.
[23]Altmannova V,Kolesar P,Krejci L. SUMO wrestles with recombination[J]. Biomolecules,2012,2:350-375.
[24]Sarangi P,Zhao X. SUMO-mediated regulation of DNA damage repair and responses[J]. Trends Biochem Sci,2015,40:233-242.
[25]Ranjha L,Levikova M,Altmannova V,et al. Sumoylation regulates the stability and nuclease activity of Saccharomyces cerevisiae Dna2[J]. Commun Biol,2019,2:174.
[26]Hietakangas V,Anckar J,Blomster H A,et al. PDSM,a motif for phosphorylation-dependent SUMO modification[J]. PNAS,2006,103:45-50.
[27]Hietakangas V,Ahlskog JK,Jakobsson AM,et al. Phosphorylation of serine 303 is a prerequisite for the stress-inducible SUMO modification of heat shock factor 1[J]. Mol Cell Biol,2003,23:2953-2968.
