基于环境DNA宏条形码的洱海鱼类多样性研究
2020-10-15林佳艳封吉猛彭作刚
舒 璐 林佳艳 徐 源 曹 特 封吉猛 彭作刚
(1. 西南大学生命科学学院, 淡水鱼类资源与生殖发育教育部重点实验室, 重庆 400715; 2. 中国科学院水生生物研究所, 武汉430072; 3. 上海交通大学云南(大理)研究院, 大理 671000)
鱼类多样性是水生生物多样性的重要组成, 保护鱼类多样性有利于水生态系统的健康与稳定。传统的鱼类多样性监测主要利用电捕、网捕与陷阱诱捕等方式采集渔获物, 通过形态学鉴定和计数称重, 识别鱼类物种种类并统计鱼类丰度与生物量[1,2]。然而这类方法普遍存在生态破坏性大、费时费力、形态鉴别困难和稀有物种捕获率低等缺点。新兴的环境DNA宏条形码(Environmental DNA metabarcoding; eDNA metabarcoding)技术能够通过直接提取环境样品(如水、沉积物、土壤等)中DNA,运用针对目标类群的通用引物, 通过PCR扩增结合高通量测序(High-throughput sequencing, HTS),对环境样本中存在的多个目标物种进行识别[3]。整个过程无需采集目标生物, 以非破坏性采样、操作简易高效和检测灵敏度高的优势弥补了传统形态学监测的不足, 对生物多样性评估具有极大的应用潜能[4]。
近年来, 环境DNA宏条形码已被广泛运用于淡水和海洋生态系统的渔业管理与鱼类多样性监测中[5]。Evans等[6]使用3对鱼类通用引物从面积为0.022 km2的水库中检测出过去传统方法监测到的所有鱼类物种, 另外还检测到11种过去未捕获过的鱼类。Sigsgaard等[7]利用12S rRNA遗传标记成功监测到丹麦沿海鱼类群落结构的季节变化。尽管环境DNA宏条形码的采样方案与分析流程的各项环节在不同的研究中有不同的策略与优化, 但成熟完整的标准化操作流程也已形成, 保障了该技术在野外调查的成功应用[8]。目前国内运用环境DNA宏条形码调查鱼类多样性的研究仍然不多[9—11]。环境DNA宏条形码在国内水生态系统的鱼类多样性监测与保护领域还具有巨大的应用空间。
洱海位于云南省大理白族自治州境内, 湖泊面积约252 km2, 是云南省第二大淡水湖泊, 具有丰富的鱼类多样性和较高的渔业利用价值[12]。洱海土著鱼类历史记录有18种, 其中土著特有种8种[13]。20世纪60年代以来, 随着过度捕捞、外来鱼类引入和水体污染等人类活动干扰, 洱海鱼类区系结构发生了重大变化, 土著鱼类多样性面临严重危机, 其中7种土著特有种成为濒危物种[13,14]。洱海鱼类多样性与生态平衡问题受到国家和政府的密切关注,近年来当地政府对洱海实施生态鱼种增殖放流, 通过土著鱼类的投放恢复, 期望改善洱海鱼类区系组成, 保护鱼类多样性, 发展可持续的生态渔业[15]。
洱海鱼类多样性保护与生态恢复迫切需要建立快速有效和环境友好的监测机制, 为渔业管理以及生态保护政策的制定与实施提供科学依据。本研究首次运用环境DNA宏条形码技术对洱海鱼类多样性进行初步评估, 旨在探讨环境DNA宏条形码技术在检测洱海鱼类多样性上的可行性, 为洱海生物多样性监测以及水生生态保护提供新的技术手段。
1 材料与方法
1.1 水样采集
本次采样于2019年10月20日至22日进行, 分别选取洱海(25°36′—25°58′N, 100°05′—100°17′E)沿岸12个采样点和湖中线4个采样点, 各采样点编号及GPS定位见图 1。每个采样点采集3个重复样品,每个样品使用容量为1000 mL的取水器采集表层水和底层水各1 L, 然后混合保存至已消毒的2 L广口瓶中。16个采样点共采集48个样品。所有样品均在24h内使用0.45 μm的混合纤维素滤膜(MCE;Whatman公司)进行真空抽滤。为评估是否存在外源DNA污染, 每次过滤设置1个阴性对照。在每份样品过滤后, 滤膜立即冷冻保存, 并对滤膜接触面进行消毒和冲洗, 防止样品间的交叉污染。冷冻滤膜置保温箱保存带回实验室备用。
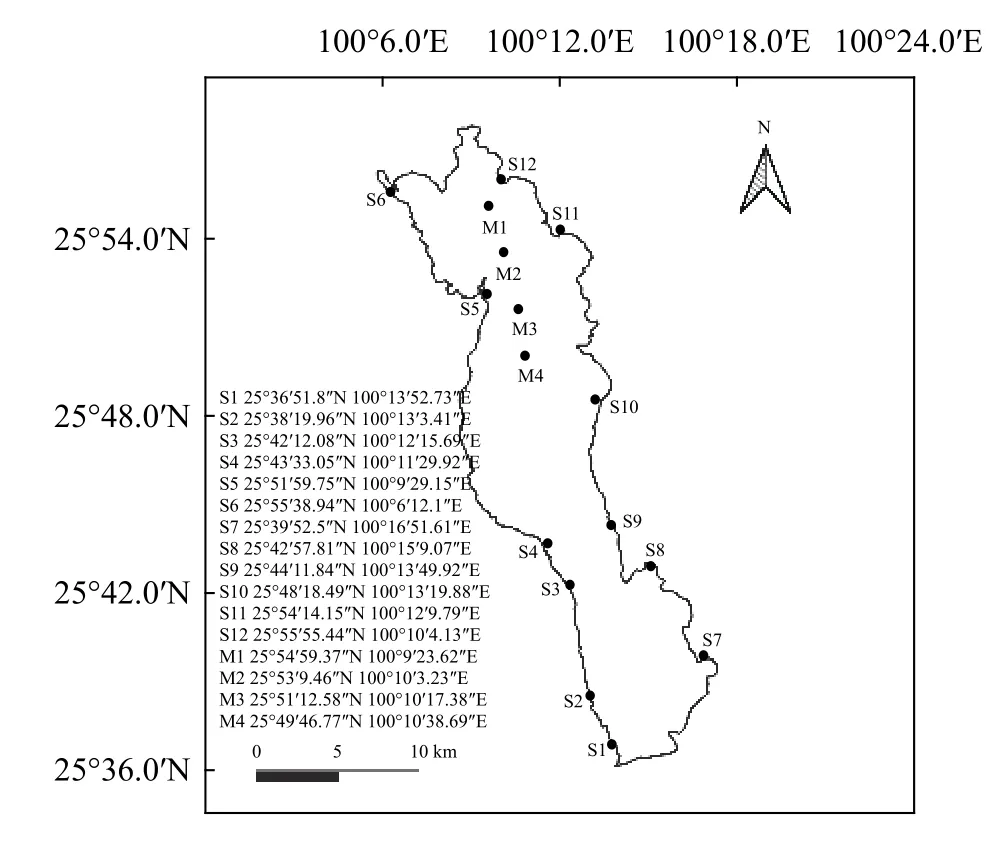
图1 采样点编号及GPS定位Fig. 1 Information of sampling locations
1.2 eDNA提取
使用PowerWater DNA Isolation Kits(Qiagen公司)按照试剂盒说明书提取滤膜DNA。每份样品独立提取, 并使用空白滤膜设置为阴性对照。所得eDNA溶液快速置于-20℃冷冻保存, 直至下一步PCR扩增。
1.3 遗传标记扩增与高通量测序
使用鱼类通用引物MiFish-U[16]对eDNA模板进行PCR扩增, 该引物被用于扩增线粒体12S rRNA基因的高变异区域, 扩增长度约163—185 bp, 是鱼类环境DNA宏条形码分析的常用引物。20 μL PCR扩增体系包括: 2—5 μL模板DNA(10 ng/μL)、正反向引物各0.8 μL (10 μmol/L)、2 μL dNTPs、4 μL 5 ×FastPfu缓冲液、0.4 μL FastPfu聚合酶和7—10 μL ddH2O。PCR反应程序为: 95℃ 预变性5min; 27个循环包括: 95℃变性30s, 55℃退火30s, 72℃延伸45s; 72℃最后延伸10min。每次PCR反应使用ddH2O为模板作PCR阴性对照。
每份样品重复扩增三次, 将同一样品的PCR产物混合后, 使用2%的琼脂凝胶电泳检测。所有阴性对照均无目的条带, 表明从采样、过滤、DNA提取至PCR扩增均无外源鱼类DNA污染。此外, 16个采样点中只有9个采样点的样品获得可检测的PCR产物, 最后经AxyPrep DNA Gel Extraction Kit(AXYGEN公司)纯化后送至上海凌恩生物科技有限公司测序。每个样品的PCR产物单独建库, 然后使用Illumina NovaSeq 6000测序平台(Illumina 公司)进行第二代高通量测序。
1.4 数据分析
各样品的原始测序reads经质控、过滤、拼接和聚类后, 将得到的一致序列与本地数据库进行blastn比对(Identity>97%,e值<10-10), 全长扩增区域以错配低于2个碱基匹配到唯一的物种序列。本地数据库包含了来自GenBank的32种鱼类全线粒体基因组序列, 占洱海历史记录鱼类种类的71.11%(洱海历史报道鱼类共45种[12—15,17,18])。各采样点的重复样品中未匹配到物种的序列被排除, 匹配到物种的序列数(Read counts)取平均值处理。9个采样点检测出的物种种类见表 1, 物种序列数见表 2。
另外, 为了比较两种采样方案(湖中线采样与湖边采样)检测出的物种种类及序列丰度是否存在差异, 本研究基于各采样点的物种序列丰度, 使用R包的pheatmap函数绘制heatmap, 以展示物种eDNA在湖中线(Midline)与湖岸边(Shoreline)的空间分布差异。为了进一步探讨不同采样点的鱼类群落组成异同, 本研究分别基于物种存在情况(Presence/absence)与物种序列丰度(Read counts)统计的Bray-Curtis相似性系数进行主坐标分析(Principal coordinates analysis, PCoA)。
2 结果
2.1 物种组成
在16个采样点中, 有9个采样点的样品获得可检测的PCR产物, 包括洱海东岸的6个采样点(S7—S12)和湖中线的3个采样点(M1、M2和M4)。9个采样点共检测出鱼类17种, 隶属于4目5科13属,其中土著种2科3属5种; 外来种4科10属12种(表 1)。9个采样点共获得已知物种序列2369773条, 各采样点的物种序列数见表 2。图 2展现出各采样点的物种组成情况(物种种类和物种相对序列丰度)。尽管不同采样点存在不同物种组成, 但仍有10种鱼类在9个采样点均被检测到, 其中鲫(Carassius auratus)、鳙(Hypophthalmichthys nobilis)、麦穗鱼(Pseudorasbora parva)、泥鳅(Misgurnus anguillicaudatus)和食蚊鱼(Gambusia affinis)在各采样点均展现出相对较高的序列丰度。
2.2 物种空间分布
图3显示了鱼类eDNA在湖中线与湖岸间没有明显的分布差异, 表明不同空间(湖中线与湖岸)采样不会对检测结果造成太大差异。此外, 泥鳅、鲫、鳙、麦穗鱼和食蚊鱼在9个采样点的总序列丰度远高于其他物种, 表明这5种鱼类为本次调查的优势种。
基于物种存在情况(图 4a)和序列丰度(图 4b)的PCoA分析分别揭示了不同采样点间鱼类组成情况的相似性。基于物种存在情况的PCoA分析(图 4a)表明采样点M2与S7具有完全一致的鱼类组成, 采样点S8具有与M2、S7相似的鱼类多样性。M1、M4与S9, S11与S12分别具有相似的鱼类组成。S10具有不同于其他采样点的鱼类组成。基于序列丰度的PCoA分析(图 4b)表明采样点S8、S10、S11、S12和M2具有相似的鱼类组成, 采样点S7、M1和M4具有相似的鱼类组成, 而S9具有与其他采样点相比不同的鱼类组成。
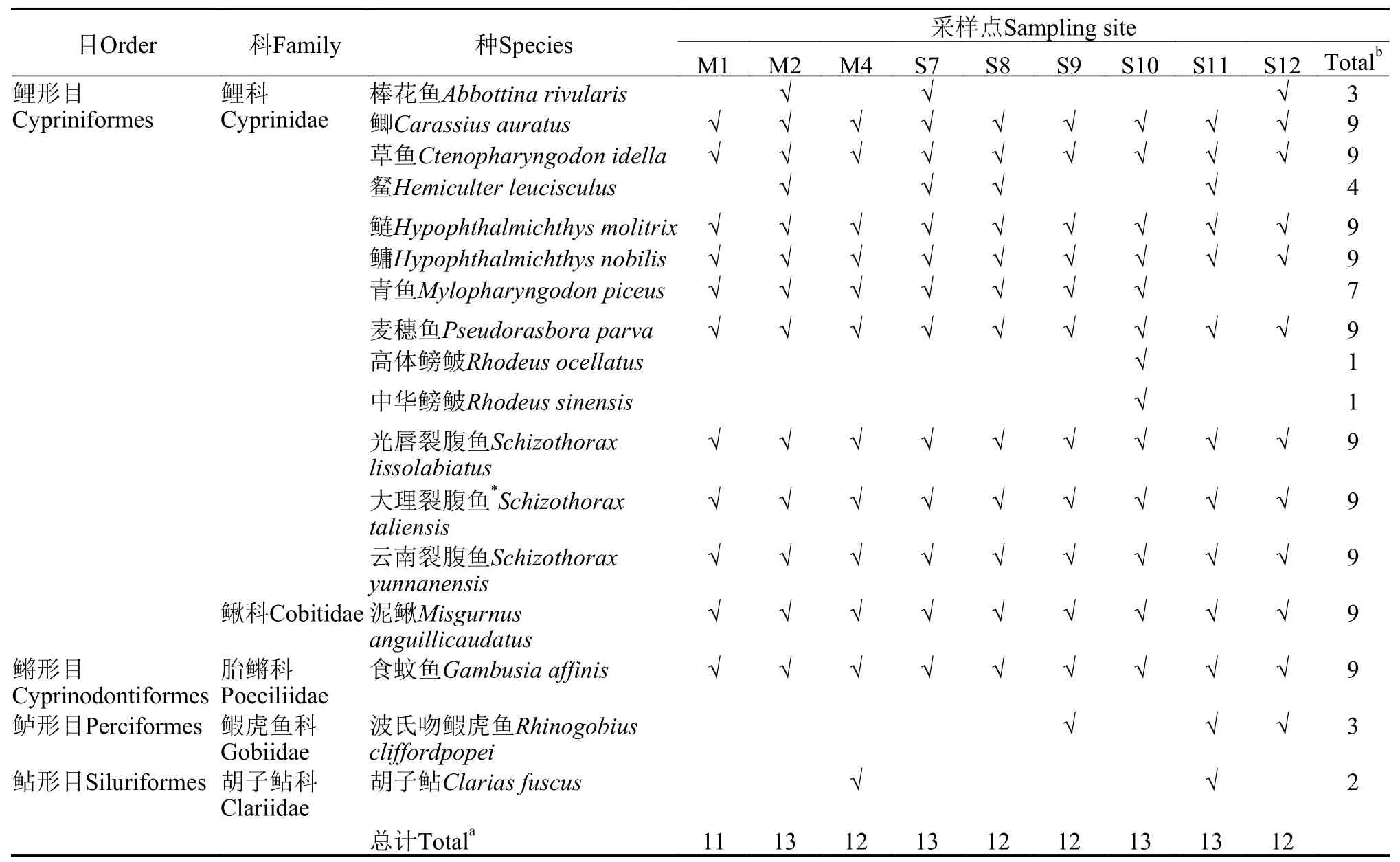
表1 基于环境DNA宏条形码在洱海9个采样点检测到的鱼类物种Tab. 1 List of fish species detected by eDNA metabarcoding at each of 9 sampling sites in Erhai Lake
3 讨论
3.1 基于环境DNA的洱海鱼类多样性检测
本研究首次使用环境DNA宏条形码技术分析了洱海鱼类多样性, 共检测出17种鱼类, 其中以鲤科鱼类居多, 总计13种, 占鱼类物种总数的76.47%;而鲫、鳙、泥鳅、麦穗鱼和食蚊鱼为优势种。虽然本研究检测出的鱼类种数不如传统渔获物方法监测的多(表 3), 但检测出的鱼类组成与过去传统方法监测的结果一致, 均表明洱海鱼类以鲤科鱼类居多, 经济型鱼类和小型鱼类为当前优势种。此外,本研究检测出了已报道的大部分外来种(12种), 说明洱海外来种丰度高于土著种。尽管低丰度的土著种DNA较难被采集到, 但通过增加采样点和采样次数, 可以提高对濒危物种的检测率[8]。
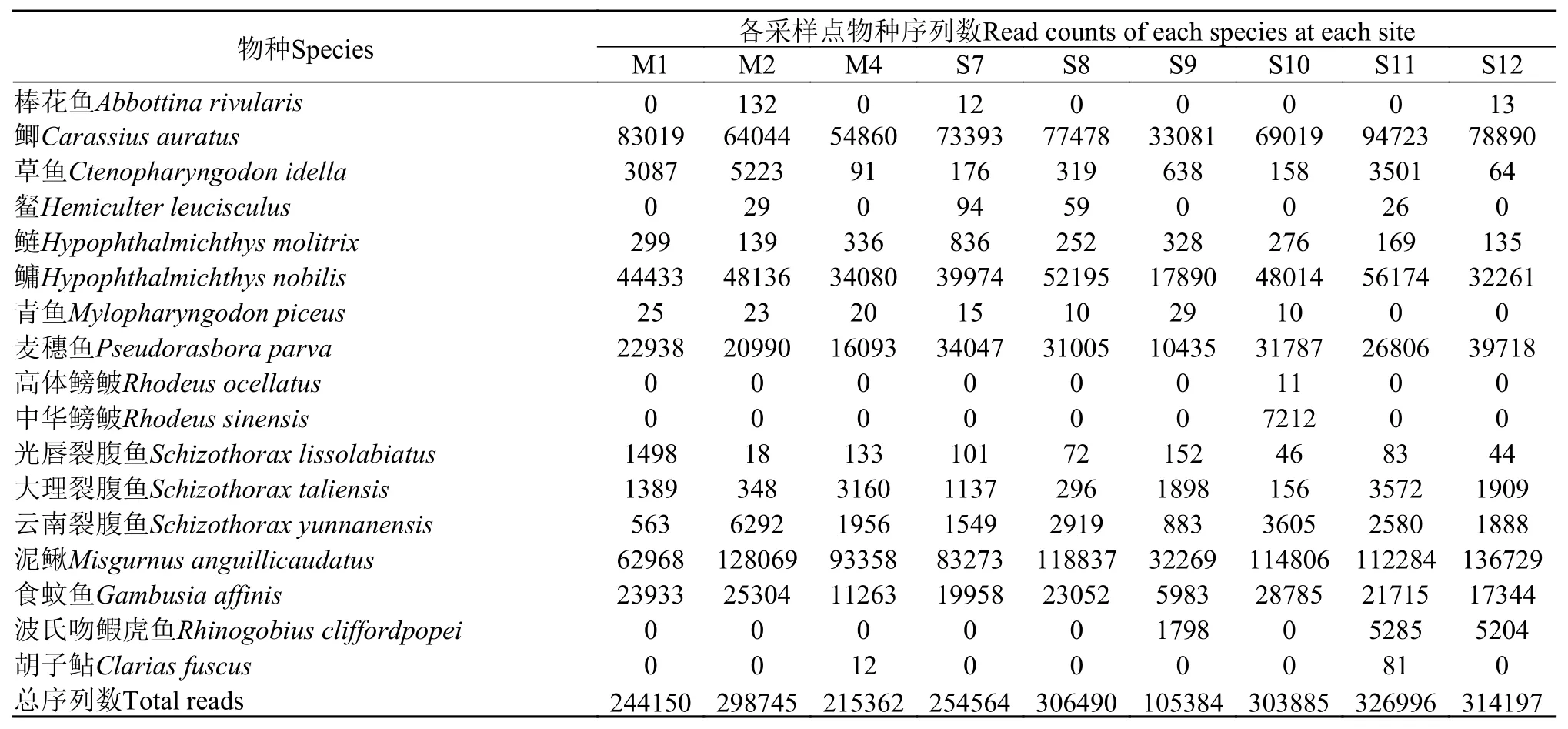
表2 洱海中9个采样点的物种序列数Tab. 2 The number of reads detected for each species at each of 9 sampling sites in Erhai Lake
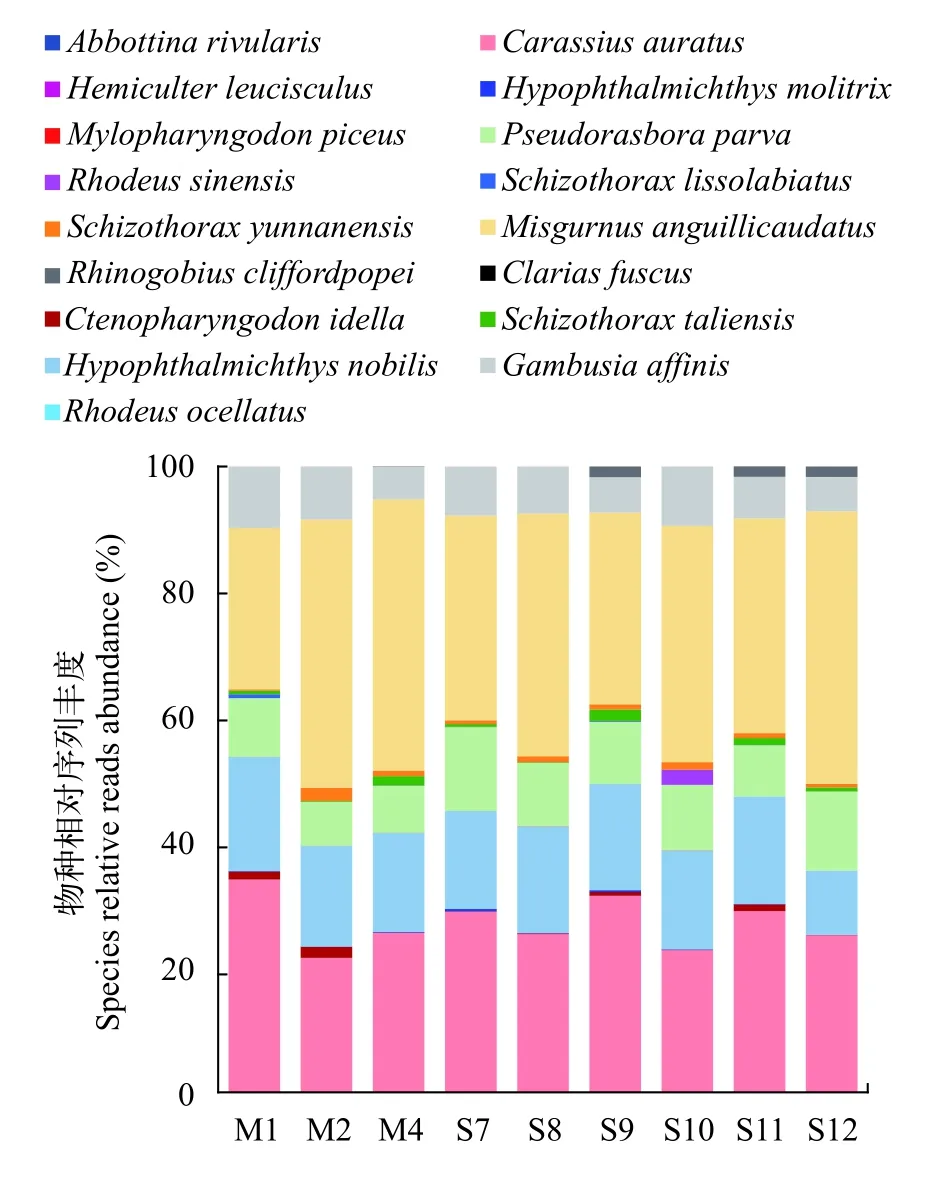
图2 各采样点的鱼类物种组成Fig. 2 The composition of fish species at each sampling site
3.2 洱海鱼类空间分布的初步探讨
除高体鳑鲏(Rhodeus ocellatus)、中华鳑鲏(Rhodeus sinensis)、波氏吻虎鱼(Rhinogobius cliffordpopei)这类栖息于浅水的小型鱼类仅在湖岸水样中被检测到, 其他鱼类在湖中线和湖岸水样中均被检测到, 且鱼类序列丰度没有明显的湖内、湖岸空间差异(图 3)。然而, 9个采样点无论是在物种种类水平还是在序列丰度水平上均揭示了鱼类空间分布的细微差异(图 4)。本研究由于缺少洱海西岸的检测结果, 只基于现有数据无法展现出完整的鱼类群落空间分布格局。费骥慧等[12]基于传统监测结果显示洱海鱼类群落在空间分布上具有明显差异, 可划分为洱海西岸与南北端湖滨带群落类型和东岸与湖心区群落类型。因此, 通过成功获取洱海东、西岸及湖心的eDNA样本, 将有助于分析洱海鱼类群落空间分布格局。总之, 本研究的结果表明只采集岸边水样已足够用于湖泊鱼类的多样性(Species richness)评估, 但广泛的空间采样能够揭示更细致的鱼类分布模式和群落结构, 这与Zhang等[9]的研究结果一致。
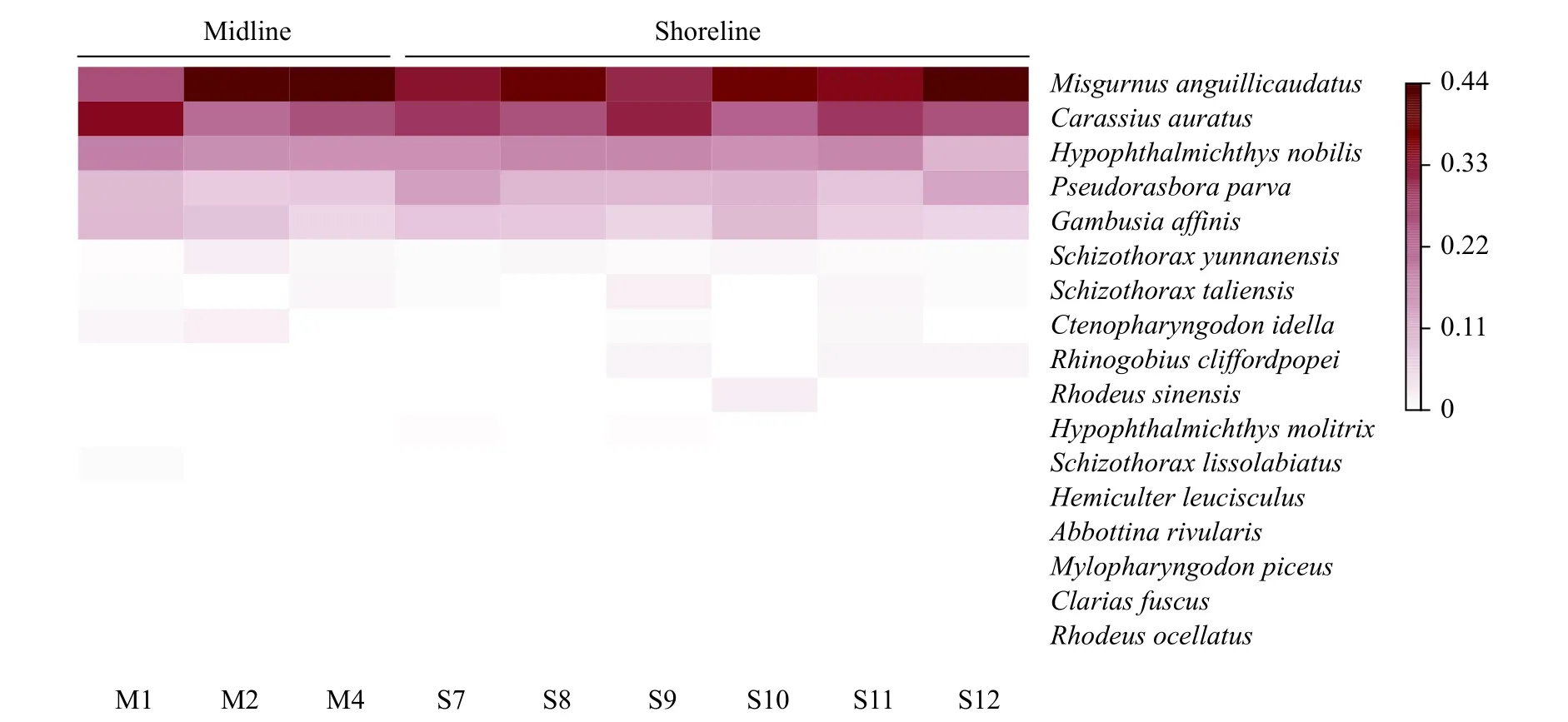
图3 湖中线和湖岸样品的鱼类相对序列丰度Fig. 3 Relative sequence abundances of fish detected in the samples from midline and shoreline
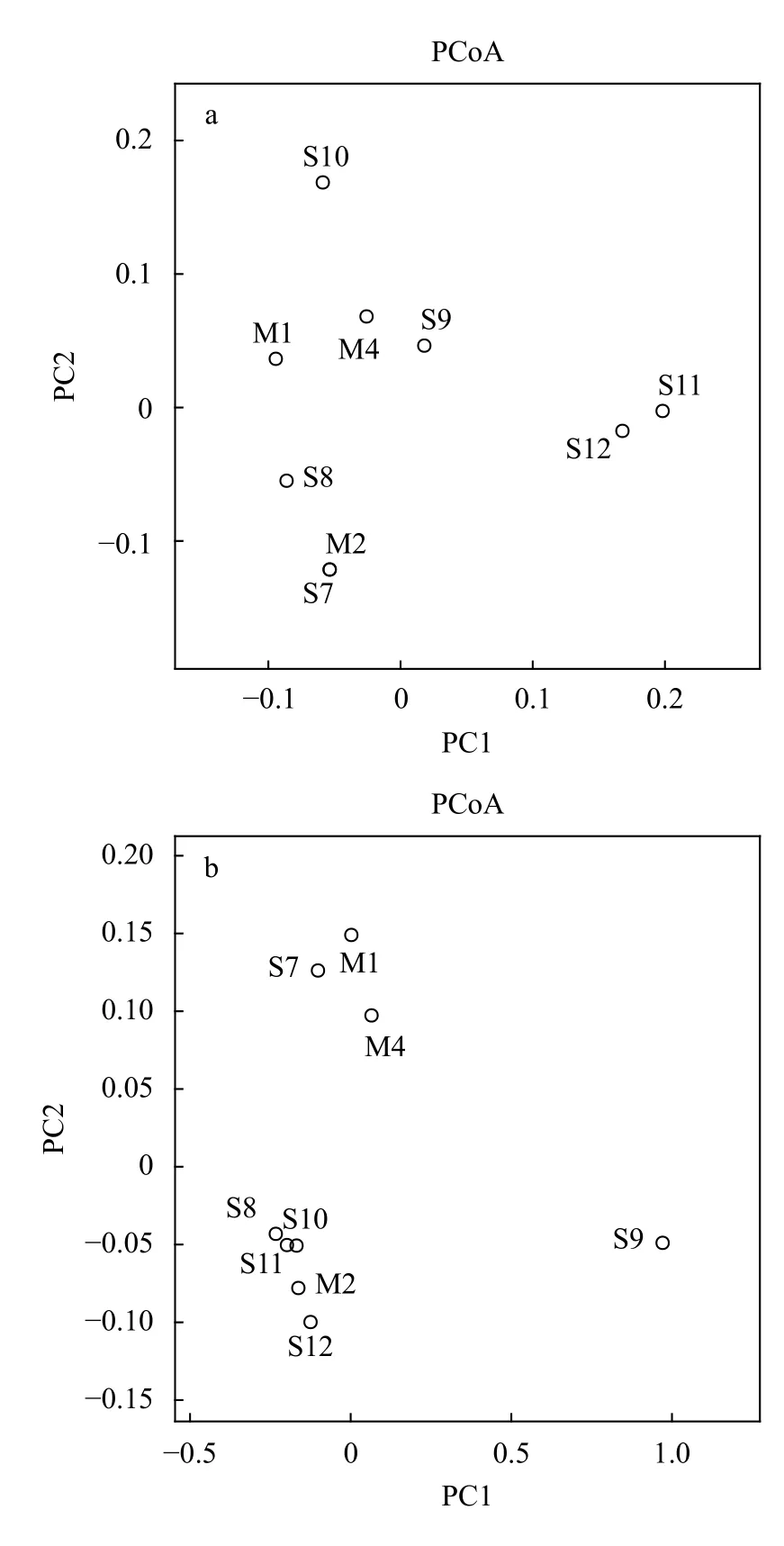
图4 基于物种存在情况(a)和序列丰度(b)统计的鱼类组成相似性Fig. 4 PCoA of the similarity in fish composition based on species occurrence (a) and read counts (b) across sampling sites
3.3 影响环境DNA检测的环境因素
鱼类DNA在水体中具有复杂的存在机制, 其产生、降解及移动过程受诸多环境因素(如紫外线、水温、pH、水体流动、水域底质等)的影响[19]。本次调查共采集洱海16个采样点, 其中只有9个采样点获得可检测的PCR产物, 另外7个采样点(S1—S6和M3)的样品没有扩增出目的条带或PCR产物浓度过低不符合测序要求。本研究认为一些环境因素的影响可能导致了鱼类eDNA在东、西两岸的检测差异。首先, 西岸采样点大多位于苍山十八溪入湖河口附近[20], 近岸水域的eDNA分子受入湖溪流自西向东的流向影响, 可能向湖心扩散。其次, 洱海秋季主导西南风向, 鱼类eDNA分子很可能向东岸聚集。再者, 东、西两岸近岸的水域底质存在差异, 东岸底质以砾石为主, 西岸底质以水草居多, 底质不同可能导致两岸边eDNA浓度存在差异[21]。以上原因可能导致西岸水样采集的鱼类eDNA浓度低于东岸水样, 从而影响了西岸鱼类eDNA的获取与扩增。此外, 已有研究表明水体中的DNA极不稳定, 容易降解[22], 因此水样采集后应尽快过滤, 用滤膜冷冻保存。本研究中西岸水样从采集到预处理的过程比东岸水样花费了更长的时间, 导致水体eDNA的降解量增加, 这可能也是西岸水样没有获得可检测的PCR产物的原因之一。
3.4 环境DNA宏条形码技术的优势与不足
相比传统鱼类监测方法, 环境DNA宏条形码在鱼类多样性评估及生态保护方面具有明显的优势,主要体现在: (1)非破坏性采样。少量的水样采集不会伤害濒危鱼类和破坏当地生态, 并且不受禁渔期和禁渔区的限制, 可以随时监测目标区域的鱼类多样性; (2)便捷高效, 节约成本。环境DNA宏条形码使用1—2 L水样便可实现多个分类单元的快速鉴定, 无需花费船只租赁或购买渔获物的费用, 无需依赖形态学和分类学的专业知识, 同时节省了调查作业和物种鉴定所需的时间。Evans等[23]通过比较电捕法与eDNA方法监测溪鳟(Salvelinus fontinalis)在Wisconsin 水域分布时所需的费用与采样工作量,发现相比电捕法, eDNA方法节省了67%的开支, 且只需较少的采样量; (3)检测灵敏度高。已有研究[24—27]表明使用环境DNA宏条形码检测鱼类多样性的结果要优于传统监测方法, 尤其对于传统方法很难捕捉到的稀有物种或隐蔽物种, 使用eDNA技术更容易检测到。
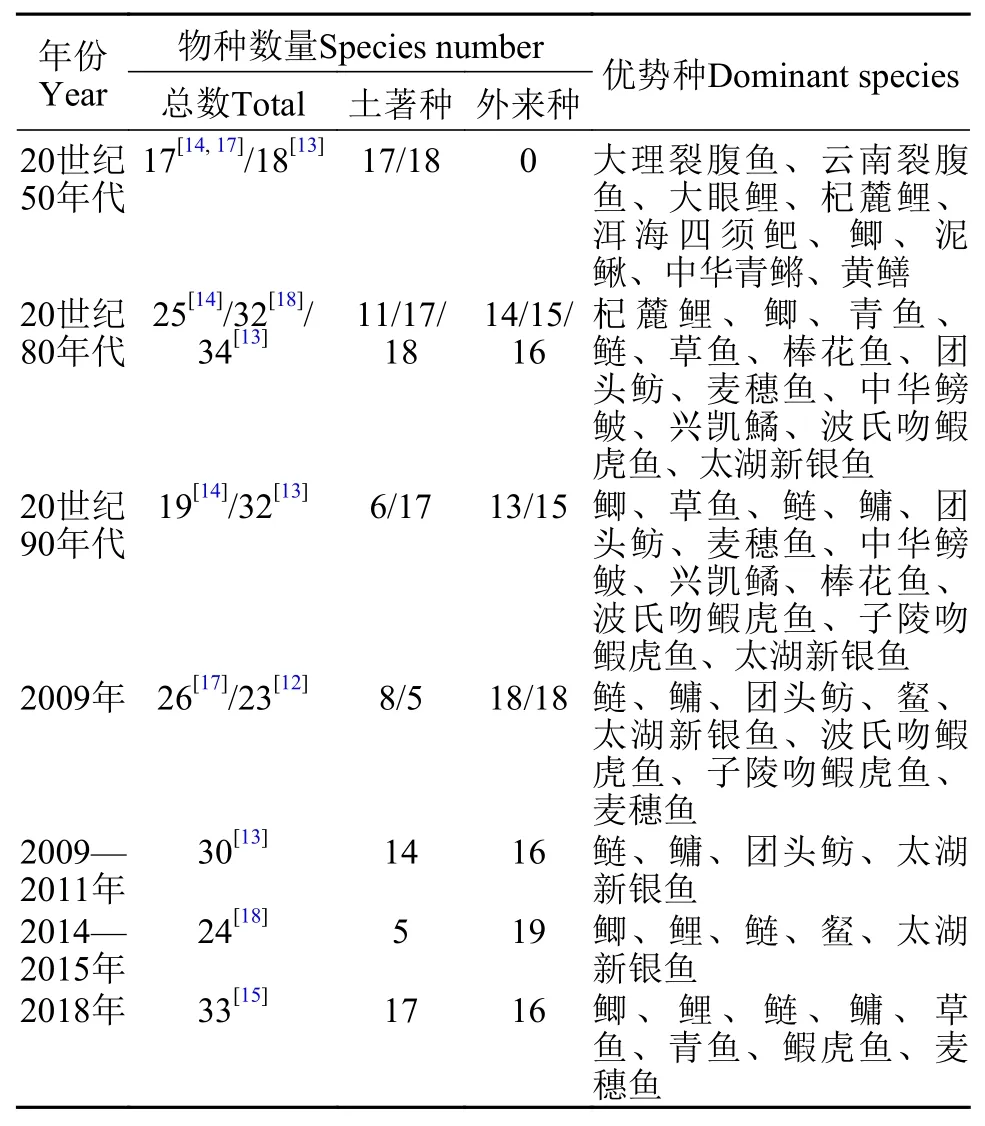
表3 基于传统方法监测到的洱海鱼类资源状况Tab. 3 Fish species composition detected by conventional monitoring methods in different years
尽管环境DNA宏条形码技术存在上述优势, 但它无法完全替代传统的鱼类调查方法[28]。首先, 环境DNA宏条形码只能凭借环境样品中的遗传信息来判断物种的存在情况, 无法获取目标物种的种群数量、年龄结构、生理状态和生长发育阶段等相关信息。其次, 环境DNA宏条形码分析依赖分子数据库的完整性, 当数据库缺少目标物种序列时则会导致假阴性检测[29]。此外, 由于水体环境中eDNA存在机制的复杂性[19], 通过eDNA序列丰度评估物种相对生物量的准确性还有待进一步的研究[30]。
4 结论
环境DNA宏条形码技术具有非破坏性采样、便捷高效以及检测灵敏度高的优势, 在鱼类多样性的监测与保护工作中具有巨大的应用前景。本研究首次使用环境DNA宏条形码技术评估了洱海鱼类多样性, 证明了环境DNA宏条形码技术检测洱海鱼类物种组成和鱼类分布情况的可行性。尽管环境DNA宏条形码无法完全替代传统的鱼类调查方法, 但可以作为一种重要的补充工具, 用于快速反映洱海鱼类多样性和鱼类空间分布, 减少传统监测对洱海生态系统的干扰, 缩短调查周期, 提高检测效率, 为洱海水生生态保护的迅速应答提供可靠数据。
致谢:
感谢袁昌波博士在水样采集工作中提供的帮助; 感谢陶晔、查富蓉在生物信息学分析工作中提供的帮助。
