调控TNBC转移的miRNAs-mRNAs网络及其预后预测价值
2020-09-23赖明芬梅家转朱俊林黄甘萍芦青青
赖明芬 梅家转 朱俊林 黄甘萍 芦青青
乳腺癌主要分为Luminal A型、Luminal B型、Her-2型、TNBC 4种分子亚型,有12%~17%的浸润性乳腺癌属于TNBC[1]。众所周知,TNBC侵袭行为强,是预后最差的一种分子亚型。大多数TNBC发现时已属晚期,常伴有远处脏器转移,从而失去根治性手术机会。由于TNBC缺乏雌激素受体(ER)、孕激素受体(PR)以及无人类表皮生长因子受体(Her-2)扩增,不能从内分泌治疗及抗Her-2靶向治疗中获益,因此放化疗是目前晚期TNBC主要的治疗手段,然而大多数晚期TNBC在放化疗过程中常常发生转移,这严重影响了患者的预后。鉴于TNBC更具侵袭转移的特性,若能明确促进TNBC发生侵袭转移的关键miRNAs及miRNAs-mRNAs调控网络,并且加予阻断,进而降低TNBC远处转移的能力,增加根治性手术的机会,使TNBC患者能够从中明显获益。miRNAs是一类广泛存在于真核生物的微小非编码RNA(ncRNA),虽占基因组的2%~5%,却能够调控大约50%的mRNAs,参与肿瘤的发生发展[2]。研究发现miRNAs能够调控肿瘤的增殖生长、侵袭转移、血管生成、细胞耐药及信号通路[3]。鉴于miRNAs在肿瘤中的作用,本研究通过生信技术筛选TNBC转移灶与原发灶之间表达差异的miRNAs,寻找促进TNBC发生转移的关键miRNAs;通过分析miRNAs-mRNAs调控网络,预测miRNAs促进TNBC转移的潜在机制;通过分析目标mRNAs对TNBC患者DMFS的影响,进而寻找TNBC治疗新靶点和预后预测指标。
1 资料与方法
我们通过GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)寻找TNBC微阵列,共找到2个发生转移的TNBC微阵列,即GSE80038(发生肺转移的TNBC)、GSE38167(发生淋巴结转移的TNBC)。设定|logFC|≥2,P<0.01作为筛选条件,分别对GSE80038、GSE38167这两个微阵列的转移灶与原发灶的miRNAs表达情况进行分析,寻找转移灶与原发灶之间表达差异的miRNAs。通过在线工具Calculate and draw custom Venn diagrams(http://bioinformatics.psb.ugent.be/webtools/Venn/)取GSE80038、GSE38167微阵列共同差异表达的miRNAs。借助targetscan数据库(http://www.targetscan.org/vert_72/)、miRDB数据库(http://www.mirdb.org/)、miWalk数据库(http://zmf.umm.uni-heidelberg.de/apps/zmf/mirwalk2/)预测miRNAs调控的mRNAs。同样借助在线工具Calculate and draw custom Venn diagrams获得miRNAs在targetscan数据库、miRDB数据库、miWalk 3个数据库均调控的目标mRNAs。借助DAIVA在线工具(https://david.ncifcrf.gov/)对mRNAs进行基因本体论(GO)富集分析、京都基因和基因组百科全书(KEGG)通路富集分析以及利用STRING(https://string-db.org/cgi/input.pl?sessionId=tjrG4d8rLiXM&input_page_active_form=single_identifier)进行蛋白互作(PPI)分析,了解转移灶及原发灶表达差异的miRNAs及其调控的mRNAs所参与的细胞功能、信号通路及蛋白之间的相互作用及关系。借助Kaplan-Meier Plotter 在线工具(http://kmplot.com/analysis/)对GO富集分析、KEGG分析筛选出来的目标mRNAs进行生存分析,以P<0.05为统计学有意义。
2 结果
2.1 表达差异的miRNAs
在GEO数据库中共找到2个发生转移的TNBC微阵列,即GSE80038(发生肺转移的TNBC)、GSE38167(发生淋巴结转移的TNBC)。在GSE80038微阵列中找到4个TNBC肺转移灶芯片,4个TNBC肺转移原发灶芯片。按照|logFC|≥2,P<0.01的筛选标准,在这8个芯片中得到264个表达差异的miRNAs,其中差异上调的miRNAs有123个,差异下调的miRNAs有141个。在GSE38167微阵列中找到13个TNBC淋巴转移灶芯片,16个TNBC淋巴转移原发灶芯片,在这29个芯片中得到84个表达差异的miRNAs,其中差异上调的miRNAs有23个,差异下调的miRNAs有61个。通过Calculate and draw custom Venn diagrams在线工具分别取在GSE80038、GSE38167微阵列均差异上调和下调的miRNAs,发现在GSE80038、GSE38167微阵列均差异上调的基因有1个(hsa-miR-548k),差异下调的基因有1个(hsa-miR-2113)(见图1)。

注:A为上调miRNAs,B为下调miRNAs。
2.2 miRNA调控的mRNAs
为了减少mRNAs预测误差,我们通过targetscan数据库、miRDB数据库、miWalk数据库这三个数据库预测hsa-miR-2113、hsa-miR-548k调控的mRNAs,然后通过Calculate and draw custom Venn diagrams在线工具分别取hsa-miR-2113、hsa-miR-548k在上述三个数据库均预测到的mRNAs,发现hsa-miR-2113在上述三个数据库均调控的mRNAs有153个,hsa-miR-548k均调控的mRNAs有8个(见表1和图2)。

注:A为hsa-miR-2113目标mRNAs.B为hsa-miR-548K目标mRNAs。

表1 3个数据库共同表达的目标mRNAs和miRNAs
2.3 mRNAs的GO富集、KEGG分析结果
hsa-miR-2113、hsa-miR-548k在上述三个数据库均预测到的mRNAs共有161个,其中hsa-miR-548k的8个目标mRNAs,hsa-miR-2113的153个目标mRNAs。借助DAIVA在线工具对这161个目标mRNAs进行GO富集分析和KEGG通路分析(以P<0.05且数目≥5作为筛选条件),预测这161个目标mRNAs的细胞组成、分子功能、生物过程及参与的信号通路,结果如下(见表2和图3)。

图3 hsa-miR-2113及hsa-miR-548K-mRNAs网络

表2 目标mRNAs 基因本体论富集分析及京都基因与基因组百科全书分析
2.4 目标mRNAs的蛋白互作图及相互作用关系
借助STRING在线工具和cytoscape 作图软件对上述161个目标mRNAs进行蛋白互作分析,经过筛选后得到25个目标mRNAs的蛋白互作图及其相互作用关系图(见图4)。

图4 目标mRNA蛋白互作分析
2.5 影响DMFS的17个目标mRNAs
通过GO富集分析、KEGG分析以及PPI分析,发现hsa-miR-548k的目标mRNAs,按照我们设定的筛选条件没有得到GO富集、KEGG分析、PPI分析结果,故最后我们借助Kaplan-Meier Plotter 在线工具只对hsa-miR-2113所靶向的153个目标mRNAs进行生存分析,我们以无远处转移生存期(DMFS)作为研究终点。以P-Value<0.05且logrankP≤0.05为筛选条件,在这161个目标mRNAs中发现只有17个目标mRNAs对DMFS的影响有统计学意义,其中包括:CNKSR2、PALM2、PAK3、PCDHA1、PCDHA12、PCDHA13、PCDHA11、PCDHA10、PCDHA2、PCDHA4、CHFR、ACTN1、S100PBP、STAB1、USP9Y、ZBTB37、NTNG1(见图5)。
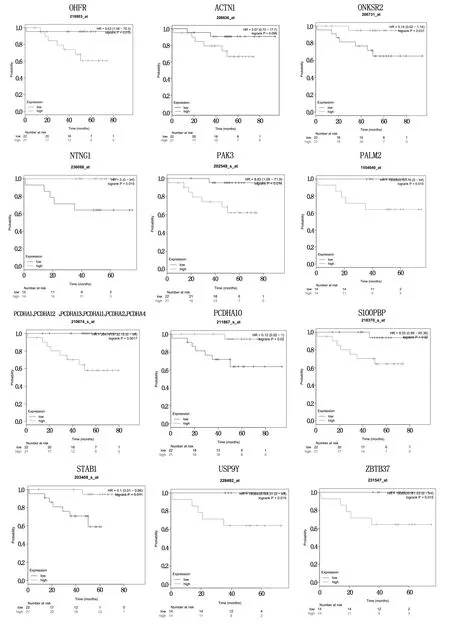
图5 目标mRNAs生存分析
3 讨论
通过对hsa-miR-2113调控的153个目标mRNAs进行生存分析,发现只有17个目标mRNAs对DMFS有统计学意义。在这17个目标mRNAs中,PCDHA家族占据了7个,推测PCDHA可能对TNBC患者的DMFS具有重要的影响作用。原钙黏附因子A(PCDHA)作为钙连蛋白超家族成员,具有与钙连蛋白共同的同源系列,能够调节细胞间的黏附作用,影响肿瘤的生长和转移。有研究发现PCDHA的表达受miRNAs的调控,在前列腺癌细胞系中,miR-193a-5p能够下调PCDHA8的表达水平,同时增加波形蛋白、降低E-钙连蛋白的表达水平,参与前列腺癌细胞转移[4]。我们的研究发现PCDHA10的表达水平与TNBC的DMFS有关,具有较高水平PCDHA10的TNBC患者具有较高的DMFS,PCDHA10可能作为TNBC良好预后的预测指标。而PCDHA1、PCDHA2、PCDHA4、PCDHA11、PCDHA12、PCDHA13表达低的TNBC患者其DMFS高,PCDHA1、PCDHA2、PCDHA4、PCDHA11、PCDHA12、PCDHA13可能是TNBC较差的预后因素,这与PCDHA作为抑癌基因发挥作用的结论相反。出现这种矛盾,我们推测不同的PCDHA亚型以及同种PCDHA亚型在不同肿瘤类型或同一肿瘤类型不同亚型中的作用可能存在差异。另外有研究发现PCDHs超甲基化出现在多种恶性肿瘤进程中[5],研究发现差异性甲基化区域(DMRs)的成簇超甲基化参与了乳腺癌基因沉默,而DMRs的成簇超甲基化与PCDH基因家族簇密切相关[6]。众所周知,miRNAs在转录后调节发挥着重要的作用,而PCDHs作为miRNAs的目标mRNAs,PCDHs的超甲基化是否是miRNAs对其转录后调节的结果,需要进一步研究证实。
S100PBP作为S100P(钙连接蛋白家族成员)的连接蛋白,通过调控组织蛋白酶Z,降低癌细胞间的黏附能力,进而参与肿瘤细胞的侵袭转移[7]。Savci-Heijink等通过分析伴有内脏转移的乳腺癌和不伴有内脏转移乳腺癌组织中基因的表达情况,发现S100PBP在伴内脏转移的乳腺癌中的表达降低,认为该基因的表达水平有助于识别乳腺癌内脏转移人群[8]。另外有研究发现miR-944能够直接与S100PBP结合,参与癌细胞增殖、迁移及侵袭[9]。而我们发现在TNBC中高水平S100PBP的DMFS越低,说明S100PBP是TNBC不良预后因素,这与S100PBP在内脏转移的乳腺癌中表达降低结论不一致。我们推测可能是由于Savci-Heijink的研究没有对乳腺癌分子亚型进行区分,S100PBP在乳腺癌中的作用可能受ER、PR、Her-2水平的影响。
SATB1是富含AT连接蛋白1序列的核基质结合蛋白,通过与染色体上富含T的序列结合,调节多种转移相关基因的表达。我们通过分析SATB1与DMFS的影响发现SATB1的水平与DMFS呈现出正相关关系,SATB1可能是TNBC较好预后的预测指标。而Wang等通过分析乳腺癌组织及癌旁组织中SATB1的表达,发现SATB1在正常组织的表达水平很低,而在肿瘤组织中表达增高,并且发现其表达水平与年龄、绝经期、PR和Her-2蛋白表达无关,而与肿瘤大小、区域淋巴结转移、组织病理级别、肿瘤分级、ER蛋白表达相关[10]。另外,近年来有研究发现SATB1通过染色体基因重组促进包括乳腺癌在内的多种恶性肿瘤生长和转移,发现在乳腺癌中高表达的SATB1显著增加了癌细胞浸润能力,其表达水平与生存呈负相关[11]。认为KI67和SATB1可能是乳腺癌总生存期预后的独立因子[12]。这与我们的研究结果不一致,推测这可能是ER蛋白对SATB1影响的结果。
CHFR是重要的细胞周期调节剂,具有丝分裂检查点和抑癌基因的作用。通过作用泛素连接酶,能够抑制细胞周期蛋白B1-Cdk的形成,进而促进细胞周期阻滞。CHFR还能够通过RNA连接锌指结构与PARP-1结合,阻断TOPK介导的AKT活化,阻断G2/M进程。在乳腺癌细胞中降低CHFR的表达能够加快癌细胞生长速度、增强侵袭和克隆的形成[13]。而我们发现表达低的CHFR具有更高的DMFS,这与乳腺癌细胞中降低CHFR的表达能够加快癌细胞生长速度、增强侵袭和克隆的形成观点不一致。这可能在不同分子亚型的TNBC中CHFR的作用是不同。
Savci-Heijink等[14]通过分析乳腺癌骨转移与乳腺癌非骨转移之间的基因表达差异情况,寻找促进乳腺癌定向性骨转移相关基因,发现15个基因与乳腺癌定向骨转移相关,能够作为乳腺癌骨转移的预测基因,其中包括PALM2。我们的研究发现PALM2越高,则TNBC发生远处转移的可能性越大,推测PALM2可能具有促进TNBC发生远处转移的能力。CNKSR2在乳腺癌组织与非癌组织中表达存在差异,并且发现CNKSR2表达水平与ER、PR、Her-2状态相关。在高HER-2、低ER和PR状态的乳腺癌亚型中,CNKSR2的表达相对较高,并且高水平的CNKSR2促进乳腺细胞发生恶变[15]。而我们的研究发现具有较高CNKSR2的TNBC患者其DMFS较高,CNKSR2是TNBC良好预后的预测指标。USP9Y编码类似于泛素特异性的蛋白酶,对保护蛋白免受蛋白酶降解具有重要的调节作用。在前列腺癌发展过程中USP9Y表达下降,认为USP9Y可能作为肿瘤预后预测指标[16]。ACTN1在乳腺癌中表达增高,能够促进细胞迁移、降低E-钙粘蛋白的黏附作用,促进乳腺癌细胞转移,并且与基底样乳腺癌不良预后相关[17]。我们的研究也发现ACTN1与TNBC不良预后相关。Liu等[18]发现p21激活的激酶3(PAK3)能够增强胸腺类癌细胞迁移和侵袭,说明PAK3可能是胸腺类癌不良预后因素,我们同样也发现PAK3是TNBC不良预后因素,PAK3与TNBC远处转移相关。ZBTB37是芳烃受体(AhR)的目标基因,调控造血干细胞髓内和髓外造血,参与多种血液系统疾病的发生[19]。我们通过对ZBTB37与DMFS的生存分析发现,ZBTB37是TNBC不良预后的预测指标,推测ZBTB37可能是通过影响造血干细胞来促进TNBC发生远处转移。通过分析这17个目标mRNAs在肿瘤中的相关研究,发现它们能够调控肿瘤侵袭转移,我们通过生存分析发现它们的表达水平与TNBC的DMFS相关,能够作为TNBC发生远处转移的预测指标,它们有可能作为TNBC治疗新靶点。
GO富集分析一般包括细胞组成(CC)、分子功能(MF)、以及生物过程(BP)。通过表4可知目标mRNAs的功能主要集中在神经系统发育、细胞间黏附以及转录调节方面。在KEGG分析结果中发现目标mRNAs主要参与轴突导向及癌症相关通路。GO富集结果中的细胞间黏附、转录调节以及KEGG分析结果中癌症通路说明miRNAs及其所调控的目标mRNAs网络与TNBC转移相关。
TNBC是乳腺癌的一种分子亚型,由于缺乏ER、PR、Her-2扩增,不能从内分泌治疗及抗Her-2靶向治疗中获益。目前TNBC的主要治疗手段包括手术、放疗以及化疗,然而大多数TNBC发现时常常处于晚期,因此放化疗成为晚期患者的主要治疗手段。TNBC因具有较强的侵袭行为,大多数TNBC患者在放化疗过程中出现脏器转移[2]。大量研究[3]发现miRNAs异常表达与肿瘤增殖生长、侵袭转移、血管生成、细胞耐药及信号通路密切相关。miR-10b、miR-26a、miR-146a、miR-15能够调控BRCA1的表达参与TNBC的发生发展。研究发现miR-10b在转移性乳腺癌中表达增高,并且能够刺激促转移基因RHOC表达,进而促进TNBC发生远处转移[20]。miR-21的表达水平与TNBC晚期临床阶段、淋巴结转移、癌细胞扩散、不良预后相关,认为miR-21能够作为TNBC患者预后的预测因素[21]。而miR-15a通过作用于CCNE1,能够调节细胞周期,促进细胞增殖,并且认为miR-15a可能是TNBC预后预测因素[20]。我们的研究发现hsa-miR-2113、hsa-miR-548k在TNBC转移灶与原发灶中存在差异,我们推测hsa-miR-2113、hsa-miR-548k可能是促进TNBC发生转移的关键miRNAs,但关于这两者在乳腺癌方面的研究尚未见报道。
综上所述,本研究发现有17个mRNAs对DMFS有统计学意义,可能作为TNBC预后预测指标和治疗新靶点。由于我们的研究是借助前人的微阵列信息进行的,缺乏相关实验研究。后期我们将进行功能实验验证上述miRNAs-mRNAs调控网络及其功能,期望能找到促进TNBC发生转移的关键miRNAs及其调控网络,为TNBC治疗提供新靶点和预后预测指标。
