基于染色体片段置换系对水稻粒形及千粒重QTL检测与稳定性分析
2020-09-14王小雷李炜星曾博虹孙晓棠欧阳林娟陈小荣贺浩华朱昌兰
王小雷 李炜星 曾博虹 孙晓棠 欧阳林娟 陈小荣 贺浩华,* 朱昌兰,*
基于染色体片段置换系对水稻粒形及千粒重QTL检测与稳定性分析
王小雷1李炜星1曾博虹2孙晓棠1欧阳林娟1陈小荣1贺浩华1,*朱昌兰1,*
1江西农业大学作物生理生态与遗传育种教育部重点实验室 / 江西省超级稻工程技术研究中心, 江西南昌 330045;2江西省农业科学院 / 江西省超级水稻研究发展中心, 江西南昌 330200
粒形及千粒重是水稻产量的重要影响因素, 通过挖掘这些性状的优异基因, 对水稻超高产育种具有重要意义。本研究利用1套以籼稻恢复系昌恢121为背景亲本, 粳稻越光为供体亲本构建的染色体片段代换系为材料, 在3个环境下对水稻粒形及千粒重进行QTL检测及稳定性分析, 共检测到59个QTL, 分布于1号、2号、3号、4号、5号、6号、7号、10号、11号和12号染色体上, 贡献率为0.77%~36.26%, 其中发现10个QTL多效位点。值得关注的是、、、、和这6个QTL能在3个环境中重复检测到, 其中为新鉴定的QTL位点。这些结果为进一步开展水稻粒形基因的精细定位、克隆和分子辅助育种奠定了一定的理论基础。
水稻; 染色体片段置换系; 粒形; 千粒重; 数量性状座位
粒形及千粒重是影响水稻产量的主要农艺性状, 受多基因控制[1-2]。目前通过构建高世代群体和高通量测序技术, 已经挖掘出大量控制水稻粒形及千粒重的QTL (quantitative trait locus)。彭伟业等[3]以粳稻魔王谷和籼稻CO39配组衍生的280个重组自交系为材料, 2年检测到17个水稻粒形QTL。周梦玉[4]利用晚粳稻品种春江16B (CJ16B)和广亲和中籼稻背景恢复系C84为亲本构建188个家系的重组自交系群体, 在海南陵水和浙江杭州两地共检测到30个籽粒相关主效QTL。丁膺宾[5]和孙妍[6]以海南普通野生稻为供体亲本, 9311为受体亲本构建的染色体片段置换系群体分别鉴定到1个粒长和粒宽相关的和, 并分别成功精细定位到第12号和第8号染色体上, 物理距离分别为15.69 kb和10 kb区间内。Feng等[7]利用全基因组关联定位技术, 在水稻4个籽粒性状中鉴定了27个显著位点, 解释了各性状表型变异的44.93%~65.90%, 总共预测了424个候选基因。迄今为止,根据国际数据库Gramene (http://www.gramene.org/)公布的数据, 水稻已有500多个控制水稻粒形及粒重性状的QTL被定位, 分布于12条染色体上。在不同遗传研究中, 已有近30个基因/QTL被克隆并被验证可以调控籽水稻粒形和粒重性状, 如[8]、[9]、[10]、[11]、[12]、[13-14]、[15]、[16-17]、[18]、[19]等。继续挖掘不同遗传资源中的水稻粒形及粒重相关QTL, 对于了解水稻粒形性状的遗传机制有着重要意义, 也可为遗传育种提供更多的优异基因资源。籼稻和粳稻是亚洲栽培稻2个亚种, 具有强大的杂种优势潜力, 其基因组存在着高度的遗传分化, 后代群体的农艺性状、生理和生态特性等各方面表现出超亲现象。利用籼稻和粳稻构建染色体片段置换系(chromosome segment substitute lines, CSSLs)群体, 可以更准确研究代换片段上所携带基因之间的互作, 同时可以将多个优良基因/QTL进行聚合育种。昌恢121是本课题组选育的强优势籼稻恢复系, 培育出农业部超级稻淦鑫688等一系列强优势杂交稻组合[20]。本研究利用昌恢121为受体亲本与粳稻越光为供体亲本构建的208个CSSLs, 在2016年江西南昌、2017年海南三亚和2017年江西南昌3个环境对水稻粒长、粒宽和千粒重进行QTL检测, 旨在进一步挖掘控制水稻粒形及粒重性状的优异等位基因/QTL, 为水稻的超高产育种提供一定的理论依据。
1 材料与方法
1.1 试验材料
研究材料为1套以籼稻昌恢121为轮回亲本和粳稻越光为供体亲本构建的染色体片段置换系群体, 包含208个株系。2016年在江西南昌(E1)、2017年海南三亚(E2)和2017年江西南昌(E3)江西农业大学实验基地种植。每个株系种植3个重复, 每个重复3行, 每行10株, 种植行株距16.7 cm × 18.7 cm, 常规田间管理。
1.2 性状测定与考察
粒长和粒宽的测定: 在水稻成熟期, 从2个亲本和每个染色体片段置换株系的株行取中间8个单株分别进行混收, 脱粒后的谷粒经自然条件风干, 去除杂质和空瘪粒, 每个株系随机选取成熟饱满谷粒约300粒, 用万深SC-G自动考种仪(杭州万深检测科技有限公司)测定粒长和粒宽, 重复测定3次, 取其平均值。千粒重的测定: 从2个亲本和每个株系中随机选取成熟饱满的谷粒500粒, 称其重量, 换算成千粒重, 重复测定3次, 取其平均值。
1.3 DNA提取与PCR扩增以及扩增产物电泳
抽穗期采集亲本和CSSLs群体各家系叶片, 采用CTAB法抽提基因组DNA。PCR扩增体系10 µL, 包括 DNA 模板 2.0 µL, 引物1 µL, 7 µL PCR混合体系(5.7 µL ddH2O; 1 µL 10×buffer; 0.2 dNTPs; 0.1 µL 5 U µL–1酶)。扩增程序如下: 94℃ 5 min, 94℃ 30 s, 55~58℃ 30 s, 72℃ 30 s, 35个循环; 72℃ 10 min, 4℃保存。PCR扩增产物用8%的琼脂糖凝胶电泳检测。
1.4 QTL定位和数据分析
试验数据整理和图表绘制采用 Microsoft Excel 2003表格完成。显著性分析使用SPSS 16.0独立样本检验分析(<0.01具有极显著性差异; 0.05>>0.01具有显著性差异;>0.05不具有显著性差异)。遗传连锁图使用MapChart[21]软件进行绘制。QTL检测利用QTL IciMapping 4.1软件中的CSL程序(ICIM-CSL)[22-23]进行检测。LOD阈值设为2.5。如果表型与标记的关联性LOD>2.5, 表明在该位置存在1个QTL。QTL的命名参照McCouch等[24]提出的方法进行。
2 结果与分析
2.1 208个株系的图示标记基因型鉴定
利用180个在两亲本间有多态性的标记对208个CSSLs进行基因分型, 该180个有多态的SSR标记, 均匀覆盖在12条染色体上, 覆盖水稻全基因组的1427.7 cM, 平均间距为7.93 cM, 最大间距为25.3 cM (图1)。208个代换系群体的单株基因型的分析结果见附表1。
2.2 亲本与群体的性状表现
2个亲本和208个CSSLs株系的粒长(grain length, GL)、粒宽(grain width, GW)、千粒重(thousand grain weight, TGW)性状表现(表1), 2亲本的粒长、粒宽和千粒重在3个环境中均存在显著或极显著差异。在CSSLs群体中, 粒长、粒宽和千粒重的变幅很大, 表型值均表现为连续变异, 且存在超亲分离现象。
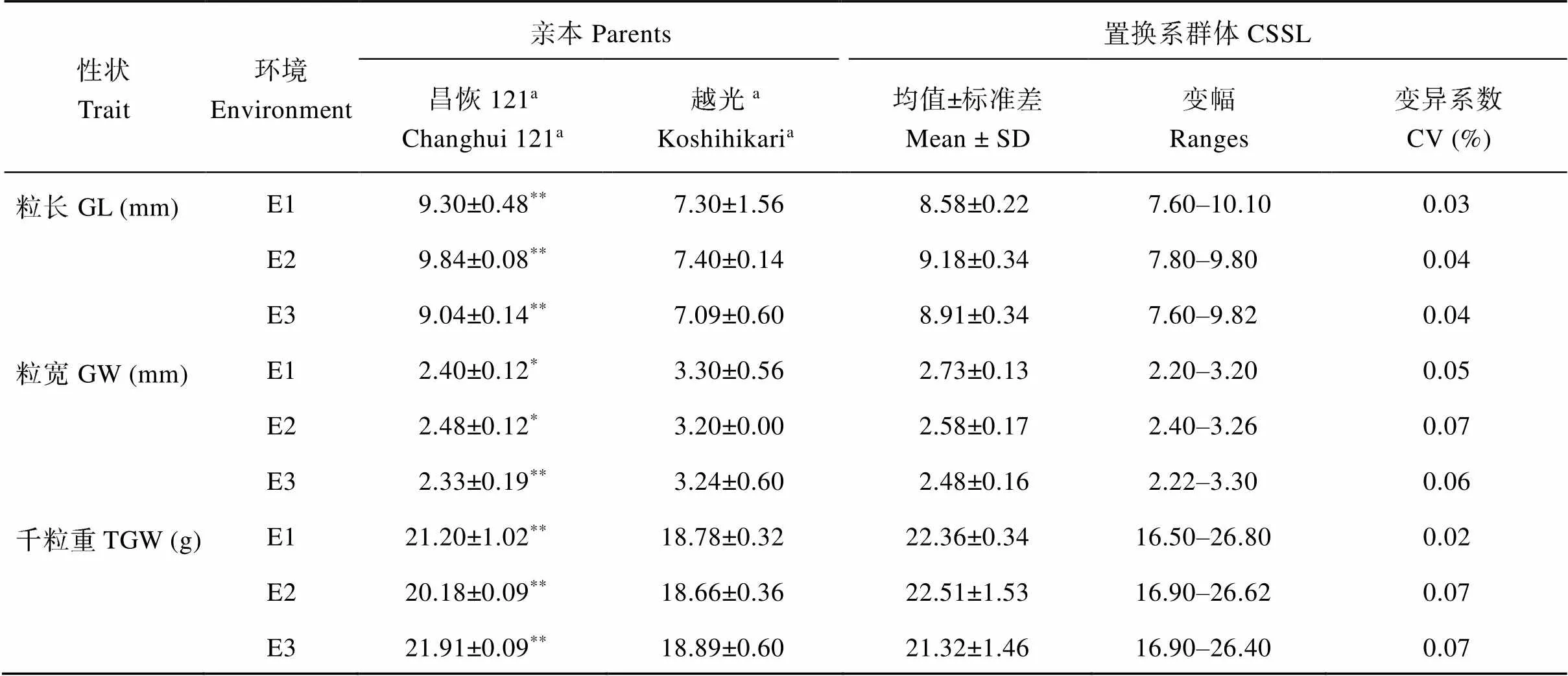
表1 CSSLs及其亲本昌恢121和越光的性状表现
E1: 2016江西南昌; E2: 2017海南三亚; E3: 2017江西南昌。a表中数值为平均值± 标准差。**和*分别表示昌恢 121 的粒形性状与越光之间的差异达1%和5%显著水平。
E1: Nanchang in Jiangxi province in 2016; E2: Sanya in Hainan province in 2017; E3: Nanchang in Jiangxi province in 2017. GL: grain length; GW: grain width; TGW: thousand grain weight; CCSL: chromosome segment substitution lines.aThe values in the column are mean ± standard deviation.**and*mean the level of 1% or 5% of significant differences between rice grain shape of Changhui 121 and Koshihikari, respectively.
2.3 QTL定位分析
利用208个CSSLs对水稻的粒长、粒宽和千粒重进行QTL检测, 共检测到59个QTL,分布于除第8号和9号染色体以外的其他10条染色体上(表2和图1)。
千粒重: 检测到16个控制千粒重的QTL, 分布于除8号、9号、10号和12号染色体以外的其他8条染色体上, 表型贡献为2.88%~36.26%, 其中、、、和在2个环境中都被检测到。、、、和的增效等位基因来自昌恢121;、、、、和的增效等位基因来自越光。
粒宽: 检测到25个控制粒宽的QTL, 分布于除7号、8号和9号染色体以外的其他9条染色体上, 表型贡献为0.77%~35.01%, 其中和在3个环境中都被检测到;、和在2个环境中被检测到。、、、、、和的增效等位基因来自昌恢121;、、、、和的增效等位基因来自越光。
粒长: 检测到18个控制粒长的QTL, 分布于1号、2号、3号、5号、6号、7号、10号和12号染色体上, 表型贡献为2.70%~23.05%, 其中和在3个环境中都被检测到;和在2个环境中被检测到。和的增效等位基因来自昌恢121;、、、、、、、和的增效等位基因来自越光。
2.4 QTL的多效性
3个环境中共检测到59个粒长、粒宽和千粒重QTL, 分布在除8号和9号染色体以外的其余10条染色体上, 发现10个QTL多效位点, 分布于1号、2号、3号、4号、5号、6号、7号和10号染色体上(表3)。其中1号染色体RM5389–RM6696区域附近的QTL控制着GW和GL; 2号染色体上有2个QTL多效位点, 分别位于RM1358–RM5812和RM8030–RM1092区间, 控制着TGW、GW和GL; 3号染色体上有2个QTL多效位点, 分别位于RM3646–RM3513和RM3513–RM2334区间, 控制着TGW、GW和GL; 4号染色体RM6089–RM5503区间, 控制着TGW和GW; 5号染色体RM3295– RM3476区域, 控制着TGW和GW; 6号染色体RM8258–RM2615区间控制着GW和GL; 7号染色体RM3186–RM3404区间控制着TGW和GL; 10号染色体RM5271–RM2125区间控制着GW和GL, 这些QTL成簇分布是一因多效或基因连锁引起, 还需要进一步实验认证。同时多个QTL能在2个及2个以上的环境中被重复检测到, 并且多个被重复检测到的位点和已克隆的QTL/基因处于相邻区域, 说明这些QTL簇确实存在并且稳定表达。
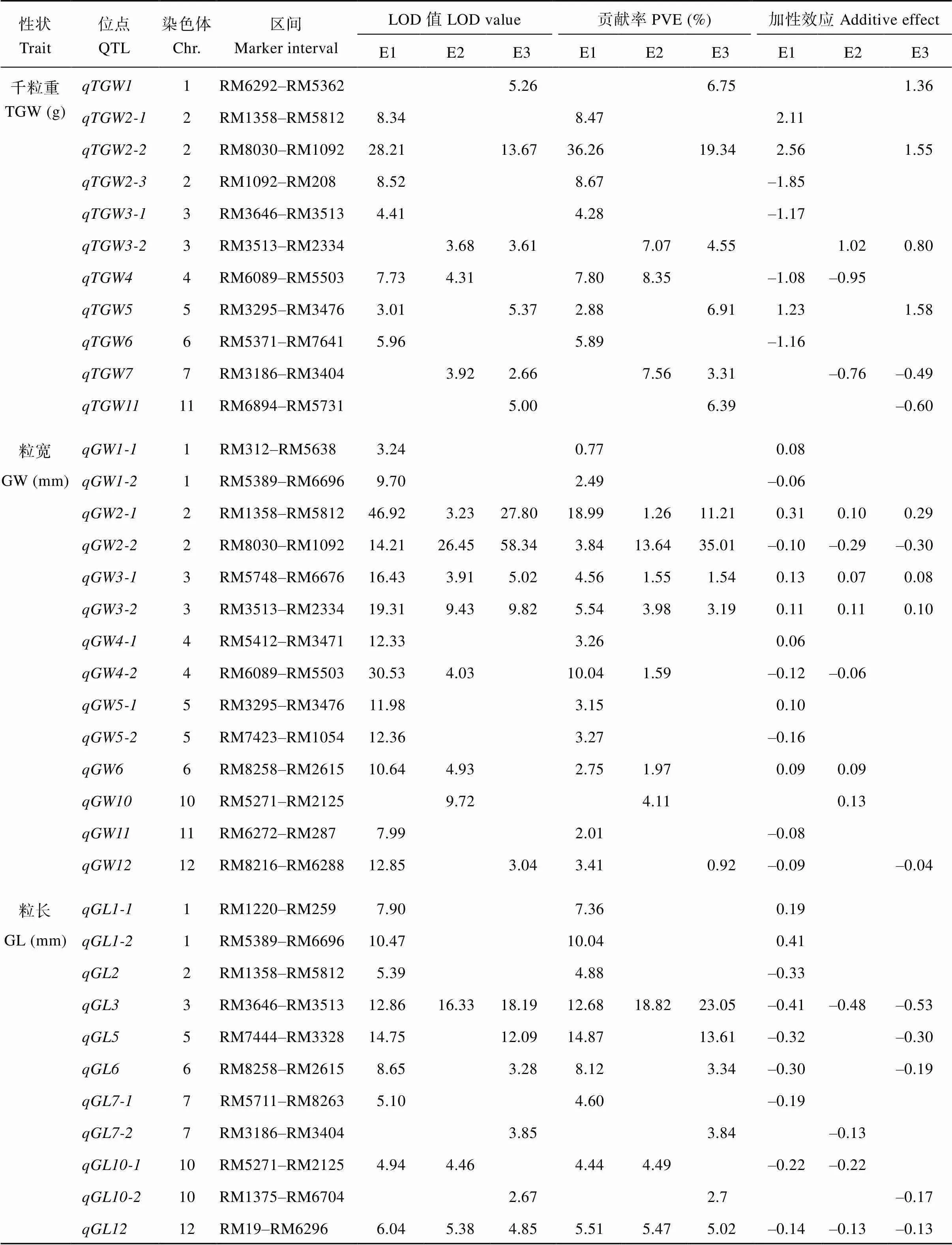
表2 利用208 个CSSLs定位到的水稻粒形及千粒重QTL
缩写同表1。Abbreviations are the same as those given in Table 1.

图1 水稻粒形及千粒重性状QTL在染色体上的分布
GL: grain length; GW: grain width; TGW: thousand grain weight.
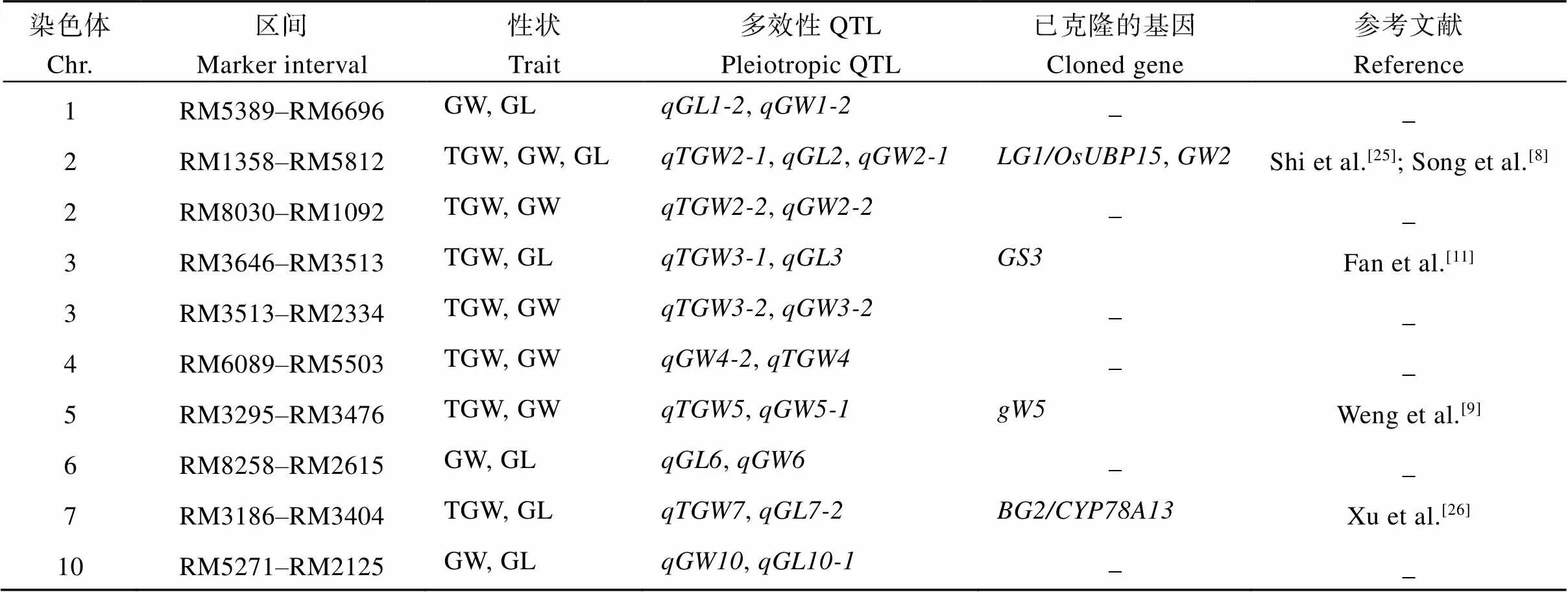
表3 多效性区域分析
GL: grain length; GW: grain width; TGW: thousand grain weight.
2.5 水稻粒形及千粒重性状QTL的稳定性分析
在3个环境中对粒形及千粒重性状QTL进行检测, 发现、、、、和均能在3个环境下检测到, 其余53个QTL在3个环境下未被重复检测到, 说明水稻粒形及千粒重性状QTL定位结果易受环境的影响。、和的增效等位基因来自昌恢121;、和增效等位基因来自越光。利用来自越光的增效等位基因对、和的加性效应和环境稳定性进行验证,定位于2号染色体RM8030–RM1092区段内, 对应的染色体片段置换系为X208, 其粒宽与昌恢121在3个环境中表型差异均达到极显著水平(图2);定位于3号染色体RM3646–RM3513区段内, 对应的染色体片段置换系为X122, 其粒长与昌恢121连续在3个环境下表型差异达到极显著水平(图2);定位于12号染色体RM19–RM6296区段内, 对应的染色体片段置换系为X8, 其粒长与昌恢121连续在3个环境下表型差异达到极显著水平(图2)。利用X208、X122和X8分别验证了、和的加性效应, 并验证了这3个QTL的环境稳定性。
3 讨论
粳稻(ssp.)和籼稻(ssp.)是亚洲栽培稻中的2个亚种, 通过挖掘和研究籼(或粳)稻的等位基因在粳(或籼)稻遗传背景中的遗传特点, 对于籼粳杂种优势的利用以及籼粳亚种间优良基因之间的互换方面等具有重要的理论和实践意义。水稻产量主要由3个因素构成: 单株穗数、每穗粒数和粒重[25]。粒形指标由粒长、粒宽、长宽比和粒厚来综合评价, 这4个性状与千粒重成正相关[26]。与大多数农艺性状一样, 水稻粒形及千粒重是受多基因控制的数量性状。Lin等[27]利用突变体克隆出基因, 该基因是的1个新的位点突变, 参与了油菜素类固醇(BR)信号通路,通过促进细胞分裂和调节颖片上表皮细胞数量来调节籽粒大小。Hu等[28]克隆出了1个新的主要数量性状位点QTL (, 它控制水稻的籽粒大小和重量。Shi等[29]最新报道1个控制大粒的基因编码1种组成性表达的泛素特异性蛋白酶, 通过调节的表达量来调控籽粒的大小。本研究利用同一套染色体片段置换系群体, 在3个环境下进行QTL的检测, 共检测到59个水稻粒形及千粒重QTL, 这些QTL大部分与前人已报道的QTL/基因处于相近区域, 如2号染色体RM1358–RM5812区域的与已克隆的控制粒形及粒重基因[29]、[8]位于相近区域;与已定位的[4]位于相近区域。3号染色体上的与已定位到的[30]处于相邻区域;与已经克隆的控制粒重和粒长的主效基因[11]都位于3号染色体着丝粒附近区域;与3号染色体上已经克隆的控制粒重和粒长的主效基因[16-17]位于相近区域。粒宽与5号染色体上已经克隆的控制稻谷的粒宽和粒重[9]基因位于相临区域。7号染色体RM3186~RM3404区域的、和Xu等[31]已经克隆的处于相邻区域。12号染色体上的与已定位到的[4]处于相邻区域。其中在多个研究中能重复检测到而尚未克隆和功能验证的QTL值得进一步重点研究。当然这些QTL与前人已报道的QTL/基因是否处于同一位点, 也还需要进一步进行验证。本研究发现了一些前人没有报道的新QTL位点, 比如能在3个环境下重复检测到的, 为1个新鉴定的能稳定遗传的QTL位点。本研究发现、、、和能在3个环境中重复检测到, 说明这6个QTL能够在多个环境条件下稳定表达。在3个环境条件下对亲本昌恢121与、和相应位点被置换株系的相关性状进行比较, 发现被置换株系的粒型性状和千粒重与轮回亲本昌恢121之间存在显著差异。如株系X208置换了, 粒宽变宽的同时, 也显著增加了千粒重; 株系X122置换了, 其粒长变短, 粒宽变窄, 千粒重减少; 株系X8置换了, 其粒长变短, 粒宽变宽, 千粒重增加。因此, 充分利用这些稳定表达的QTL对于水稻高产和优质育种是有利的。
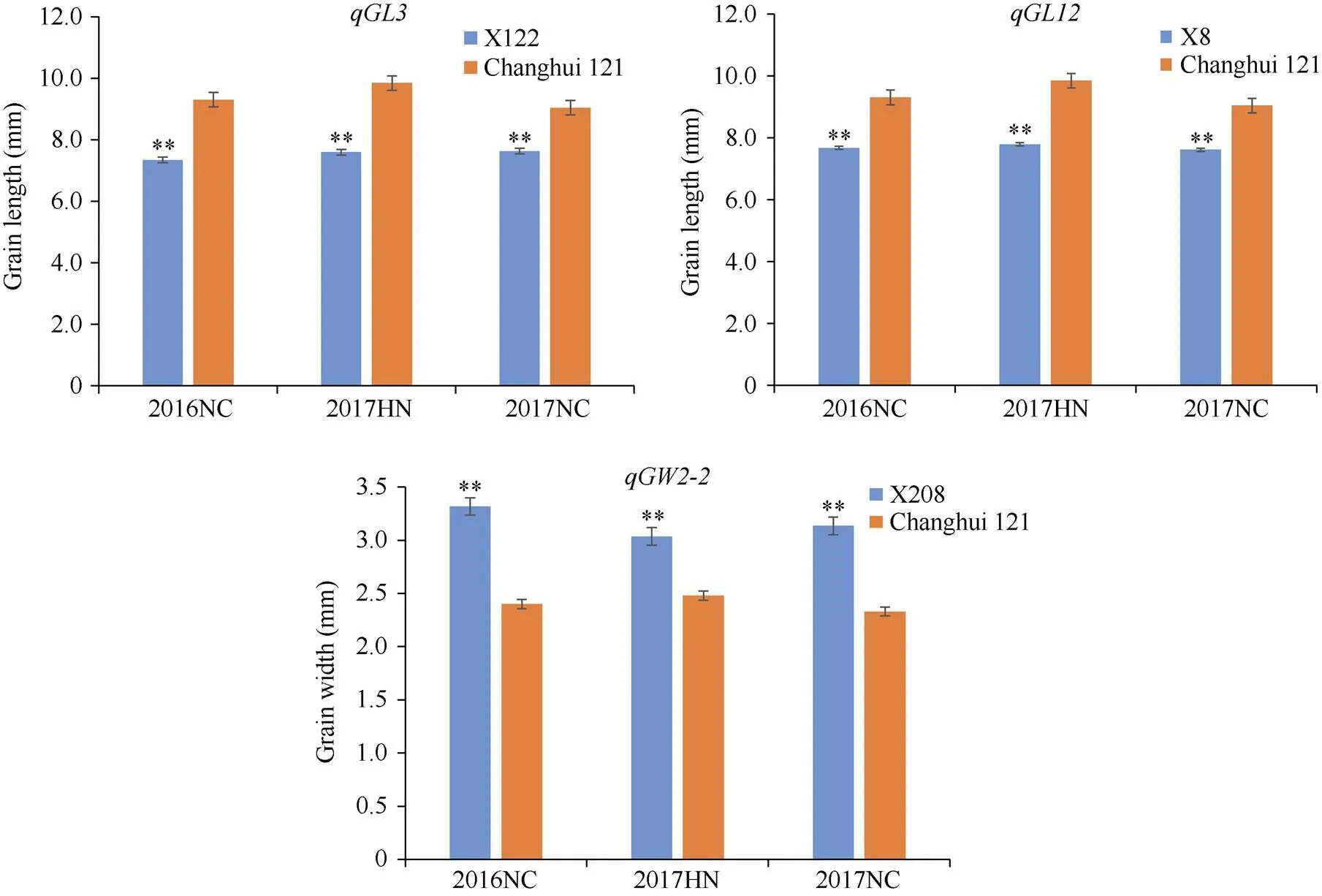
图2 水稻粒形性状QTL对应片段置换系与背景亲本昌恢121的表型差异比较
NC和HN分别表示南昌和海南。**表示昌恢121的粒形性状与目标片段置换系之间的差异达0.01显著水平,检验。
NC and HN represent Nanchang and Hainan, respectively.**means significant differences by Student’s-test (< 0.01) between rice grain shape traits of Changhui 121 and the CSSLs harboring the QTL alleles.
4 结论
本研究利用籼稻恢复系昌恢121为受体亲本和粳稻越光为供体亲本构建的BC3F6、BC4F5和BC5F4共208个CSSLs, 在栽培方式相同条件下, 对3个环境下的水稻粒长、粒宽和千粒重进行QTL检测, 共有59个水稻粒形及千粒重QTL被检测到, 分别为粒长18 QTL、粒宽25 QTL和千粒重16 QTL。其中发现、、、和能在3个环境中重复检测到, 其中为新鉴定的QTL位点。这些定位结果为进一步开展相关基因的精细定位、克隆和分子辅助育种奠定了一定的理论基础。
[1] 邢永忠, 谈移芳, 徐才国, 华金平, 孙新立. 利用水稻重组自交系群体定位谷粒外观性状的数量性状基因. 植物学报, 2001, 43: 840–845. Xing Y Z, Tan Y F, Xu C G, Hua J P, Sun X L. Quantitative trait genes of grain appearance traits were identified by rice recombinant inbred population., 2001, 43: 840–845 (in Chinese with English abstract).
[2] 余守武, 樊叶杨, 杨长登, 李西明. 水稻第1染色体短臂粒长和粒宽QTL的精细定位. 中国水稻科学, 2008, 22: 465–471.Yu S W, Fan Y Y, Yang C D, Li X M. Detailed mapping of QTLs for short arm length and grain width in rice chromosome 1., 2008, 22: 465–471 (in Chinese with English abstract).
[3] 彭伟业, 孙平勇, 潘素君, 李魏, 戴良英. 水稻品种魔王谷水稻粒形、剑叶性状和株高QTL定位. 作物学报, 2018, 44: 1673–1680. Peng W Y, Sun P Y, Pan S J, Li W, Dai L Y. Mapping of QTL for grain shape, leaf character and plant height of rice variety Mowanggu., 2018, 44: 1673–1680 (in Chinese with English abstract).
[4] 周梦玉, 宋昕蔚, 徐静, 付雪, 李婷, 朱雨晨, 肖幸运, 毛一剑, 曾大力, 胡江, 朱丽, 任德勇, 高振宇, 郭龙彪, 钱前, 吴明国, 林建荣, 张光恒. 籼稻C84和粳稻春江16B重组自交系遗传图谱构建及籽粒性状QTL定位与验证. 中国水稻科学, 2018, 32: 207–218. Zhou M Y, Song X W, Xu J, Fu X, Li T, Zhu Y C, Xiao X Y, Mao Y J, Zeng D L, Hu J, Zhu L, Ren D Y, Gao Z Y, Gou L B, Qian Q, Wu M G, Lin J R, Zhang G H. Construction of genetic map ofrice C84 andrice Chunjiang 16B recombinant inbred lines and mapping and verification of QTL for grain traits., 2018, 32: 207–218 (in Chinese with English abstract).
[5] 丁膺宾, 张莉珍, 许睿, 王艳艳, 郑晓明, 张丽芳, 程云连, 吴凡, 杨庆文, 乔卫华, 兰进好. 基于染色体片段置换系的野生稻粒长QTL-的精细定位. 中国农业科学, 2018, 51: 3435–3444. Ding Y B, Zhang L Z, Xu R, Wang Y Y, Zheng X M, Zhang L F, Cheng Y L, Wu F, Yang Q W, Qiao W H, Lan J H. Fine localization of wild rice grain length QTL-based on chromosome fragment replacement line., 2018, 51: 3435–3444 (in Chinese with English abstract).
[6] 孙妍, 苏龙, 乔卫华, 郑晓明, 齐兰, 丁膺宾, 许睿, 张丽芳, 程云连, 兰进好, 杨庆文. 基于染色体片段置换系的野生稻粒宽QTL-的精细定位. 植物遗传资源学报, 2018, 19: 135–142. Sun Y, Su L, Qiao W H, Zheng X M, Qi L, Ding Y B, Xu R, Zhang L F, Cheng Y L, Lan J H, Yang Q W. Precise mapping of grain width QTL-in wild rice based on chromosome fragment replacement line., 2018, 19: 135–142 (in Chinese with English abstract).
[7] Feng Y, Lu Q, Zhai R, Zhang M, Xu Q, Yang Y, Wang S, Yuan X, Yu H, Wang Y, Wei X. Genome wide association mapping for grain shape traits inrice., 2016, 244: 819.
[8] Song X J, Huan W, Shi M, Zhu M Z, Lin X. A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase., 2007, 39: 623–630.
[9] Wan X Y, Weng J F, Zhai H Q, Wang J K, Lei C L, Liu X L, Guo T, Jian L, Su N, Wan J M. Quantitative trait loci (QTL) analysis for rice grain width and fine mapping of an identified QTL allelein a recombination hotspot region on chromosome 5., 2008, 179: 2239–2252.
[10] Wang S K, Wu K, Yuan Q B, Liu X Y, Liu Z B, Lin X Y, Zeng R Z, Zhu H T, Dong G J, Qian Q, Zhang G Q, Fu X D. Control of grain size, shape and quality byin rice., 2012, 44: 950–954.
[11] Fan C C, Xing Y Z, Mao H L, Lu T T, Han B, Xu C G, Li X H, Zhang Q F., a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein., 2006, 112: 1164–1171.
[12] Li Y B, Fan C C, Xing Y Z, Jiang Y H, Luo L J, Sun L, Shao D, Xu C J, Li X H, Xiao J H, He Y Q, Zhang Q F. Natural variation inplays an important role in regulating grain size and yield in rice., 2011, 43: 1266–1269.
[13] Wang S K, Li S, Liu Q, Wu K, Zhang J Q, Wang S S, Wang Y, Chen X B, Zhang Y, Gao C X, Wang F, Huang H X, Fu X D. Theregulatory module determines grain shape and simultaneously improves rice yield and grain quality., 2015, 47: 949–954.
[14] Wang Y X, Xiong G S, Hu J, Jiang L, Yu H, Xu J, Fang Y X, Zeng L J, Xu E B, Xu J, Ye W J, Meng X B, Liu R F, Chen H Q, Jing Y H, Wang Y H, Zhu X D, Li J Y, Qian Q. Copy number variation at thelocus contributes to grain size diversity in rice., 2015, 47: 944–948.
[15] Wu W G, Liu X Y, Wang M H, Rachel S M, Luo X J, Marie N N, Tan L B, Zhang J W, Wu J Z, Cai H W, Sun C Q, Wang X K, Rod A W, Zhu Z F. A single-nucleotide polymorphism causes smaller grain size and loss of seed shattering during African rice domestication., 2017, 3: 1–7.
[16] Qi P, Lin Y S, Song X J, Shen J B, Huang W, Shan J X, Zhu M Z, Jiang L W, Gao J P, Lin H X. The novel quantitative trait locuscontrols rice grain size and yield by regulating., 2012, 22: 1666–1680.
[17] Zhang X J, Wang J F, Huang J, Lan H X, Wang C L, Yin C F, Wu Y Y, Tang H J, Qian Q, Li J Y, Zhang H S. Rare allele ofassociated with grain length causes extra-large grain and a significant yield increase in rice., 2012, 109: 21534–21539.
[18] Ken I, Naoki H, Yuka M, Naomi M, Nao H, Haruko O, Takayuki K, Kazuhiro U, Bunichi S, Atsuko O, Hisashi M, Etsuko K. Loss of function of the IAA-glucose hydrolase geneenhances rice grain weight and increases yield., 2013, 45: 707–711.
[19] Ying Z J, Ma M, Bai C, Huang X H, Liu J L, Fan Y Y, Song X J., a major QTL that negatively modulates grain length and weight in rice., 2018, 11: 750–753.
[20] 贺浩华, 傅军如, 朱昌兰. 香型超级杂交稻新组合淦鑫688. 杂交水稻, 2008, (3): 80–82.He H H, Fu J R, Zhu C L. Ganxin 688, a new combination of fragrant super hybrid rice., 2008, (3): 80–82 (in Chinese with English abstract).
[21] Voorrips R E. MapChart: software for the graphical presentation of linkage maps and QTLs., 2002, 93: 77–78
[22] Wang J K, Wang X Y, Crossa J, Crouch J T, Weng J F, Zhai H Q, Wan J M. QTL mapping of grain length in rice (L.) using chromosome segment substitution lines., 2006, 88: 93–104.
[23] Meng L, Li H H, Zhang L Y, Wang J K. QTL IciMapping: integrated software for genetic linkage map construction and quantitative trait locus mapping in biparental populations., 2015, 3: 269–283.
[24] McCouch S R. Gene nomenclature system for rice., 2008, 1: 72–84.
[25] 刘健, 牛付安, 江建华, 孙程, 陈兰, 郭媛, 付淑换, 洪德林. 多环境下粳稻产量及其相关性状的条件和非条件QTL定位. 中国水稻科学, 2012, 26: 144–154.Liu J, Niu F A, Jiang J H, Su C, Chen L, Gou Y, Fu S H, Hong D L. Location of conditional and unconditional QTL forrice yield and its related traits in multiple environments., 2012, 26: 144–154 (in Chinese with English abstract).
[26] 梁云涛, 潘英华, 徐志健. 利用野栽分离群体定位水稻粒型相关QTL. 西南农业学报, 2017, 30: 2161–216.Liang Y T, Pan Y H, Xu Z J. Rice grain type-related QTLs were identified by isolated populations in wild cultivation., 2017, 30: 2161–216 (in Chinese with English abstract).
[27] Lin Z, Yan J, Su J, Liu H, Hu C, Li G, Wang F, Lin Y. Novelmutant,, positively regulates grain shape in rice ()., 2019, 46: 857–868.
[28] Hu Z J, Lu S J, Wang M J, He H H, Sun L, Wang H R, Liu X H, Jiang L, Sun J L, Xin X Y, Kong W, Chu C C, Xue H W, Yang J S, Luo X J, Liu J X. A novel QTLencodes the GSK3/SHAGGY-like kinasethat interacts withto negatively regulate grain size and weight in rice., 2018, 11: 736–749.
[29] Shi C L, Ren Y L, Liu L L, Wang F, Zhang H, Tian P, Pan T, Wang Y F, Jing R N, Liu T Z, Wu F Q, Lin Q B, Lei C L, Zhang X, Zhu S S, Guo X P, Wang J L, Zhao Z C, Wang J, Zhai H Q, Cheng Z J, Wan J M. Ubiquitin specific protease 15 has an important role in regulating grain width and size in rice., 2019, 180: 381–391.
[30] Thomson M J, Tai T H, McClung A M, Lai X H, Hinga M E, Lobos K B, Xu Y, Martinez C P, McCouch S R. Mapping quantitative trait loci for yield, yield components and morphological traits in an advanced backcross population betweenand thecultivar Jefferson., 2003, 107: 479–493.
[31] Xu F, Fang J, Ou S J, Gao S P, Zhang F X, Du L, Xiao Y H, Wang H R, Sun X H, Chu J F, Wang G D, Chu C. Variations incoding region influence grain size and yield in rice., 2015, 38: 800–811.
QTL detection and stability analysis of rice grain shape and thousand-grain weight based on chromosome segment substitution lines
WANG Xiao-Lei1, LI Wei-Xing1, ZENG Bo-Hong2, SUN Xiao-Tang1, OU-YANG Lin-Juan1, CHENXiao-Rong1, HE Hao-Hua1,*, and ZHU Chang-Lan1,*
1Key Laboratory of Crop Physiology, Ecology and Genetic Breeding (Jiangxi Agricultural University), Ministry of Education / Jiangxi Super Rice Engineering Technology Research Center, Nanchang 330045, Jiangxi, China;2Jiangxi Super Rice Research and Development Center of Jiangxi Academy of Agricultural Sciences, Nanchang 330200, Jiangxi, China
Grain shape and 1000-grain weight are the important factors affecting rice yield. Discovering the excellent genes of these traits is of great significance for super high yield rice breeding. In this study, a set of chromosome segment substitute lines (CSSLs), derived from a cross between Koshihikari (acultivar, as donor patent) and Changhui 121 (anrestorer line, as a background patent), were used to quantitative trait locus (QTLs) detection and stability analysis in three environments. The results showed that a total of 59 QTLs were identified on chromosomes 1, 2, 3, 4, 5, 6, 7, 10, 11, and 12, respectively, whose contribution rate was 0.77%–36.26%. Among them, 10 pleiotropic QTLs were found, and,,,,, andcould all be detected in three environments. Furthermore,is a novel identified QTL locus. These results lay a foundation for further fine mapping, cloning and marker-assisted breeding of grain shape genes.
rice; chromosome segment substitution lines (CSSLs); grain shape; 1000-grain weight; quantitative trait locus (QTL)
10.3724/SP.J.1006.2020.02008
本研究由国家自然科学基金项目(31860373), 国家转基因生物新品种培育重大专项(2016-ZX08001-002)和江西省“5511”优势科技创新团队项目(2016-5BCB19005)资助。
This study was supported by the National Natural Science Foundation of China (31860373), the National Major Project for Developing New GM Crops (2016-ZX08001-002), and the “5511” Superior Science and Technology Innovation Team Project of Jiangxi Province, China (2016-5BCB19005).
朱昌兰, E-mail: zhuchanglan@163.com;贺浩华, E-mail: hhhua64@163.com
E-mail: wxl0vip@163.com
2020-02-13;
2020-06-02;
2020-06-23.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20200622.1834.018.html
