生物信息学分析长链非编码RNA在结肠癌中的调控网络
2020-09-11韩泽平李艳黎毓光吕钰冰何思华何金花
韩泽平 李艳 黎毓光 吕钰冰 何思华 何金花
结肠癌是全球常见的消化道恶性肿瘤之一,其发病率居常见恶性肿瘤的第3位,病死率也为癌症死亡的第3位[1],严重威胁人类的生命和健康。由于早期结肠癌患者临床症状较隐匿,多数结肠癌患者被发现时已至中晚期。目前,手术切除仍是治疗结肠癌的首选方法,但术后高复发率、高转移率和5年低生存率等难题一直未有效解决,结肠癌患者的预后仍然很差[2],因此寻找新的诊断和治疗方法尤为重要。近年发现,一些长链非编码RNA(long non-coding RNA,lncRNA)可作为结肠癌诊断、预后的生物标志物,广泛影响结肠癌细胞增殖、侵袭、凋亡、转移和耐药能力[3]。lncRNA是一类转录本长度>200 nt的非编码 RNA(non-coding RNA,ncRNA),其中绝大多数不具有或仅具有有限的蛋白质编码能力,但是它们能对mRNA转录后加工,包括5忆加帽、剪接和聚腺苷酸化,参与机体生理及病理过程[4]。
生物信息学是在生命科学的研究中,以计算机为工具对生物信息进行储存、检索和分析的科学。利用生物信息学方法可预测lncRNA在结肠癌发生、发展中的调控网络,可大大节省实验室验证的工作量。本研究运用生物信息学方法,从lncRNA出发探索其与上游转录因子、下游靶微小 RNA(microRNA,miRNA)及 miRNA 靶基因间的调控关系,以更好理解lncRNA在结肠癌发生、发展过程中的作用机制,并为下一步实验验证其分子调控网络机制提供线索。
1 材料和方法
1.1 材料 本研究所用到的生物学信息软件和数据库如下:lncRNA 疾病数据库(http://www.cuilab.cn/lncr原nadisease),治疗lncRNA疾病关联数据的实验资源,是一种用于预测新型lncRNA相关疾病的综合工具[5]。Starbase v2.0(http://starbase.sysu.edu.cn/),系统地识别和确立 RNA-RNA、protein-RNA、miRNA-mRNA 和 miR原NA-lncRNA的交互网络[6]。人类miRNA疾病数据库(HMDD v3.0,http://www.cuilab.cn/hmdd),集人 miRNA的实验验证支持和疾病关联的数据库[7]。PubMed(https://www.ncbi.nlm.nih.gov/pubmed);Consite(http://consite.genereg.net/),通过基因结合位点进行预测的在线数据工具,用于查找基因组序列中的顺式调控元件[8]。Long Noncoding RNA Database v2.0(http://www.lncrnadb.org/);miRBase(http://www.mirbase.org/);miRwalk2.0(http://zmf.umm.uni-heidelberg.de/apps/zmf/mirwalk2/),一个关于miRNAs的在线软件,该数据库包含了关于人类、小鼠和大鼠等所有已知基因的信息,可用于预测和验证miRNA的结合位点[9]。Cytoscape软件。
1.2 方法
1.2.1 预测与结肠癌相关的lncRNAs 在lncRNA疾病数据库中通过“Browse”进入浏览界面,在疾病选项栏中选取“colon cancer”作为研究对象,筛选出和结肠癌相关的lncRNAs,并挑选出前3个研究较多的lncRNAs作为下一步的研究对象。
1.2.2 预测与lncRNAs相关的miRNAs 运用Starba原sev2.0在线软件可预测出与lncRNAs相关的miRNAs。通过“miRNA Target”进入“miRNA-lncRNA”调控网络预测界面,提交目的lncRNA后可获得预测相互结合的miRNAs。
1.2.3 查询已证实与结肠癌相关的miRNAs 通过HMDD v3.0查询已证实的与结肠癌相关的miRNAs。
1.2.4 筛选与lncRNAs和结肠癌相关的miRNAs 将上述所得出与lncRNAs相关的miRNAs和已证实与结肠癌相关的miRNAs作交集并筛选出共同的miRNAs。
1.2.5 预测与lncRNAs相关的转录因子 在Consite数据库中预测出各lncRNA的转录因子并作交集筛选出它们共同的转录因子,通过Pubmed查询转录因子在结肠癌中的调控机制。
1.2.6 预测各miRNA的转录因子 运用Consite数据库预测出各miRNA的转录因子并进行分析研究,筛选出上述miRNAs的共同转录因子,并通过Pubmed数据库查询转录因子与结肠癌的关系。
1.2.7 预测各miRNA的靶mRNA 通过miRwalk v2.0查询上述miRNAs的靶mRNA,并且进行交集后筛选出共同靶基因。并利用Pubmed数据库筛选出与结肠癌相关的共同靶基因。
1.2.8 绘制网络调控图 综合以上结果,运用Cytoscape软件绘制出lncRNA在结肠癌中的调控网络图。
2 结果
2.1 与结肠癌相关的lncRNAs 通过lncRNA疾病数据库查询得出与结肠癌相关的lncRNAs为H19、HO原TAIR、MALAT1、MEG3、GSEC、CYTOR、TUG1、RBM5-AS1、LINC-ROR、UCA1、UPAT、UHRF1、ATB、FER1L4、CCAT1、SNAR、CCAT2等 17个(表 1)。如表 1所示,该数据库收录的H19、HOTAIR、MALAT1与结肠癌的研究相对较多,因此本研究选取H19、HOTAIR和MALAT1作为进一步的研究对象。
2.2 与lncRNAs相关的miRNAs 在Starbase v2.0中预测得出与H19相关的miRNAs共39个,与HOTAIR相关的miRNAs共30个,与MALAT1相关的miRNAs共113个。其中 H19、HOTAIR、MALAT1 3个共同的靶miRNAs 为 miR-17、miR-20a、miR-20b、miR-93、miR-106a、miR-106b、miR-519d等7个miRNAs(图 1)。
2.3 已证实的与结肠癌相关的miRNAs 经HMDDv2.0输入“colon cancer”查询得已证实过的与结肠癌相关的miRNAs共90个(图 2)。
2.4 与lncRNAs和结肠癌相关的miRNAs 将 H19、HOTAIR、MALAT1共同的7个靶miRNAs与上述所得的已证实与结肠癌相关的90个miRNAs作交集后筛选得共同靶miRNAs miR-17、miR-20a、miR-106a 和miR-106b等与结肠癌密切相关,并将它们纳入后续研究对象(图3)。
2.5 与lncRNAs相关的转录因子 在Consite中分别预测3个lncRNAs的转录因子,进而筛选出它们的共同转录因子;在设定cut-off为90%的条件下预测得H19共有12个转录因子,HOTAIR的转录因子共18个,MALAT1的转录因子共31个;其中 E74A、ARNT、Thing1-E47、Hunchback、Snail为lncRNAs的共同转录因子(图 4)。
2.6 与miRNAs相关的转录因子 在Consite分别预测各miRNA的转录因子,并且筛选出它们的共同转录因子;在设定cut-off为70%的条件下预测得到miR-17的转录因子共14个,miR-20a的转录因子共18个,miR-106a的转录因子共15个,miR-106b的转录因子共7个;取交集后 Sox-5、FREAC-4、Sox-17、ARNT、Snail为miRNAs的共同转录因子(图5)。
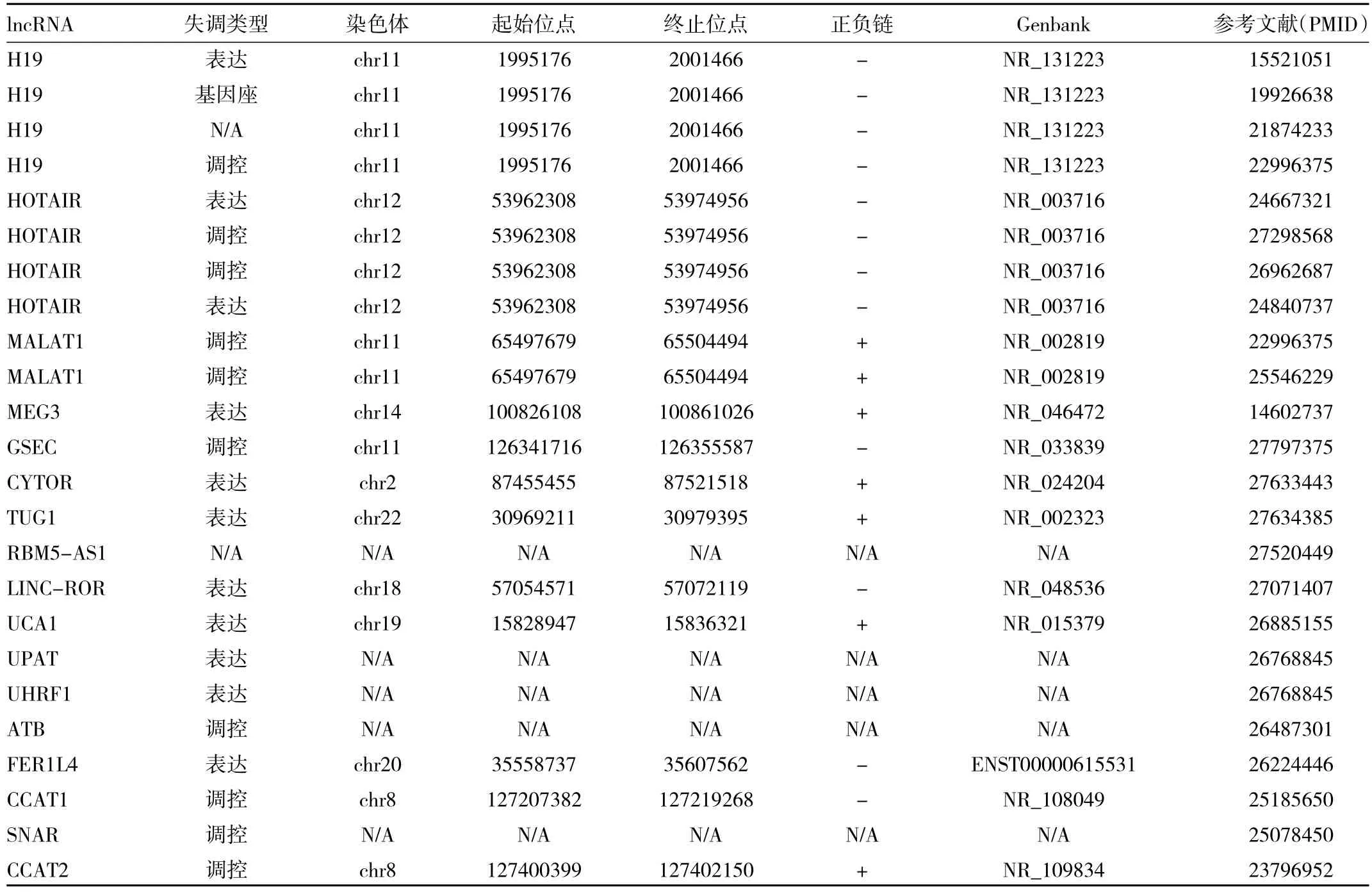
表1 与结肠癌相关的lncRNAs
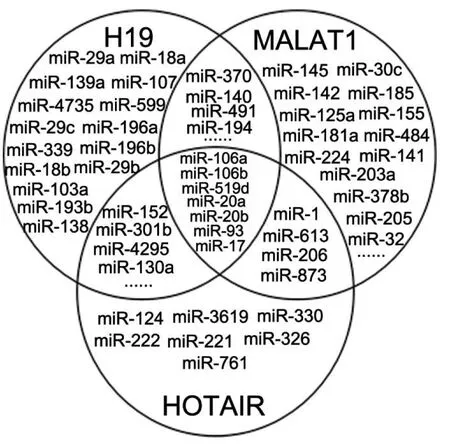
图1 与lncRNAs相关的miRNAs
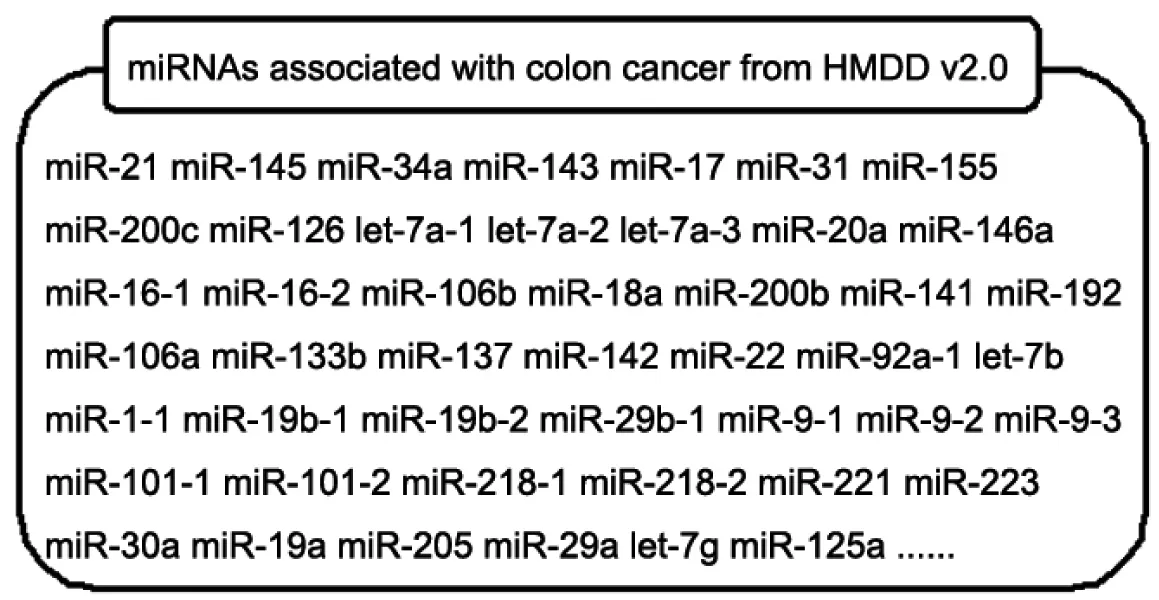
图2 已证实的与结肠癌相关的部分miRNAs
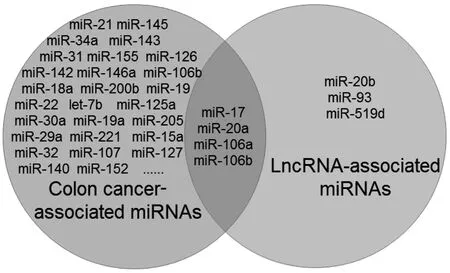
图3 与lncRNAs和结肠癌相关的miRNAs
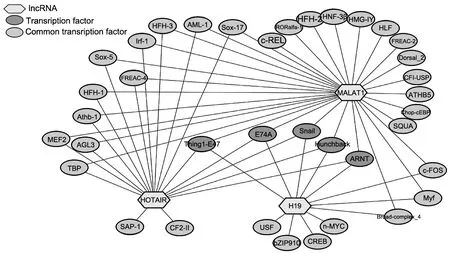
图4 各lncRNA的转录因子及其共同转录因子
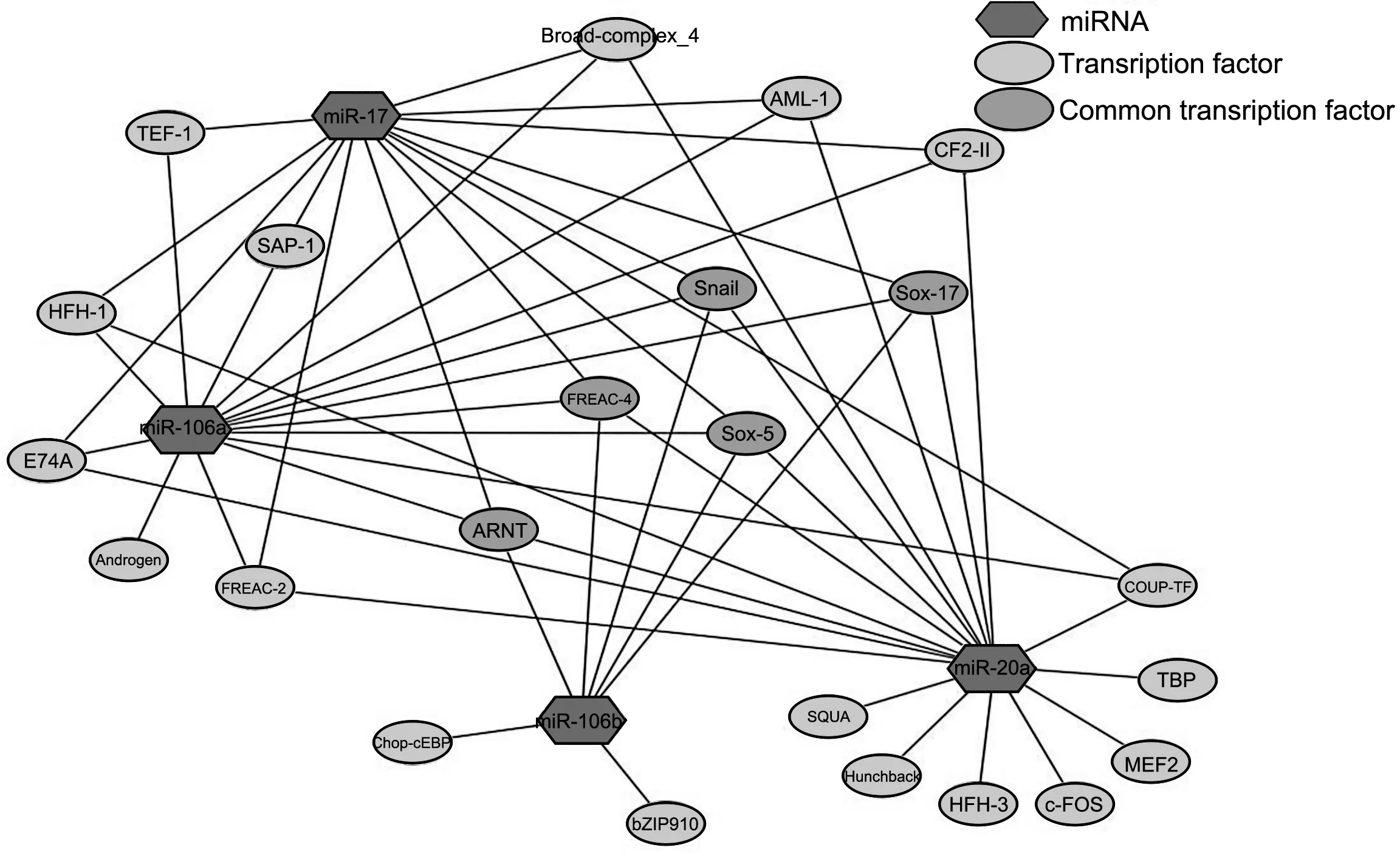
图5 各miRNA的转录因子及其共同转录因子
2.7 与结肠癌相关的转录因子 通过Pubmed查阅文献找到已有文献证实与结肠癌相关的转录因子,其中已证实与结肠癌相关的lncRNAs转录因子为ARNT和Snail,miRNAs转录因子中 ARNT、Snail、Sox-17 参与结肠癌的发生和发展。其中,ARNT和Snail为lncRNAs与miRNAs的共同相关转录因子(图6)。
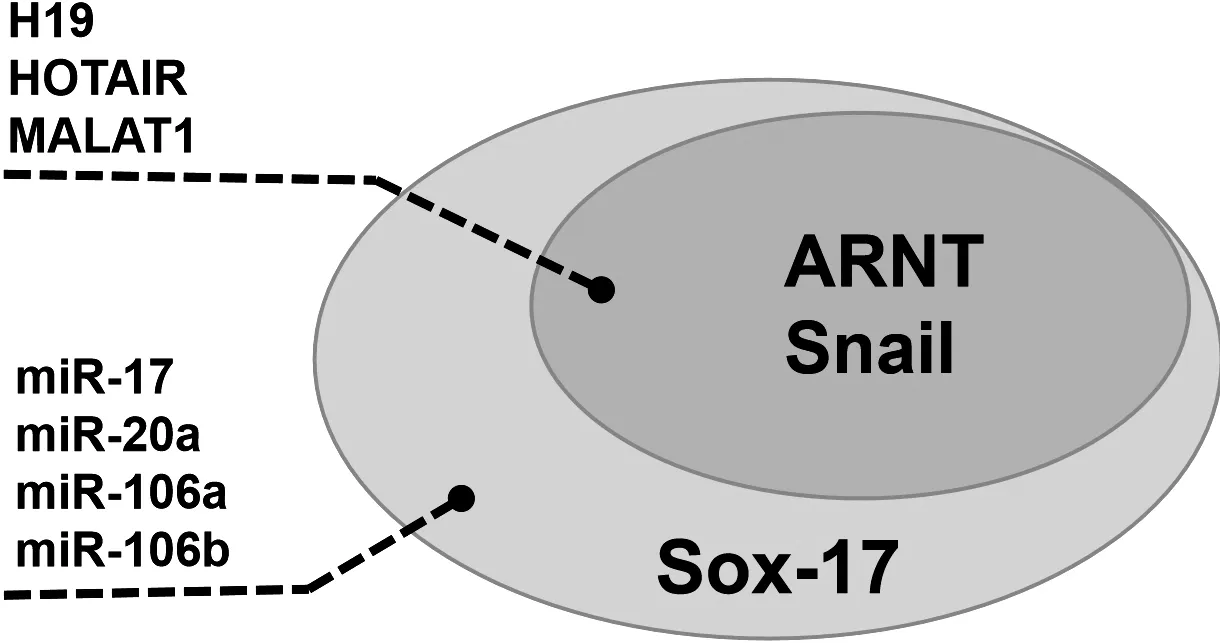
图6 与结肠癌相关的转录因子
2.8 与结肠癌相关miRNA的靶mRNA 运用miRwalk v2.0经Predicted Target Module寅MicroRNA-geneTargets查询页面进入,分别输入 miR-17、miR-20a、miR-106a、miR-106b预测得各miRNA的靶mRNA。其中同时满足11个或以上预测模块的条件下预测得miR-17共42个靶基因,miR-20a共50个靶基因,miR-106a共42个靶基因,miR-106b共36个靶基因。进一步运用一致法筛选得到 4个 miRNAs的共同靶基因为 HLF、SORL1、ZFYVE26、PFN2、PKD2、PKN2、PTPN4、RBL2、WEE1、FZD3、RGL1、MKRN1、C7orf43、NTN4、ZFP91等15个靶基因(见表2)。经Pubmed数据库查询,筛选得PKD2、PKN2、WEE1、ZFP91等4个靶基因与结肠癌相关。

表2 与结肠癌相关的miRNA的靶基因
2.9 绘制miRNA在结肠癌中的分子调控网络图 根据上述结果,在lncRNA这一环,通过数据库预测并筛选得到H19、HOTAIR、MALAT1等3个与结肠癌相关的lncRNAs,进一步分别预测出它们的下游miRNAs并且用一致法筛选得到共同的miRNAs有miR-17、miR-20a、miR-20b、miR-93、miR-106a、miR-106b、miR-519d 等 7个。同样运用数据库查询得到90个与结肠癌相关的miRNAs。与上述 H19、HOTAIR、MALAT1 等 lncRNAs相关的 miRNAs 取交集得 miR-17、miR-20a、miR-106a、miR-106b等4个与结肠癌密切相关的miRNAs。同时,在转录因子水平预测出3个lncRNAs共同的转录因子为 E74A、ARNT、Thing1-E47、Hunchback、Snail 5 个。此外,预测出4个miRNAs的共同转录因子为Sox-5、FREAC-4、Sox-17、ARNT、Snail 5 个,在 Pubmed 中查阅有文献证明与结肠癌相关的转录因子有ARNT、Sox-17、Snail,并且ARNT和Snail同时与lncRNAs 和miRNAs相关。最后预测得 miR-17、miR-20a、miR-106a、miR-106b分别可共同调控HLF、SORL1、ZFYVE26、PFN2、PKD2、PKN2、PTPN4、RBL2、WEE1、FZD3、RGL1、MKRN1、C7orf43、NTN4、ZFP91等15个靶基因,其中PKD2、PKN2、WEE1、ZFP91 可找到相关文献证实与结肠癌有关。综合以上,构建出miRNA在结肠癌中的分子调控网络图(图苑)。
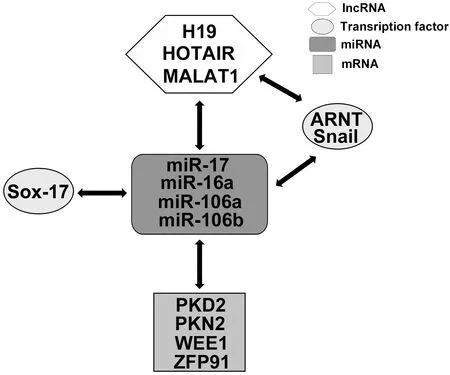
图7 miRNA在结肠癌中的分子调控网络图
3 讨论
生物信息学是一门新兴的学科,通过综合数学、统计学、计算机科学与工程、生物学的工具与技术研究生物信息的采集、处理、存储、分析和解释各种信息的生物学意义,并予以管理和利用的一门科学,其目标是发展和利用先进的计算技术解决生物学难题[10]。生物信息学依靠计算机工具和互联网技术开发出各种用于基因研究的数据库,通过分析、处理实验数据和公共数据可预测及筛选出研究者想要的信息,加快了研究进度,缩短了科研时间;利用实验数据分析所得的结论可设计下一阶段的实验,并可用计算机管理实验数据并预测新基因及其结构和功能,从而一步步破解复杂的生物信息,为各种疾病的诊断、治疗提供更明确的方向。
本研究运用生物信息学分析lncRNA在结肠癌发生、发展中的分子调控网络。利用lncRNA疾病数据库查询得与结肠癌相关的lncRNAs有17个(见表1),并且选取H19、HOTAIR、MALAT1等前3个与结肠癌相关性最高的lncRNAs为研究对象。研究报道,lncRNA H19在结肠癌组织中表达上调,并通过MAPK信号通路促进结肠癌细胞的迁移和侵袭[11];Luo等[12]从80个结肠癌组织和肿瘤邻近正常结肠组织中检测HOTAIR的表达发现HOTAIR在结肠癌组织中的表达显著高于匹配的肿瘤相邻正常结肠组织,在淋巴结转移病例中的表达明显高于无转移病例,在低分化和未分化病例中高于中度分化病例,HOTAIR在结肠癌可能扮演癌基因角色。MALAT1是结肠癌中的一个致癌基因,可通过上调H3K27三甲基化和抑制GSK-3蛋白表达进而抑制茁-catenin降解,促进结肠癌的生长[13]。
lncRNA可通过竞争性结合miRNA,抑制miRNA表达及其对下游靶基因的负向调控作用[14]。本研究进一步在 Starbase v2.0中预测得 H19、HOTAIR、MALAT1 3个共同的 miRNA 为 miR-17、miR-20a、miR-20b、miR-93、miR-106a、miR-106b、miR-519d等 7个。HMDD v2.0数据库查询得出与结肠癌相关的miRNAs共90个,进一步将H19、HOTAIR、MALAT1共同的7个靶miRNAs与上述已证实与结肠癌相关的90个miRNAs作交集后筛选出共同的 miRNAs,分别为 miR-17、miR-20a、miR-106a和miR-106b。通过Pubmed文献分析,miR-17在结肠癌早期表达显著上调[15-16]并且在结肠癌晚期通过抑制其下游基因PTEN的表达而促进了结肠癌细胞的增殖和侵袭[17]。miR-20a在结肠癌中表达上调,它通过负调控结肠间质成纤维细胞中其靶CXCL8 mRNA的表达抑制肿瘤细胞生长[16,18]。miR-106a结肠癌中处于高表达状态[16,19],并且有研究证明其与PTEN存在靶向调控关系,在结肠癌组织中起着致癌作用[19]。miR-106b表达增强,负向调控其靶DLC1并诱导结肠癌细胞的迁移和侵袭,为结肠癌患者的独立预后因素[20]。
众所周知,转录因子是参与目的基因表达的重要调控因子。笔者围绕转录因子与lncRNAs和miRNAs的靶向调控关系在Consite中分别预测各lncRNAs与miRNAs的转录因子并得到lncRNAs共同的转录因子为 E74A、ARNT、Thing1-E47、Hunchback、Snail等 5 个,miRNAs共同的转录因子为 Sox-5、FREAC-4、Sox-17、ARNT、Snail等5个。取交集后,在Pubmed中查阅文献发现已证实与结肠癌相关的转录因子为Sox-17、ARNT、Snail。Sox-17在结肠癌中通过Wnt信号通路发挥肿瘤抑制作用,在肿瘤早期阶段被Wnt激活诱导,在恶性进展过程中被甲基化下调[21]。ARNT在肿瘤早期通过诱导参与肿瘤生长和血管生成的基因广泛参与肿瘤的生长,但在肿瘤的侵袭和转移过程中起着负向作用,这说明ARNT在肿瘤不同时期的靶向治疗中有不同的提示作用[22],但尚未找到研究ARNT与结肠癌调控关系的资料。有关文献表明,Snail在结肠癌中表达上调,参与结肠癌的病程进展,与其晚期淋巴结转移、预后不良有关[23-24]。
通过miRwalk v2.0在线软件预测得出与结肠癌密切相关的miRNAs的共同靶mRNAs有 HLF、SORL1、ZFYVE26、PFN2、PKD2、PKN2、PTPN4、RBL2、WEE1、FZD3、RGL1、MKRN1、C7orf43、NTN4、ZFP91 等 15 个。有研究表明,PKD2是PKD在结肠癌中表达最丰富的亚型,其通过表达上调在肿瘤细胞的生长和增殖中起着积极作用[25]。PKN2在结肠癌中表达降低,其在正常结肠组织中表达高于息肉、腺瘤和转移性腺癌,并且在肿瘤中较高的表达倾向提示预后良好,PKN2在结肠癌中起着肿瘤抑制作用[26]。WEE1是一种癌基因,它在结肠癌中的表达上调与恶性程度高、预后差有关,通过抑制WEE1可以有明显的抗肿瘤作用,并且提示WEE1可能是P53突变型结肠癌有效的治疗靶点[27-28]。ZFP91在结肠癌中表达显著上调,并且通过正向调控HIF-1琢促进肿瘤细胞的血管生成、增殖和转移[29]。然而未找到HLF、SORL1、ZFYVE26、PFN2、PTPN4、RBL2、FZD3、RGL1、MKRN1、C7orf43、NTN4等与结肠癌的相关文献报道。
综上所述,本研究构建出了结肠癌相关的lncRNAs分子调控网络。通过这个相对系统的网络,我们可知在结肠癌中 H19、HOTAIR、MALAT1、miR-17、miR-20a、miR-106a、miR-106b、Snail、PKD2、WEE1、ZFP91 等 表达上调,促进了结肠癌细胞的生长和增殖,Sox-17、PKN2表达下调并且起着抑制作用,而ARNT在肿瘤生长期表达上调并起促进作用,在肿瘤转移和侵袭期表达下调并有着抑制功能。基于此调控网络,进一步的研究方向中能更有针对性地寻找治疗方法来降低H19、HOTAIR、MALAT1、miR-17、miR-20a、miR-106a、miR-106b、Snail、PKD2、WEE1、ZFP91 和肿瘤细胞生长期的ARNT的表达水平,以及提高Sox-17、PKN2、转移和侵袭期的ARNT的表达以达到有效治疗结肠癌的目的。
生物信息学分析技术始终运用于整个分子调控网络的构建过程中,虽然所得到的调控网络仅仅是从理论层面上推导而来,其可行性尚还需要进一步的实验加以证明,但为下一步的实验验证指明了方向。生物信息学分析与实验验证的结合为我们更准确高效地研究结肠癌提供了更多的依据和可能。
