新疆早熟陆地棉种质资源遗传多样性及纤维品质性状SSR 关联分析
2020-08-08徐濉喜王旭文田琴孔宪辉刘丽司爱君王娟余渝
徐濉喜,王旭文,田琴,孔宪辉,刘丽,司爱君,王娟,余渝*
(1.新疆农垦科学院棉花研究所/ 农业农村部西北内陆区棉花生物学与遗传育种重点实验室,新疆石河子832000;2. 中国农业大学农学院,北京100094)
新疆植棉历史悠久,是我国适宜种植棉花的区域之一,目前已成为我国棉花的主产区,每年80%以上的棉花销往全国各地和用于出口, 棉花种植业已经成为新疆经济的重要支柱及植棉区棉农的主要收入来源。 棉花品种作为棉花生产的基础,品种好坏直接影响到新疆棉区棉花产量与品质。 新疆棉花品种演变经历了从前苏联引进陆地棉阶段、 新疆自育陆地棉品种为主的阶段、以自育品种和内地引进品种并存3 个阶段。 位于天山北坡的北疆棉区,生长季节短,热量条件相对不足, 从70 年代末新陆早1 号在北疆大面积推广开始,北疆棉区进入了长期以自育早熟陆地棉为主的阶段。 受早熟性和棉花种质资源创新能力不足的限制,早熟陆地棉的遗传基础狭窄,适应新疆干旱多灾环境变化的能力十分脆弱; 因此,为拓宽新疆早熟陆地棉的遗传基础,对新疆早熟陆地棉种质资源的研究已迫在眉睫。
棉花的许多重要性状如产量、品质相关性状都属于数量性状,是由微效多基因控制,极易受环境影响,传统选择方法选择效率较低、周期长,分子标记辅助育种 (Marker-assisted selection,MAS)的出现,使育种家能够将分子育种与传统育种方法相结合,利用与目标基因紧密连锁的分子标记从分子水平上进行选择,从而减少选择的盲目性,有助于实现作物性状的综合改良。 随着分子标记技术的发展, 限制性片段长度多态性(Restrictionfragmentlength polymorphism,RFLP)、扩增片段长度多态性 (Amplified fragment length polymorphism,AFLP)、 随 机 扩 增 多 态 性DNA(Random amplified polymorphic DNA,RAPD)、简单序列重复(Simple sequence repeat,SSR)等分子标记技术相继用于棉花种质资源遗传多样的评价与关联分析中。其中SSR 标记,以其重复性好、多态性高等特点,目前被广泛用于种质资源遗传多样性的评价和关联分析。 近年随着高通量测序技术的发展, 单核苷酸多态性位点 (Single nucleotide polymorphism,SNP)得到广泛的应用,但成本较高。 因此,SSR 标记仍然是资源创制与评价中常用的标记之一。
本研究对219 份早熟陆地棉种质资源群体进行表型与基因型数据收集研究,用关联分析的方法检测与纤维品质性状关联的SSR 标记,发掘特异的种质资源和优异等位变异,为揭示现有种质资源优异基因、育种亲本的配制和棉花纤维品质性状分子标记辅助育种提供参考。

本研究材料来源于新疆农垦科学院棉花研究所早熟陆地棉育种中多年收集的219 份亲本材料,信息参见附表1(印刷版省略,本刊网站可查阅)。

本试验于2015 年安排在新疆农垦科学院棉花研究所石河子试验点种植,2016 年在新疆农垦科学院棉花研究所石河子、库尔勒2 个试验点种植,株距10 cm,平均行距库尔勒为38 cm,石河子为44 cm,2 次重复,各材料人工点播,膜下滴灌栽培,田间管理遵循当地常规田间管理。 试验采用随机区组设计,每个材料均种植2 行,2 次重复。 2015 年石河子田间试验中行长为4 m,种植材料189 份;2016 年石河子田间试验中行长为2.2 m,种植材料219 份;2016 年库尔勒田间试验中行长为2 m,种植材料219 份。

植株表型鉴定标准参照棉花行业标准NY/T 2673—2015[1]。8 月中下旬进行植株表型性状调查,选取每小区连续5~10 株, 调查株高(Height of plant,HP)、 第一果枝节位高度 (Height of first fruiting branch node,HFFBN)、 第一果枝节位(First fruiting branch node,FFBN)、单株果枝数(Number of fruit branch per plant,NFBP)、单株铃数 (Boll number per plant,BN);9 月中旬收取各材料中部10 个铃进行室内考种, 调查铃重(Boll weight,BW)、 衣分 (Lint percentage,LP)、 衣指(Lint index,LI)、籽指(Seed index,SI)、小区籽棉产量(Seed yield per plot,SYP);轧花后取10 g 以上皮棉用印度Premier 电子有限公司生产的全自动棉花纤维测试仪HFT9000 检测其品质指标,包括纤维上半部平均长度(Fiber upper half mean length,FUHML)、断裂比强度(Breaking tenacity,BT)、 长度纤维整齐度指数 (Uniformity index,UI)、断裂伸长率(Breaking elongation,BE)、马克隆值(Micronaire,Mic)。
DNA 提取参照Paterson 等[2]发表的CTAB法,SSR 实验操作程序,PCR 参照张小娟等[3]方法, 采用毛细管电泳分析仪Fragment Analyzer-XL960 SSR/Tilling 分析PCR 产物。
本研究所用SSR 标记为新疆农垦科学院棉花研究所实验室保存标记引物,由上海生工公司合成。 本研究标记来源:参考Liang 等[4]发表的四倍体棉花遗传图谱, 并进一步结合钱能[5]、Song等[6]、薛艳等[7]、Sun 等[8]、艾先涛等[9]的研究结果,选取557 对引物,经过自然群体筛选过后得到的298 对有多态性的标记。选取12 份地理来源差异大的材料对298 对SSR 引物进行筛选,将条带差异大、清晰易读有多态性引物留下备用,共得到128 对多态性引物。
对电泳结果使用ProSize2.0 软件查看, 采用0、1 统计法,依据读胶视图中Ladder 判断样品不同位点片段大小,同一位点,有条带记为“1”,无条带记为“0”,用“a”、“b”、“c”、“d”、“e”(对应片段大小由大到小)区别一个标记在材料中的多个多态性位点。

利用R 语言将不同环境的表型数据进行最佳线性无偏估计 (Best Linear Unbiased Prediction,BLUP)分析,并用SPSS 19.0 对其进行描述性统计; 利用MS Excel 对引物的多态性信息含量(Polymorphism information content,PIC)计算。计算公式如下:PIC=1-∑Pij2,Pij表示SSR 引物i的第j个等位基因出现的频率。
通过NTsys-pc2.1 计算材料两两之间的遗传相似系数(Genetic similarity,GS),通过MS Excel计算遗传相似系数分布的区间,绘制频率分布图。
利用Structure 2.3.1 软件进行群体结构估计,Structure 参数设置分别是:K 值选取1~10,重复次数为5; 将MCMC (Markov chain Monte Carlo)起始时的不作数迭代(Length of burn in period)设为10 000 次,不作数迭代后将MCMC 设为100 000 次,其余参数采用软件默认的设置。最后, 依据最大似然值的原则确定合适的K值,如果似然值随K值持续增大,则根据ΔK来确定合适的K值[10],ΔK计算公式如下:ΔK=m[|L(K+1)-2L(K)+L(K-1)|]/S|L(K)|,L(K)表示每个K的对数值,S表示标准差,m表示平均值得到该K值对应的Q 矩阵(遗传相似性比例,即第i材料的基因组变异来源于第K群体的概率)。

利用Tassel 5.0[11]软件将基因型数据生成亲缘(Kinship)关系矩阵(K 矩阵),以Q值+亲缘关系作为协变量,利用混合线性模型(Mixed linear model,MLM)进行性状和标记之间的关联分析,同时计算标记位点在P=0.01 时对表型变异的贡献率(R2)。
在已获得关联位点的基础上参照钱能[5]的方法,计算SSR 位点等位变异表型效应,统计和分析与表型性状显著关联的等位变异位点、表型效应及典型材料。 表型变异计算公式:ai=Σxij/ni-ΣNK/nK。
其中ai代表第i个等位变异的表型效应值,xij为携带第i个等位变异的第j材料性状表型测定值,ni为具有第i等位变异的材料数。NK为所有材料的表型测定值,nK为材料数。若ai为正,则认为该等位变异为增效等位变异,反之为减效等位变异。 最终获得与表型性状显著关联的位点等位变异、表型效应及典型材料。

由表1 可知,在15 个性状中,第一果枝节位高度变异系数最大,为12.69%;长度整齐度指数变异系数最小,为0.64%。 从株型相关性状、产量相关性状和品质相关性状整体来看,有2 个株型相关性状异系数小于7%, 有4 个产量相关性状变异系数小于7%, 而所有品质相关性状的变异系数均小于7%。 因此,整体来看,此群体株型相关性状的多样性大于产量相关性状,大于品质相关性状。 BLUP 之后各性状偏度系数绝对值都小于1,除衣指、衣分与长度整齐度指数的峰度绝对值大于2 之外,其余各性状峰度绝对值均小于2,均属正态分布,可用于后续分析。
对15 个性状2015—2016 年3 个环境的表型数据进行方差分析,结果如表2。 可以看出,15
个性状都在P=0.01 显著水平上受到遗传效应和环境效应的影响。
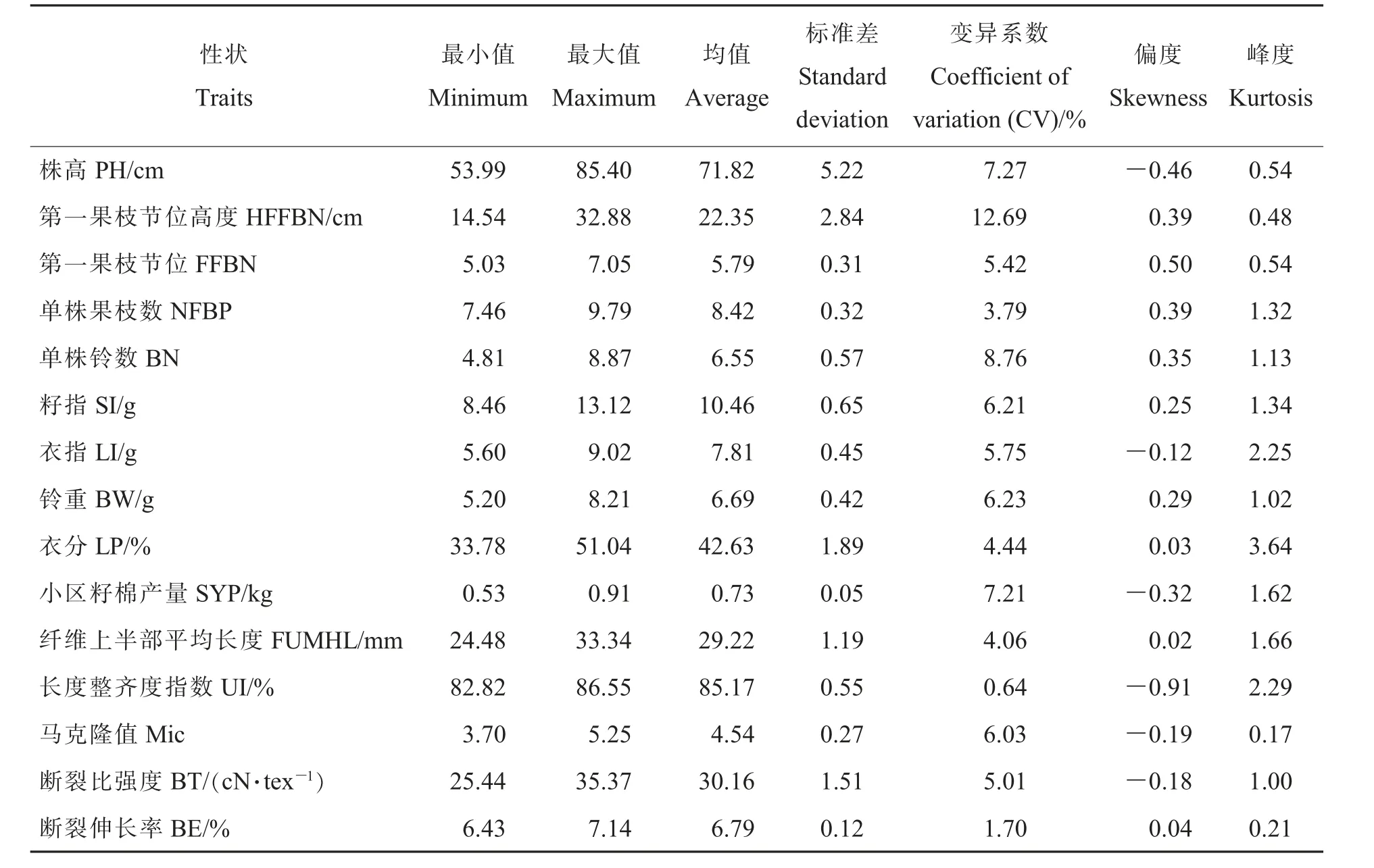
表1 各性状BLUP 结果描述统计Table 1 The statistical analysis of different traits after BLUP
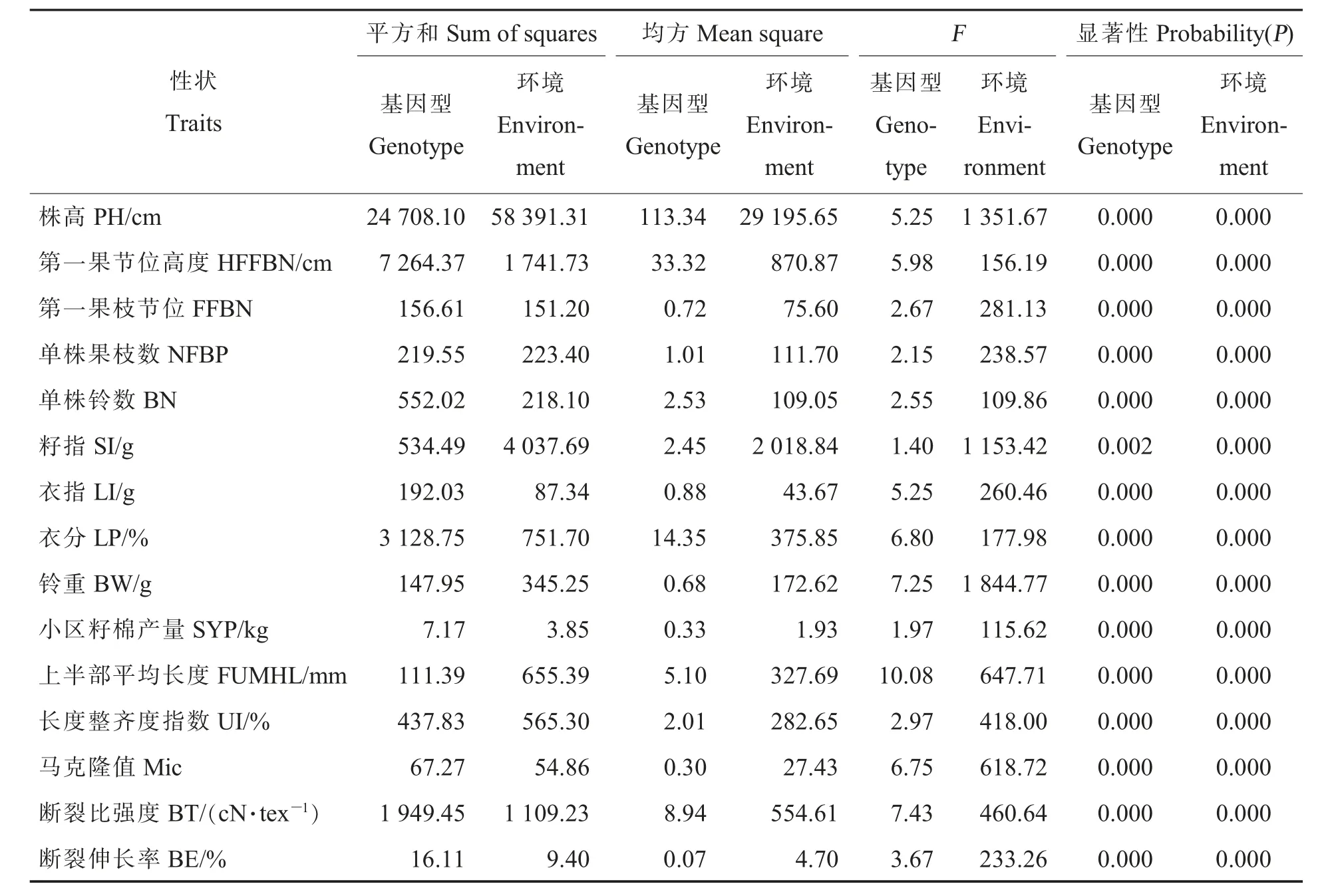
表2 各性状在多环境中的方差分析Table 2 ANOVA for different traits from multi-environments

用筛选出的多态性好的128 对引物对219份供试材料进行扩增, 共检测到244 个等位变异,平均每对引物检测出1.91 个等位变异。 扩增条带数范围为1~5 个,其中JESPR153、HAU1952扩增出5 个条带,扩增出1 条带的引物37 个,占总引物的28.9%,扩增出2 条以上条带的引物91对,占总引物的61.1%,所占比例较高。 PIC 值最大的引物是NAU3736 和NAU4926,PIC 值最小的引物是NAU1350(表3)。引物多态信息含量范围为0.13~0.86,平均为0.63。说明选取的标记引物多态性较高,可用于关联分析。
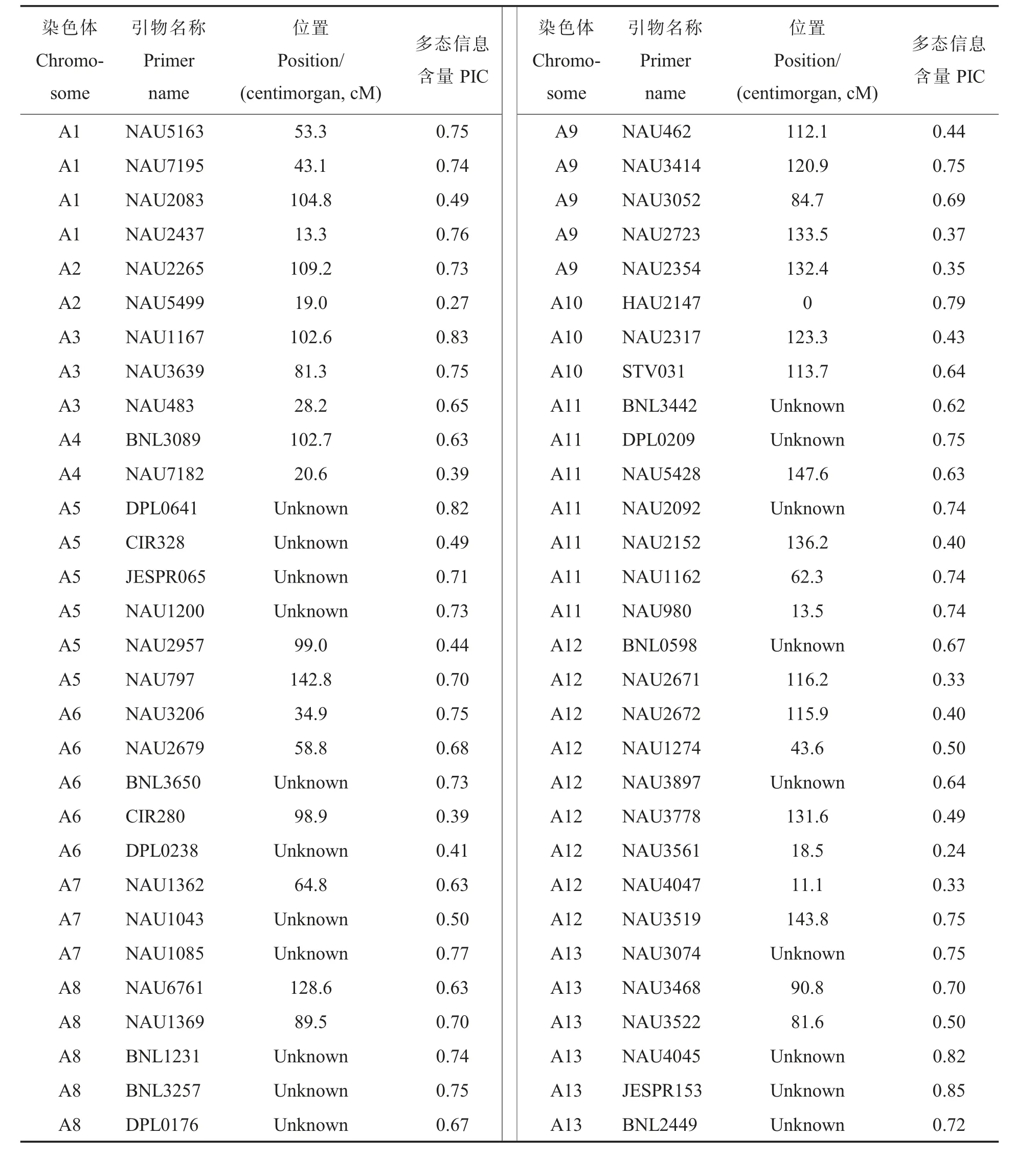
表3 128 对引物信息Table 3 The information of 128 pairs of polymorphic markers
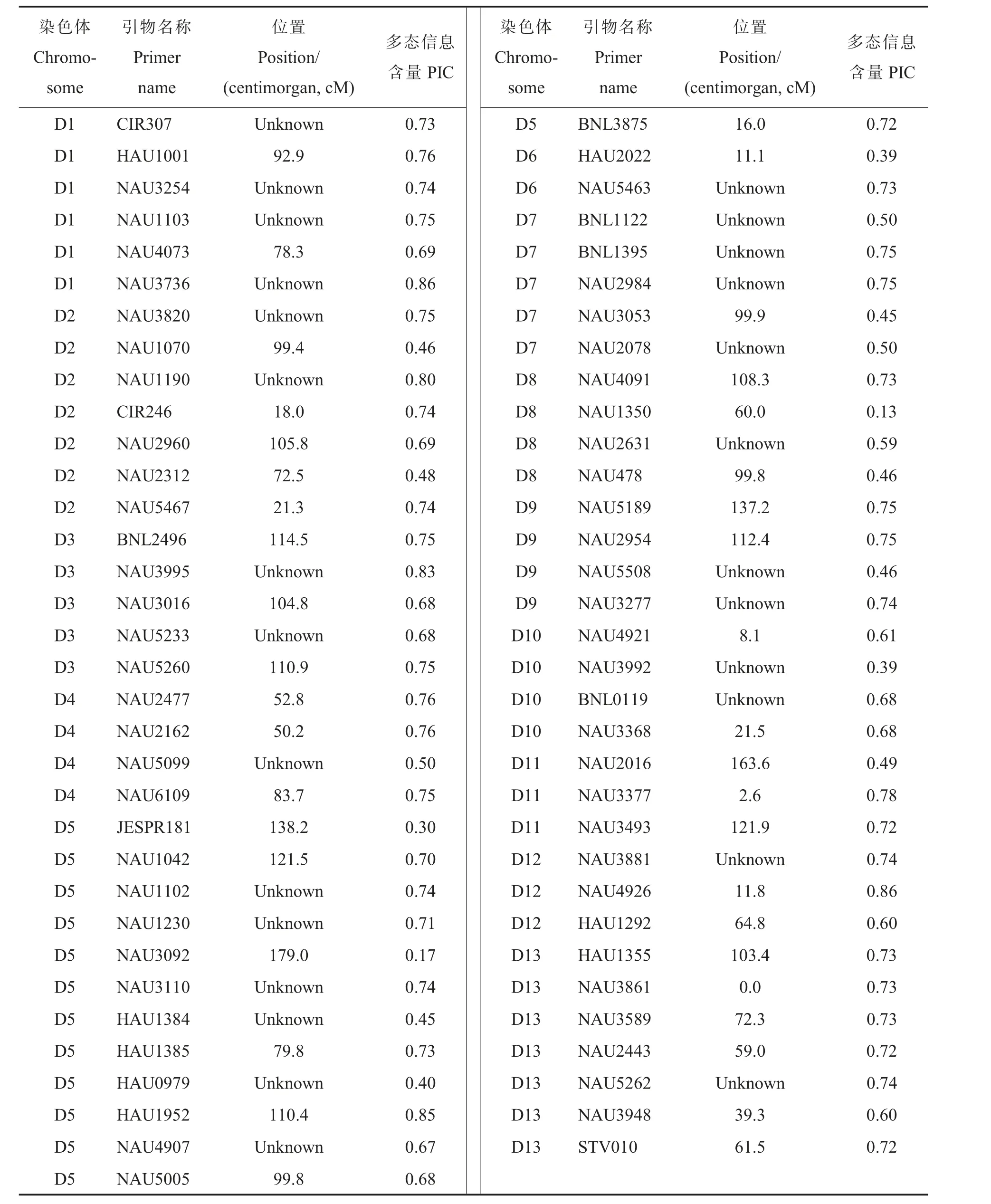
表3 (续)Table 3 (Continued)

用NTsys-pc2.1 计算219 份材料两两之间SM 遗传相似性系数,用MS Excel 绘制遗传相似性系数频率分布图见图1。 此群体遗传相似性系数分布范围为0.42~0.99, 平均为0.61, 处于0.4~0.5 的占2.01%, 处于0.5~0.6 的材料占44.66%,处于0.6~0.7 的占46.63%,处于0.7~0.8 的占5.74%,处于0.8~0.9 的占0.64%。 此群体中, 遗传相似系数在0.5~0.7 的材料数占90.19%。 说明此自然群体遗传基础狭窄,遗传多样性低。
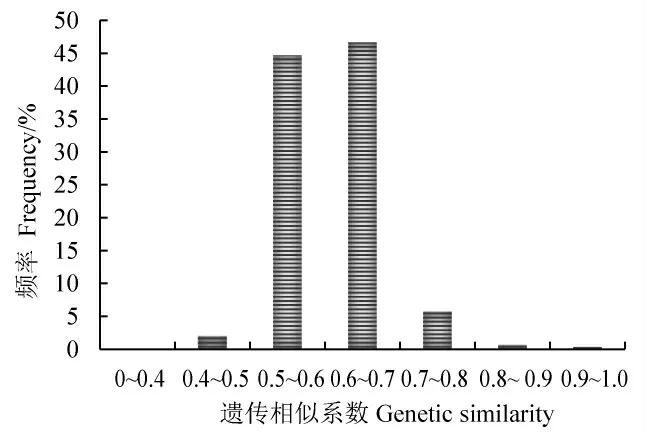
图1 219 份材料遗传相似性系数频率分布Fig. 1 Distribution of genetic similarity of 219 materials
由K与ln(P(D))的折线图(图2A)可以看出,随着K值的增大,ln(P(D))也持续增大,由于整个折线图非常平滑,无法确定K的取值。 由ΔK随K变化的曲线图(图2B)可以看出,当K=2 时,ΔK的值最大, 即认为此自然群体可以分为2 个亚群。Structure 软件依据计算得到的Q值大于或等于0.6,将各个材料划分到亚群中,第一亚群包括28 份材料,第二亚群包括156 份材料,另外35个材料的Q值在2 个类群中都不大于0.6, 被分到混合亚群中(图2C、表4)。 对此自然群体进行基于基因型的主成分分析结果(图2D),可以看出在群体数为2 时可以较好区分群体,因此更加确定K=2 为真实K值。
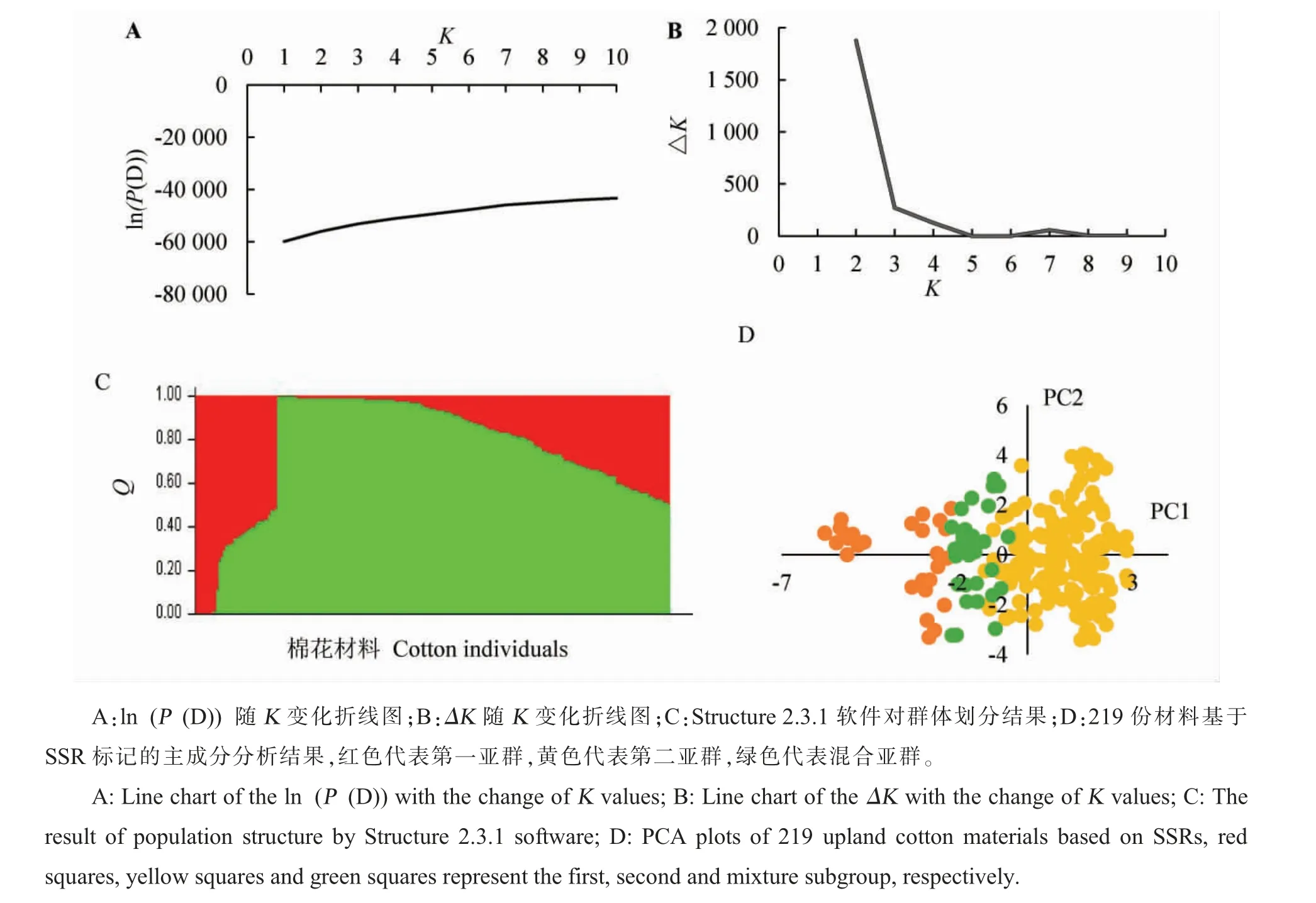
图2 供试材料的群体结构分析Fig. 2 Population structure analysis of experimental materials

2.5.1纤维品质性状显著关联标记。 5 个棉花纤维品质性状在3 个环境下共检测到29 个显著关联位点(表5)。其中,断裂比强度在3 个环境下共检测到10 个显著关联位点, 是5 个性状中检测到显著关联位点最多的,马克隆值检测到4 个显著关联位点,断裂伸长率检测到9 个显著关联位点,纤维上半部平均长度和长度整齐度指数均检测到3 个显著关联位点。

表4 219 份材料群体划分Table 4 The population division of 219 experimental materials
3 个环境的纤维品质数据BLUP 之后与基因型数据关联分析, 共检测到13 个与纤维品质性状显著关联的标记。 检测到4 个与断裂比强度显著关联的标记, 分别是JESPR153e、NAU5499、NAU5233a 和NAU3881b, 解释的表型变异分别为5.38%、5.24%、3.69%和3.63%; 检测到2 个与马克隆值显著关联的标记,分别为NAU7195a 和NAU7195b, 解释的表型变异为3.36%和3.92%;检测到3 个与断裂伸长率显著关联的标记,分别为DPL0641b、DPL0641a 和NAU4073a, 解释的表型变异分别为8.29%、6.95%和3.19%; 检测到2 个与纤维上半部平均长度显著关联的标记,NAU5499 和JESPR153e,解释的表型变异分别为5.02%和3.19%; 检测到2 个与长度整齐度指数显著关联的标记,分别为NAU3016b 和NAU5499,解释的表型变异分别为3.60%和3.32%。
同一标记扩增位点中,有2 个及以上位点与同一性状显著关联的标记有NAU7195 和DPL0641,其中NAU7195a 和NAU7195b 与马克隆 值 显 著 关 联,DPL0641a、DPL0641b 与 断 裂 伸长率显著关联。 与多个性状显著关联的标记有JESPR153 和NAU5499。 其中,JESPR153e 与纤维上半部平均长度和断裂比强度显著关联,NAU5499 同时与纤维上半部平均长度、 断裂比强度、断裂伸长率、长度整齐度指数显著关联,这两个标记可用于纤维品质综合性状的改良。
2.5.2稳定的显著关联位点与前人定位的QTL位点比较。 在2 个及以上的环境显著关联到的位点被认为是具有一定稳定性的位点, 结果如表6所示, 共有11 个位点在2 个及2 个以上环境与表型显著关联。其中:4 个位点与断裂比强度显著关联, 对应的分子标记位点为JESPR153e、NAU3881b、NAU5233a、NAU5499;3 个位点与断裂伸长率显著关联;2 个位点与马克隆值显著关联;NAU5499 与纤维上半部平均长度和长度整齐度指数显著关联。 在这些位点中,JESPR153e与纤维断裂比强度显著关联,且在3 个环境及在性 状 BLUP 后 都 可 以 检 测 到;DPL0641b、NAU5499、NAU7195b 在两个环境与BULP 结果中可以检测到;其余7 个标记均只在1 个环境和在BLUP 中检测到。
另外,与已报道定位的棉花QTL 进行比较,从上述11 个标记位点中鉴定出4 个标记在他人研究结果中重复出现, 前人研究结果显示:JESPR153 关联纤维强度[12],强度、马克隆值[5],纤维强度[13],铃重[14];NAU5233 关联衣分、籽棉产量[15];NAU5499 关联铃重[16];NAU4073 关联强度、马克隆值[5]以及生育期、霜前籽棉、霜前皮棉[17]。
依据表型BLUP 结果共挖掘出7 个携带特异等位变异位点的典型材料,如表7。27 号、65 号材料是典型的携带断裂比强度变异位点的材料,其中JESPR153e 位点使27 号材料的表型值降低4.72 cN·tex-1,NAU3881b 位点使65 号材料的表型值增加4.88 cN·tex-1; 材料10、189 是典型的携带马克隆值变异位点材料,其中NAU7195a 位点使10 号材料的马克隆值降低0.84,NAU7195b使189 号材料的马克隆值增加0.71;172、189 是典型的携带断裂伸长率变异位点的材料, 其中DPL0641a 使172 号材料的断裂伸长率增加0.49%,DPL0641b 使189 号材料的断裂伸长率减少0.36%;NAU5499 使35 号材料的纤维上半部平均长度增加4.12 mm; 同时,NAU5499 使164号材料的长度整齐度指数增加1.38%。
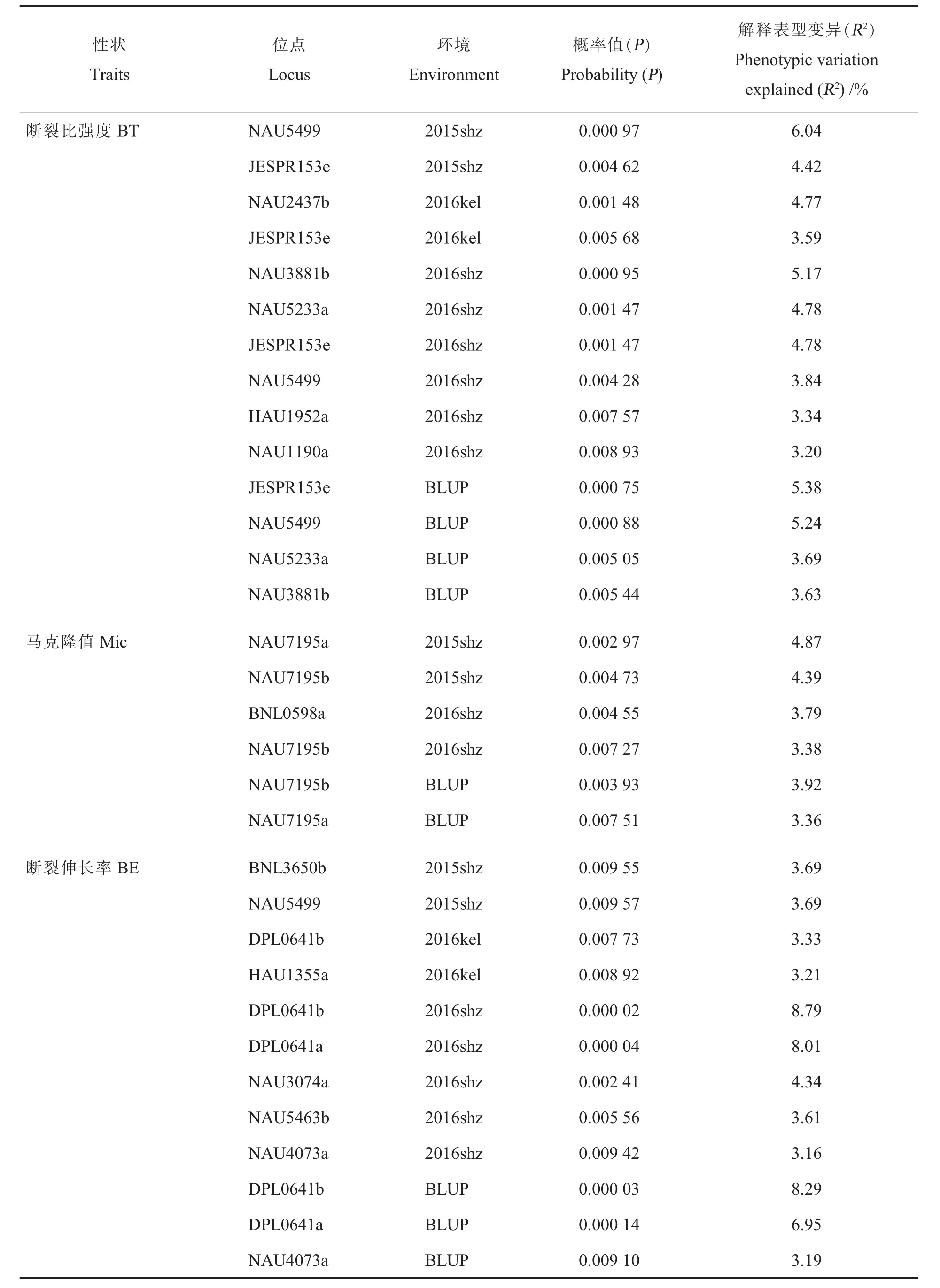
表5 各环境下与纤维品质性状显著关联的SSR 位点Table 5 SSR loci significantly associated with fiber quality traits under different environments
表5 (续)Table 5 (Continued)

表6 在2 个及以上环境检测到的显著关联的位点Table 6 Significantly associated loci detected in two or more environments
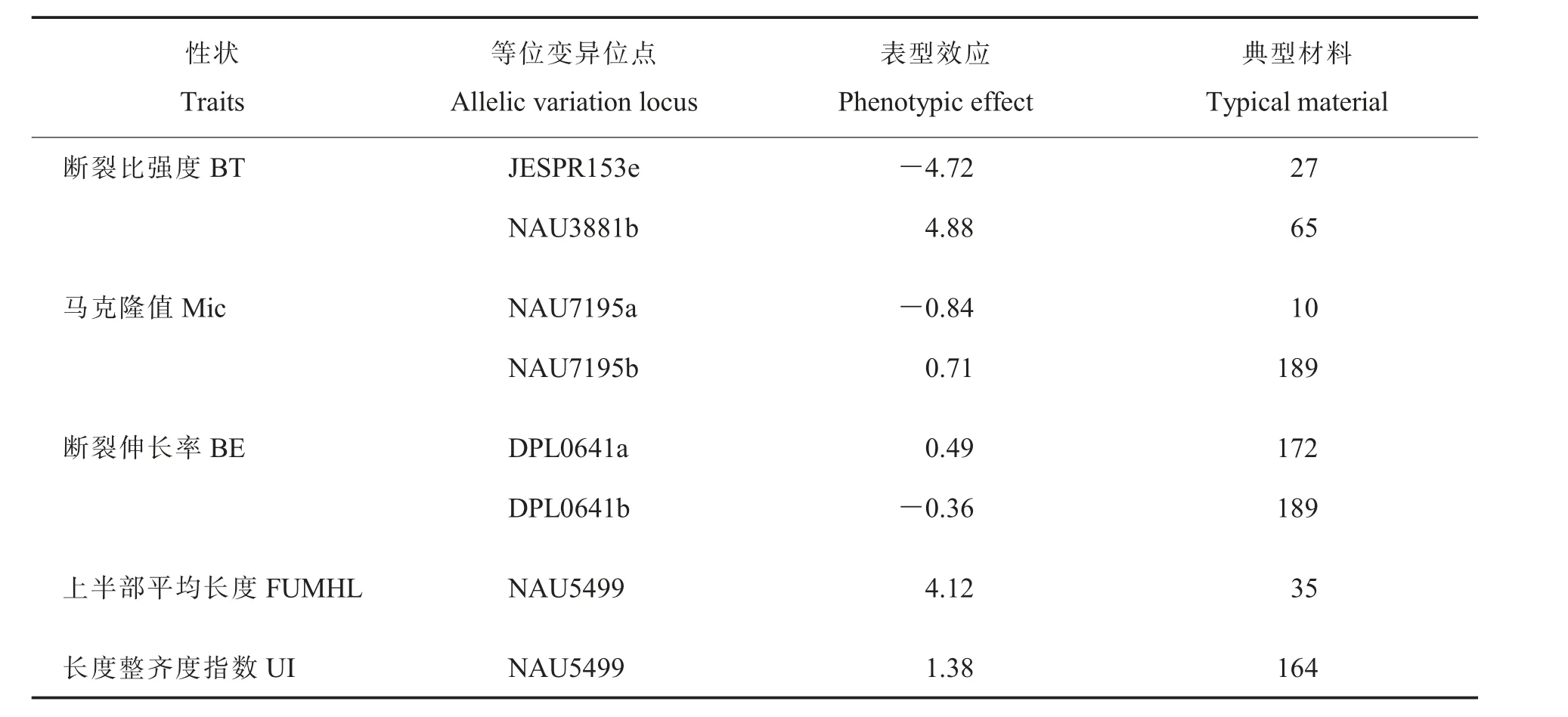
表7 携带特异等位变异的材料Table 7 Materials carrying distinctive allelic variation

陆地棉种质资源是陆地棉品种选育与改良的基础。 育种家只有对现有种质资源有全面的了解,才能更好地利用收集到的种质资源,并进行有目的的亲本组配与品种改良,减少盲目性。 对于不同种质资源群体, 前人已做了许多研究,聂新辉等[18]对新陆早品种的遗传多样性进行了评价, 结果表明51 个新陆早品种遗传相似系数变化范围是0.426 9~0.987 3,平均值为0.707 1,在2016 年对503 份种质资源进行了评价,发现遗传相似系数分布范围为0.337~0.921,平均为0.552[19]。 遗传相似系数的大小与收集材料的多少和收集范围有关,只收集一个地区种质资源难免会出现遗传基础狭窄情况,何陈述等[20]、贾子昉等[21]、艾先涛等[22]、郭志军等[23]的研究结果同样验证了这一结果。 总体来看,随着种质资源来源范围的增大,其遗传多样性呈增大趋势。 本研究使用分布在26 条染色体上的128 对SSR 标记对219 份种质资源进行分子水平的遗传多样性鉴定,结果表明此群体遗传相似性系数分布范围为0.42~0.99,平均为0.61,表现出遗传相似性高,与前人得出的陆地棉种内遗传基础狭窄的结果[20-23]相一致。 此群体作为育种亲本,应当通过增加群体材料数量和扩充群体材料地理位置来源,来增加群体的遗传多样性。

种质资源群体结构划分是遗传多样性的基础[24],错误的群体划分往往导致关联结果的错误,准确划分的群体表现出组内遗传差异小,组间遗传差异大[25]。因此,关联分析结果的准确性取决于群体结构划分的合理性。 前人的研究表明,Q 矩阵[26]、Structure 软件[27]、主成分分析(PCA)[28]都可以用来划分群体结构。 核心种质资源一般由各地引进和本地驯化的种质组成,因此群体内存在大量遗传重组。 为使得群体结构的划分更为准确,本研究同时采用Q 矩阵、Structure 软件和主成分分析划分群体结构,最终将此群体划分为2 个亚群。 也有学者将地理来源作为群体结构划分的依据之一[27]。本研究中由于大多数材料来源于新疆,所以并未使用地理来源这一因素对群体结构进行划分,但群体结构划分的结果与群体内材料地理来源也表现出一致性,即地理来源中大多数材料来源于我国新疆,被划分为一类,少部分材料来源于国内其他省份和国外,还有一部分材料由遗传相似性较为接近, 无法被划分到具体亚群。因此, 认为此群体被划分为2 个类群是合理的,有助于消除关联分析的假阳性结果。

通过对群体基因型和纤维品质性状进行关联分析,分别进行了3 环境中纤维品质数据与基因型的关联分析和3 环境纤维品质数据表型BLUP 结果与基因型的关联,在P=0.01 水平下,棉花纤维品质5 个性状在3 个环境下共检测到29 个显著关联位点。其中在3 个环境下断裂比强度检测到的显著关联位点最多, 共检测到10 个显著关联位点,马克隆值检测到4 个显著关联位点,断裂伸长率检测到9 个显著关联位点,纤维上半部平均长度和纤维长度整齐度指数均检测到3 个显著关联位点。
3 个环境表型数据BLUP 之后与基因型数据关联分析, 共检测到13 个与纤维品质性状显著关联的标记。 根据前人研究[5,12-17],将检测到的纤维品质性状显著关联位点(P≤0.01)与棉花已报道定位的QTL 进行比较, 发现JESPR153、NAU5233、NAU5499 和NAU4073 与已报道的显著关联位点重复出现。 获得已报道定位的标记位点,特别是相同性状标记,说明这些标记具有可重复性和稳定性。 与多个性状显著关联的标记有JESPR153 和NAU5499,其中JESPR153e 与纤维上半部平均长度和纤维断裂比强度显著关联,NAU5499 同时与纤维上半部平均长度、 断裂比强度、断裂伸长率、长度整齐度指数显著关联,这两个标记可用于纤维品质综合性状的改良。 在2个及以上的环境与表型显著关联的位点共有11个。 并根据表型BLUP 结果共挖掘7 个携带特异等位变异的典型材料,将为棉花分子设计育种的应用提供一些参考。

调查了3 环境下收集的219 份早熟陆地棉遗传群体的15 个表型数据, 评价了其表型多样性, 用128 对分布在26 对染色体上的SSR 分子标记评价了其DNA 水平的遗传多样性,同时,用这些分子标记对此群体棉花纤维品质性状在P=0.01 水平下进行了关联分析。
群体基因型遗传多样性的分析结果表明,此自然群体遗传基础狭窄,遗传多样性低。
通过关联分析, 检测到与纤维品质性状在3个环境下的显著关联位点29 个, 在5 个纤维品质性状中, 断裂比强度的显著关联位点最多,其中11 个位点在2 个及2 个以上环境中重复出现。 JESPR153e 位点在3 个环境及BLUP 数据中都检测到与断裂比强度显著关联。 NAU5499 与纤维上半部平均长度、断裂比强度、断裂伸长率、长度整齐度指数均显著关联。 获得携带优异等位变异基因的典型材料共计7 份。
