尼罗罗非鱼干扰素调节因子4基因的克隆与表达
2020-07-24罗国玲马嘉霖莫芳玲陈俊羲罗伟涛
罗国玲,马嘉霖,莫芳玲,陈俊羲,罗伟涛,杨 琴,黄 瑜
尼罗罗非鱼干扰素调节因子4基因的克隆与表达
罗国玲1,2,3,马嘉霖1,莫芳玲1,陈俊羲1,罗伟涛1,杨 琴1,黄 瑜1,2,3
(1. 广东海洋大学水产学院 // 2. 广东省水产经济动物病原生物学及流行病学重点实验室 // 3. 水产经济动物病害控制广东省普通高等学校重点实验室,广东 湛江 524088)
【】探究干扰素调节因子(IRF)在尼罗罗非鱼()免疫中的作用。从尼罗罗非鱼中鉴定IRF4同源物,命名为。使用生物信息学分析软件分析的氨基酸序列。对健康尼罗罗非鱼腹腔注射浓度为3×107CFU/mL的无乳链球菌200 μL,用实时荧光定量PCR法分析在健康鱼中的表达模式及其对细菌感染的生物学效应。【】序列的开放阅读框为1 350 bp,编码449个氨基酸。OnIRF4与其他物种的IRF4蛋白的同源性为52% ~ 98%。推导的肽链具有干扰素因子典型的DNA结合结构域(DBD)、干扰素相联结构域(IAD)。亚细胞定位结果表明,主要定位在细胞核。表达分析表明,在健康尼罗罗非鱼皮肤、头肾、鳃、脑、肠、肌肉、肝、胸腺和脾中均有分布。在无乳链球菌()感染后,在肝脏、脾脏、头肾、脑中的表达显著上调(< 0.05),表明参与了尼罗罗非鱼对细菌感染的免疫反应。
尼罗罗非鱼;干扰素调节因子4;基因表达;无乳链球菌
尼罗罗非鱼() 为我国重要的经济鱼类。近年来,随着养殖规模的不断扩大,高密度养殖以及水体污染原因导致病害频发,经济损失巨大[1]。研究尼罗罗非鱼免疫机制可为其病害防治提供基础。
干扰素(Interferon,IFN)是一种多功能细胞因子,在抗病毒反应,免疫调节,细胞生长、分化、凋亡方面发挥重要的作用[2],其分泌受干扰素调节因子(Interferon regulatory factors,IRFs)调节。IRF家族可分为IRF1、IRF3、IRF4和IRF5等4个亚家族(IRF4、IRF8、IRF9、IRF10基因隶属于IRF4亚家族[3-4])。IRF4(亦称Pip、LISIRF、LCSAT、MUM1)有助于树突状细胞的发育[5],参与先天免疫和适应性免疫的调节[6]。其通过调节与辅助性T细胞17(Helper T cells 17,Th17)分化相关的3个关键转录因子Foxp3、RORa和RORgt的表达模式,在白细胞介素21(IL-21)介导的Th17发育过程中有重要作用。IRF4在哺乳动物中研究较多,最近发现IRF4也存在于虹鳟()、黄鳝()和岩鲷()中[8-10]。虹鳟在受病原刺激后其肝脏和脾脏中表达显著上调,IRF可能在淋巴组织吞噬细胞吞噬、清除病原菌过程发挥重要作用;而受病毒和细菌感染的岩鲷肾脏和脾脏中大量表达,表明不仅对病毒和细菌病原入侵有保护作用,而且可能是硬骨鱼类的免疫调节因子。研究IRFs的调控机制对研究尼罗罗非鱼机体免疫机制及其疾病防治有重要作用[11]。笔者克隆尼罗罗非鱼IRF4基因()的开放阅读框片段,构建亚细胞定位表达载体,以研究其在细胞中的定位,并用qRT-PCR法研究在健康个体组织分布情况,以及无乳链球菌刺激后的表达模式,为进一步探究参与的机体免疫反应提供参考。
1 材料与方法
1.1 材料
实验用尼罗罗非鱼取自于广东海洋大学东海岛海洋生物研究基地,体质量(50±10)g。克隆载体pMD18-T、rTaq聚合酶、Extaq聚合酶,TaKaRa公司(日本);柱式RNA提取试剂盒(TransZol Up Plus RNA Kit)、反转录试剂盒(TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix)、实时荧光定量试剂盒(TransStart Green qPCR SuperMix)以及DH5α感受态细胞,北京全式金生物技术有限公司;Thermo Scientific GeneJET凝胶回收试剂盒、核酸marker,Thermo Fisher公司。引物由生工生物工程(上海)股份有限公司合成。无乳链球菌()及FHM细胞(胖头鲤肌肉细胞系)由广东省水产经济动物病原生物学及流行病学重点实验室保存。
1.2 方法
1.2.1 病原刺激 尼罗罗非鱼于水箱中暂养1周,随机选取5尾,对其肝脏、脾脏及头肾等组织进行病原微生物检测,确定尼罗罗非鱼处于健康状态。于无菌环境下取健康鱼皮肤、头肾、鳃、脑、肠、肌肉、肝、胸腺和脾于2 mL冻存管,置液氮速冻后于–80 ℃冰箱保存,用于基因克隆和后续使用。
无乳链球菌于37℃培养6 h后,取菌液离心,用磷酸盐缓冲液(PBS)稀释至终浓度为1×107CFU/mL。参考文献[12],对尼罗罗非鱼腹腔注射100 μL的无乳链球菌,在注射后0、6、12、24、48、72、96 h各取尼罗罗非鱼3尾,收集其脾脏、头肾、鳃和脑,液氮冷冻后于- 80 ℃冰箱保存备用。
1.2.2 RNA提取及第一cDNA链合成 样品用TransZol up裂解(每mL 50 mg),按照RNA提取试剂盒说明书步骤提取RNA。按照反转录试剂盒说明书将各样本的组织RNA反转录成第一链 cDNA,反转录体系:RNA 500 ng, Anchored Oligo(dT)18 引物(0.5 μg /μL) 1 μL,2×TS Reaction Mix 10 μL, TransScript® RT/RI Enzyme Mix 1 μL,gDNA Remover 1 μL,补无RNA酶水至20 μL。将混合物在45 ℃下反应30 min后,于85 ℃下孵育5 min至酶失活。反转录产物于– 20 ℃下保存备用。
1.2.3开放阅读框克隆及测序 根据NCBI上公布的基因序列(GeneBank登录号:XP_005476429.1),利用Primer 5.0 设计一对开放阅读框(Open Reading Frame, ORF)扩增引物(IRF4-ORF-F/R)(表1)。以脾脏的第一链cDNA为模板,通过PCR扩增IRF4的ORF片段,反应体系:Ex Taq 10 μL,ddH2O 7 μL,cDNA 模板1 μL,IRF4-ORF-F/R 1 μL。反应条件:94 ℃ 5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 90 s,35个循环;72 ℃ 10 min,16 ℃下保存扩增产物。PCR产物经琼脂糖凝胶电泳检测达标后,进行切胶回收及PCR产物纯化。将纯化后的PCR产物与pMD18-T载体相连接,转入DH5α感受态细胞,涂布于含氨苄的LB琼脂培养基,倒置于37 ℃下培养12 h。挑取单菌落,用pMD18-T载体的通用引物M13-R/F进行菌落PCR检测,阳性菌落送至生工生物工程(上海)股份有限公司测序。

表1 引物
1.2.4基因生物信息学分析 将NCBI预测的全长序列及测序结果用DNAMAN进行片段连接和序列比对,运用ORF-finder在线程序寻找潜在的ORF区,用ExPASy ProtParam tool和DNAMAN推导的相关氨基酸序列并分析相关理化性质。根据推导的氨基酸序列,通过SMART(http://smart.embl-hei delberg.de/)搜索结构域。使用swiss模型预测算法(http://SWISSMODEL. expasy.org/)建立潜在的三维结构,并由Deepview/ SWISS Pdb Viewer version 4.0显示。用PSORT Prediction预测的亚细胞定位。用ClustalW多重比对程序(http://www.ebi.ac.uk/ ClustalW/)创建多重序列比对。采用MEGA 6.0的邻接法进行系统进化树分析,Bootstrap值设置为1 000。
1.2.5亚细胞定位 设计一对亚细胞定位表达引物IRF4-XHol-F、IRF4-EcoRI-R,以脾脏第一链cDNA为模板克隆目的片段,反应程序同1.2.3。用Ⅰ和Ⅰ限制性快切酶对PCR产物及亚细胞定位表达质粒PGEFP-N1进行双酶切,酶切产物纯化后,将目的基因片段与PGEFP-N1空质粒连接,连接产物转入DH5α感受态细胞。转化产物均匀涂布于LB琼脂糖固体平板上,挑取单菌落,经菌落PCR鉴定后挑选阳性样品送至生工生物工程(上海)股份有限公司测序。
将无菌盖玻片放入24孔细胞板,每孔接种500 μL FHM细胞。FHM细胞在含体积分数15%胎牛血清的L-15细胞培养基、28 ℃条件下培养至80%细胞贴壁后,对测序正确的菌株进行去内毒素质粒提取及浓度测定,按照Lip3000试剂使用说明书,将空的PGEFP-N1质粒或成功构建的IRF4-PGEFP-N1转染至FHM细胞内。24 h后吸出玻片边缘的培养基,用PBS洗涤细胞,加入150 μL体积分数4%多聚甲醛固定液,静置30 min。吸去多聚甲醛固定液,用PBS再次洗涤,加入100 μL 4,6-二脒基-2-苯基吲哚(DAPI) 进行细胞核染色。染色10 min后,用水溶性封片剂封片,置于荧光倒置显微镜下观察。
1.2.6基因的表达分析 设计一对荧光定量引物qIRF4-F/R,用β-actin作为内参基因。对健康尼罗罗非鱼的组织样品及病原刺激后的组织样品进行RNA提取及cDNA合成,方法同1.2.2。利用LightCycler®96实时荧光定量PCR仪进行基因的表达分析。实验参照TransStart® Top Green qPCR SuperMix试剂盒说明书进行,反应体系如下:qPCR SuperMix 5 μL,cDNA 0.5 μL,dH2O 3.5 μL,上下游引物0.5 μL。反应程序:94 ℃ 30 s;94 ℃ 5 s,60 ℃ 15 s,72 ℃ 10 s,40个循环;95 ℃ 15 s [溶解曲线分析(Dissociation Stage)]。每个样本设置3个复孔,利用2-ΔΔCt方法计算相对表达量。使用SPSS统计学软件进行显著性检验。
2 结果
2.1 OnIRF4开放阅读框扩增及序列的表征
克隆的基因片段长度约1 350 bp,与pMD-18T连接后,测序,经比对发现,其与的ORF区100%重合。ORF片段长度为1 350 bp,编码449个氨基酸(图1)。cDNA有141 bp的5′UTR和1 952 bp的3′UTR,3′UTR中有1个poly(A)加尾信号(AATAAA)。理论分子质量为51.783 ku,等电点为6.21。该蛋白平均亲水值(GRAVY)为-0.817,属亲水性蛋白。SMART分析可知,IRF4氨基酸序列含有1个N端DNA结合结构域(N-terminal DNA binding region,DAB)和1个C端干扰素相关结构域(C-terminal interferon related region,IAD)(图2)。经预测可知,IRF4主要分布于细胞核;有N-糖基化位点4个、蛋白激酶C磷酸化位点10个、酪蛋白激酶II磷酸化位点12个、N-肉豆蔻酰化位点6个、异戊二烯基结合位点(CAAX框)3个、微抗体C端靶向信号10个。

方框部分是起始密码子和终止密码子;阴影部分为DNA结合结构域;单下划线为IRF相关结构域;双下划线为poly(A) 加尾信号
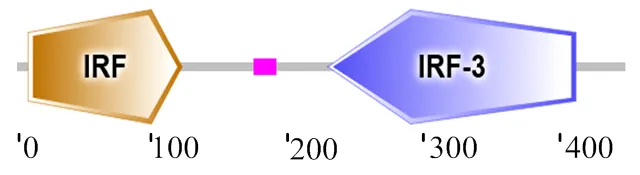
图2 尼罗罗非鱼OnIRF4基因结构域
2.2 OnIRF4蛋白空间结构预测
图3可见,OnIRF4蛋白449个氨基酸残基中,包含23.16%的α螺旋(蓝色h)、12.25%的延伸链(红色e)、4.68%的β转角(绿色t)及59.91%的无规则卷曲(黄色c),其中,无规则卷曲是最多的结构元件。通过构建的蛋白三维立体结构模型(图4)发现,该蛋白与人类的同源物有一定同源性。

图3 OnIRF4蛋白二级结构预测

图4 尼罗罗非鱼IRF4蛋白与人类的三维结构
2.3 OnIRF4多重序列比对分析
图5表明,OnIRF4氨基酸序列与其他物种IRF4的相似性为98.00% ~ 52.12%。与鲈形目慈鲷科的淡水鱼类美妊丽鱼()同源性最高,为98%,与鳃鱼科黄鳝属动物同源性较低,为64.46%。此外,所比对序列均有IRF的DAB和IAD保守结构域结构,且保守结构域的同源性高。

虚下划线,DNA结合结构域;实线下划线,IRF相关结构域;GenBank登录号:尼罗罗非鱼,XP_005476429.1;美妊丽鱼,XP_026005407.1; 斑马拟丽鱼,XP_004542815.1; 原鸡,NP_989630.1; 牛,NP_001193091.1; 人,AAH15752.1; 小鼠,AAI37714.1; 黄鳝,AFQ22942.1
2.4 系统进化分析
图6可见,与斑马拟丽鱼()基因聚为一支,亲缘关系较近,与库舍剑尾鱼()聚为一类。哺乳动物聚为一簇,鱼类聚为一簇。
2.5 OnIRF4的亚细胞定位
图7可见,在尼罗罗非鱼PGEFP-N1空质粒转染的细胞中,绿色荧光均匀分布于整个细胞。而在OnIRF4-PGEFP-N1融合蛋白转染的细胞中,绿色荧光蛋白主要分布于细胞核,与PSORT Prediction预测的结果相符;在细胞质中也有少量分布,推测细胞质中的绿色荧光蛋白在核糖体中合成,未能及时转移至细胞核内。

图6 尼罗罗非鱼与其他物种IRF4的系统进化树分析

图7 OnIRF4在FHM细胞中的亚细胞定位
2.6 IRF4在尼罗尼罗罗非鱼不同组织中的表达
图8显示,在脑、肝脏、肠道、头肾、肌肉、脾、皮肤、鳃和胸腺等9个组织中均有表达,但相对表达量差异较大。在脑中表达量最高,肝脏、肠道和头肾中的表达水平较高,在肌肉、脾、皮肤、鳃相对表达量较低,在胸腺中表达量最低。
2.7 OnIRF4在感染无乳链球菌后的表达模式
图9显示,在感染无乳链球菌后,尼罗罗非鱼的mRNA表达水平在3个免疫相关组织中均显著上调(< 0.05)。头肾于感染后6、12 h相对表达量明显上调(图9A),鳃于感染后6、24 h相对表达量显著上调 (图9B),脾脏于感染后6、48、72、96 h相对表达量明显上调 (图9C)。脑于感染后12、48 h表达量明显上调(图9D)

TH,胸腺;GI,鳃;SK,皮肤;SP,脾;M,肌肉;HK,头肾;IN,肠;LI,肝;BR,脑
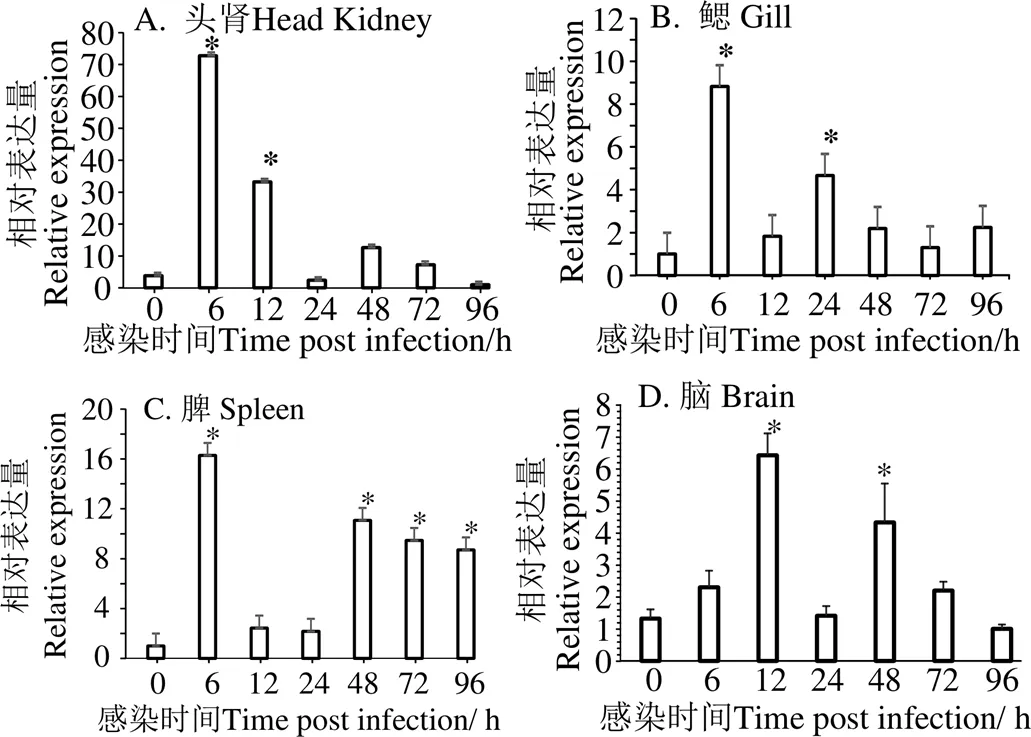
*,与0 h对照组有显著性差异(P < 0.05)
3 讨论
本研究从尼罗罗非鱼脾脏中克隆得基因ORF片段,根据推导的氨基酸序列,ORF片段包含1 350 bp,可编码449个氨基酸;理论分子质量为51.783 ku,理论等电点为6.21;含有IRF4家族的典型结构域,即1个N端的DAB和1个C端的IAD,鱼类IRF4的DAB、IAD序列与哺乳动物的同源性较高,表明IRF4的作用方式在进化上较为保守。此外预测在DBD内含有色氨酸五重肽重复区,这是针对病毒诱导的磷酸化,以促进与其他IRF家族成员的相互作用和随后激活病毒清除信号通路的结构域,表明IRF4可能在尼罗罗非鱼中作为免疫应答的干扰素发挥作用。系统进化树反映的进化关系与传统分类地位基本一致。
OnIRF4的亚细胞定位结果与斑马鱼IRF4a[13]相似,主要在细胞核内表达。尼罗罗非鱼IRF4在没有刺激的情况下主要分布在细胞核中,表明IRF4主要在细胞核中发挥调控细胞凋亡与生长及免疫应答等作用。
可在巨噬细胞中表达,且在B细胞、T细胞、树突状细胞和巨噬细胞的发育和分化中发挥重要的作用[14-17],脑中含有大量的巨噬细胞[18],本研究中,在脑中表达量最高,暗示着参与了鱼脑部巨噬细胞的发育和分化。在其他硬骨鱼类中,也在富含淋巴细胞的器官或黏膜免疫组织(肝脏、肠道、头肾和脾脏)中大量表达,如在黄鳝[8]的肠、头肾中表达量较高,在褐牙鲆()[19]的脾脏、头肾、鳃中表达量较高,在虹鳟[10]的脾脏、肾脏和鳃组织中表达量较高,表明在先天性和适应性免疫中起重要作用。然而,在健康尼罗罗非鱼鳃中IRF4的表达量相对于同为微生物感染入口的肠道低,推测OnIRF4主要参与抵御肠道微生物入侵。此外,在肌肉中也有相对较高的表达水平,表明在非免疫组织中亦有潜在的作用。
在免疫应答中发挥一定作用[9]。鳃是鱼类黏膜免疫系统的第一道屏障[20],极易受病原感染;头肾和脾脏是硬骨鱼类的主要免疫器官[21],而鱼的脑中含有大量的吞噬细胞[18]。本研究中,在无乳链球菌感染后基因表达在头肾、脾脏、鳃和脑均在6 h内表达显著上调,提示尼罗罗非鱼的参与黏膜免疫,并在免疫相关组织感染后可快速响应[22],提示在抗菌反应中起重要作用。在头肾中观察到最大的转录反应(72.9倍,< 0.01),头肾为硬骨鱼类重要的内分泌和造血器官[23],在头肾中最大响应进一步表明其在免疫应答中的重要作用。此外,在无乳链球菌注射后的48、72、96 h后,脾脏的表达量再次显著上调,可能是由于组织中的巨噬细胞聚集,可将抗原保留更长时间[24]。研究表明,尼罗罗非鱼因感染无乳链球菌引起的脑膜炎时间变化为:在感染的2 d内,未见明显的神经症状;而在感染3 d至以后几天鱼体出现异常活动的神经症状及眼突、角膜浑浊等病理变化[25];而这与感染了无乳链球菌后脑中的表达模式相符合,在脑中的表达量主要在48 h内显著上调,即在感染的12 h表达量到达峰值(6.4倍,< 0.01),在48 h出现表达量的次高峰,而在72 h和96 h表达量趋于平稳。综上可见,参与了尼罗罗非鱼细菌感染的免疫应答。
4 结论
从尼罗罗非鱼中克隆并鉴定了干扰素调节因子4。OnIRF4有干扰素调节因子的典型结构域:DBD和IAD。亚细胞定位显示主要在细胞核中发挥功能。健康尼罗罗非鱼的脑、肝脏、肠道和头肾中的mRNA含量丰富。在细菌感染后的体内表达呈时间依赖性上调。表明参与了细菌感染的免疫应答。
[1] 梁筱燕. 尼罗罗非鱼血清免疫球蛋白单克隆抗体的制备及鉴定[D]. 重庆:西南大学, 2013.
[2] PFEFFER L M, DINARELLO C A, HERBERMAN R B. et al. Biological properties of recombinant alpha-interferons: 40thanniversary of the discovery of interferons[J]. Cancer Research, 1998, 58(12): 2489-2499.
[3] HUANG B, QI Z T, XU Z, et al. Global characterization of interferon regulatory factor (IRF) genes in vertebrates: Glimpse of the diversification in evolution[J]. BMC Immunology, 2010, 11(1): 1-28.
[4] 付建平, 陈丹婷, 赵昕, 等. 中华鳖IRF4基因克隆及免疫功能研究[J]. 水产科学, 2019, 38(3): 368-379.
[5] TAMURA T, TAILOR P, YAMAOKA K, et al. IFN regulatory factor-4 and -8 govern dendritic cell subset development and their functional diversity[J]. Journal of Immunology, 2005, 174(5): 2573-2581.
[6] TAILOR P, TAMURA T, OZATO K. IRF family proteins and type I interferon induction in dendritic cells[J]. Cell Research, 2006, 16(2): 134-140.
[7] BISWAS P S, GUPTA S, CHANG E, et al. Phosphorylation of IRF4 by ROCK2 regulates IL-17 and IL-21 production and the development of autoimmunity in mice[J]. The Journal of Clinical Investigation, 2010, 120(9): 3280-3295.
[8] XU Q Q, YANG D Q, TUO R, et al. Gene cloning and induced expression pattern of IRF4 and IRF10 in the Asian swamp eel ()[J]. Zoological Research, 2014, 35(5):380-388.
[9] BATHIGE S D N K, WHANG I, UMASUTHAN N, et al. Interferon regulatory factors 4 and 8 in rock bream,: Structural and expressional evidence for their antimicrobial role in teleosts[J]. Fish & Shellfish Immunology, 2012, 33(4): 857-871.
[10] HOLLAND J W, KARIM A, WANG T H, et al. Molecular cloning and characterization of interferon regulatory factors 4 and 8 (IRF-4 and IRF-8) in rainbow trout,[J]. Fish & Shellfish Immunology, 2010, 29(1): 166.
[11] 黄瑜, 马嘉霖, 周棉鑫, 等. 罗非鱼干扰素诱导蛋白35基因克隆及表达分析[J]. 广东海洋大学学报, 2020, 40(2): 13-19.
[12] NIU J Z, HUANGY, NIU J M, et al. Characterization of Galectin-2 from Nile tilapia () involved in the immune response to bacterial infection[J]. Fish & Shellfish Immunology, 2019, 87: 737-743.
[13] LI S, GUO X, LU L F, et al. Regulation pattern of fish irf4 (the gene encoding IFN regulatory factor 4) by STAT6, c-Rel and IRF4[J]. Developmental & Comparative Immunology, 2015, 51(1): 65-73.
[14] HUBER M, BRÜSTLE A, REINHARD K, et al. IRF4 is essential for IL-21-mediated induction, amplification, and stabilization of the Th17 phenotype[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(52): 20846-20851.
[15] HONDA K, TANIGUCHI T. IRFs: master regulators of ignaling by Toll-like receptors and cytosolic pattern-recognition receptors[J]. Nature Reviews Immunology, 2006, 6(9): 644-658.
[16] BRÜSTLE A, HEINK S, HUBER M, et al. The development of inflammatory TH-17 cells requires interferon-regulatory factor 4[J]. Nature Immunology, 2007, 8(9): 958-966.
[17] PAUN A, PITHA P M. The IRF family, revisited[J]. Biochimie, 2007, 89(6/7): 744-753.
[18] 曾山. 小胶质细胞/脑巨噬细胞与胶质瘤相互关系的研究进展[J]. 国际神经病学神经外科学杂志, 2016, 43(3): 255-258.
[19] LIU D H, CHEN J J, ZHANG H Y, et al. Interferon regulatory factor 4b (IRF4b) in Japanese flounder,: Sequencing, ubiquitous tissue distribution and inducible expression by poly(I:C) and DNA virus[J]Developmental & Comparative Immunology, 2016, 62(4): 127-133.
[20] MALLATT J. Fish gill structural changes induced by toxicants and other irritants: A statistical review[J]. Canadian Journal of Fisheries & Aquatic Sciences, 1985, 42(4): 630-648.
[21] STENVIK J, SCHRØDER M B, OLSEN K, et al. Expression of immunoglobulin heavy chain transcripts (VH-families, IgM, and IgD) in head kidney and spleen of the Atlantic cod (L.)[J]Developmental & Comparative Immunology, 2001, 25(4): 291-302.
[22] MU L L, YIN X X, XIAO Y H, et al. A C-type lectin (CL11X1-like) from Nile tilapia () is involved in host defense against bacterial infection[J]. Developmental & Comparative Immunology, 2018, 84(2): 230-240.
[23] GEVEN E J W, KLAREN P H M. The teleost head kidney: Integrating thyroid and immune ignaling [J]. Developmental& Comparative Immunology, 2017, 66: 73-83.
[24] WANG Y, KE F, MA J J, et al. A tandem-repeat galectin-9 involved in immune response of yellow catfish,, against[J]. Fish & Shellfish Immunology, 2016, 51(2): 153-160.
[25] 崔静雯. 尼罗罗非鱼无乳链球菌脑膜炎病理损伤的动态研究及无乳链球菌透明质酸酶多克隆抗体制备[D]. 雅安:四川农业大学,2016.
Cloning and Expression Analysis of Interferon Regulatory Factor 4 Gene () in
LUO Guo-lin1,2,3, MA Jia-ling1, MO Fang-ling1, CHEN Jun-xi1, LUO Wei-tao1, YANG Qing1, HUANG Yu1,2,3
(1.//2.// 3.524088,)
【】Interferon regulatory factor 4 (IRF4) plays a key role in innate and adaptive immune responses in mammals. 【】the IRF4 homologous from, namedwas identified. The amino acid sequence ofwas analyzed by bioinformatics analysis software. The expression pattern ofwas analyzed by Quantitative Real-time PCR in healthy fish and its biological effect was analyzed on bacterial infection by intraperitoneal injection of 200 μL(3×107CFU/mL) on healthy fish. 【】The open reading frame ofsequence is 1 350 bp, encoding 449 amino acids. It has a 52% - 98% homology with other IRF4 proteins. The derivedmature peptide has typical DNA binding domain (DBD) and interferon-associated domain (IAD) of interferon factors. The results of subcellular localization showed thatwas mainly located in the nucleus. Expression analysis showed thatwas distributed in all tissues of healthy tilapia. Afterinfection, the expression ofin the liver, spleen, head kidney and brain was significantly up-regulated (< 0.05), suggesting thatwas involved in the immune response of Nile tilapia to bacterial infection.
; interferon regulatory factor 4; gene expression;
Q78;Q959.483
A
1673-9159(2020)05-0019-08
10.3969/j.issn.1673-9159.2020.05.003
2020-03-24
大学生创新创业训练计划(CXXL2019141)
罗国玲(1996-),女,硕士,研究方向为鱼类免疫学。E-mail:1433792702@qq.com。
黄瑜(1986—),男,博士,研究方向为鱼类免疫学。E-mail:huangyu@gdou.edu.cn。
罗国玲,马嘉霖,莫芳玲,等. 尼罗罗非鱼干扰素调节因子4基因的克隆与表达[J]. 广东海洋大学学报,2020,40(5):19-26.
(责任编辑:刘庆颖)
