基于BERT的医疗电子病历命名实体识别
2020-07-23梁文桐朱艳辉冀相冰
梁文桐,朱艳辉,詹 飞,冀相冰
(1.湖南工业大学 计算机学院,湖南 株洲 412007;2.湖南工业大学 智能信息感知及处理技术湖南省重点实验室,湖南 株洲 412007)
1 研究背景
在自然语言处理(natural language processing,NLP)的任务中,命名实体识别(named entity recognition,NER)是具有挑战的基础性工作[1]。从狭义上来说,一般的命名实体识别任务的目的,是从文本中识别出3种类型的实体提及,包括人名、地名和机构名。在医学领域中,医务工作人员通过医疗机构信息系统将病人的临床诊断信息存储在计算机中,得到电子病历(electronic medical records,EMR)。电子病历命名实体识别是命名实体识别在电子病历文本分析研究中的重要应用和扩展,其目的是自动地识别并且分类电子病历中的医疗命名实体。这些命名实体对象能够被用于后续医疗电子病历信息的分析和研究中,比如构建临床信息决策系统、构建医疗领域的知识图谱等。
早期的电子病历命名实体识别方面的研究主要运用基于词典和规则的方法,仅仅依赖于现有的词典和手工编辑的规则来识别医疗命名实体[2]。后来,基于统计机器学习的方法被运用到电子病历命名实体识别中。如于楠等[3]采用基于多特征融合的CRF (conditional random fields)模型进行了中文电子病历NER的研究。A.Kulkarni[4]从生物医学文本中完成DNA、RNA和蛋白质等生物医学术语的识别,该任务使用CRF 统计模型完成。许源等[5]基于CRF 以及RUTA(rule-based text annotation)规则,建立了一个医学命名实体识别模型,该模型在识别脑卒中患者入院记录的医学命名实体时取得了良好的效果。王润奇等[6]利用半监督学习方法,将Tri-Training 算法进行了改进,使得中文电子病历实体识别模型的效果得到了提升。
近年来,随着硬件计算能力的大幅度提高,基于深度神经网络的方法已经被成功地应用到电子病历命名实体识别中,该方法是一种端到端的方法,不需要特殊的领域资源(如词典)或者构建本体,可以从大规模的标注数据中自动地学习和抽取文本特征。在电子病历NER 任务中,杨红梅等[7]利用一种基于BiLSTM(bidirectional long short-term memory)与CRF的实体识别模型,抽取了入院记录和出院小结中的医学命名实体。万里等[8]提出了一种基于字词联合训练的BiLSTM模型,能够有效识别中文电子病历中疾病、症状等相关实体。Wang Q.等[9]将词典特征加入深度神经网络中,提出了5种不同特征的表示方式和基于BiLSTM 两种不同的神经网络结构。S.Chowdhury 等[10]提出了一种新型的、多任务的双向循环神经网络(recurrent neural network,RNN)模型,该模型可以从中文的电子病历中抽取出医疗实体。杨文明等[11]使用BiLSTM-CRF和IndRNN-CRF 等模型,抽取了在线医疗问答文本中疾病、治疗、检查和症状4类医疗实体。与此同时,也有很多学者利用卷积神经网络(convolutional neural network,CNN)的方法,将其应用到医疗电子病历NER 任务中。如Gao M.等[12]利用一种结合词序和局部上下文特征的基于注意力的IDCNN(iterated dilated convolution neural networks)-CRF模型,完成了对临床电子病历中医学实体术语的抽取。
但是,以上基于深度神经网络的NER 方法,都存在无法准确表示字符或者词语多义性的问题。例如,“张三和李四的身高差得很远”和“小明的学习成绩很差”,两个句子中的“差”字在各自的语境中是两个完全不同的含义,但是在上下文无关的词嵌入表示方法(如Word2Vec)中,两个“差”字映射成完全相同的向量,因此这种向量无法考虑到句子的上下文语义。近年来,学术界提出了许多与上下文有关的词嵌入表示方法,比如EMLo(embeddings from language models)方法和OpenAI-GPT(generative pre-training)方法[13]。但是,上述两种语言模型的语言表示都是单向的,无法同时获取前后两个方向电子病历文本的语义信息。
当前,医疗电子病历的命名实体识别面临着训练语料不足和标注质量不高的问题,由于医疗领域的专业性,导致其缺少高质量的标注语料[14]。此外,医疗电子病历中的命名实体有着特殊和严谨的语言结构,使得该领域命名实体识别具有一定的挑战性。
为了解决上述问题,本研究拟将可以表示双向丰富语义的BERT(bidirectional encoder representations from transformers)预训练语言模型引入电子病历NER 任务中,提出了BERT-IDCNN-MHA(multi-head attention)-CRF命名实体识别模型。并利用该模型对医疗电子病历中预定义的疾病和诊断、影像检查、实验室检验、手术、药物以及解剖部位6类实体进行命名实体识别,并且将该6类实体正确归类到预定义类别中。
2 BERT-IDCNN-MHA-CRF命名实体识别模型
BERT-IDCNN-MHA-CRF命名实体识别模型的整体结构如图1所示。
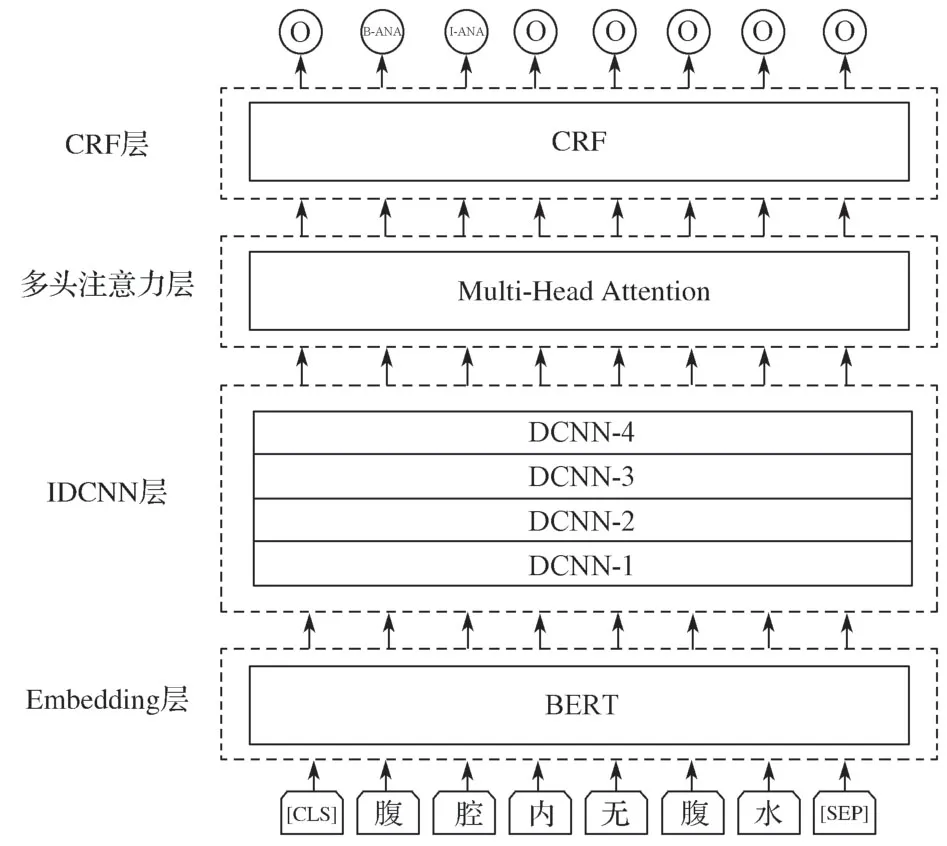
图1 BERT-IDCNN-MHA-CRF命名实体识别模型结构图Fig.1 BERT-IDCNN-MHA-CRF NER model structure diagram
整个识别模型由4个部分组成:首先,输入电子病历中的每一个字,经过Embedding层即BERT模型,得到与每个字的上下文相关的向量表示。其次,经过IDCNN层,将上层输入的每个字的向量进行膨胀卷积编码来提取局部特征,再将获取到的特征向量输入到多头注意力层,多次计算每个字和所有字的注意力概率来获取电子病历句子的长距离特征,得到新的特征向量。因为多头注意力层无法考虑标签之间的依赖关系,比如“I-ANA”标签不能紧接在“B-DIS”标签的后面,所以最后经过CRF层约束预测标签之间的依赖关系,对标签序列进行建模,从而获取全局最优序列。为了提高该模型的泛化能力,在Embedding层与IDCNN层之间加入了dropout层。
本研究通过上述命名实体识别模型识别电子病历中的医疗命名实体,具体步骤如下:
1)预处理原始电子病历文本数据集。将电子病历文本集合D={d1,d2,…,dN}及其对应的预定义类别C={c1,c2,…,cM}按照字符级别进行分割并进行标注,标注时字符和预定义类别用空格隔开。
2)构建电子病历文本训练数据集。按照比例,将分割并标注好的电子病历训练数据分为训练集、验证集和测试集。
3)训练生成命名实体识别模型。基于深度学习技术,训练BERT-IDCNN-MHA-CRF命名实体识别模型。4)识别电子病历文本测试数据集,计算识别率。以电子病历测试文本集合Dtest={d1,d2,…,dN}为输入,文本中医疗实体提及和所属预定义类别的集合其中,mi是出现在文档di中的实体提及,表示所属的预定义类别)为输出,再根据精确率、召回率和F1值来计算其识别率。
2.1 BERT预训练语言模型
BERT模型是一个深度双向编码的包含字符级、词语级和句子级特征的预训练语言模型[15]。针对医疗电子病历的NER 任务,只需要调用该预训练模型的相应接口,就能够得到电子病历中每个字的嵌入表示,且能更准确地表示电子病历中与上下文相关的语义信息。本文构建BERT预训练语言模型的网络结构如图2所示。
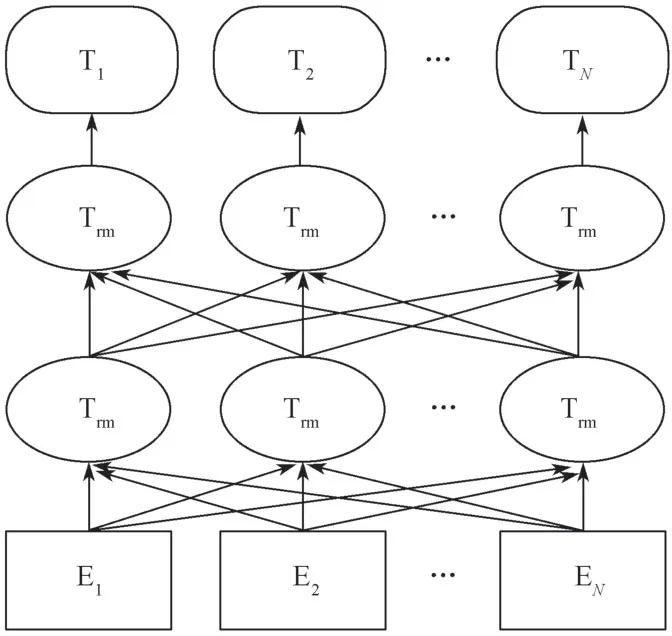
图2 BERT预训练语言模型的网络结构图Fig.2 Network structure diagram of BERT pre-training language model
BERT模型使用“Masked语言模型”来预训练该语言模型,以获取字词级别的上下文相关语义表示。“Masked语言模型”的核心思想来自于完形填空。传统的语言模型以句子中某个给定词语的下一个词语来预测该词语,而“Marked语言模型”则是把句子中随机选择的15%的词语盖住,通过上下文的内容预测被盖住的词语,但是这一方法会导致微调时模型无法准确地预测某些100%被盖住的词语。为解决这一问题,本研究在BERT预训练实验中采取了如下策略:
1)80%的时间,用“[MASK]”标记来替换被盖住的词语。
2)10%的时间,用一个任意的词语来替换被盖住的词语。
3)剩余10%的时间,保持被盖住的词语不变。
同时,BERT模型的预训练利用“下一个句子预测”任务来获取句子级别的上下文相关语义表示。该任务的目标,是判断句子N是否是句子M的下一句。传统的语言模型不能直接反映两个句子之间的关系,在NLP 领域的许多任务中,都需要在理解两个句子之间关系的基础上进行,如问答和自然语言推理等,因此无法直接使用传统的语言模型。两个句子之间的关系通过BERT预训练一个模型学习得到,训练的输入是句子M和N两个句子,然后利用模型来预测句子N是否是M的下一句。
BERT预训练语言模型的输入是电子病历文本中的每一个字,输出是该字的总特征向量,总特征向量由字(词)向量、句子切分向量和位置向量3种不同的特征向量相加得到,位置向量的计算公式如式 (1)和(2)所示。其中,编码使用的是正弦函数和余弦函数,pos代表的是电子病历文本中第几个字,i代表第几维,编码后的向量维度是dmodel。
BERT模型输入示例如图3所示,第一个标记的标签是一种特殊嵌入[CLS],代表电子病历文本的开始位置;其后的特殊嵌入[SEP],代表电子病历文本的结束位置。
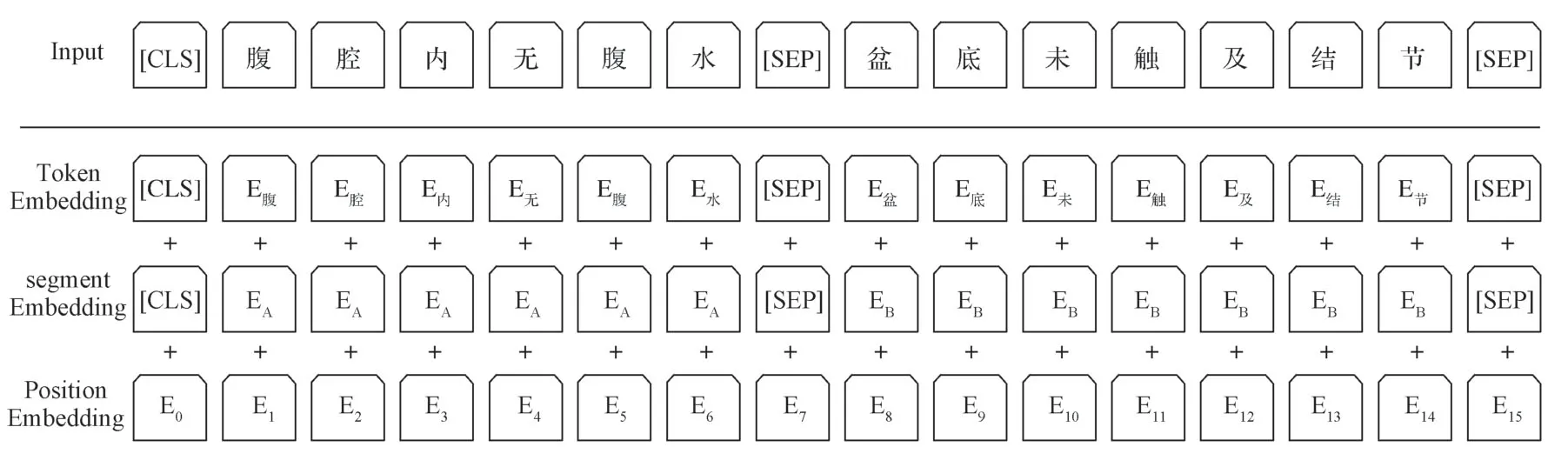
图3 BERT模型输入示例Fig.3 Samples of BERT model input
2.2 IDCNN层
相关研究结果表明,相对于BiLSTM的长距离依赖关系编码,IDCNN 对局部实体的卷积编码可以达到更好的医疗实体识别效果,同时其训练速度和预测的效率都有所提高[12]。因此,本研究采用IDCNN模型对电子病历文本的特征进行提取。一般的CNN滤波器,都是通过在输入矩阵的区域上不断地滑动来做卷积运算,且这种区域通常是连续的。而DCNN(deep convolutional neural networks)则是因在滤波器上添加了膨胀宽度,导致此时输入矩阵的区域不再连续,每次做卷积运算时都会跳过所有膨胀宽度中间的输入数据。在膨胀卷积运算过程中,输入矩阵上更多的数据被滤波器获取,但是滤波器本身的大小并没有发生变化,反而扩大了其感受域,看上去像是“膨胀”了一般,因此称作膨胀卷积神经网络。与一般的CNN 相比,DCNN 没有通过池化操作也可以获得较大的感受域,而且减少了信息损失。DCNN的膨胀示意图如图4所示。
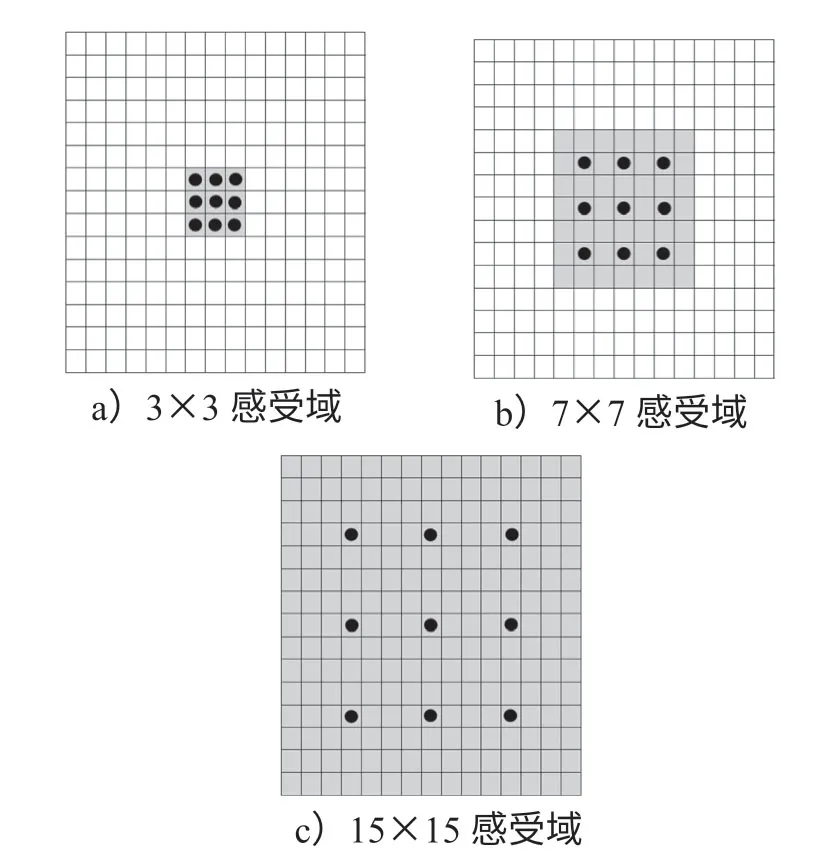
图4 DCNN的膨胀示意图Fig.4 Dilated schematic diagram of DCNN
图4中,图中心点的1×1 区域是开始的感受域,卷积核的大小为3,从感受域的中心点出发,以步长为1 向外部扩散,得到图a中大小为3×3的新感受域;再从新感受域的中心点出发,以步长为2 向外部扩散,得到图b中大小为7×7的新感受域;接下来从这一新感受域的中心点出发,以步长为4 向外扩散,得到图c中大小为15×15的新感受域。膨胀卷积的感受域计算公式见式(3),式中i代表步长。
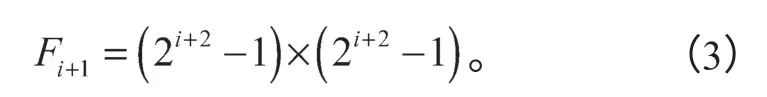
在逐步扩大感受域、层数不断增加的过程中,神经网络参数呈线性增加,而感受域呈指数级增加。如图4所示,仅经过3 步膨胀变化后,感受域就已扩散至输入矩阵中的全部数据。这种膨胀卷积神经网络结构,每层的参数都是相互独立且数量相同的,可有效减少训练时的参数,从而可加快训练速度。
IDCNN模型则是将4个结构相同的膨胀卷积块进行堆叠,相当于进行了4次迭代,每次迭代将前一次的结果作为输入,这种参数共享可有效防止模型过拟合,每个膨胀卷积块有膨胀宽度分别为1,1,2的3层膨胀卷积。通过IDCNN模型,将电子病历中的每个字进行膨胀卷积编码,自动提取文本中特征,输出为对应的特征向量。虽然IDCNN可使感受域变大,但提取的特征仅是局部的,因此还需经多头注意力层进行电子病历文本的长距离特征提取。
2.3 多头注意力层
注意力机制(attention mechanism)首先被应用在数字图像处理领域,后来逐渐被应用于NLP 领域的多种任务中。可以将注意力函数看作一个查询(Q)到一系列键(K)-值(V)对的映射。在NLP 领域的多种任务中,K和V通常取相等值。在计算自注意力时,通常取Q=K=V,可以计算输入句子中每个字符和所有字符的注意力概率。本研究利用注意力机制中的多头注意力,从电子病历文本的内部结构中得到字符之间的长距离依赖关系。多头注意力模型的结构如图5所示,其中,拼接k次自注意力计算结果,将拼接结果进行线性变换后,即可以得到本次注意力计算结果。
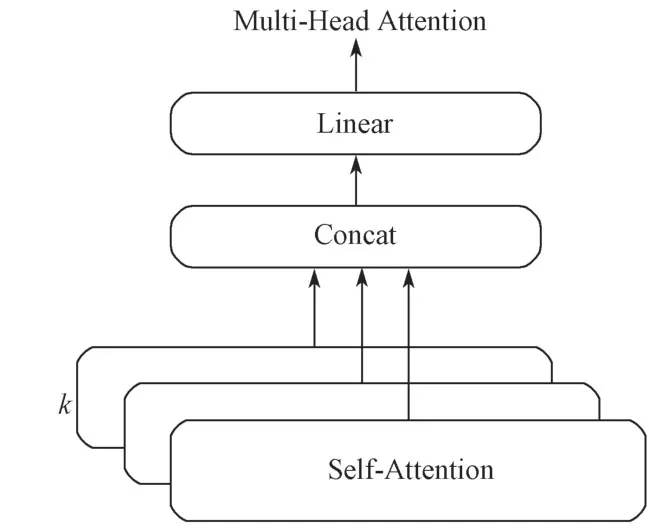
图5 多头注意力模型结构图Fig.5 Multi-Head Attention model structure diagram
与自注意力模型相比,多头注意力模型实质上是进行多次自注意力计算,每一次算一个头,可以使模型在不同的表示子空间里学习到相关的信息而且具有优于RNN的并行计算性能。
首先,在电子病历NER 任务中,对于输入的一个句子X=(x1,x2,···,xt,···,xn),通过IDCNN层后的输出是Y=(Y1,Y2,···,Yt,···,Yn),对于句子中的第t个字符的输出状态Yt,通过式(4)进行单头自注意力计算。其中,共进行i次计算,即有i个头,第i次计算的结果是headi。
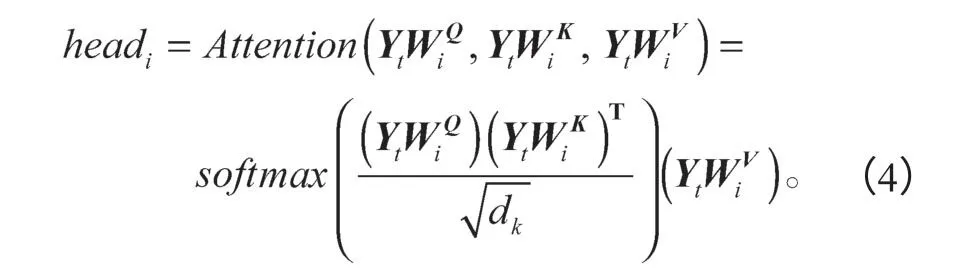
softmax( )为归一化因子。
然后,拼接这i次的计算结果,再进行一次线性变换,即可以得到句子中第t个字符的多头注意力计算结果,具体的计算公式如式(5)所示,其中W O为权重参数。

2.4 CRF层
CRF模型是一种经典的判别式概率无向图模型,该模型经常被应用于序列标注任务中,即在给定观察序列C=(c1,c2,···,cn)的情况下,计算状态序列Y=(y1,y2,···,yn)的条件概率P(y|c),具体计算公式如式(6)所示,其中,fk为特征函数,wk为特征函数的权重,Z(c)为归一化项。
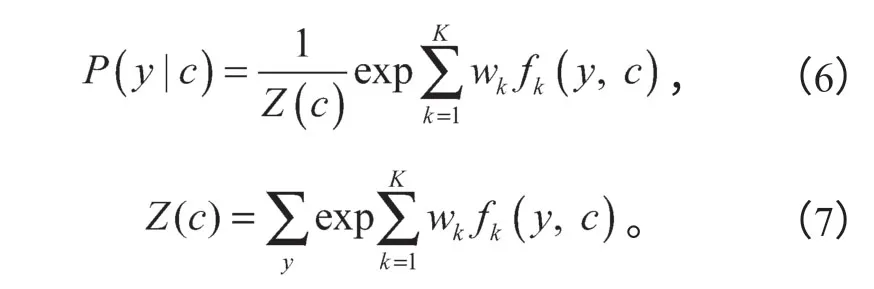
在医疗电子病历NER中,多头注意力层无法考虑标签之间的依赖关系,比如“I-ANA”标签不能紧接在“B-DIS”标签的后面。CRF层可以有效地约束预测标签之间的依赖关系,对标签序列进行建模,从而获取全局最优序列。多头注意力层的输出是电子病历句子中每个字对应的各个标注符号的分数,记矩阵P为打分矩阵,Pi,j为第i个字符分类到第j个标签的概率值,Ti,j为第i个到第j个标签的状态转移打分。对于输入句子X=(x1,x2,···,xn),句子标签序列y=(y1,y2,···,yn)的打分为
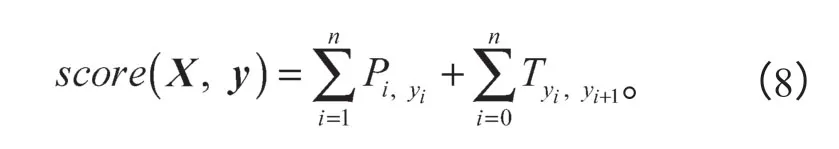
使用最大化对数似然函数对CRF模型进行训练,通过式(9)和(10)计算在给定句子X的情况下标签序列y的条件概率,其中yX为给定的句子X全部可能的标签序列,L为定义的损失函数。
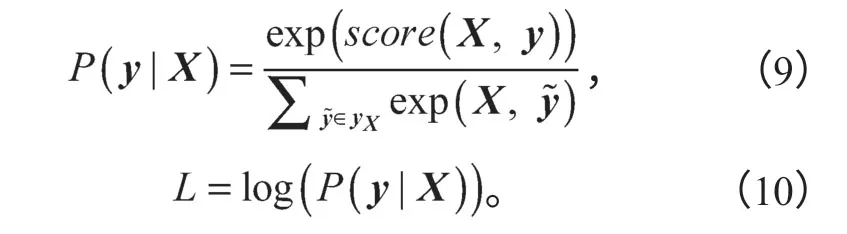
在CRF模型预测过程中,采用维特比(Viterbi)算法来求解全局最优序列,公式如式(11)所示,其中y*为集合中使得分函数取得最大值的序列。
3 实验及结果分析
3.1 实验数据及标注策略
本研究采用的电子病历医疗实体识别中文数据集由CCKS2019 评测任务一“面向中文电子病历的医疗实体识别及属性抽取”提供,所有电子病历语料由专业的医学团队进行人工标注。该标注数据集分为训练集和测试集,其中训练集包含1 000份医疗电子病历,共计7 717个句子;测试集共包含379份医疗电子病历。表1是各类医疗实体个数统计信息,总共为5 363个文档。
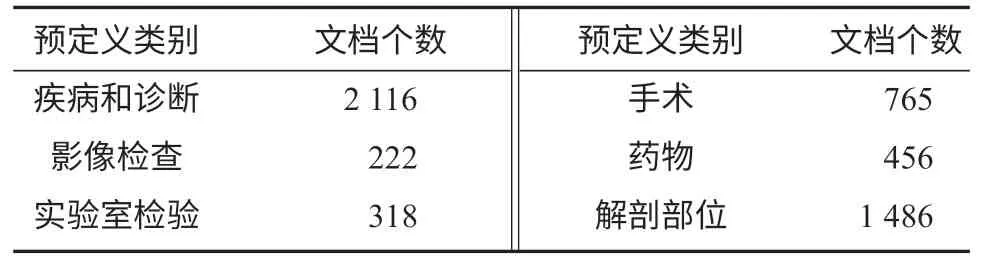
表1 医疗实体类别数据统计Table1 Statistics of medical entity categories
每份电子病历详细地标注了医疗实体的名称、起始位置、结束位置和预定义实体类别,并进行脱敏处理。具体分为疾病和诊断、影像检查、实验室检验、手术、药物和解剖部位6类预定义类别,各类预定义类别及其含义信息如下:
1)疾病和诊断。即医学上定义的疾病和医生在临床工作中对病因、病生理、分型分期等所作的判断,如胃癌、肠胃炎等。
2)影像检查。包括影像检查、造影、超声、心电图,如CT、MRI(magnetic resonance imaging)等。
3)实验室检验。指在实验室进行的物理或化学检查,特指临床工作中检验科进行的化验,不含免疫组化等广义实验室检查,如血红蛋白、CA199 等。
4)手术。指医生在患者身体局部进行的切除、缝合等治疗,如腹腔镜根治性全胃切除术、经腹直肠癌切除术(DIXON)等。
5)药物。指用于疾病治疗的具体化学物质,如伊立替康、格列卫等。
6)解剖部位。指疾病、症状和体征发生的人体解剖学部位,如口腔、十二指肠等。
本研究选择字标注方法完成对数据集的标注,采用BIO(begin,inside,outside)标注体系,其具体格式为B-X、I-X和O。B代表医疗实体开始位置的字符,I代表医疗实体剩余部分的字符,O代表非医疗实体的字符。X代表医疗实体的类别,记为DIS、IMG、LAB、OPE、MED和ANA,分别代表疾病和诊断、影像检查、实验室检验、手术、药物和解剖部位6类医疗实体。该任务共有13种不同的标签,分别为
B-DIS、I-DIS、B-IMG、I-IMG、B-LAB、I-LAB、B-OPE、I-OPE、B-MED、I-MED、B-ANA、I-ANA和O。各类别的实体标注符号及示例如表2所示。
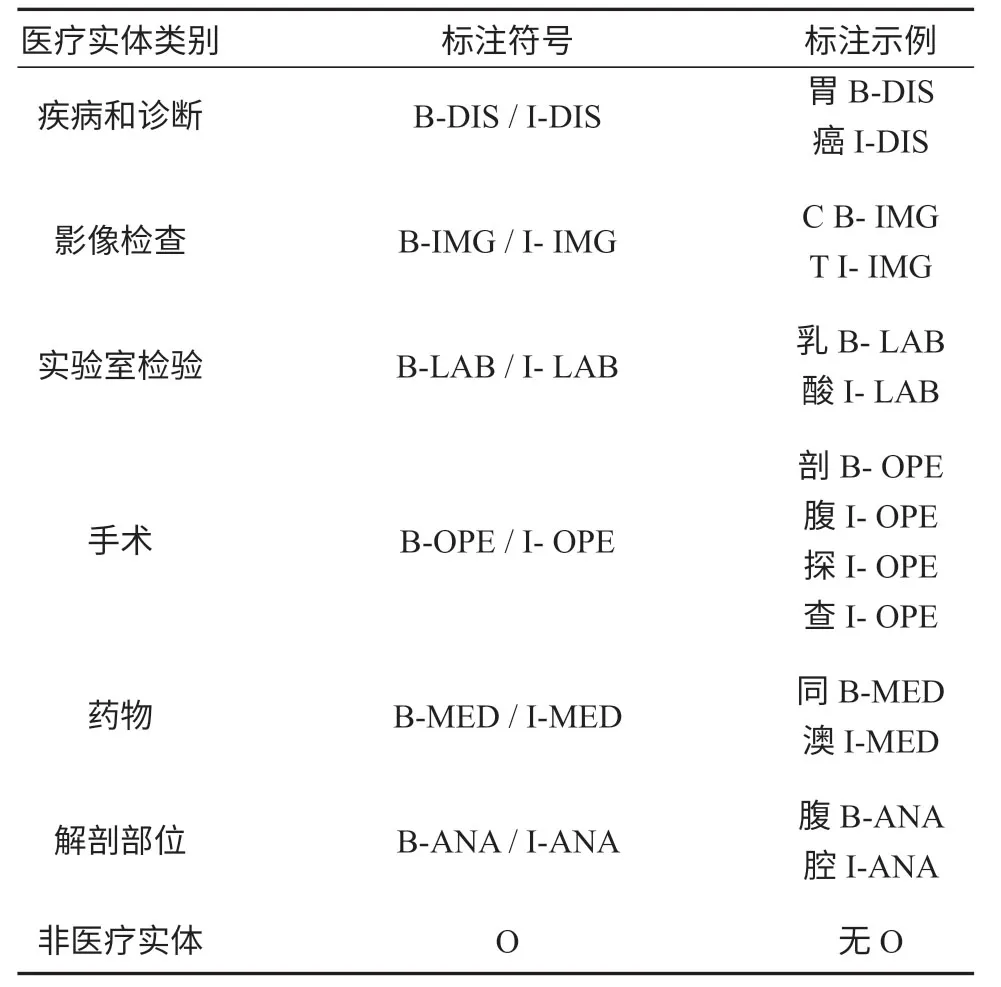
表2 医疗实体类别标注符号及示例Table2 Classification labeling symbols and examples of medical entities
虽然电子病历语料由专业的医学团队进行人工标注,但是不可避免地会出现实体类别或者开始、结束位置的标注错误以及标注前后不一致等问题。比如,在一段电子病历文本“直肠癌术后,拟行第4次化疗”中,“直肠癌术后”被人工标注为“疾病和诊断”类别的医疗实体,而在另一段电子病历文本“食管癌术后、肝癌介入术后”中,“食管癌”被人工标注为“疾病和诊断”,与前一段文本中的标注存在前后不一致的问题,这种标注不一致会导致实体识别过程中错误预测实体边界,从而影响实体识别的效果。本研究针对实体类别或者开始、结束位置的标注错误问题,在数据集的预处理中采取人工纠错的方式,将标注错误的实体进行纠正。
3.2 评价指标
医疗电子病历命名实体识别的评价指标采用精确率(precision)P、召回率(recall)R以及F1-Measure,其中F1-Measure是精确率和召回率的加权调和平均值,具体公式为(12)~(14)。

式(12)~(14)中:TP为正确识别医疗实体的个数;
FP为识别到不相关医疗实体的个数;
FN为未识别到相关医疗实体的个数。
在预测时,判断医疗实体预测完全正确的标准是实体的边界和类别同时预测正确。
3.3 实验环境
本文实验的命名实体识别模型基于TensorFlow框架,具体实验环境设置如表3所示。
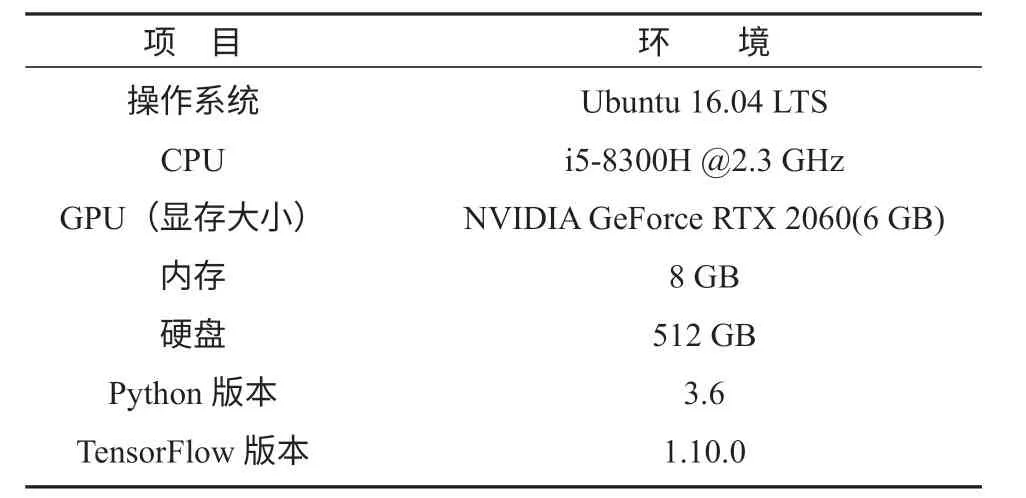
表3 实验环境设置Table3 Experimental environment settings
3.4 实验参数设置
BiLSTM-CRF模型参数设置如下:Word2Vec的预训练字嵌入向量维数为100,窗口大小为3,最小词频为10;LSTM(long short-term memory)隐藏层的单元个数为128;学习率为0.000 5,批大小(batchsize)为20,dropout为0.5,clip为5,优化算法使用自适应时刻估计法(Adam)。
IDCNN-CRF模型的参数设置如下:IDCNN 隐藏层的滤波器个数为128个;其余参数的设置与BiLSTM-CRF模型保持一致。
BERT-IDCNN-CRF模型的参数设置如下:采用BERT-Base版预训练语言模型,该模型由Google提供,为12 头模式,共有12层和110 M个参数,隐藏层为768维;最大序列长度(max_seq_len)为128;学习率为0.000 5,批大小(batchsize)为20,dropout为0.5,clip为5,优化算法使用自适应时刻估计法(Adam)。
BERT-IDCNN-MHA-CRF模型的参数设置如下:采用BERT-Base版预训练语言模型,该模型由Google提供,为12头模式,共有12层和110 M个参数,隐藏层为768维;多头注意力层头数为4;最大序列长度(max_seq_len)为128;其余参数设置与BERTIDCNN-CRF模型保持一致。
3.5 实验设计与结果分析
本研究将CCKS2019 提供的电子病历数据,采用交叉验证的方法,以7:2:1的比例划分为训练集、验证集和测试集。为验证BERT-IDCNN-MHA-CRF模型的有效性,将该模型和以下模型进行对比:
1)BiLSTM-CRF模型。即基于BiLSTM的特征抽取和CRF 约束的模型,在该模型中,使用100维的Word2Vec预训练字向量。
2)IDCNN-CRF模型。即基于IDCNN的特征抽取和CRF 约束的模型,IDCNN 能够更好地抽取句子的局部特征,且有更快的并行计算速度。在该模型中,使用100维的Word2Vec 预训练字向量。
3)BERT-IDCNN-CRF模型。即在IDCNN-CRF模型的基础上加入BERT预训练语言模型。
在该项实验中,epoch 默认设置为80次,表4是不同模型的实验结果。对比表4中各模型的实验结果,可以看出BERT-IDCNN-MHA-CRF模型的精确率、召回率和F1值相比于BiLSTM-CRF 基线模型的分别提高了1.80%,0.41%,1.11%,该模型在疾病和诊断、检查、手术、药物和解剖部位5类医疗实体上的F1值是最高的。检验实体最高的F1值为87.82%,出现在BiLSTM-CRF模型中。
在所有模型中,“疾病和诊断”类型医疗实体的F1值较低,该类型实体普遍长度较长,而且存在括号等补充说明信息,例如“(直肠)腺癌(中度分化),浸润溃疡型”,因此在预测该类实体时存在边界预测错误的问题,从而导致实体识别错误。此外,一些“疾病和诊断”医疗实体和“手术”医疗实体在文本结构上相似,这会导致该类型实体被错误分类,比如“脾脏切除术后”和“脾脏切除术”,前者属于“疾病和诊断”实体,而后者属于“手术”实体,虽然两个实体仅一字之差,却是预定义类别不同的两类实体。“解剖部位”类型医疗实体F1值也较低,该类实体的数量是6类实体中最多的,而且特征众多,识别时存在较大的难度。
BiLSTM-CRF模型和IDCNN-CRF模型的F1值分别为81.32%和81.44%,说明两种模型的识别效果相当。但是,IDCNN的并行计算能力比BiLSTM的要强,IDCNN-CRF模型与BiLSTM-CRF模型相比,训练一轮的时间要少25 s。因此,本文实验选择在IDCNN-CRF模型的基础上加入BERT预训练语言模型,相比于IDCNN-CRF模型,BERT-IDCNNCRF模型的识别效果有小幅度提升,F1值提高了约0.42%,这说明BERT预训练语言模型对于电子病历句子中的上下文语义有更准确的表示,从而可以提高实体识别效果。BERT-IDCNN-MHA-CRF模型是在BERT-IDCNN-CRF模型的基础上,加入多头注意力机制,多次计算句子中每个字和所有字的注意力概率,实验结果表明,该模型的精确率为82.63%,F1值为82.43%,是所有模型中最高的;同时,其召回率为82.23%,相比于BERT-IDCNN-CRF模型的F1值,提高了0.57%。
综上所述,本研究提出的BERT-IDCNN-MHA- CRF模型的总体性能最好,可以被成功地应用于医疗电子病历命名实体识别中。
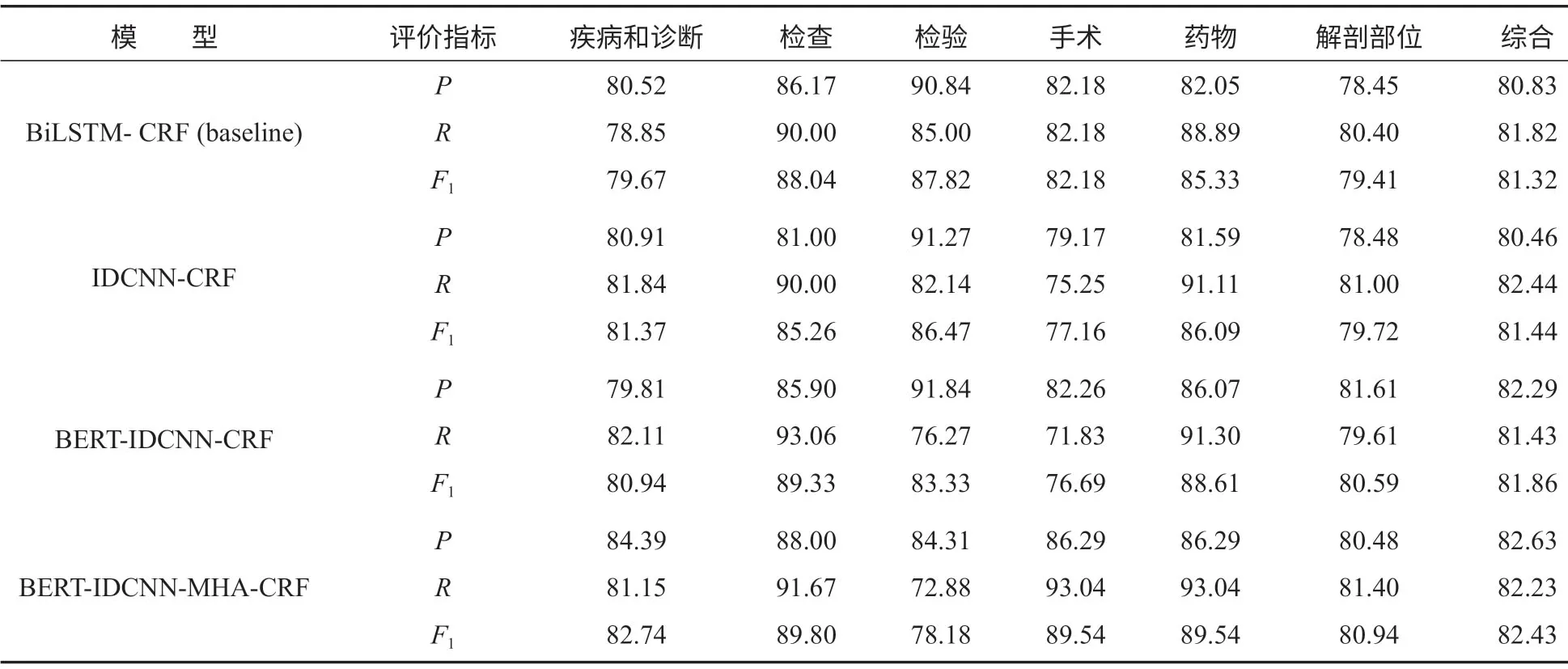
表4 不同模型的实验结果Table4 Experimental results of different models %
4 结语
采用基于BERT的医疗电子病历命名实体识别模型,能够较好地识别电子病历中的医疗实体。其中BERT预训练语言模型可以更准确地表示电子病历句子中的上下文语义,IDCNN 对局部实体的卷积编码相对于BiLSTM的长距离依赖关系编码,可以达到更好的医疗实体识别效果,训练速度和预测的效率都有所提高。多头注意力可以获取电子病历句子中的长距离依赖特征。实验结果表明,模型能够较好地完成医疗电子病历的命名实体识别任务。接下来将该命名实体识别模型进行改进,再应用到其它领域的命名实体识别研究中。
