全基因组关联分析显示基因ANXA8和C10orf11为影响肌少症的候选基因
2020-07-06蒋自璇刘宝林邓红文
冉 姝, 何 笑, 蒋自璇, 刘宝林, 邓红文
(1. 上海理工大学 医疗器械与食品学院,上海 200093;2. 杜兰大学 公共健康医学院,美国)
肌少症(sarcopenia)是以骨骼肌质量和功能随年龄增长而减少为特征的多基因复杂疾病,肌少症还与一系列其他健康问题相关,如跌倒和骨折、运动障碍、感染、代谢紊乱等,这些都是老年人残疾和死亡的主要原因之一[1-2]。在70岁以下的白人老年人中,有13%~24%的老人患有肌少症,而在80岁以上的老年人中,这一比例则高达50% 或更多[3]。瘦体重(lean body mass,LBM)收缩性的下降也会导致骨骼负荷的下降,使骨骼长期处于闲置状态,容易诱发骨质疏松症[4]。肌少症是遗传和环境因素共同作用的复杂疾病。遗传因素可分别解释个体间肌肉强度、下肢功能和日常生活能力变异的36%~65%、57%和34%[5-6];此外,骨骼肌能量代谢中主要酶的活性都具有家族遗传[7]。LBM是肌少症重要的性状[8-10]。LBM由骨骼肌(60%)、脏器等结缔组织[11-12]组成,遗传率50%以上[13],是预测肌少症的遗传特征。利用双能X射线吸收仪(DXA)可以准确地测量LBM。
既往研究表明,肌少症与骨质疏松症、肥胖症均具有较高的遗传易感性,且具有遗传相关性,可能存在多效性基因影响这两种疾病的发病风险。骨和肌肉在生长过程中都对旁分泌和内分泌刺激有共同的反应[14]。肌肉收缩的减少导致骨骼负荷的减少,从而导致骨骼完整性的丧失[15]。在胚胎发育过程中,成骨细胞和肌肉细胞共享一个共同的间充质干细胞。基于骨密度(bone mineral density,BMD)与LBM相互作用的生物学信息,预测两者可能存在共同的遗传背景,即多效性的基因[16]。此外,肌少症常与体脂增加一起出现,这种情况被称为“骨骼肌减少性肥胖”[17],它是伴随肌量减少和肥胖增加而出现的一种重要的老年性疾病[18]。肌少症和肥胖症均具有较高的遗传易感性,可能存在多效位点影响这两种疾病的发病风险。
候选基因连锁分析和关联法是早期鉴定肌少症遗传因子的两种主要方法。但研究结果并不一致,其中种族、样本大小不同等因素造成试验的可重复性低、所涉及的染色体区域范围较宽可能是其重要原因。全基因组关联分析(genome-wide association study,GWAS)通过包括单核苷酸多态性变异(single nucleotide polymorphisms,SNPs)和拷贝数变异(copy number variation,CNV)在内的全基因组高密度遗传标记并分型,已成为鉴定复杂疾病致病基因的热点研究方法之一[19]。最近的GWAS研究发现许多SNPs与LBM相关。2009年,有研究对1 000个无亲缘关系美国白人进行LBM的GWAS分析,发现亚甲基四氢叶酸还原酶(MTHFR)多个SNP与肌肉量显著关联[20]。Livshits等[21]在3 180个英国妇女研究中发现,肌量与MTHFR基因所在的染色体1p36位点存在连锁(LOD (log odds score)值为 2.17,p值为 0.001 6)。Sun等[22]在中国人群双变量GWAS发现HK2等基因的变异可以同时引起股骨颈骨几何学参数和四肢LBM的变化。Urano等[23]对1 081个日本绝经妇女的研究发现rs12409277位点与LBM显著关联,该位点影响转录调节因子PRDMl6的转录活性。Livshits等[24]对1 550例英国孪生子全基因DNA甲基化的研究发现,一些基因DNA甲基化与肌肉量变异相关。Medina-Gomez等[25]对10 414名儿童通过双变量GWAS荟萃分析发现,8个基因位点与LBM和头骨密度关联。
为鉴定与肌少症相关联的基因,本研究对1 000个不相关的白人样本进行GWAS分析。验证样本包括中国汉族样本和白人样本。
1 研究方法
1.1 样 本
a. 全基因研究样本。
本研究通过了密苏里堪萨斯大学(University of Missouri Kansas-City)伦理委员会的审查,并在开展研究前和所有参与者签署了知情同意书。所有研究对象完成了包括各项生理指标、生活习性、疾病史、家族史、运动史等相关内容的调查问卷。本次全基因组关联分析从本实验室建立和正在扩大的遗传资源数据库中的6 000多个样本中随机抽取了1 000个独立样本。所选择样本均来自具有欧洲血统的美国白种人。
b. 验证样本包含中国汉族样本和美国白人样本。
中国汉族样本是在湖南省长沙市征集的1 625个随机样本,包括823个女性样本和802个男性样本。本研究得到西安交通大学与湖南师范大学审查委员会批准。样本的筛选采取了和堪萨斯独立样本征集相同的标准。美国白人样本包含2 283个高加索裔无血缘关系的成年人,包括556个男性和1 727个女性。所有样本来源于美国中部密苏里州堪萨斯城和内布拉斯加州奥马哈城城区及其周边地区。本研究得到了密苏里大学堪萨斯分校伦理委员会(Institutional Review Board, IRB)的批准。验证样本与第一阶段的研究样本没有重复。
1.2 表型测量
采用Hologic公司的双能X射线(DXA)骨密度仪(Hologic Inc., Bedford, MA, USA)扫描仪对研究对象进行表型测量。让受测者卧位于测量床上,从头到脚进行扫描。DXA能够精确测量四肢、全身瘦体重量,以及全身脂肪量。同时测量受试者的身高(m)和体重(kg)。
1.3 基因分型和质量控制
使用商业分离试剂盒(Gentra systems, Minneapolis,MN, USA,按说明书步骤操作)从人体血液中提取基因组DNA。基因分型采用Affymetrix 250KNsp和Affymetrix 250K,由美国范德比尔大学医学中心范德比尔共享中心完成,过程按标准步骤进行。
在个体和SNP水平上,严格执行基因型质量控制(QC)步骤。在个体水平上,使用PLINK根据X染色体上的基因型数据推断出个体的性别,并与问卷中记录的性别作比较,将不明确的以及与报告性别不一致的个体删除。在SNP水平上,在最初的500 568个SNP中,舍弃了32 961个成功率小于95%的SNP、36 965个等位基因频率显著偏离 Hard-Weinberg(HWE)平衡的 SNPs(p<0.001)、51 323个等位基因频率小于1%的SNPs,同时把具有孟德尔错误的SNPs设为缺失值。因此,最后共有379 319个SNPs用于关联分析。
用于验证的两个样本分别采用Affymetrix Gene Chip Human Mapping SNP 6.0芯片进行基因分型。
1.4 统计分析
采用逐步回归法(stepwise regression)检验年龄、年龄的平方、身高、体重是否对全身LBM具有显著影响,然后采用Minitab(Minitab Inc., State College, PA, USA)来校正身高、体重等因素对LBM的影响。GWAS分析中,为了最大限度地减少因群体分层而导致的假阳性或假阴性结果,采用主成分分析(principal component analysis, PCA)法来校正1 000个白人样本中潜在的种群分层。首先,在样本中挑选一组无关样本并计算它们的主成分,作为他们的遗传背景信息。然后,选用前5个主成分,利用这5个主成分值对表型进行校正,再利用校正后的表型进行关联分析。
关联分析使用Plink软件(http://zzz.bwh.harvard.edu/plink/summary.shtml)的加性遗传模型分析。
为分析GWAS和验证研究的整体关联证据,采用Fisher’s方法[26]的荟萃分析将研究样本和验证样本的p值整合,得到合并p值。
2 结 果
研究样本和验证样本的基本信息列于表1。
在发现样本中,共有379 319个SNPs用于关联分析。Bonferroni校正全基因组显著性水平(GWAS,0.05/379 319=1.31×10-7),在 GWAS 水平上,没有SNP低于这个显著性阈值标准,p值最低的是 rs7086719(p=1.23×10-6)。在研究样本中发现了809 个与LBM 相关的SNPs(p<1×10-3,call rate>90%,最小等位基因频率(minor allele frequency,MAF)>0.01)。在1 625个不相关的中国汉族样本中验证,发现40个SNPs与LBM高度关联。将这40个SNPs在2 283个不相关的白人中进行验证,最终有4个 SNPs得到验证(rs7905603:p=9.67×10-4;rs9416083:p=3.17×10-4;rs4409772:p=4.39×10-4;rs2894310:p=4.37×10-4),如表 2 所示。
通过其网页门户搜索国际小鼠表现型数据库[27](international mouse phenotyping consortium,IMPC)(http://www.mousephenotype.org/),发现敲除基因ANXA8纯合子的老鼠比对照组老鼠BMD增加,同时身体脂肪量也出现异常(p<0.05)。
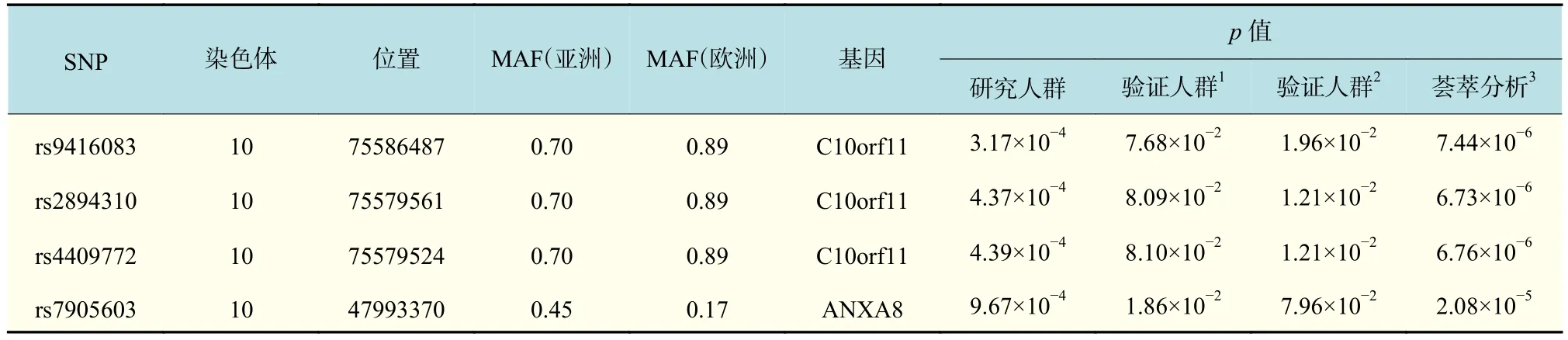
表 2 在GWAS研究中显示和瘦体重相关联的SNPsTab.2 SNPs associated with LBM in GWAS
使用STRING蛋白互作网络(https://stringdb.org/)对基因ANXA8进行注释,所得到的蛋白质-蛋白质相互作用网络显示基因ANXA8与MYOD,MYOG,MYF6等调控肌肉表达的基因存在某些联系(见图1),ANXA8是膜联蛋白家族中调控Ca2+结合蛋白的一个成员,它与急性早幼粒细胞白血病(APL)有关[28]。
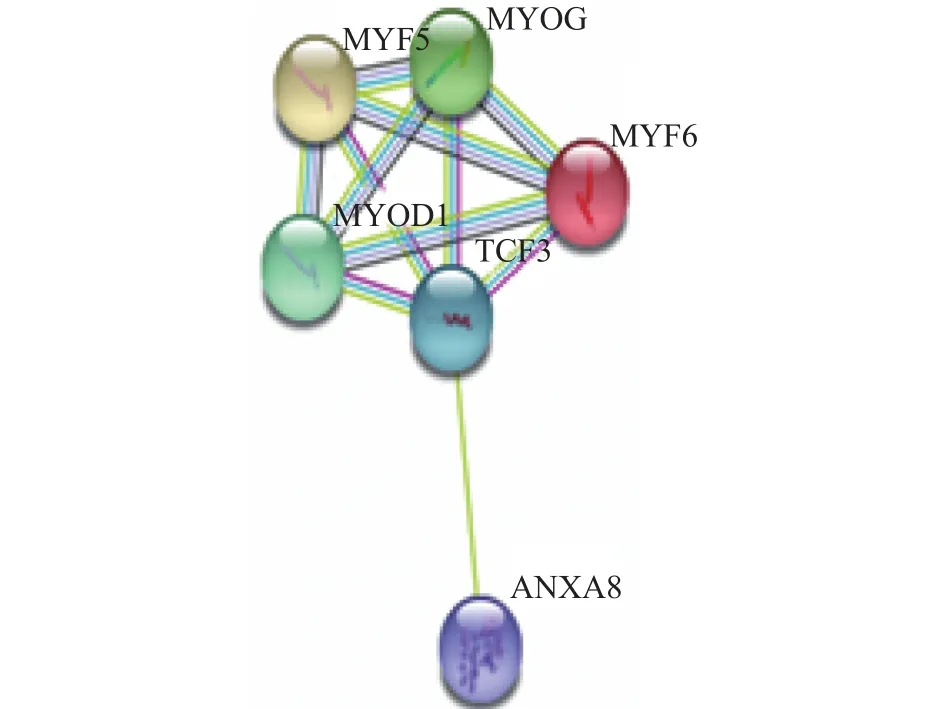
图1 ANXA8基因与肌肉代谢蛋白的STRING蛋白质互作网络图Fig. 1 STRING protein interaction network between ANXA8 and muscle metabolic proteins
3 讨 论
ANXA8(annexin A8)是在破骨细胞分化后期强烈诱导的基因和调节细胞特有肌动蛋白环形成的蛋白。膜联蛋白家族是多基因蛋白质家族,最初被称为内酯环[29]。迄今为止,已经有几个膜联蛋白家族的成员被发现其表达与骨骼肌有关系。例如在骨骼肌中有膜联蛋白annexin A8和annexin A5的表达[30]。annexin A1通过促进卫星细胞的迁移来促进骨骼肌的分化,从而调节成肌细胞的分化有助于骨骼肌组织的再生[31];另一个膜联蛋白家族的成员annexin A6,它是一种主要的骨骼肌蛋白,在骨骼肌中高度表达[32-34]。ANXA8基因在各种白血病和淋巴细胞中活化[35-36],ANXA8在这些细胞的增殖和/或分化中发挥着独特的作用[37]。此外,STRING蛋白互作网络还发现基因ANXA8与骨骼肌肌肉相关代谢蛋白TCF3有着直接的联系。TCF3蛋白靶基因的表达限于特定的细胞谱系,通过含有TCF3和组织限制性螺旋-环-螺旋(bHLH)因子的异二聚体来激活细胞特异性基因表达并促进细胞分化[37]。
rs9416083, rs2894310和rs4409772均位于基因C10orf11(leucine rich melanocyte differentiation associated),该基因编码是一种富含亮氨酸的重复蛋白[38]。图2中的haploview图显示C10orf11基因在白人和中国人中的LD结构,运用Haploview 4.2软件和从1 000个基因组计划中获取的链接数据,对 C10orf11基因的 3个 SNPs(rs9416083,rs2894310和rs4409772)分别在中国人群和欧洲白人中进行连锁不平衡分析。图2(a)为中国人的分析结果,图2(b)为欧洲白人的分析结果。图中颜色的深浅代表对应的两个SNP间的LD强度(r2)。红色区域表示r2=1。该区域仅包括一个LD块,由带有黑色线条的三角形表示。所有图均使用Haploview软件,根据HapMap国际计划Phase 1和Phase数据生成。目前,对基因C10orf11的生物学功能仍知之甚少,Wada等[39]的研究表明,人类C10orf11的同源基因参与了早期肠胚中的β-连环蛋白(MIM 116806)的信号传导,并表明了C10orf11的功能在β-连环蛋白的上游或平行于β-连环蛋白。

图2 基因C10orf11中国人和欧洲白人中的LD结构图Fig.2 LD structure of the C10orf11 gene in the Chinese and Caucasusian sample
采用GWAS分析方法,在1 000名不相关的美国高加索人中发现,位于基因ANXA8和C10orf11的4个SNPs与LBM有关,它们可能会影响或者导致肌少症。
