食管鳞状细胞癌相关miRNA的生物信息学分析
2020-07-02曹诗茹周荣秒霍向然
曹诗茹, 李 琰, 周荣秒, 黄 茜, 霍向然, 王 娜
(河北医科大学第四医院分子生物学研究室, 河北 石家庄 050011)
食管鳞状细胞癌(Esophageal squamous cell carcinoma,ESCC)是食管癌的一种主要组织学类型,发病率和病死率均居我国恶性肿瘤前列。早期诊断率低和侵袭性强是食管癌高死亡率的主要原因。所以,需要进一步研究食管鳞癌的生物标志物和分子机制。MicroRNAs是一种单链非编码RNA,约23个核苷酸。miRNAs的失调可导致恶性肿瘤,而miRNAs发挥致癌作用或抑癌作用因其靶向的mRNAs类型而异。随着基因芯片和RNA测序技术的迅速发展,基因表达综合数据库在生物信息学分析中发挥着重要作用,它可以为我们发现功能性miRNA提供线索。本研究利用GEO数据库中食管鳞癌miRNA表达谱芯片数据GSE114110,筛选食管鳞癌及正常食管上皮组织的差异表达miRNA(differentially expressed miRNAs, DE-miRNAs),并预测其靶基因,对靶基因进行聚类分析及功能富集分析,利用Cytoscape软件构建蛋白相互作用(protein-protein interaction, PPI)网络,筛选Hub基因并构建miRNA-Hub gene网络,旨在进一步鉴定食管鳞癌发生发展中的关键miRNA及其靶基因,为食管鳞癌筛选分子标志物及进一步明确发病分子机制提供理论指导。
1 材料与方法
1.1研究工具和对象:通过对美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)GEO数据库中食管鳞状细胞癌miRNA表达谱数据进行筛选,选取GSE114110 miRNA表达谱芯片数据为研究对象,该芯片为Agilent-038169 human miRNA,安捷伦GPL24967平台。GSE114110数据集包括30例食管鳞癌组织及10例正常食管上皮组织。
1.2差异miRNA分析:应用GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r/)对GSE114110数据集进行分析,对两组实验条件相同的样品进行对比,筛选DE-miRNAs。本研究所设定的阈值为P<0.05,|logFC|≥2。
1.3筛选验证数据集并对差异miRNA进行验证:在GEO数据库中输入关键词 “Esophageal squamous cell carcinoma”和“ESCC”进行搜索,设定非编码RNA数据并设定物种为人类,确定32个数据集。纳入标准:每个数据集包括ESCC样本组和对照样本组(健康食管组织、相邻非癌组织)。排除标准:①仅有lncRNAs和circRNAs数据,无miRNAs数据;②缺乏正常对照组。最后纳入GSE97049, GSE59973, GSE55856 和 GSE43732。
1.4预测靶基因:通过miRNet(https://www.mirnet.ca/miRNet/faces/home.xhtml)分别对前三位上调和下调的差异miRNAs进行靶基因预测。
1.5靶基因的功能富集:将筛选所得的6个DE-miRNAs预测的靶基因数据导入DAVID ( https://david.ncifcrf.gov/)进行功能注释和通路富集分析,包括GO分析和KEGG通路分析。P<0.05差异有统计学意义。
1.6PPI网络和miRNA-Hub gene网络的构建:分别构建PPI网络和miRNA-Hub gene网络。首先将靶基因映射到STRING数据库(http://string-db.org),以评估靶基因之间的功能关联,并选择可信度评分>0.7的交互作用进行下一步研究。利用Cytoscape软件分析PPI网络的连通性,获得Hub基因,建立miRNA-Hub gene网络。
1.7Hub基因的表达验证:starBase(http://starbase.sysu.edu.cn/panCancer.php)是一种包含了来源于TCGA数据库中肿瘤组织表达数据的在线分析网站。本研究应用starBase数据分析工具对Hub基因进行表达水平的验证。
1.8统计学处理:使用上述各种数据分析软件,对所得的各种数据信息进行统计分析。
2 结 果
2.1食管鳞癌和正常食管上皮组织中DE-miRNAs的筛查及其靶基因的预测:使用GEO2R分析GSE114110数据集,结果显示共筛查出159个差异倍数达4倍以上的DE-miRNAs,其中上调的miRNA86个,下调的miRNA73个,并绘制了这些DE-miRNAs的火山图(图1),列出了上调幅度最大的10个miRNA和下调幅度最大的10个miRNA(表1、表2)。根据fold change (FC), 上调幅度最大的3个miRNA分别是hsa-miR-196a-5p、hsa-miR-196b-5p和hsa-miR-34c-5p;下调幅度最大的3个miRNA分别是hsa-miR-133a、hsa-miR-1和hsa-miR-4770。将这6个DE-miRNAs在食管鳞状细胞癌miRNA表达谱芯片数据集GSE97049, GSE59973, GSE55856 和 GSE43732中进行验证,与前期分析一致(表3)。通过miRNet预测出3个上调miRNA的514个潜在靶基因及3个下调miRNA的1186个潜在靶基因(图2)。
2.2靶基因的功能富集:利用DAVID对上述靶基因进行功能富集分析,包括生物学过程(BP)、细胞组成(CC)和分子功能(MF)三类GO功能注释分析。如图3a1-a3所示,三种上调miRNA的靶基因主要参与的生物学过程(BP)为调控细胞增殖、调节细胞周期、转录调控等;主要参与的细胞组成(CC)为膜结构、核质等;主要参与的分子功能(MF)为蛋白的结合、转录因子的活性、DNA结合等。图3b1-b3展示了三种下调miRNA的靶基因主要参与的生物学过程(BP)为蛋白质的定位和运输、肌动蛋白细胞骨架组织、细胞凋亡的调节等;主要参与的细胞组成(CC)为各种膜结构等;主要参与的分子功能(MF)为肌动蛋白结合、细胞骨架内蛋白结合、转运蛋白活性等。为了进一步分析这些靶基因的富集途径,我们进行了KEGG通路富集分析。上调miRNA的靶基因富集到的KEGG通路包括癌症通路、黏着斑、p53信号通路等(图4a)。下调miRNA的靶基因富集到的KEGG通路包括癌症通路、黏着斑等(图4b)。重要的是,上调miRNA的靶基因和下调miRNA的靶基因都富集于黏着斑通路,黏着斑调节异常在细胞侵袭和迁移中起着重要作用。因此,上调miRNA的靶基因中与黏着斑通路相关的21个基因(ACTB, PIK3CG, CAV1, VAV3, MAP2K1, MET, ITGA11, ITGB3, FLNA, VCL, AKT1, MAPK1, IGF1R, CCND1, LAMB2, CCND2, ITGAV, BCL2, PDGFRA, PDGFRB, PTENP1)和下调miRNA的靶基因中与黏着斑通路相关的32个基因(PDGFB, XIAP, ERBB2, ITGB4, CDC42, IGF1R, SOS2, PIK3CA, THBS1, THBS2, PIK3R2, FN1, EGFR, ACTB, ACTN4, MET, ITGA1, ACTN1, IGF1, ITGA3, FLNC, FLNB, FLNA, VASP, MAPK1, CCND1, ITGA6, VEGFA, RAP1B, COL1A1, CRK, PARVA)和上述筛选出的6个miRNA相互作用,从而调节食管鳞状细胞癌的侵袭和转移。
2.3PPI网络和miRNA-Hub基因网络的分析:将筛选获得的6个DE-miRNAs的靶基因输入STRING网站,构建PPI网络。进一步应用Cytoscape软件分析靶基因之间的连通度,并筛选出前十名,为Hub基因(表4)。上调DE-miRNAs的hub基因为MAPK1、MYC、AKT1、HSP90AA1、POLR2D、CCND1、SMAD4、HIST1H2BB、HIST1H2BD、CDKN1B。下调DE-miRNAs的hub基因为MAPK1、EGFR、CDC42、POLR2K、PIK3CA、KRAS、POLR2I、HIST2H2AC、FN1、VEGFA。在这些基因中,MAPK1节点度最高。结果表明,MAPK1可能是食管鳞状细胞癌的关键靶点。随后,利用Cytoscape软件构建miRNA-Hub gene网络,如图5所示。从图5a中我们发现,hsa-miR-196a-5p可能调控10个hub基因中的7个(MAPK1、MYC、POLR2D、CCND1、HIST1H2BB、HIST1H2BD、CDKN1B)。6个hub基因和5个hub基因可能分别被hsa-miR-196b-5p和hsa-miR-34c-5p调控。此外,hsa-miR-1可能调控10个hub基因中的9个(EGFR、CDC42、POLR2K、PIK3CA、KRAS、POLR2I、HIST2H2AC、FN1、VEGFA)。其中4个hub基因和1个hub基因可能被hsa-miR-133a和hsa-miR-4770调控(图5b)。这些数据支持hsa-miR-196a-5p和hsa-miR-1可能是食管鳞状细胞癌的两个潜在调控因子。
2.4Hub基因的表达水平验证:应用starBase数据库进一步验证hsa-miR-196a-5p和hsa-miR-1在ESCC中靶基因的表达情况,如图6所示。分析结果表明,hsa-miR-196a-5p的7个靶基因中5个(MYC、POLR2D、CCND1、HIST1H2BB、HIST1H2BD)的表达在ESCC组织中较正常组织明显上调。而hsa-miR-1的9个靶基因中7个(CDC42、POLR2K、PIK3CA、POLR2I、HIST2H2AC、FN1、VEGFA)的表达在ESCC组织中较正常组织明显上调, miRNA表达与靶基因表达之间存在着负相关关系,这一点已被广泛认识,因此,显著上调的7个基因可能是下调hsa-miR-1的靶点。

表2 正常上皮组织和ESCC组织中差异表达miRNA下调前10位

表3 DE-miRNAs在食管鳞状细胞癌miRNA表达 谱芯片数据集GSE97049 GSE59973 GSE55856 GSE43732中的差异表达

图1 火山图(黑点代表30个ESCC样品和10个NE样品中无差异表达的miRNA,红点和绿点分别代表ESCC样品中上调和下调的miRNA)

图2 前三位上调和前三位下调miRNA的靶基因(a前三个上调miRNA;b前三个下调miRNA)

图3 前3位上调和前3位下调miRNA靶基因的GO功能

图4 六个DE-miRNA靶基因的KEGG通路富集分析

表4 PPI网络中连通度位列前十的Hub基因

图5 前3位上调和前3位下调miRNA及其Hub基因的调控网络
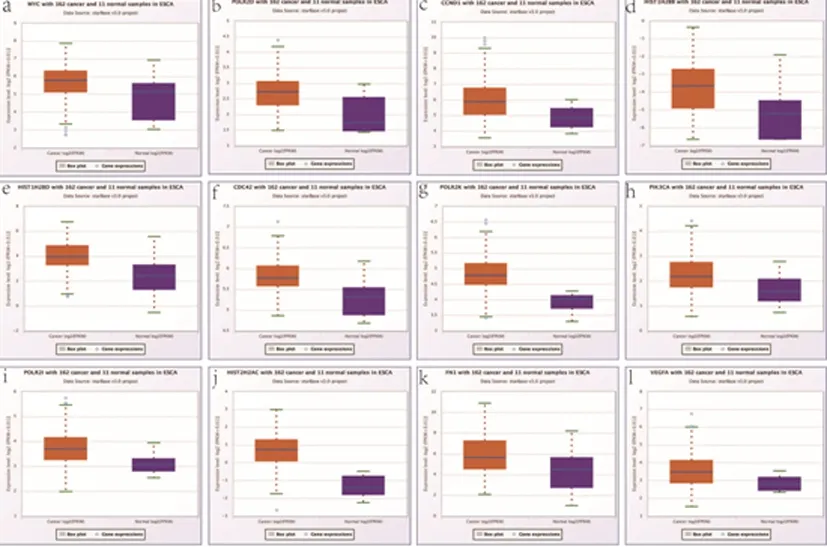
图6 starbase工具验证分析hsa-miR-196a-5p和hsa-miR-1靶基因的mRNA表达
3 讨 论
随着科技的进步,越来越多的新技术被应用于医学领域,尤其是基因芯片和高通量测序技术。通过生物信息学方法分析肿瘤相关大数据,为肿瘤研究提供了帮助。本研究通过从GEO数据库下载食管鳞状细胞癌miRNA表达谱芯片数据集GSE114110,分析差异表达miRNAs,得到6个与食管鳞癌相关的miRNA,预测其靶基因,并对靶基因进行GO和KEGG富集分析。结果显示三种上调miRNA的靶基因主要参与了细胞增殖、细胞周期、转录调控等生物学过程,这些生物学过程的异常会造成细胞的过度增殖、低分化细胞的出现和蛋白的异常表达。三种下调miRNA的靶基因主要参与了肌动蛋白细胞骨架等细胞组成;蛋白质的定位和运输、细胞凋亡等生物学过程;肌动蛋白结合、细胞骨架内蛋白结合、转运蛋白活性等分子功能。这些都是调节细胞增殖、迁移、侵袭的生物学基础。KEGG通路还发现上调miRNA的靶基因和下调miRNA的靶基因都富集于癌症通路和黏着斑通路。因此,上调miRNA的靶基因中与黏着斑通路相关的21个基因与下调miRNA的靶基因中与黏着斑通路相关的32个基因和筛选出的6个miRNA相互作用,从而调节食管鳞状细胞癌的侵袭和转移。
筛选连通度最高的前十个靶基因为Hub基因,构建了miRNA-Hub gene网络。我们发现hsa-miR-196a-5p和hsa-miR-1可调控大部分Hub基因。研究表明miR-196a在食管鳞状细胞癌中表达上调,通过靶向ANXA1调控食管癌细胞的增殖、侵袭和迁移,miR-196a/ANXA1轴可能是食管癌潜在的治疗靶点[1]。Mona等指出miR-196a在ESCC患者唾液样本中较健康组表达上调,其靶基因多富集在黏着斑通路和p53信号通路,为研究食管癌非侵袭性检测标志物指明了方向[2]。hsa-miR-1在许多恶性肿瘤中均低表达,是一种抑癌基因。研究显示hsa-miR-1可以靶向Notch2基因调节EMT信号通路,抑制食管癌细胞迁移、侵袭[3]。杜彦艳等证实食管癌中hsa-miR-1的表达与淋巴结转移、组织学分型和血管浸润有关,并通过结合靶基因LASP1和TAGLN2抑制癌细胞的增殖、侵袭和转移[4]。本文通过筛选和表达验证,确定CDC42、POLR2K、PIK3CA、POLR2I、HIST2H2AC、FN1、VEGFA在ESCC组织中明显上调,与hsa-miR-1存在负相关关系,可能是其靶点。纤维连接蛋白FN主要在食管鳞癌间质中高表达,并与患者预后不良密切相关。肿瘤间质FN高表达改变食管鳞癌细胞的形态和迁移能力,促进肿瘤细胞的转移[5]。hsa-miR-1可以靶向FN1基因抑制食管癌细胞增殖、侵袭,促进肿瘤细胞凋亡[6]。PI3K-Akt是细胞内重要的信号传导途径,在细胞生长、增殖、凋亡、血管生成等过程中发挥重要的生物学功能。PIK3CA是PI3K-Akt信号通路中关键的原癌基因,PIK3CA突变可导致激酶活性增强,刺激下游AKT,增加细胞侵袭和转移能力[7]。有研究证明miR-1高表达抑制了食管癌细胞的生长,使细胞周期阻滞在G1期,促进了细胞凋亡,同时降低了PIK3CA蛋白的表达水平。此外,miR-1过表达增加了食管癌细胞对抗癌药物吉非替尼的敏感性,其机制可能是PIK3CA信号通路失活[8]。Chou等发现miR-1能与CDC42 3'UTR结合并抑制其表达,在诱导乳腺癌转移中发挥重要作用;MALAT1也可通过竞争性结合miR-1影响CDC42的表达,MALALT1、miR-1和CDC42共同调节乳腺癌细胞的迁移、侵袭[9]。血管内皮生长因子VEGF-A是血管生成的关键调控因子,通过抑制血管生成信号通路从而影响血管生成是许多实体肿瘤的重要治疗方法。hsa-miR-1可通过抑制VEGFA和EDN1的表达,降低胃癌细胞的恶性度和血管内皮的生成,从而发挥抑癌作用[10]。
综上所述,通过分析miRNA表达谱芯片数据GSE114110获得差异表达miRNA hsa-miR-1及与其表达呈负相关的靶基因CDC42、POLR2K、PIK3CA、POLR2I、HIST2H2AC、FN1、VEGFA,这些基因在某些肿瘤的发生发展过程中起着重要作用,可能成为ESCC基因治疗的潜在靶点,为进一步研究提供理论指导。
