不同放牧对滇西北高原泥炭沼泽土壤氨氧化微生物群落的影响
2020-06-03郑荣波郭雪莲侯亚文
陈 梨,郑荣波,郭雪莲,*,侯亚文
1 西南林业大学国家高原湿地研究中心,昆明 650224

放牧过程伴随着植物的啃食,牲畜践踏及排泄物的输入[18],使土壤的环境因子发生改变[6,19],势必影响AOA和AOB群落结构和多样性,进而影响土壤氮循环过程。大量研究表明,放牧影响土壤微生物多样性和群落组成[6, 20- 22],Olivera等[23]的研究发现不同放牧强度对不同植被覆盖的区域土壤细菌多样性的影响不同。Suleima等[24]的研究发现猪粪肥(多种动物的排泄物和水混合)的施用降低土壤AOB的多样性,改变其群落结构,Orwin等[25]研究发现在草原生态系统中,牛尿液的施用改变土壤AOB的群落结构。但目前关于不同放牧类型对泥炭沼泽土壤氨氧化微生物群落组成及其多样性的影响鲜见报道。
滇西北是云南高原湿地的集中分布区,地处少数民族聚集的农牧交错带,放牧是当地湿地资源利用的主要途径。滇西北高原纳帕海湿地是受放牧干扰最严重最典型的区域,主要的放牧类型是牦牛放牧和藏香猪放养。牦牛放牧主要是践踏土壤、取食植物地上部分,藏香猪放养主要是对原位土壤进行剧烈翻拱扰动,破坏地表植物,使表层土壤裸露,破坏地下草根层造成土壤结构发生改变[26]。目前,纳帕海藏猪的载畜量达到115头/km2,已远超过当地的承载率是40头/km2,藏香猪放养破坏纳帕海湿地面积约总面积的5%[27]。放牧干扰会影响湿地土壤理化性质和PNR,影响土壤AOA和AOB群落,进而导致高原湿地氮循环的过程发生改变。因此,研究放牧活动对AOA和AOB群落结构及其多样性的影响对于预测放牧对湿地氮循环的影响至关重要。本研究选取滇西北高原泥炭沼泽湿地围栏禁牧区和长期放牧区为研究区,研究放牧干扰(牦牛放牧和藏香猪放养)对泥炭沼泽土壤理化性质,PNR,AOA和AOB群落结构及其多样性的影响,并通过分析土壤理化性质、PNR和AOA和AOB群落结构及其多样性之间的关系,阐明放牧对泥炭沼泽土壤氨氧化功能影响的内在机制,为湿地土壤微生物对放牧干扰的响应机制研究提供理论基础,为滇西北高原湿地放牧管理提供理论依据。
1 材料和方法
1.1 研究区的概况
研究区位于滇西北高原的纳帕海国际重要湿地(99°35′—99°43′ E, 27°47′—27°55′ N)内的泥炭沼泽湿地。纳帕海湿地属于青藏高原与亚热带季风气候区和中南半岛热带季风区的结合部,行政上隶属云南省迪庆藏族自治州香格里拉市,平均海拔3260m。纳帕海发育于石灰岩母质的中甸高原上,受喀斯特作用的强烈影响,湖盆底部被蚀穿而形成落水洞,湖水在地下汇集后从北部穿过小背斜出露形成支流汇入金沙江。湖盆四周山岭环绕,从湖盆中心至湖岸生长着大量的水生和陆生植被,湖滨有较大面积的沼泽和沼泽化草甸,周围山上生长硬叶常绿阔叶林和云杉(Picealikiangensis)冷杉(Abiesgeorgei)针叶林以及灌丛。具有高寒、年均温低、霜期长、气温年较差和日较差大、干湿季节分明等特点。年均温为5.4℃,年降水量为619.9mm,主要集中在6—8月[28]。土壤类型以草甸土、沼泽化草甸土、沼泽土和泥炭土为主。
1.2 实验样地设计
实验地选择在纳帕海国际重要湿地哈木谷村神湖附近的泥炭沼泽地(图1)。该区域土壤类型为典型泥炭沼泽土,地表无积水,土壤饱和,植被主要有水葱(Schoenoplectustabernaemontani)、苔草(Carex)、杉叶藻(Hippurisvulgaris)、矮地榆(Sanguisorbafiliformis)等。本研究选取土壤类型、水文和地貌特征相对一致的区域设置样地。选择地表植被未受啃食,土壤未受到干扰,禁止放牧的区域设置为对照区(CK)。在受牦牛放牧干扰,地表植被被啃食,土壤被牦牛践踏压实,且地上部分植物群落结构遭受到严重破坏的区域设置牦牛放牧区(YT);在地表植物和土壤草根层受到藏香猪翻拱扰动破坏,表层土壤裸露,具有典型猪拱斑块的区域藏香猪放养区(PA)。在对照区、牦牛放牧区和藏香猪放养区分别设置10m×10m的样地,沿样地的对角线分别选取3个1m×1m的小样方。每个小样方以“s”形采样,用直径5cm的土壤钻取土柱0—10cm的土壤,每5个小样方混合为一个样品,每个处理有3个重复样品。剔除土壤中可见的石块和动植物残体后,土样用无菌自封袋保存于液氮中,带回实验室过2mm筛后一部分保存于80℃超低温冰箱,用于分子生物学分析;另一部分土样自然风干,用于测定土壤基本化学性质。
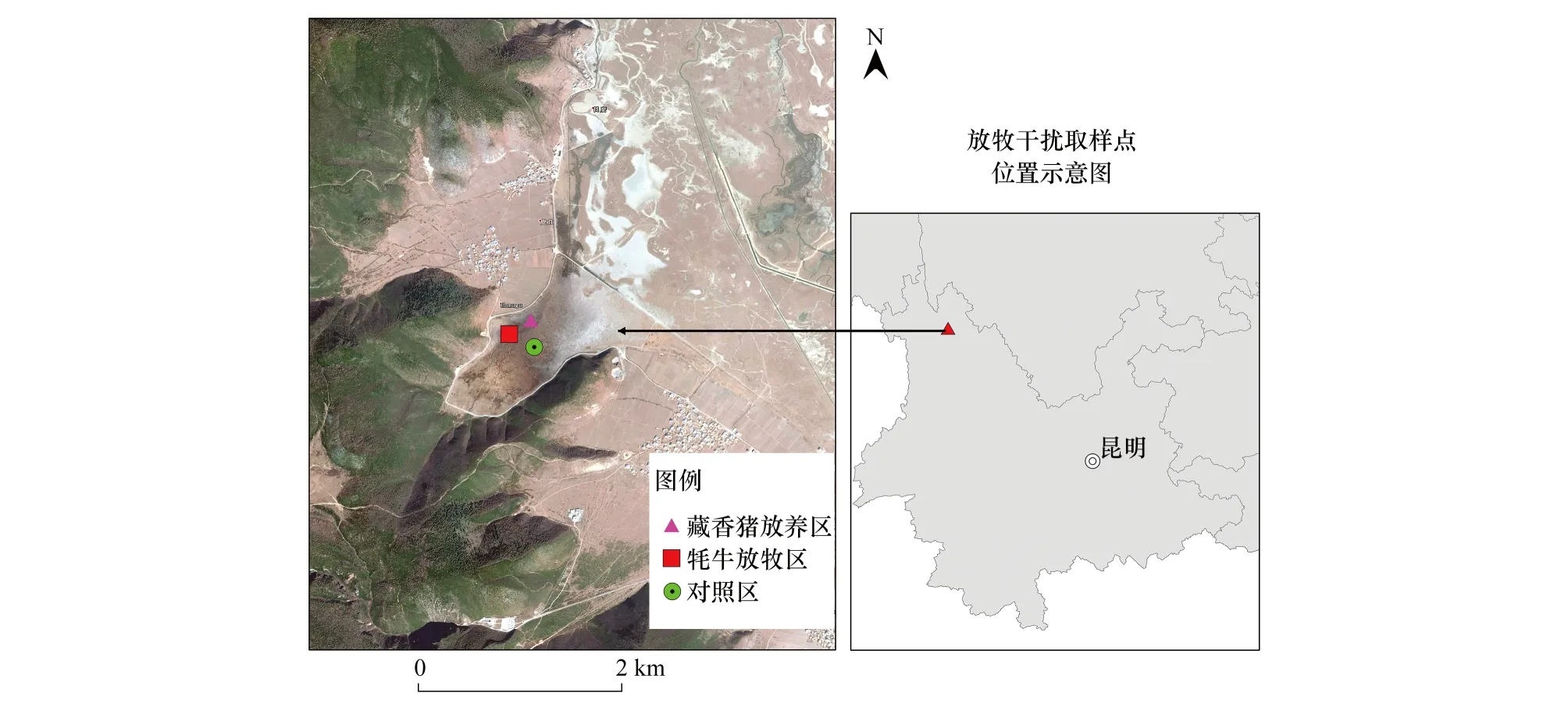
图1 实验样地Fig.1 Experimental plots
1.3 土壤基本理化性质和潜在硝化势的测定
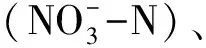
1.4 土壤DNA提取
土壤样品的总DNA 提取采用Power Soil DNA Isolation Kit 试剂盒(MOBIO公司,美国)试剂盒,用0.5g 新鲜土壤完成基因组DNA抽提后,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA。进一步进行AOA 和AOB 的PCR 扩增。
1.5 氨氧化微生物的测定
AOA 的扩增引物设计为Arch-amoA-F (5′-STAATGGTCTGGCTTAGACG- 3′)/Arch-amoA-R (5′-GCGGCCATCCATCTGTATGT- 3′)[30]和AOB的扩增引物为amoA- 1F (5′-GGGGTTTCTACTGGTGGT- 3′)/amoA- 2R (5′-CCCCTCKGSAAAGCCTTCTTC- 3′)[31]。所有PCR反应均采用TransStart Fastpfu DNA Polymerase,20μL反应体系:4μL5*FastPfu 缓冲液,2μL 2.5mmol/L dNTPs,0.8μL引物(5μmol/L),0.4μLFastPfu聚合酶;10ng DNA模板以及约10ng DNA模板,其余的用灭菌水补足。AOB和AOA-amoA基因PCR 反应条件如下:95℃变性5min,然后27个循环(95℃变性30s,在55℃30s,在72℃延伸1min)。最后72℃延伸10s。全部样本按照正式实验条件进行,每个样本3个重复,将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳回收产物,采用AxyPrepDNA 凝胶回收试剂盒(AXYGEN公司)进行纯化。并使用用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行检测定量,之后按照每个样本的测序量要求,进行相应比例的混合。纯化的PCR产物使用NEBNext®UltraTMDNA文库制备试剂盒(Illumina,NEB,USA)生成测序文库,最后在Illumina MiSeq平台上进行高通量测序(凌恩,中国上海)。
原始测序序列使用 Trimmomatic 软件质控,使用 FLASH 软件进行拼接:使用UPARSE软件(version 7.1 http://drive5.com/uparse/),根据97%的相似度对序列进行操作分类单元(Operational Taxonomic Units,OTU)聚类,并在聚类的过程中去除单序列和嵌合体。利用RDPclassifier(http://rdp.cme.msu.edu/) 对每条序列进行物种分类注释,比对Silva 数据库(SSU123),设置比对阈值为70%。应用软件Mothur(version v.1.30.1),计算Chao1和ACE指数,用来估计微生物丰富度,并使用Shannon和Simpson指数来估计微生物多样性。所有这些指数均使用QIIME计算。使用Weighted unifrac主坐标分析(PCoA)以分析微生物群落β多样性的差异(R.3.3.2)。
1.6 统计分析
使用SPSS 软件进行统计分析,以P<0.05为统计差异。用单因素方差分析(ANOVA)方法,检验处理之间土壤理化性质和氨氧化微生物功能基因多样性的差异显著性。Spearman等级相关分析用来评估土壤理化性质,PNR与土壤功能微生物的α多样性(Chao1、ACE、Shannon和Simpson)的相关性。采用R.3.3.2软件对 AOA和AOB amoA基因序列OTU 进行维恩(Venn) 分析。利用Canoco(4.5)多元统计模型分析样本空间差异特征,利用蒙特卡罗置换检验分析环境因子对氨氧化微生物群落影响显著水平,用典型对应分析(Canonical correspondence analysis, CCA),找出对环境变化的敏感微生物。
2 结果与分析
2.1 不同放牧对土壤理化性质和硝化潜势的影响
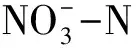
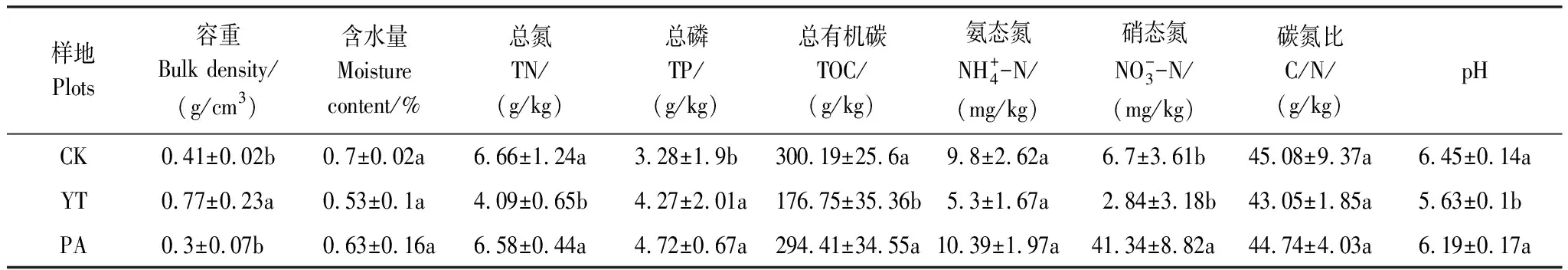
表1 不同放牧对泥炭沼泽土壤理化性质的影响
列内不同的小写字母表示显著性水平P<0.05;表中数据为平均值±标准差(n=3); CK:对照样地Control;YT:牦牛放牧区Yak-grazing; PA: 藏香猪放养区Pig -grazing;TN: Total nigrogen; TP: Total phosphorous;TOC: Total organic carbon
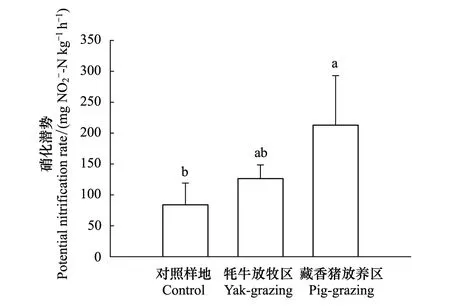
图2 不同放牧对沼泽湿地土壤硝化潜势的影响 Fig.2 The influence of different grazing on soil potential nitrification rates of peatland CK:对照样地Control;YT:牦牛放牧区Yak-grazing;PA: 藏香猪放养区Pig-grazing
2.2 不同放牧对土壤氨氧化微生物多样性的影响
2.2.1α多样性
总体上,平均每一个样本的AOA和AOB 的测序深度分别为34306、39180条序列。AOA及AOB的稀释曲线都趋向于平坦的状态,说明测序是合理的(图3)。
对照区、牦牛放牧区和藏香猪放养区的土壤均基于97%的相似度下,每个样本的AOA和AOB文库覆盖度都达到99%(表2)。AOA的OTU数目、Chao1和ACE指数均表现为CK>YT> PA,PA与CK和YT差异均显著,而CK和YT差异不显著。AOA的Shannon指数表现为YT>CK>PA,而AOA的Simpson指数表现为PA> YT>CK(P>0.05)。AOB的OTU数目、Chao1和ACE指数均表现为CK> PA>YT(P<0.05),AOB的Shannon指数表现为CK> PA>YT,AOB的Simpson指数表现为YT高于 PA和CK(P>0.05)。表明牦牛放牧和藏香猪放养降低土壤AOA和AOB 的丰富度,藏香猪放养对AOA丰富度的影响更显著,牦牛放牧对AOB丰富度的影响更显著。牦牛放牧降低了AOB的多样性。藏香猪放养降低了AOA和AOB的多样性。
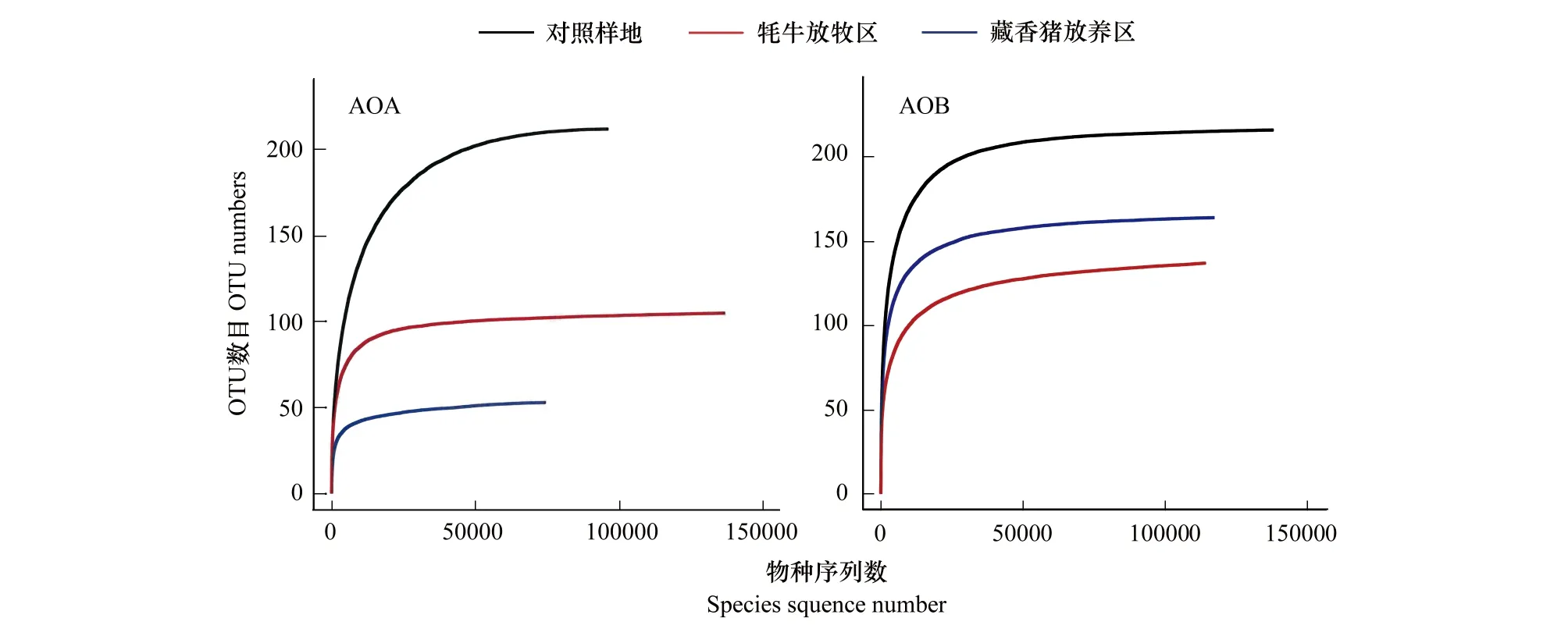
图3 基于OTU 数目97%相似度的AOA和AOB 的稀释曲线Fig.3 Rarefaction curve of the OTU number at 97% similarity for AOA (a) and AOBOTU:操作分类单元,Operational Taxonomic Units;AOA: 氨氧化古菌,ammonia-oxidizing archaea;AOB: 氨氧化细, ammonia-oxidizing bacteria
表2 不同放牧对沼泽湿地土壤AOA和AOB的amoA基因多样性指数的影响
Table 2 The α-diversity indices of AOAamoAandAOBamoAof peat swamp soil with influence of different grazing
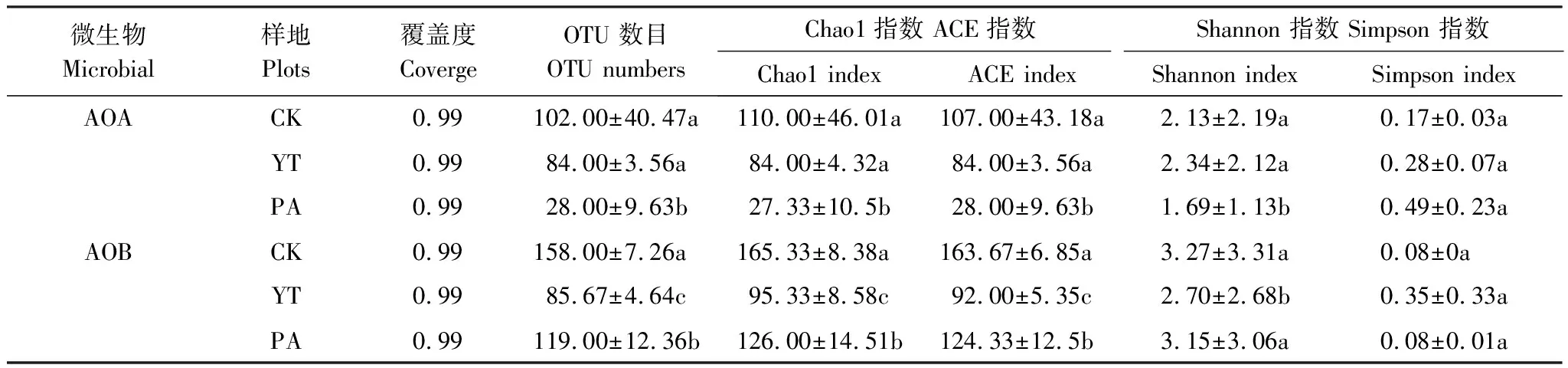
微生物Microbial样地Plots覆盖度CovergeOTU 数目OTU numbersChao1指数 ACE指数Shannon 指数 Simpson 指数Chao1 indexACE indexShannon indexSimpson indexAOACK0.99102.00±40.47a110.00±46.01a107.00±43.18a2.13±2.19a0.17±0.03aYT0.9984.00±3.56a84.00±4.32a84.00±3.56a2.34±2.12a0.28±0.07aPA0.9928.00±9.63b27.33±10.5b28.00±9.63b1.69±1.13b0.49±0.23aAOBCK0.99158.00±7.26a165.33±8.38a163.67±6.85a3.27±3.31a0.08±0aYT0.9985.67±4.64c95.33±8.58c92.00±5.35c2.70±2.68b0.35±0.33aPA0.99119.00±12.36b126.00±14.51b124.33±12.5b3.15±3.06a0.08±0.01a
同一列内不同的小写字母表示显著性水平P<0.05;表中数据为平均值±标准差(n=3); AOA:氨氧化古菌,ammonia-oxidizing archaea; AOB:氨氧化细菌,ammonia-oxidizing bacteria;OTU:操作分类单元, Operational Taxonomic Units;ACE:基于盖度的估计量,abundance-based coverage estimator
2.2.2β多样性指数
基于weighted unifrac PCoA的分析,AOA的PC1和PC2分别为53.01%和32.19%(图4)。CK和YT组间的样本在PC1的方向上区分开,而CK与PA土壤组间的样本在PC1和PC2方向上都区分开。排序结果较好的反映了土壤微生物群落在对照区、牦牛放牧区和藏香猪放养区的整体状况,且区分度高,表明牦牛放牧和藏香猪放养均对土壤的AOA的β多样性的影响比较显著。AOB的PC1和PC2分别为62.40%和19.82%。CK在PC1的方向与YT组间的样本区分开,但与PA没有区分开(图4)。表明牦牛放牧对土壤的AOB的β多样性的影响比较显著,而藏香猪放养对AOB的β多样性的影响相对比较小。
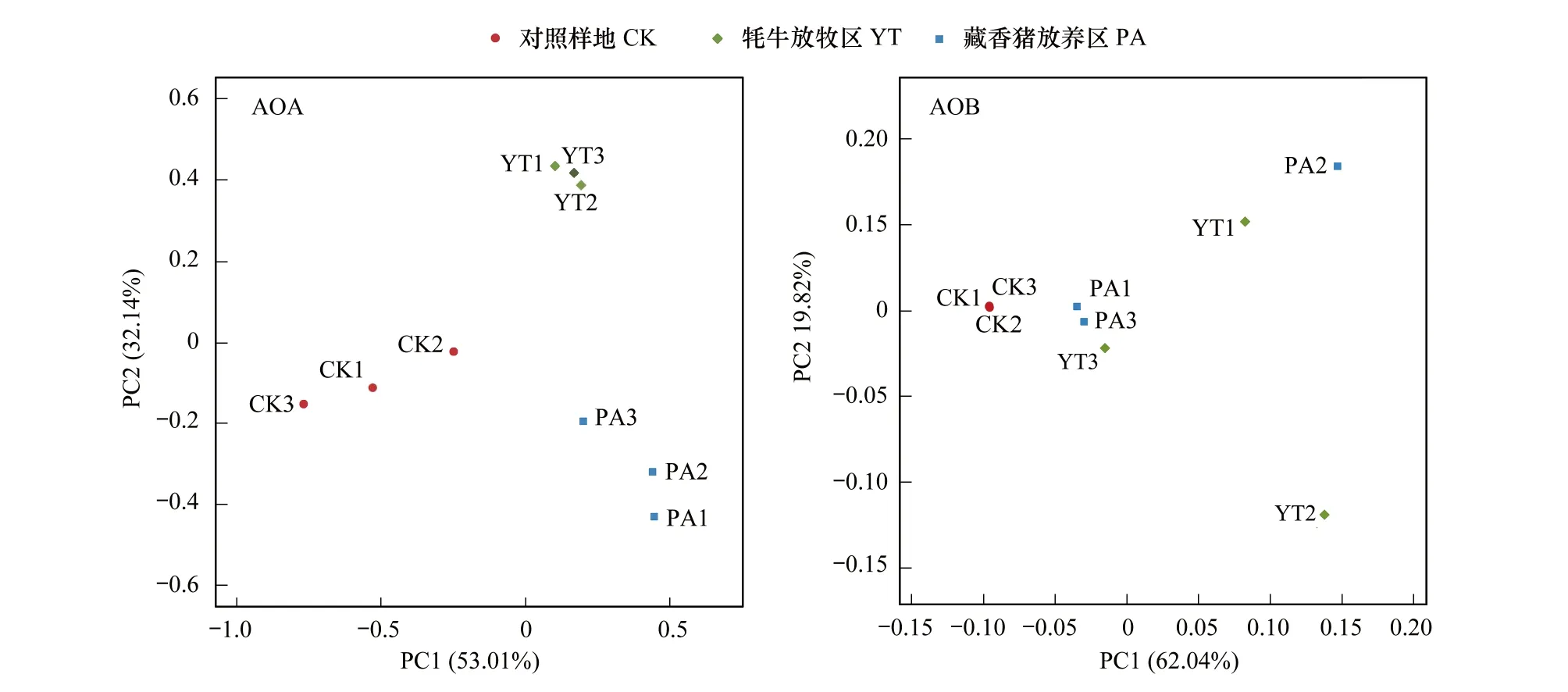
图4 不同放牧对泥炭沼泽土壤AOA及AOB 的β多样性分析Fig.4 The β diversities of AOA and AOBof peat swamp soil with influence of different grazing
2.3 不同放牧对土壤氨氧化微生物群落结构的影响
在AOA-amoA基因的门分类水平上,泉古菌门(Crenarchaeota)在对照区土壤中相对比重最高,约占62.8%,在牦牛放牧区和藏香猪放养区土壤中分别占6.5%和2.7% (图5)。在AOA-amoA基因的属分类水平上,对照区土壤中泉古菌门(Crenarchaeota_norank)的属占62.8%,但在牦牛放牧和藏香猪放养区土壤中分别为6.5%和2.7%。在AOB-amoA基因的门分类水平上,变形菌门(Proteobacteria)在对照区土壤中约占99.9%,在牦牛放牧区和藏香猪放养区土壤中分别占97.6%和99.0%。在AOB-amoA基因的属分类水平上,亚硝化螺菌属(Nitrosospira)在对照区土壤中约占99.9%,在牦牛放牧区土壤中和藏香猪放养区占96.1%和98.0%。可见,牦牛放牧和藏香猪放养均对土壤AOA群落组成影响比较显著,对土壤AOB群落组成的影响比较小。
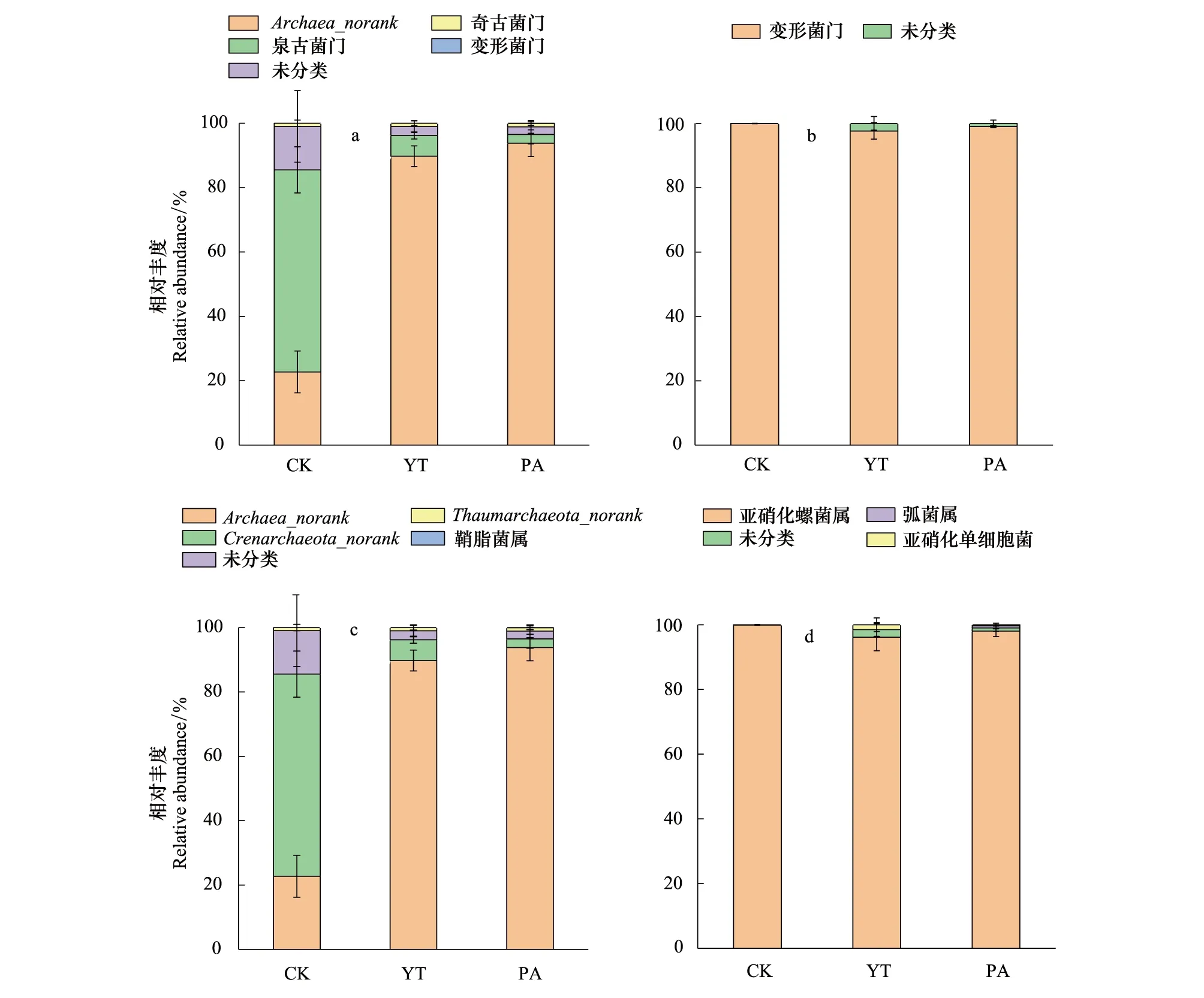
图5 不同放牧对沼泽湿地土壤AOA门(a),AOB门(b),AOA属(c)及AOB属(d)的相对丰度Fig.5 Relative abundance of AOA phyla (a), AOB phyla (b), AOA genera (c) and AOB genera (d) of peat swamp soil with influence of different grazing
不同放牧处理中AOA-amoA和AOB-amoA基因序列OTU的Venn分析(图6)。不同放牧影响下AOA-amoA基因共获得259个AOA-amoA基因序列OTU,不同放牧共有OTU数目为10个,占总OTU数目的3.8%。特有的OTU数目表现为CK>YT>PA,不同放牧影响下AOB-amoA基因序列OTU共获得287个,不同放牧共有OTU为82个,占总OTU的28.5%。其中,特有的OTU表现为CK>YT>PA。总体上,不同放牧对AOA-amoA基因的OTU数量和分布影响比AOB-amoA基因更显著,藏猪放养比牦牛放牧区的特有的OTU数目更少。
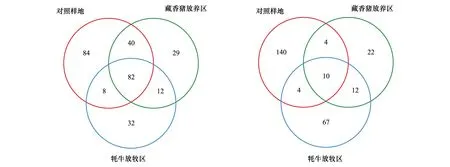
图6 不同放牧AOA和AOB amoA 基因序列OTU 的Venn图Fig.6 Venn diagram of AOA and AOB amoA genes based on OTU in different grazing
2.4 土壤理化性质、PNR 与氨氧化微生物群落的关系
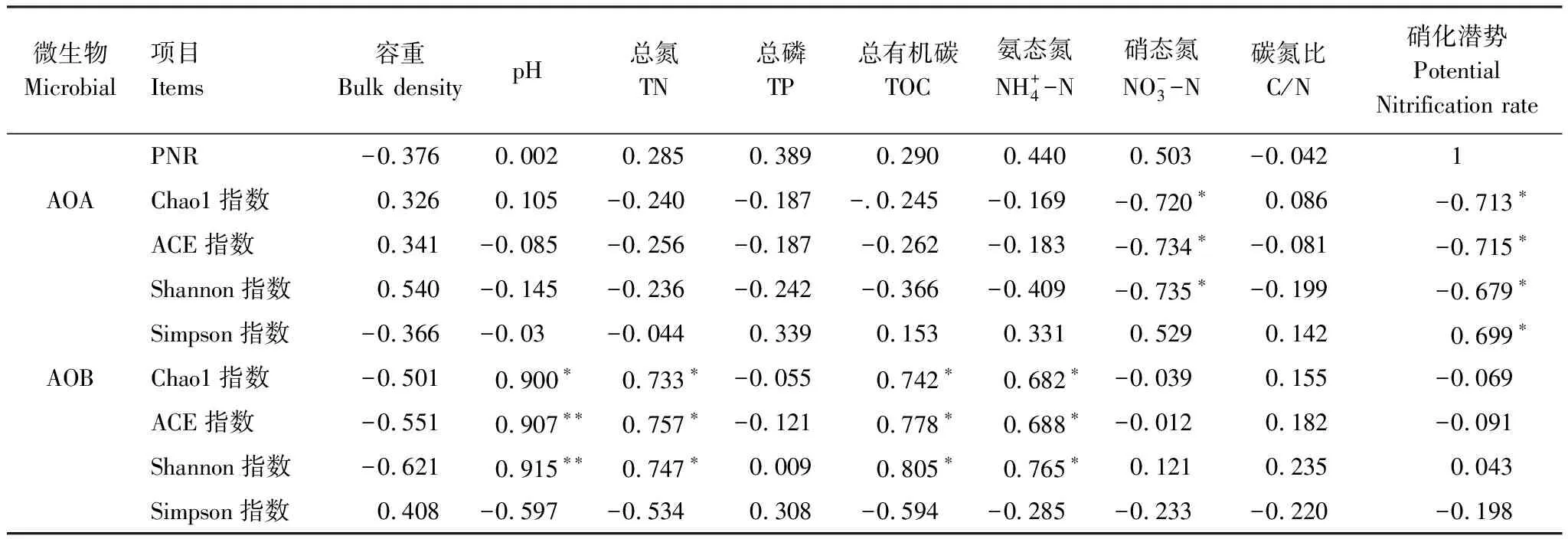
表3 土壤理化性质,PNR及AOA与AOB 多样性的关系
*相关系数的显著水平P<0.05 **相关系数显著性P<0.01. ACE:基于盖度的估计量,abundance-based coverage estimator

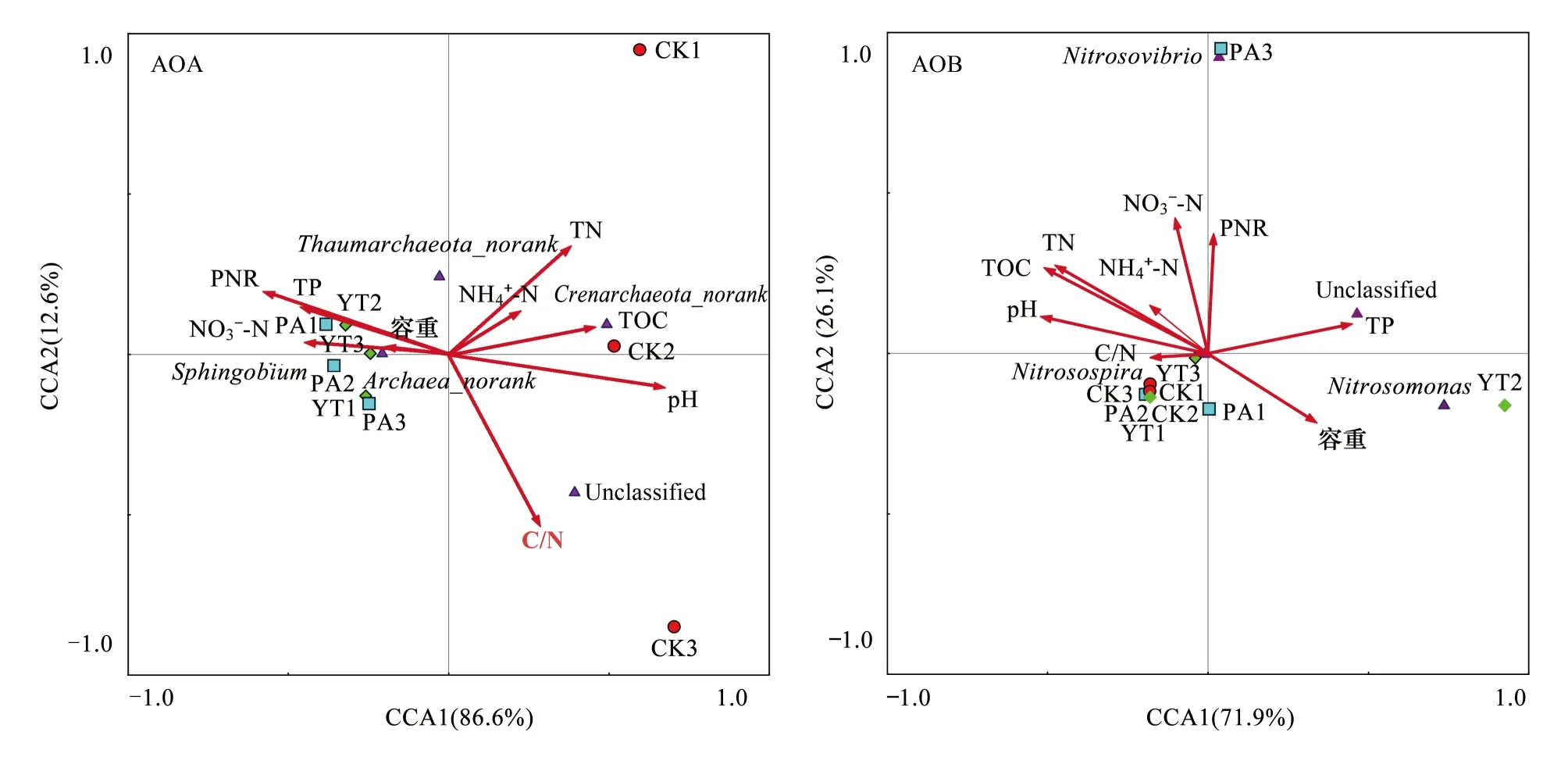
图7 土壤AOA-amoA和AOB-amoA基因群落组成和环境因子的典型对应分析Fig.7 Canonical correspondence analysis of relationship between the environmental factors and community structures of AOA-amoA gene and AOB-amoA gene Archace_norank:属于古菌门;Crenarchaeota_norank:属于泉古菌门;unclassfied: 未分类的菌;Thaumarchaeota_norank:属于奇古菌门;sphingobium:鞘脂菌属;Nitrosospira:亚硝化螺菌属;Nitrosomonas:亚硝化单细胞菌;Nitrosovibrio:弧菌属; pH: 土壤pH; TN:总氮Total nitrogen content;TP:总磷Total phosphorus;TOC:总有机碳Total organic 铵态氮Ammonium 硝态氮Nitrate content;PNR:硝化潜势Potential nitrification rate
3 讨论
3.1 放牧对土壤理化性质和PNR的影响
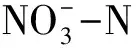
3.2 放牧对土壤氨氧化微生物多样性的影响
放牧对微生物多样性的影响依赖于环境条件、土壤特性、植凋落物质量和数量、放牧强度和动物类型的综合作用[39]。藏香猪放养和牦牛放牧均降低土壤AOA的多样性,但牦牛放牧对AOA的多样性的影响不显著。研究表明,在放牧最严重的地区,AOA的多样性最低[40];适量的尿素可增加AOA的多样性,而长期施加尿素降低AOA的多样性[41],动物尿液中含有90%的尿素[42]。牦牛放牧和藏香猪放养过程中由于干扰强度和排泄物的输入不同会对土壤AOA多样性产生不同的影响。牦牛放牧显著降低土壤AOB的多样性(P<0.05),但藏香猪放养对土壤AOB的多样性的影响不显著。这与研究发现在放牧强度最严重地区,AOB的多样性最高不一致[40],原因是在本研究中受放牧影响的土壤pH,TOC和总氮含量没有出现向高水平转化,而是比未受干扰的对照区的土壤更低。也有研究表明,放牧降低了土壤微生物的多样性[33]。牦牛放牧和藏香猪放养均降低土壤AOA和AOB的丰富度,藏香猪放养和牦牛放牧分别对AOA和AOB丰富度的影响更显著。这主要是不同的放牧方式对土壤养分的影响不同,微生物多样性的变化还会受到其他综合环境因素的影响。
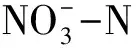
AOA与AOB的群落组成随环境因素变化而发生改变[43]。牦牛放牧和藏香猪放养活动改变了氨氧化微生物的群落组成,原因是这两种放牧活动导致土壤环境因子发生了改变。
3.3 放牧对氨氧化微生物群落结构的影响
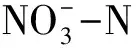
4 结论
(2)牦牛放牧降低土壤AOA的丰富度和AOB的α多样性,改变土壤AOA和AOB的β多样性;藏香猪放养降低土壤AOB的丰富度和AOA的α多样性,二者均降低Crenarchaeota的相对丰度。藏香猪放养对土壤α多样性和群落结构的影响比牦牛放牧更显著。
