人工智能在识别肿瘤切片中DNA错配修复的应用
2020-05-29朱维健
朱维健
【摘 要】:目的:通过查询文献资料来分析当前人工智能在病理学诊断DNA错配修复方面的应用与进展 方法:检索文献资料和信息选定了深度残差学习算法,研究者从癌症基因组图谱中获取了病理切片数据,包括315个胃癌样本,360个结直肠癌样本和378个冷冻结直肠癌样本。样本进一步经过自动肿瘤检测仪处理,分别生成了100570、93408、60894个颜色归一化色块,作为深度学习的“原料”,随后分别对DNA错配修复和MSS(微卫星稳定)样本进行DNA错配修复评分。结果:直接从病理切片识别DNA错配修复,利用深度残差学习算法识别结直肠癌中DNA错配修复准确率达84%。结论:这意味着我们或许可以越过免疫组化或基因检测,直接利用人工智能机器提取肿瘤组织特征,对病理切片进行分析,更快捷地识别出那些适合使用免疫治疗的患者。考虑到DNA错配修复在各癌种中的普遍性,后续人工智能的应用可能潜力无限。
【关键词】:人工智能 ;DNA错配修复 ;肿瘤切片
Abstract: Objective : Application and Progress of Artificial Intelligence in Pathological Diagnosis DNA Mismatch Repair by Searching Literature. Methods : Searching literature and information to select a deep residual learning algorithm, the researchers obtained pathological section data from the cancer genome atlas, including 315 gastric cancer samples ,360 colorectal cancer samples and 378 frozen colorectal cancer samples. the samples were further processed by an automatic tumor detector to generate 100570,93408,60894 color-normalized color blocks as “raw materials” for deep learning, followed by DNA mismatch repair and MSS( microsatellite stable) samples with a DNA mismatch repair score, respectively. Results : Detection DNA mismatch repair directly from pathological sections using deep residual learning algorithm to identify colorectal cancer DNA mismatch repair accuracy reached 84%. Conclusion : This means that we may be able to go beyond immunohistochemistry or genetic testing, directly using artificial intelligence machines to extract tumor tissue features, analyze pathological sections, and more quickly identify patients suitable for immunotherapy. considering the universality of DNA mismatch repair in various cancer species, subsequent ai applications may have unlimited potential.
keywords: artificial intelligence ; DNA mismatch repai ; Tumor sections
1 資料与方法
1.1 一般资料:
通过查询近年来文献资料包括《中华医学遗传杂志》、《中华医学杂志》、《肿瘤研究与临床》、《癌症》《吉林医学》、《现代预防医学》、《中国医学论坛报》等核心期刊和互联网医学信息追踪关于肿瘤DNA错配修复和不稳定的最新进展。中美两国的科研人员在国际期刊《美国科学院院报》(PNAS)发表了DNA错配修复的最新研究成果,利用精确地单分子研究技术报告了DNA错配修复研究的突破性发现。来自北卡大学教堂山分校的杜克医学中心和北卡州立大学的研究人员发现DNA错配位置处形成的钳状复合物沿着DNA链移动如何修复错配碱基。 查阅德国亚琛工业大学、美国德克萨斯大学的研究报告,利用人工智能从病理切片中识别DNA错配修复。研究者从癌基因图谱(TCGA)中获取病理切片数据进行三次外部验证,数据分别来自DACHS研究的378例样本和来自日本KCCH研究队列(185列)样本和UCEC研究队列(327)例样本,以及基因检测和免疫组化检测结果等资料。
1.2 方法:检索文献资料和信息选定了深度残差学习算法,研究者从癌症基因组图谱中获取了病理切片数据,包括315个胃癌样本,360个结直肠癌样本和378个冷冻结直肠癌样本。样本进一步经过自动肿瘤检测仪处理,分别生成了100570、93408、60894个颜色归一化色块,作为深度学习的“原料”,随后分别对MSI(DNA错配修复)和MSS样本进行MSI评分。
2 结果
直接从病理HE切片中识别DNA错配修复(MSI)利用深度残差学习算法,识别直肠癌DNA错配修复(MSI)准确率达到84%。考虑到DNA错配修复(MSI)在各种癌症中的普遍性,人工智能的后续应用潜力无限。
3 结论
这意味着我们或许可以跳过免疫组化或基因检测,直接利用人工智能机器提取肿瘤组织特征,对病理切片进行分析,更快捷地识别出那些适合使用免疫治疗的患者。
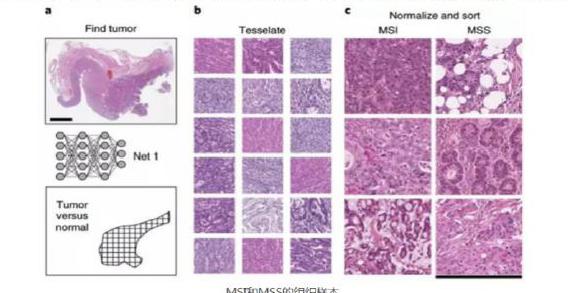
研究者在一个小样本数据集中测试了5种不同的算法,最终名为Resnet18的深度残差学习(deep residual learning)算法脱颖而出,曲线下面积(AUC)竟然高于0.99。深度残差学习采用跳跃链接方法,对有效数据保留更好,同时结构更加简单,训练速度也更快。选定了算法,研究者从癌症基因组图谱(TCGA)中获取了病理切片数据,包括315个福尔马林固定石蜡包埋(FFPE)胃癌样本(TCGA-STAD),360个FFPE结直肠癌样本(TCGA-CRC-DX)和378个冷冻结直肠癌样本(TCGA-CRC-KR)。这些样本又进一步经过自动肿瘤检测仪处理,分别生成了100570、93408、60894个颜色归一化色块,作为深度学习的“原料”,随后分别对(DNA错配修复)MSI和MSS(微卫星稳定)样本进行(DNA错配修复)MSI评分。
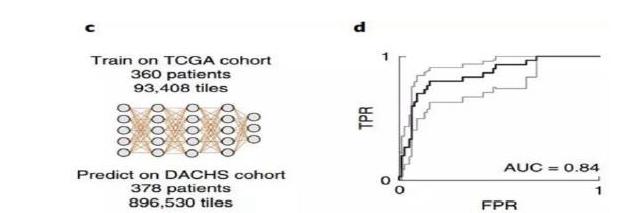
测试结果显示,针对FFPE结直肠癌样本,(DNA错配修复)MSI图像的MSI评分为0.61,MSS图像的(DNA错配修复)MSI评分为0.29;对于冷冻的结直肠癌样本,二者数据分别为0.5和0.22。可见该算法并不受样本处理方法的限制,都有足够的区分度。再看准确性,对胃癌、FFPE结直肠癌和冷冻结直肠癌三组样本,AUC则分别达到0.81、0.84、0.77。接下来,研究者对算法进行了三次外部验证。第一组样本数据来自DACHS研究,包括378份样本,使用FFPE结直肠癌样本训练出的模型,AUC达到了0.84。研究者还尝试了用冷冻样本训练出的模型和胃癌样本训练出的模型,效果均略有不足,说明模型准确性受到组织类型和样本处理方法的影响。
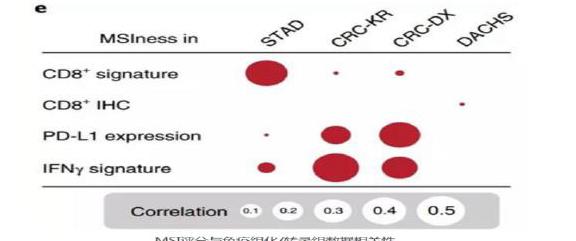
第二组数据来自日本横滨的KCCH队列。选择它的原因主要是亚裔的胃癌组织学和临床过程与非亚裔差别很大。来自TCGA的胃癌样本中有80%是非亚裔,100%亚裔的KCCH队列(n=185)验证结果AUC为0.69。考虑到(DNA错配修复)MSI是一种泛癌种的生物标志,研究者选了第三组数据,来自UCEC队列的327个子宫内膜癌样本。经训练的模型在剩余样本中AUC为0.75。由此可见,这种算法模型可能受癌种、样本类型、患者人种的影响,如果要拓展使用,可能需要更大的数据集来训练。研究者还计算了该检测需要的组织样本大小,结果显示下限约为100×256μm见方。这个大小的组织通过活检就可以取得,可以说是非常方便了。最后,研究者对比了模型计算的(DNA错配修复)MSI评分与转录组和免疫组化数据的差异。相关区域胃癌的淋巴细胞基因特征、结直肠癌的PD-L1和IFN-γ表达、肿瘤低分化、免疫细胞浸润等特征都是符合组织病理学理论的。
癌症的免疫治疗开拓了一片新天地,不过至今寻找能从免疫治疗获益的患者仍旧是关键难题。正在召开的ASCO也把寻找新的免疫治疗生物标志物列为2020年研究首要任务。而现有的生物标志物檢测也远还没有惠及到大多数患者。人工智能可能是解决这一问题的最好方案。低成本、无需额外的实验过程、样本方便获取……相信经过更大数据集的训练和前瞻性验证,它能够真正把免疫治疗带向更多人。
参考文献
Rahman, R., Asombang, A. W. & Ibdah, J. A. World J. Gastroenterol. 20, 4483–4490 (2014).
Le, D. T. et al. N. Engl. J. Med. 372, 2509–2520 (2015).
He, K., Zhang, X., Ren, S. & Sun, J. In Proc. IEEE Conference on Computer Vision and Pattern Recognition 770–778 (2016).
Bonneville, R. et al. JCO Precis. Oncol. 2017, 1–15 (2017).
Darvin, P., Toor, S. M., Sasidharan Nair, V. & Elkord, E. Exp. Mol. Med. 50, 165 (2018).
Coudray, N. et al. Nat. Med. 24, 1559–1567 (2018).
Schaumberg, A. J., Rubin, M. A. & Fuchs, T. J. Preprint at
Chang, P. et al. AJNR Am. J. Neuroradiol. 39, 1201–1207 (2018).
错配修复/微卫星不稳定性对晚期结直肠癌化疗敏感性及预后影响的研究[D]. 扬州大学,2018.
中华实验外科杂志,2010,(8).doi:10.3760/cma.j.issn.1001-9030.2010.08.007.
