红外光谱数据融合对美味牛肝菌产地鉴别
2020-05-07胡翼然李杰庆刘鸿高范茂攀王元忠
胡翼然,李杰庆,刘鸿高,范茂攀*,王元忠
1. 云南农业大学资源与环境学院,云南 昆明 650201 2. 云南农业大学农学与生物技术学院,云南 昆明 650201 3. 云南省农业科学院药用植物研究所,云南 昆明 650200
引 言
近年来由于环境污染造成的食品安全问题频繁发生,食品质量安全引起全国高度重视[1]。 地理标志产品来源于特定地区,有特定的加工方法,解决了消费者和生产者之间信息不对等的问题,其产品质量安全受到消费者的信任[2]。 作为牛肝菌之王的美味牛肝菌(BoletusedulisBull.: Fr.)富含维生素、膳食纤维、矿质元素、人体必需的氨基酸、不饱和脂肪酸等,是高蛋白、低脂、低钠的健康食品同时还具有抗氧化、抗疲劳、抗突变、增强免疫力等药用活性[3]。 美味牛肝菌品质受生长环境(温度、降水等)影响巨大[4],导致不同地区之间的美味牛肝菌营养成分有显著差异[5-6],进而导致食药用价值的差异。 我国云南省地形复杂,晴雨交错的气候生态环境特征有利于美味牛肝菌的生长,美味牛肝菌产量丰富,约占全国出口量的70%。 根据各地气候、地理、文化等特点,可以建立相应的野生食用菌地理标志,形成独具特色的品牌。 为此,现阶段急需一种高效、廉价的美味牛肝菌产地鉴别技术,以确定美味牛肝菌的真实产地,防止假冒伪劣产品进入市场,保护消费者身体健康和权益。
近红外和中红外光谱具有无损、快速、便捷、廉价等优点,广泛应用于食品质量控制中[7-8]。 但利用单一来源的光谱信息无法完全表征样品中的化学信息,产地鉴别效果弱。 而数据融合可以使不同来源的光谱信息相互补充,从而增强模型性能,实现样品的精确鉴别。 近年来融合近红外和中红外光谱在食品质量控制方面有许多成果,如Li等[9]融合近红外与中红外的光谱数据,使用偏最小二乘判别(partial least squares discriminant analysis, PLS-DA)模型进行低级融合、中级融合、高级融合,成功鉴别3种植物油掺假6个不同来源橄榄油。 Li等[10]融合近红外与中红外的光谱数据,使用随机森林进行低级融合、中级融合、高级融合,成功鉴别三七地理位置。
现阶段,野生牛肝菌的产地鉴别以中红外光谱为主,基于近红外光谱对野生牛肝菌产地鉴别未见报道。 本研究的主要目的是探讨融合近红外光谱与中红外光谱鉴别不同产地美味牛肝菌的可行性,对4个单一信息矩阵: 近红外的菌柄(N-b)、近红外的菌盖(N-g)、中红外的菌柄(M-b)、中红外的菌盖(M-g),结合PLS-DA进行低级、中级、高级数据融合,选出美味牛肝菌产地鉴别最佳方法,为野生牛肝菌产地鉴别提供参考。
1 实验部分
1.1 材料
141份美味牛肝菌采自云南8个产地,分别为昆明、楚雄、玉溪、迪庆、大理、保山、文山、曲靖(表1)均由云南农业大学刘鸿高教授鉴定。 样品采集后去除土样等杂质,用纯净水清洗干净,置于50 ℃烘箱烘干至恒重,高速粉碎机(FW-100型)粉碎,过80目标准筛盘,分别储存于自封袋中,保存于避光处。
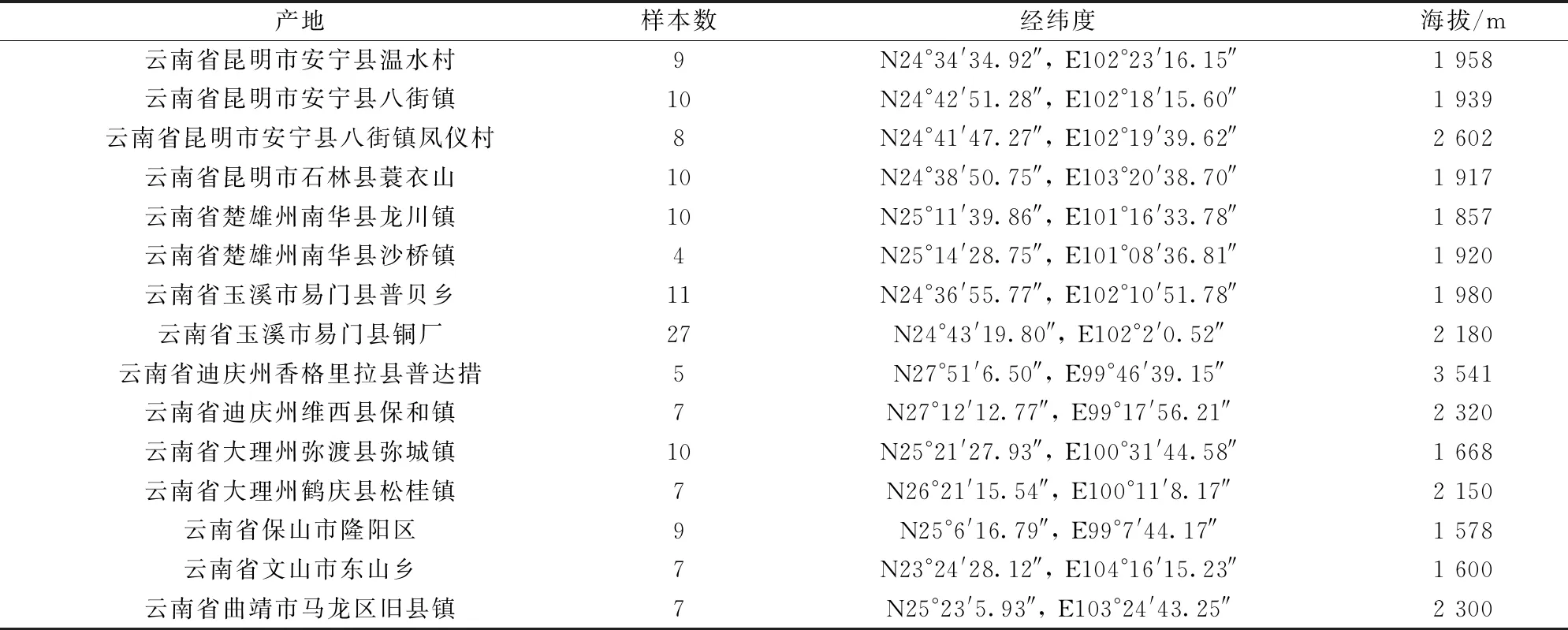
表1 美味牛肝菌产地信息
1.2 光谱信息采集
1.2.1 近红外光谱
近红外光谱是由Antaris Ⅱ型傅里叶变换近红外光谱仪(Thermo Fisher公司,USA)用漫反射显微镜采集。 称取20 g样品混合均匀后,置于玻璃器皿中压缩,扫描。 扫描波数范围10 000~4 000 cm-1,分辨率4 cm-1,信号扫描累计64次。 每个样本重复扫描3次,取平均光谱。
1.2.2 中红外光谱
中红外光谱是由Frontier型傅里叶变换红外光谱仪(Perkin Elmer 公司,USA)采集。 取(1.5±0.2)mg美味牛肝菌样品和(150±20)mg KBr 粉末在研钵中磨细混匀,再将细粉倒入磨具中压成薄片,扫描。 扫描波数范围4 000~400 cm-1,分辨率4 cm-1,信号扫描累计16次,每个样本重复扫描3次,取平均光谱。
1.3 偏最小二乘判别分析及评价
PLS-DA是在标准PLS回归算法的基础上建立的线性监督分类方法,从变量矩阵(x)中寻找与分类矩阵(y)有最大协方差的变量,其中y分为2类,y=1代表样本属于特定分类,y=0代表样品不属于特定分类,最终得出每个样品分类到每一类的概率。 该算法解释样本观测数目少且可以降低样本间多重共线性的影响。 使用Kennard-Stone算法划分数据集(141)的2/3为训练集(97)和1/3为预测集(44)以提高模型识别能力,避免随机选择的不可重复性。
根据交叉验证均方根误差(root mean square error of cross validation, RMSECV)和预测均方根误差(root mean square error of prediction, RMSEP)两个重要参数评价模型稳定性,RMSECV≥RMSEP且数值越小,表明模型稳定且预测能力越好。 综合训练集和预测集的正确率再加上非错误率(non-error rate, NER)评价模型分类性能。 NER是灵敏度和特异性的平均值,NER越高模型分类性能越强。
1.4 数据融合
低级融合又名数据级融合,直接将多个数据矩阵串联得到一个新的数据矩阵,再建立判别模型; 中级融合又名特征级融合,将多个特征值数据矩阵串联得到一个新的数据矩阵,再建立判别模型。 潜在变量(latent variable, LV)是一种常见的特征提取方法,根据Q2第一次到达最大值时的因子数确定最优LV个数,最大程度的表征样品信息; 高级融合又名决策级融合,各数据源进行分析获得独立结果,再根据一定准则对结果进行融合,最终得到整体一致的决策。 本文基于“模糊集合论”,首先提取各原始数据的LV结合PLS-DA建模,把模糊现象(同一样品在不同光谱信息来源下有不同分类结果)通过最小值(Min)、最大值(Max)、平均值(Avg)和乘积(Prod)这4种运算符连接,再进行多数投票,表决出最终样品分类结果[11]。
2 结果与讨论
2.1 光谱分析


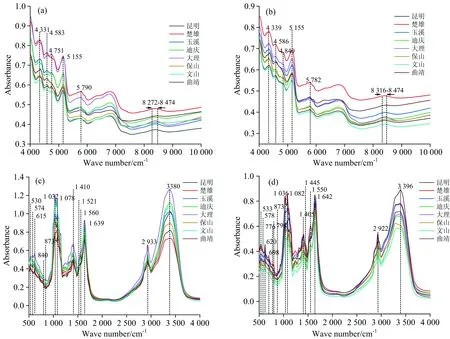
图1 美味牛肝菌不同产地的近红外和中红外平均光谱图
不同地区美味牛肝菌的光谱之间有相同趋势,且峰出现的位置也相近,代表不同产地间美味牛肝菌所含化学成分相似,但吸光度差异明显,代表不同产地之间化学成分含量不同。 中红外光谱的特征峰数量远多于近红外光谱的特征峰数量,且吸光度值也更大,表明中红外光谱能更好的表征美味牛肝菌化学信息。 从图1(a)和(c)比较(b)和(d)可以看出菌柄的吸光度高于菌盖的吸光度,代表菌柄积累的化学物质更多。 杨天伟等利用聚类分析发现美味牛肝菌的菌柄和菌盖之间有明显差异[14],从光谱图证明了这一点。
2.2 单一光谱分析
表2为所建模型的主要参数。 其中,N-b代表141×3 112(141个样本×3 112个变量)的数据矩阵,提取前10个潜在变量; 有第二高的训练集正确率为80.4%和第三高的预测集正确率为75%。 N-g代表141×3 112的数据矩阵,提取前6个LV,有最低的训练集正确率66%和最低的预测集正确率70.5%。 M-b代表141×1 867(141个样本×1 867个变量)的数据矩阵,提取前7个LV,有第三高的训练集正确率76.3%和第二高的预测集正确率79.6%。 M-g代表141×1 867的数据矩阵,提取前13个LV,有最高的训练集正确率91.8%和最高的预测集正确率88.6%。
表2中4个单一光谱(N-b,N-g,M-b,M-g)所建立的模型的预测集正确率在70.5%~88.6%之间。 预测效果优劣依次为M-g(88.6%),M-b(79.6%),N-b(75%),N-g(70.5%)。 中红外光谱的预测效果优于近红外光谱的预测效果,表明中红外光谱相对于近红外光谱有更好的预测能力。 其原因可能是近红外光谱的吸光度弱且谱带重叠严重,中红外光谱相对于近红外光谱有更高的灵敏度,能表征更多的样本化学信息。
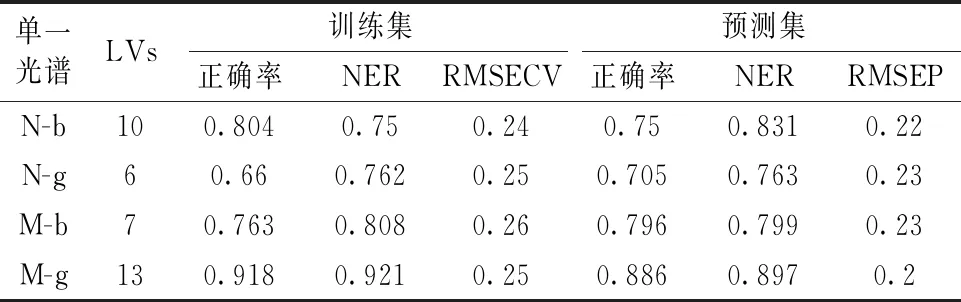
表2 单一光谱模型主要参数
2.3 数据融合分析
2.3.1 低级融合
将四个单一光谱矩阵((N-b),(N-g),(M-b)和(M-g))进行低级融合形成一个141×9 958(141个样本×9 958个变量)的矩阵,其中N-b提供3 112个变量,N-g提供3 112个变量,M-b提供1 867个变量,M-g提供1 867个变量。 如表3所示,变量数为9 958,训练集正确率为97.9%,RMSECV为0.238; 预测集正确率为93.2%,RMSEP为0.188。 相较于单一光谱,低级融合策略有更多的信息量,对产地的预测正确率更高。
2.3.2 中级融合
提取四个单一光谱((N-b),(N-g),(M-b),(M-g))的LV形成一个141×49(141个样本×49个变量)的矩阵,其中N-b提供13个LV,N-g提供15个LV,M-b提供8个LV,M-g提供13个LV。 如表3所示,变量数为49,训练集正确率为100%,RMSECV为0.193预测集正确率为100%,RMSEP为0.133。 相较于单一光谱和低级融合策略,中级融合策略效果更好。 低级融合不仅融合了有效信息,还融合了很多干扰信息。 而中级融合策略在提取特征值的过程中去除了无效信息,不仅降低运算成本,而且提高了产地预测正确率。
2.3.3 高级融合
提取四个单一光谱((N-b),(N-g),(M-b)和(M-g))的LV进行高级融合,如表3所示,预测集正确率为97.5%。 表4为高级数据融合统计表。 其中,总共141组样品中有55组样品需要进行高级融合,其中有2组分类错误(88,90),3组分类歧义(91,92,114),49组分类正确。 表为5部分样品高级融合结果表。 其中,37号样品被N-b,N-g错误分类为class1,M-b,M-g正确分类为class3,经高级融合后正确分类到class3,表明高级融合有很好的纠错效果,可以有效提高产地鉴别效果。 92号样品被N-b错误分类为class1,N-g,M-b,M-g正确分类为class7,经高级融合后分类为class1和class7,表明基于LV的高级融合策略受错误分类结果影响,混淆了最终决策结果。 高级融合策略相较于单一光谱和低级融合策略有更高的产地预测正确率,其原因可能是提取特征值的过程中去除了无效信息。 高级融合策略汲取了中级融合策略的优点,再利用“模糊集合论”进行决策,理论上高级融合策略应当取得比中级融合策略更好的产地鉴别效果,但在本研究过程中,有3.5%的模型受错误分类结果影响了最终决策结果,其原因可能是高级融合策略不擅长处理异常值。
2.4 数据分析
7个数据矩阵(N-b),(N-g),(M-b),(M-g),(Low-level),(Mid-level),(High-level)构建的模型预测正确率依次为88.6%,79.6%,75%,70.5%,93.2%,100%和97.5%。 三种数据融合策略模型预测集正确率均高于单一光谱模型预测集正确率,结果表明数据融合策略可以提高美味牛肝菌产地鉴别模型分类效果。 比较三种数据融合策略((Low-level),(Mid-level),(High-level)),基于特征值LV的中级融合策略在产地鉴别效果上优势最明显,用最少的变量数(49)取得最高的产地鉴别正确率(100%)。 其次是高级融合策略,其建立的PLS-DA模型产地鉴别正确率为97.5%,有少量异常数据无法鉴别。 最后是低级融合策略模型有最多的信息量(9958)但受干扰因素影响其产地鉴别正确率仅为93.2%。 与姚森等鉴别绒柄牛肝菌产地溯源研究结果相似[15]。
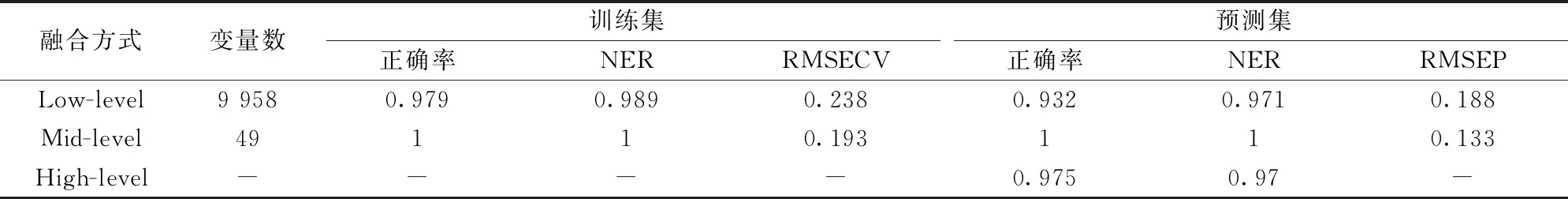
表3 数据融合主要参数
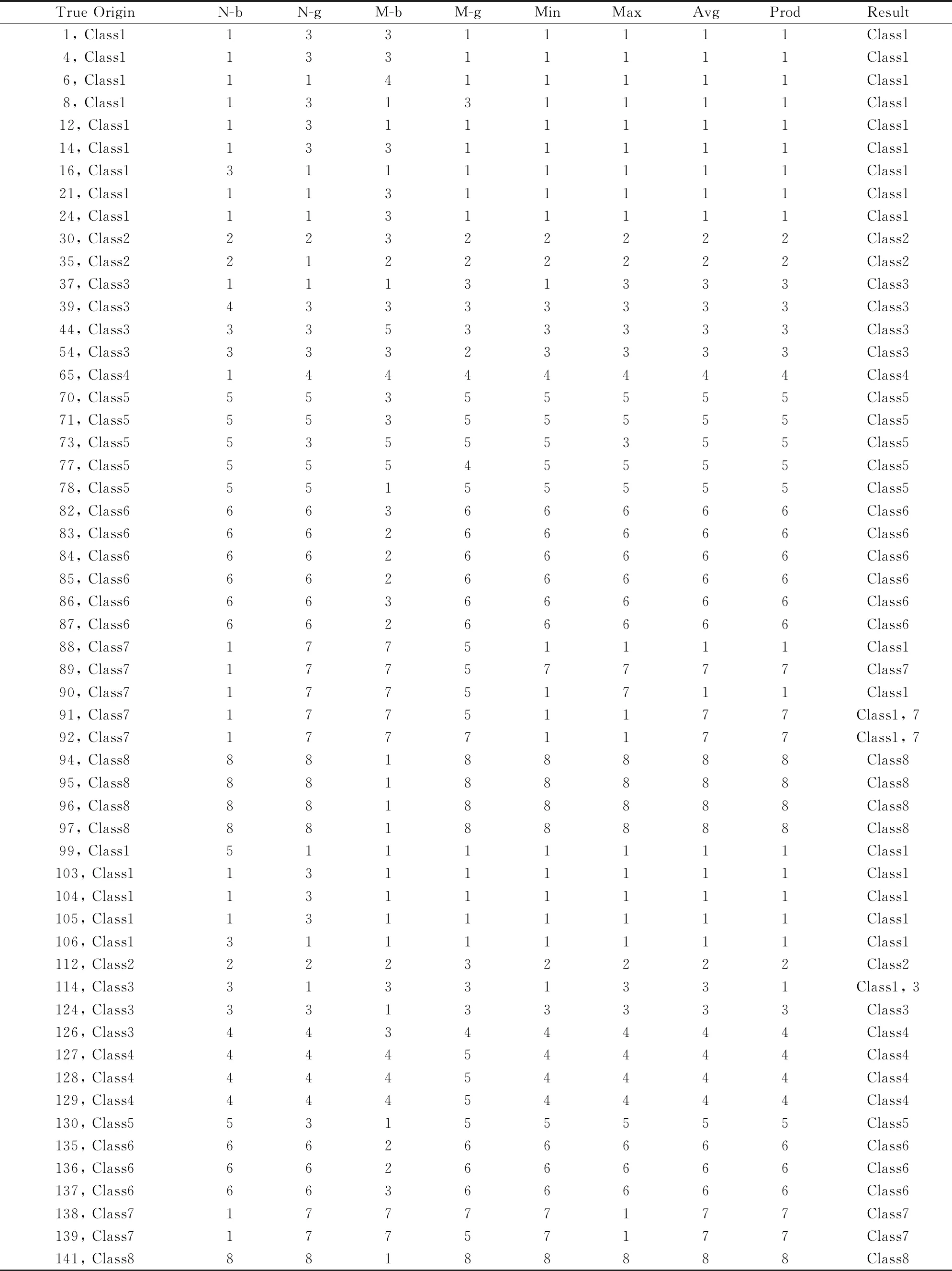
表4 高级数据融合结果统计
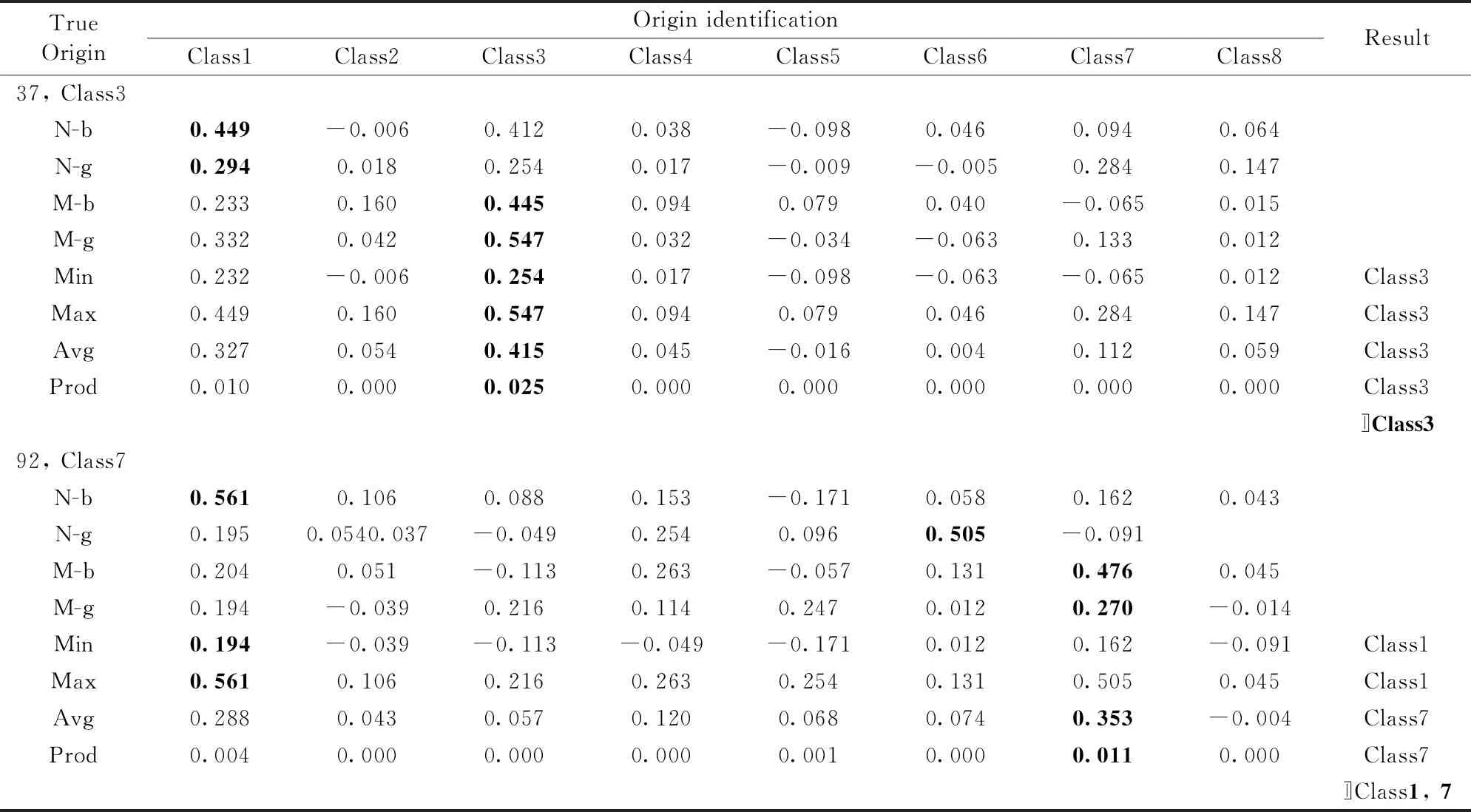
表5 部分样品高级融合结果
3 结 论
探讨了美味牛肝菌不同部位近红外光谱和中红外光谱及三种数据融合策略(低、中、高)对产地溯源的可行性。 结果表明: (1)近红外和中红外光谱均能鉴别美味牛肝菌产地。 (2)中红外光谱所建立的模型优于近红外光谱所建立的模型。 (3)三种融合策略均可提高美味牛肝菌的产地鉴定效果,产地鉴别效果优劣依次为中级融合、高级融合、低级融合、单一光谱。
通过融合近红外和中红外光谱结合PLS-DA进行基于特征值LV的中级融合策略,建立不同产地美味牛肝菌鉴别模型,有最少的变量数(49),最高的产地训练集正确率(100%),最高的产地预测集正确率(100%),最低的RMSEP(0.133),实现了美味牛肝菌产地的快速、准确鉴别,可以作为美味牛肝菌产地溯源的一种可靠方法。
猜你喜欢
杂志排行
光谱学与光谱分析的其它文章
- The Characterization on Organic Sulfur Occurrence in Coking Coal and Mechanism of Microwave Actionon Thiophene
- Elemental Analysis of Tea Leaves (Camellia Sinensis) and Their Own Soils at Eastern Black Sea Region in Turkey
- 基于高光谱开发滩羊肉中高铁肌红蛋白含量的定量函数
- 不同耕法下秸秆还田对土壤WSOC荧光特性的影响
- 高盐样品中锂的电感耦合等离子体发射光谱法(ICP-OES)测定研究
- 西坡遗址出土器物的光谱学分析
