侵染贵州火龙果的仙人指X病毒的基因组序列变异
2020-03-26郑乾明王小柯马玉华
郑乾明, 王小柯, 马玉华
(贵州省农业科学院 果树科学研究所, 贵州 贵阳 550006)
火龙果又称红龙果或仙蜜果,为仙人掌科(Cactaceae)量天尺属(Hylocereus)的多年生果树。当前在我国南方和西南地区种植,常使用成熟茎进行营养繁殖,导致病毒积累。仙人掌X病毒(CactusvirusX,CVX)、蟹爪兰X病毒(ZygocactusvirusX,ZyVX)和仙人指X病毒(SchlumbergeravirusX,SchVX)等侵染火龙果后会产生局部坏死、黄化和褪绿等症状,在我国台湾[1-2]、海南[3-4]等省及巴西[5]、韩国[6]和美国[7]等国家均有报道。
CVX、ZyVX和SchVX均为正义单链RNA病毒,属甲型线形病毒科(Alphaflexiviridae)马铃薯X病毒属(Potexvirus)[5]。CVX(Genbank登录号:AF308158)基因组全长约6.6 kb,5′端含帽子结构,3′端含poly A序列[2],含有5个开放读码框(Open Reading Frame,ORF),分别为依赖RNA的RNA聚合酶(RNA-dependent RNA polymerase,RdRp)、TGB(Triple gene block)蛋白1~3和外壳蛋白(Coat protein,CP)。ZyVX的B1分离物(Genbank登录号:AY366208)分离自蟹爪兰[8],P39分离物(Genbank登录号:JF930326)分离自火龙果,其基因组均具有上述类似结构。目前从仙人掌科仙人指属(Schlumbergera)[8]和胭脂掌属(Opuntia)[9-10]寄主分离SchVX基因组全长,从锦绣玉属(Parodia)寄主分离CP序列(Genbank登录号:KY581588)[11],从巴西火龙果分离RdRp部分序列(Genbank登录号:EU670721)[5]。课题组近年来对采自贵州省的疑似病毒感染火龙果样品开展相关研究,郑乾明等[12-13]利用高通量测序结合PCR扩增,获得CVX-NTU等贵州分离物基因组序列,并分析其遗传变异。目前从我国种植的火龙果中分离SchVX并研究其遗传变异的报道鲜见。为研究侵染火龙果的仙人指X病毒SchVX)发生情况和遗传变异,笔者等利用基于Illumina平台的高通量测序对6份疑似病毒感染贵州火龙果样品进行分析,以了解SchVX发生情况,获得SchVX贵州分离物的相关序列,分析其遗传变异,以期为后续病毒分子检测与防控提供理论基础。
1 材料与方法
1.1 材料
供试样品:2017年8月在贵州省罗甸县和镇宁县的火龙果果园(品种均为紫红龙),采集茎表面有褪绿、黄化斑点等疑似病毒病表现的样品6份(LR、ZNF、ZYP、RF、ZNS、LW),置于冰盒中带回实验室,使用无菌水清洗茎表面并擦干,切取感病组织液氮速冻,-80℃保存待用。
主要试剂:Trizol试剂购自Invitrogen公司;去真核生物核糖体RNA试剂Ribo-ZeroTMrRNA Removal Kit购自Epicentre公司。
1.2 方法
1.2.1 高通量测序 利用Trizol试剂提取6份样品的总RNA,使用琼脂糖凝胶电泳和紫外光分光光度计检测质量与浓度。后续高通量测序和数据分析委托广州基迪奥生物科技有限公司完成。合格样品使用Ribo-ZeroTMrRNA Removal Kit去除核糖体RNA,剩余RNA打断成短片段。使用随机引物合成第1条cDNA链,然后加入缓冲液、dNTPs、RNase H和DNA polymerase I合成第2条cDNA链。经过纯化、洗脱、末端修复、连Poly(A)、连测序接头及片段大小筛选,最后进行PCR扩增。测序文库使用Illumina HiSeq 4000进行测序,测序策略为PE150。
1.2.2 数据处理 测序获得的raw read进行质量控制,过滤低质量的read,产生的clean read利用Trinity v 2.1.1进行组装。获得的contig序列在NCBI非冗余核酸和蛋白质数据库中分别使用BLASTn和BLASTx进行检索和注释。
1.2.3 序列分析 使用ORF Finder程序(https://www.ncbi.nlm.nih.gov/orffinder/)查找contig序列中的ORF。与参考基因组序列的一致性比对使用BLAST(http://blast.ncbi.nlm.nih.gov/)工具在线比对。使用Lasergene计算核苷酸和推导的氨基酸序列一致性。核苷酸和推导的氨基酸序列使用Clustal W程序进行序列比对,比对结果用MEGA 7.0构建基于邻接法的系统进化树,Bootstrap值计算进行1 000次重复。
2 结果与分析
2.1 SchVX相关序列
对6份样品获得的clean read分别进行组装,获得大量contig序列。通过在NCBI中进行BLASTn和BLASTx检索,筛选出SchVX相关的contig序列结果(表1),6份样品中SchVX相关contig数量为18~52条。样品LR中SchVX相关的clean read数量最少,仅3 269条,占该样品中所有clean read数量的0.003%。其余5份样品中,SchVX相关clean read数量为3 540 797~17 792 691条。样品LW中SchVX相关的clean read数量最多,达17 792 691条,占该样品中所有clean read数量的17.5%。
表1火龙果样品的SchVX相关clean read和contig序列
Table 1 SchVX-related clean read and contigs from six pitaya samples
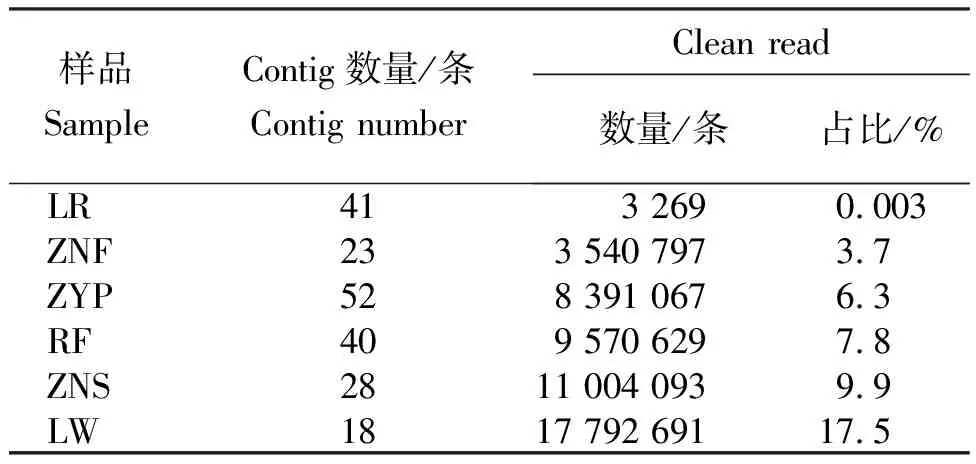
样品SampleContig数量/条Contig numberClean read 数量/条占比/% LR413 2690.003 ZNF233 540 7973.7 ZYP528 391 0676.3 RF409 570 6297.8 ZNS2811 004 0939.9 LW1817 792 69117.5
注:占比指占该样品中所有clean read数量的百分比。
Note:Proportion means percentage of all clean read number in each sample.
2.2 SchVX相关序列与参考基因组的差异
参试样品中的SchVX相关contig序列与参考基因组SchVX-Palma-PE(Genbank登录号:KP090203)进行序列比对和一致性计算的结果(表2)显示,6份样品中,contig长度最短为201bp,最长为5 617 bp。每份样品中的contig序列均覆盖99.2%~99.6%的参考基因组,说明6份样品获得的SchVX相关contig序列基本覆盖完整的参考基因组。ZYP样品中的contig序列与参考基因组序列的一致性为82.8%~100.0%,其他5份样品的序列一致性为85.9%~96.7%。
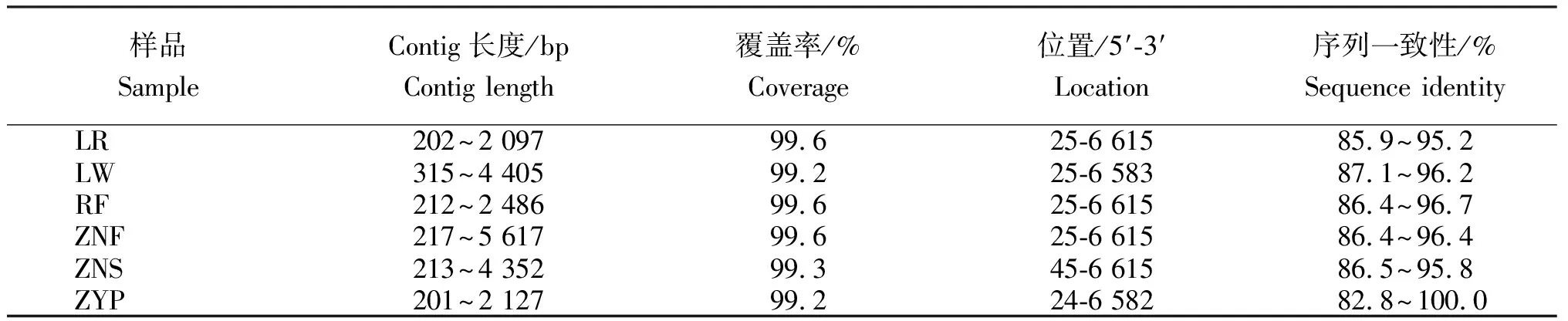
表2 SchVX贵州分离物相关contig与参考基因组序列比对
2.3 CP序列差异
从表3看出,SchVX贵州分离物CP的核苷酸序列和推导的氨基酸序列一致性分别为92.5%~97.3%和96.0%~99.1%。与NI分离物(Genbank登录号:KY581588)序列一致性分别为93.7%~95.1%和97.3%~98.2%,与K11分离物(Genbank登录号:AY366207)序列一致性分别为93.5%~95.0%和95.1%~96.9%,与Palma-PE分离物序列一致性分别为92.9%~95.6%和96.9%~98.2%,与nopal verdure 1分离物(Genbank登录号:KU854929)序列一致性分别为87.3%~88.5%和95.6%~97.3%。
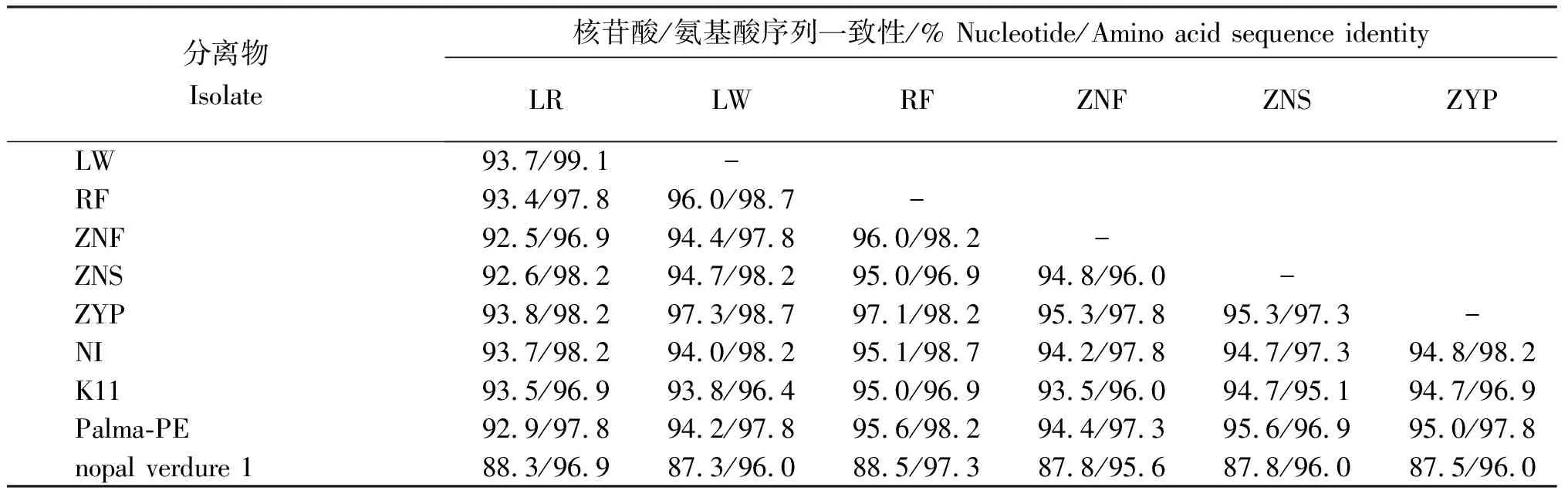
表3 SchVX贵州分离物CP序列与其他分离物序列的一致性
2.4 基于CP序列的系统进化
利用SchVX贵州分离物CP序列和NI等6个分离物,以及侵染火龙果的PiVX、CVX、ZyVX、CVX-NTU等CP序列共同构建的系统进化树(图1)显示,外类群OpVX单独为一类,SchVX各分离物均聚为一类,与PiVX、CVX、ZyVX和CVX-NTU明显分开。
基于核苷酸序列的系统进化树(图1A)表明,SchVX-LW、SchVX-RF、SchVX-ZNF、SchVX-ZNS、SchVX-ZYP与Palma-PE分离物亲缘关系较近,SchVX-LR与SchVX-LW、SchVX-RF、SchVX-ZNF、SchVX-ZNS、SchVX-ZYP、Palma-PE、K11和NI分离物的亲缘关系较远。基于推导的氨基酸序列的系统进化树(图1B)表明,6个SchVX贵州分离物与NI和Palma-PE分离物亲缘关系较近,与K11分离物亲缘关系较远,与nopal verdure 1分离物亲缘关系最远。
3 结论与讨论
基于Illumina平台的高通量测序技术具有深度高、准确率高、读长较短的特点,广泛用于果树作物病毒挖掘和鉴定[14]。笔者等对贵州2个火龙果主产县果园采集的6份疑似病毒侵染样品进行高通量测序,获得并统计SchVX序列。在5份样品中检测到大量SchVX相关序列,其含量占总read数量的3.7%~17.5%,说明采样地点的火龙果受SchVX侵染的比例较高。
对6份样品获得的SchVX序列单独组装,产生的contig序列基本覆盖完整的参考基因组序列,为了解SchVX基因组分子变异提供了基础。6份样品中SchVX相关contig序列与参考基因组相比,序列一致性最低为82.8%,最高为100.0%,多为85.9%~96.7%。与前期分离自贵州火龙果的CVX-NTU和ZyVX的遗传变异相比[12-13],SchVX的序列一致性水平略低,说明存在一定程度的遗传变异。
注:A为基于CP核苷酸序列的系统进化树,B为基于推导的CP氨基酸序列的系统进化树。
Note: A,Phylogenetic tree based on CP nucleotide sequence; B,Phylogenetic tree based on deduced CP amino acid sequence.
图1SchVX贵州分离物与其他病毒的系统进化树
Fig.1 Phylogenetic trees of SchVX Guizhou isolates and other viruses
马铃薯X病毒属病毒的CP序列编码外壳蛋白,在包装病毒核酸物质和病毒胞间移动的过程中发挥重要功能[15]。侵染火龙果的SchVX贵州分离物的CP核苷酸和推导的氨基酸序列一致性分别为92.5%~97.3%和96.0%~99.1%,与来源于仙人指K11分离物[8]、胭脂掌Palma-PE分离物[9]和金晃NI分离物[11]序列一致性较接近,明显高于来自梨果仙人掌nopal verdure 1分离物。系统进化分析也表明,nopal verdure 1分离物与上述分离物的亲缘关系较远。CHEN等[16]研究表明,病毒的变异主要是由病毒与寄主之间的互作关系所决定。当前来源于仙人掌科各类寄主的SchVX分离物表现不同程度的变异,可能与其侵染的寄主种类有关。
综上所述,样品采集地的火龙果普遍受SchVX侵染,其基因组序列存在一定程度的遗传变异。研究获得的序列未完全覆盖完整的SchVX基因组,采集的样品数量也不够丰富。在未来的研究中需要进一步获得SchVX基因组全长序列,同时增加样品采集数量,以更准确分析侵染火龙果的SchVX群体的遗传变异情况,为其检测和防控提供理论基础。
