桉树杂交种与其亲本的近红外光谱判别
2020-03-20卢万鸿王楚彪罗建中
卢万鸿,李 鹏,王楚彪,林 彦,罗建中
国家林业和草原局桉树研究开发中心,广东 湛江 524022
引 言
研究桉树(桉属Eucalyptus、伞房属Corymbia和杯果木属Angophora)控制授粉后目标性状的基因作用方式,有助于分析亲本组合时的基因重组规律,为开发优良杂交组合的亲本选配提供理论支撑。常规的数量标记分析精度往往不高[1-2],而分子生物学方法[3-4]又需要强的专业知识,且程序繁复,很难满足林木育种改良研究中对大量群体材料的快速分析。
化学计量学和光谱学的发展促进了近红外光谱(near infrared spectroscopy, NIRs)用于植物来源分类及其物质成分快速预测研究的繁荣。研究人员采集了伞房属(Corymbia)桉树纯种及其杂交种叶片的NIRs信息,建立的伞房属桉树NIRs模型的判别准确率为76%-90%[5]。采用人为混合松树(Pinus)松针模拟杂交种,建立了纯种松树的NIRs判别模型,其判别准确率也达到了90%以上[6]。伞房属桉树杂交子代与其亲本萜烯含量的差异非常小,但NIRs对这种极微小的差异反映很敏感,能检测到常规方法检测不到的微小差异[7]。关于玉米自交系遗传距离与其NIRs光谱距离间关系的研究显示,其光谱距离与其遗传距离间的相关性超过0.9,表明NIRs可以反映玉米自交系间的遗传关系。Diepeveen等的研究更具体定位了影响NIRs特征的含有遗传信息的小麦染色体片段[8]。
不同来源的物种在特定条件下内在的遗传物质差异从根本上决定了其组织成分的差异,这是NIRs用于物种成分预测和判别分析的主要依据[9-11]。本研究以桉树控制授粉材料为对象,分析桉树叶片NIRs与其遗传基础间的关系,并用簇类独立软模式(soft independent modeling of class analog, SIMCA)和偏最小二乘判别分析(partial least squares discrimination analysis, PLS-DA)两种判别分析方法进行桉树杂交种及其亲本的分类判别,探索NIRs用于桉树杂交种与其亲本判别的可行性及准确性。
1 实验部分
1.1 材料
试验材料包括粗皮桉(E.pellita)、尾叶桉(E.urophylla)、细叶桉(E.tereticonis)3个纯种及其5个杂交种,杂交种分别为3个亲本树种间的杂交F1代,外加一个目前商用的桉树无性系DH3229(见表1)。从田间试验林中采集各基因型的叶片用于NIRs的扫描。
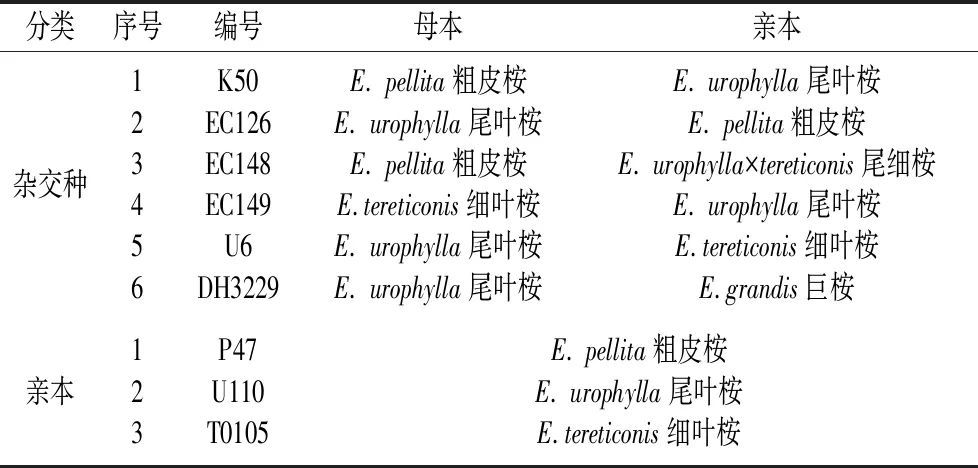
表1 测试桉树杂交组合及其亲本信息Table 1 The details of the hybrids and their parents of eucalypt
1.2 仪器
手持式近红外仪Phazir Rx (1624) (Polychromix, Thermo Scientific, USA)用于NIRs数据的采集。Phazir Rx (1624)波长范围为1 600~2 400 nm,光学分辨率12 nm,内置基于MEMS技术的可编程微衍射光栅,自带背景校正片。
1.3 方法
1.3.1 光谱采集
每种基因型分别选取10个单株,采集树冠中上部的新鲜健康叶片。每单株选10片完全生理成熟的健康叶片,用Phazir Rx(1624)扫描其正面光谱共5次,以均值代表单个叶片的NIRs信息[10]。每种基因型获得10条NIRs。
1.3.2 NIRs数据的预处理和分析
对原始NIRs进行二阶多项式S.G一阶导数预处理[10-11]。通过主成分分析直观判断NIRs对桉树不同基因型的分类效果。建立簇类独立软模式(SIMCA)和偏最小二乘判别分析(PLS-DA)两种有监督方式的判别模型检验NIRs的树种判别效果。建立PLS-DA模型时,分别对3个亲本树种人为赋值,即1,2和3[12]。数据预处理和分析过程均在The Unscrambler x10.4(CAMO, Oslo, Norway)中实现,主成分分析(principal component analysis, PCA)和PLS过程均采用全交互式内部交叉验证算法。
2 结果与讨论
2.1 桉树杂交种叶片的NIRs信息的差异
NIRs主要是物质有机分子的倍频与合频吸收光谱,不仅能得到物质的分子结构、组成和状态信息,也能反映密度、粒度、高分子物的聚合度及纤维形态等物质的物理状态信息[10]。图1是6个桉树杂交组合原始NIRs的平均值曲线,通过NIRs原始光谱的直观变化很难发现其特征峰,6种组合的NIRs信息在全波段变化趋势基本一致,且存在明显的重叠。在波长1 860 nm之前和波长1 940 nm之后,6个杂交组合的NIRs反射率在一定程度上存在差异,但不足以据此进行树种判别。
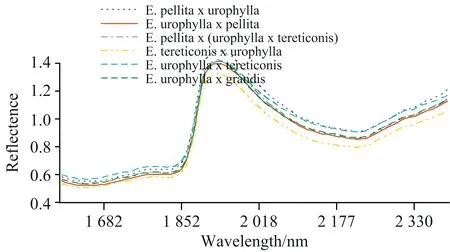
图1 桉树杂交种的原始NIRs反射率
2.2 桉树杂交种及其亲本的PCA聚类
PCA可以简化多维数据中大量重叠的信息,因子得分可以反映受试样本间的距离关系。图2是6个桉树杂交种叶片NIRs数据的PCA因子得分图。杂交种EC126,EC148和EC149能清晰地聚类[图2上],U6, K50和DH3229也能清晰地分开[图2下],表明NIRs能够区分不同的基因型,可对遗传差异做出响应。

图2 桉树杂交子代的PCA因子得分图
将6个杂交种同时进行PCA分析时,各杂交种存在一定程度的重叠(未展示),这主要与其亲本的遗传亲缘关系有很大的关系。另外,因子得分的聚集度也反映了杂交种遗传变异的大小,聚集度高的变异小,反之亦然,如图2中EC126,K50和EC149的变异可能较大,EC148和U6的变异则相对较小。
图3是3个亲本NIRs数据PCA的因子得分图。从图中可以看出,粗皮桉相对较为分散,细叶桉聚集度最高,尾叶桉聚集度居中。从分类的聚集度来看,粗皮桉的变异最大,细叶桉的变异则最小。总体来看,因子得分图能够将3个亲本树种清晰地分开,真实地反映了不同树种内在的遗传差异。
2.3 桉树亲本模型对其杂交种的SIMCA判别
用3个亲本样本建立PCA模型,设定临界概率水平为0.05。图4为每组亲本PCA模型对杂交子代进行SIMCA判别的结果,结果以杂交种样本与亲本PCA模型中心间的距离表示。图4显示,6个杂交种均可以与其亲本清晰地分开,其中杂交种K50与其母本粗皮桉的距离相对较近,EC126与其父本粗皮桉间的距离较近,EC148与其母本粗皮桉间的距离非常接近,EC149距其父本尾叶桉较近,U6基本居于其父本尾叶桉和母本细叶桉之间,商用杂交种DH3229基本居于尾叶桉和粗皮桉中间(未采集到巨桉样本)。SIMCA模式判别中的样本距离更直观地反映了杂交种与其亲本样本间的遗传相似性(遗传距离)。
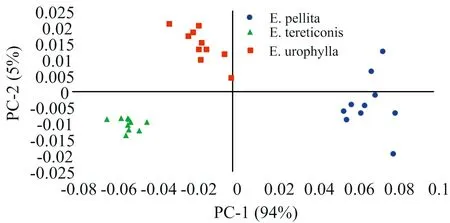
图3 桉树杂交亲本的PCA因子得分图
图4 SIMCA分析中6个桉树杂交种样本到亲本模型中心距离的Cooman图
2.4 桉树亲本模型对其杂交种的PLS-DA判别
图5展示了5个组合的桉树杂交亲本PLS模型对亲本和杂交子代样本的预测结果。结果显示,每个亲本的预测值都集中在各自响应变量周围(1,2和3),且集中度很高。杂交种K50的预测值为1.5~1.7,EC126的预测值为1.1~1.2,EC148的预测值为2.1~2.3,EC149的预测值为1.2~1.5,U6的预测值为1.5~1.8。预测值显示,EC126和EC148的预测值高度重叠,EC149预测值的变异幅度最大,K50和U6预测值的变幅居中。PLS-DA判别可以清晰地将不同基因型区分开来,不过,任何判别方法都需要专业知识来配合解读分析结果。
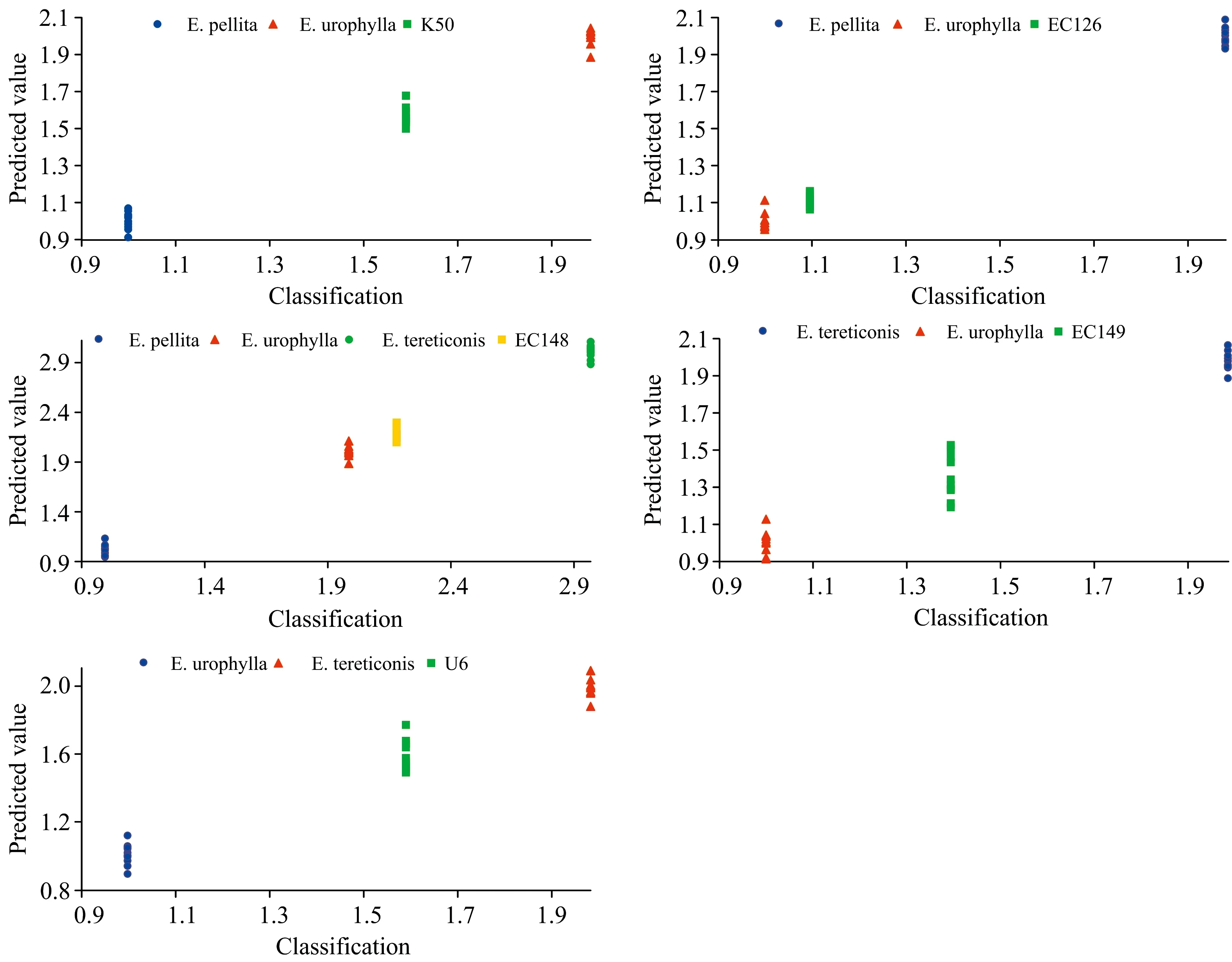
图5 PLS-DA模型对桉树亲本与其杂交种样本响应变量的预测
图6是PLS-DA判别分析时所建模型第一个主因子的载荷,第二和第三个主因子的载荷分布与第一个相似,强度略有差异,本文没有展示。图中小方块指此处波段所对应的有机化合物的NIRs特征峰。1 890 nm处为化学键O—H和C—O的伸缩振动(stretching)吸收峰,对应的化合物主要为纤维素。1 980~2 000 nm处为O—H键伸缩振动、水分子中O—H键变形(deformation)吸收峰[13-14]。
3 结 论
采用SIMCA和PLS-DA两种有监督的判别模型,有效地解决了桉树叶片NIRs信息复杂、重叠的问题。两种模式的判别效果均显示,NIRs可以将桉树杂交种、亲本、杂交种与其亲本清晰地区分。
结果表明NIRs真实地反映了不同基因型的遗传信息。其次,NIRs可以反映桉树的遗传变异程度,即桉树杂交F1代来自亲本的加性遗传效应的大小。所以,NIRs能够用于桉树树种的区分,也可根据PCA聚类离散度或PLS的预测响应变量分析基因重组过程中的加性遗传效应。
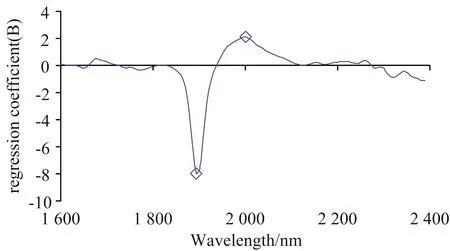
图6 PLS-DA判别模型第一主因子不同波段的载荷
The small square ◇ indicates NIRs characteristic peaks of the chemical bands
