正交试验设计优化近红外检测牛乳中蛋白质的建模条件
2020-03-11刘亚丽李林青毕艳兰
彭 丹,刘亚丽,李林青,毕艳兰
(河南工业大学粮油食品学院,河南 郑州 450001)
蛋白质作为生命的物质基础,在生理功能调节中起着重要作用。牛乳是提供人体蛋白质的重要途径之一,已成为人们生活中的必需品。国家标准中明确规定了牛乳中蛋白质含量的最低要求,如《巴氏杀菌乳》《灭菌乳》中蛋白质含量不得低于2.9%。近年来,有关乳品中蛋白质含量不达标的事件时有发生,一些不法商贩向乳品中添加高氮物质如三聚氰胺等提高蛋白质含量牟取利益,严重影响乳品行业的发展和消费者的身体健康,引起了社会关注,也对质监部门和检验工作者提出了更高的要求,即寻找快速、准确检测乳品中蛋白质含量的方法。
目前,常用的蛋白质检测方法有凯氏定氮法、紫外吸收法[1]、电泳法[2]、低场核磁共振法[3]和近红外光谱法[4-7]等。凯氏定氮法是通过测量总氮量计算得到蛋白质的含量,该方法测量准确,但操作复杂、费时长;紫外吸收法具有简便、灵敏等优点,但分析精度不高,干扰物质较多;近年来出现的电泳法、低场核磁共振法等检测准确、快速,然而这些方法还不够成熟。相比其他方法,近红外光谱法具有快速、无损、可在线检测等特点,已广泛应用于食品、农业、化工和制药领域中[8-12]。近红外光谱技术属于间接分析方法,需要通过多元校正方法建立光谱信息与待测成分间的关联,由于仪器性能、外界环境、自身特性等因素影响,使得光谱数据中往往含有共线性、噪声及外界干扰信息等,导致建模结果误差较大甚至无法使用,故此需要优化条件。目前,在乳蛋白质检测中主要开展了3 个方面的研究:1)通过不同测量方式检测乳品中蛋白质含量,对于含有悬浮颗粒的液体,多采用漫透射[13]或漫反射[14-15]方式进行测量;2)选择不同特征波段开展蛋白质含量检测[16],如短波段(780~1 100 nm)、中波段(1 100~1 700 nm)和长波段(1 700~2 500 nm)等,以此剔除冗余信息,增强检测的针对性;3)确定蛋白质检测的最佳化学计量学方法,包括预处理方法[17-18]和建模方法[19-20]。由于牛乳介质中存在颗粒不均匀性,使其对近红外光谱有很强的散射作用,多采用正交信号校正、多元散射校正、求导等对光谱进行处理。在已有的建模条件研究中往往只考虑某一方面的优化,而实际问题中各因素完全独立的情况极为少见。因此,本实验针对各因素间存在相互作用影响的情况,在单一建模条件研究的基础上,运用正交试验设计对建模条件进行优化,建立一个准确度高、重复性好、适用于牛乳蛋白质含量分析的优化检测体系。
1 材料与方法
1.1 材料与试剂
实验所用的牛乳均来自厂家直营店。为使蛋白质含量在较大范围内变化,采取添加蛋白粉的方式,并对样品进行均质操作以保证样品的均匀性和稳定性,共得到180 个样本。通过K-S方法选取135 个样本的光谱数据作为校正集,其余45 个样本的光谱数据作为验证集。
1.2 仪器与设备
XDS型近红外光谱仪 丹麦FOSS公司;ZS90粒度电位分析仪 英国Malvern公司;AH-Pilot高压均质机德国APV公司。
1.3 方法
1.3.1 蛋白质含量的测定
参考GB 5009.5ü2016《食品中蛋白质的测定》测定。
1.3.2 近红外光谱检测
采用XDS型近红外光谱仪对样品的吸收光谱进行测试,光谱采集范围780~2 500 nm,扫描次数32 次,分辨率2 nm,检测器为硅(780~1 100 nm)和硫化铅(1 100~2 500 nm)。
1.3.3 光谱数据处理
以蛋白质含量为外部微扰,进行二维相关同步谱和自相关谱解析,寻找与蛋白质含量相关的敏感波段。具体过程:1)依据蛋白质含量的真实值,从小到大顺序均匀选取10 个代表性样品用于一维近红外牛乳光谱的测量;2)采用正交信号校正对测得的光谱进行预处理,消除与被测成分无关的光谱信息;3)根据二维相关光谱理论,对处理后的一维光谱进行分析、计算,获得其二维相关同步谱及自相关谱;4)通过解析二维相关同步谱和自相关谱,确定与蛋白质含量相关的最佳光谱波段。上述二维相关光谱计算过程利用Matlab R2018a软件完成。
利用CAMO公司的Unscrambler 10.5软件对光谱进行预处理,分别建立多元线性回归(multiple linear regression,MLR)、主成分回归(principal component regression,PCR)、偏最小二乘(partial least squares,PLS)法和支持向量机(support vector regression,SVR)校正模型,模型性能通过决定系数(R2)、校正均方根误差(root mean square error of calibration,RMSEC)和预测均方根误差(root mean square error of prediction,RMSEP)评价。
1.3.4 正交试验设计
在单因素试验的基础上,选取检测波段、预处理方法和建模方法为考察因素,以目标函数F[21]为评价指标,采用L16(45)正交表进行试验设计,确定蛋白质含量最佳的建模条件。
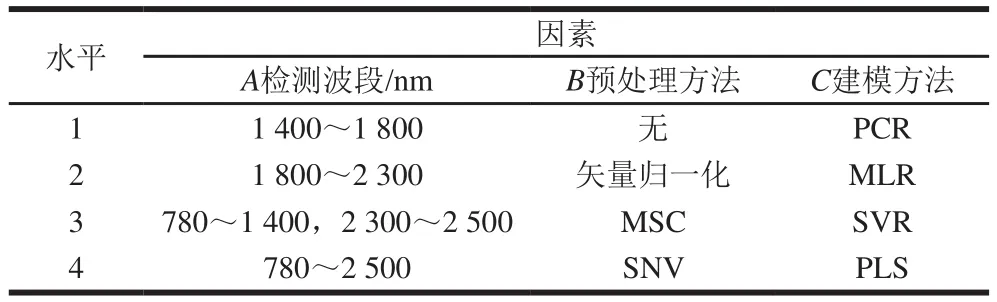
表1 正交试验因素与水平Table 1 Code and level of independent variables used for orthogonal array design
1.4 数据统计
采用Matlab R2018a、Origin 9.0软件和Excel 2016软件进行数据统计和图表绘制,利用SPSS 22.0软件对数据进行方差分析。
2 结果与分析
2.1 牛乳近红外光谱及二维相关分析
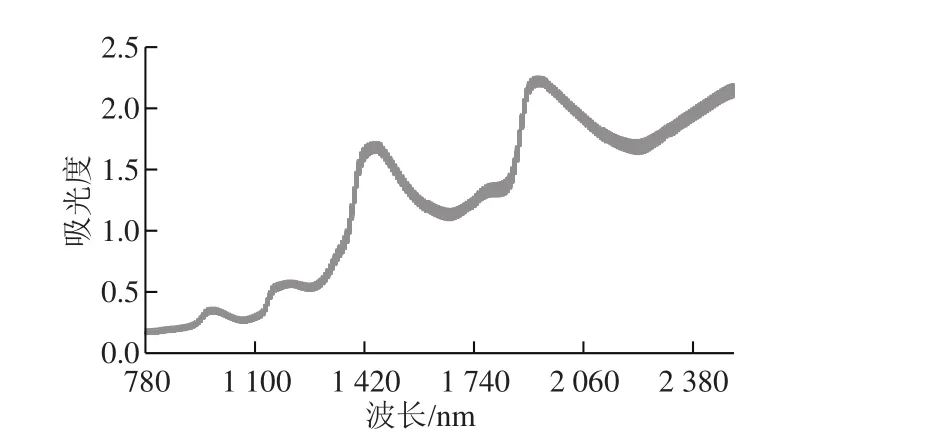
图1 牛乳的近红外光谱图Fig. 1 NIR spectra of milk samples
如图1所示,脂肪、蛋白质和乳糖等成分近红外吸收分布于整个光谱区域,且相互重叠。在1 460、1 974 nm波长附近有2 个主要吸收峰,分别为牛乳中水、蛋白质、脂肪和乳糖等成分的结构和组成信息。其中,1 460 nm波长处与—OH、—NH2等基团的倍频吸收有关[22],1 974 nm波长处为水的吸收信息。为确定蛋白质的主要吸收位置,以蛋白质含量为外扰,对牛乳近红外光谱进行同步二维相关分析,结果如图2所示。
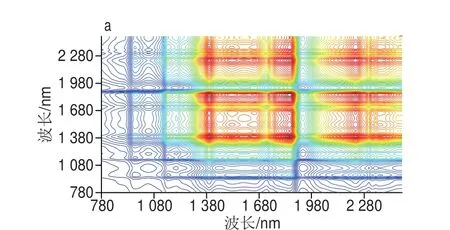
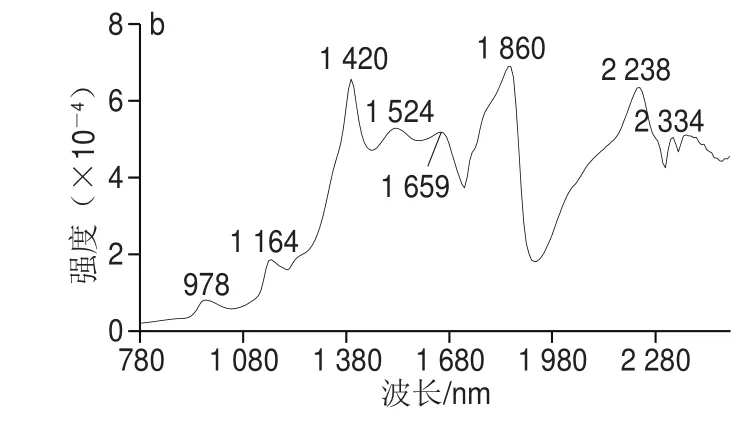
图2 牛乳二维近红外光谱相关同步谱(a)和自相关谱(b)Fig. 2 Two-dimensional correlation spectra of samples (a) and autocorrelation spectrum (b)
图2 a中的峰有自相关峰和交叉峰两类,位于对角线上的峰为自相关峰,位于对角线外为交叉峰。自相关峰的强度反映了不同波长下光谱信号随外部扰动变化的程度[23-25],即对蛋白质含量变化的敏感程度,其值均为正;交叉峰则表示不同波长下光谱强度变化的相似性,其值有正有负。自相关谱是由对角线上自相关峰构成的谱图(图2b)。由图2可以看出,在波长978、1 164、1 420、1 524、1 659、1 860 nm和2 238 nm处存在较强的自相关峰,其中2 238 nm处的自相关峰与蛋白质有关;在主对角线以外,在(1 420 nm,1 860 nm)、(1 860 nm,2 238 nm)、(1 420 nm,2 238 nm)、(1 524 nm,2 238 nm)位置处存在明显正交叉峰,表明波长1 420、1 524、1 860 nm和2 238 nm处的吸收峰来源相同,均是由蛋白肽键和氨基中NüH键对光谱吸收形成[26]。可见,1 400~2 300 nm波段的光谱随蛋白质含量变化极显著。该波段与Kalinin等[27]牛乳蛋白质测量所用的光谱区域(800~1 065 nm)有所不同,这可能与测量方式、仪器硬件等有关,本实验采用了漫反射方式测量牛乳蛋白质含量,而文献[27]中利用了透射方式。虽然短波区域(780~1 100 nm)的光透射性强,但吸收系数较小[28],与长波相比蛋白质含量信息量相对较少(图2b),增加了数据分析和建模的难度,结合二维相关分析,本研究选择波段1 400~2 300 nm作为蛋白质检测的研究区域。
2.2 单因素试验结果
2.2.1 预处理方法的对比
为降低干扰信息的影响和提升模型预测精度,分别采用矢量归一化、多元散射校正(multiplicative scatter correction,MSC)、一阶导数(1st)、二阶导数(2nd)、标准正态变量变换(standard normal variate,SNV)方法对原始光谱进行预处理后建立PLS模型,结果如表2所示。除2nd方法外,矢量归一化、MSC、1st、SNV方法处理后模型的相关系数和预测准确性均有明显提高,表明这4 种方法能够扣除光谱中与待测成分无关的干扰信号,保留有用信息。其中,经SNV法处理后的蛋白质模型的预测效果最佳,其次为MSC法。与预处理前相比,经SNV法和MSC法处理后模型的RMSEP分别降低了31.1%和27.8%。通过粒径分析可知,均质后牛乳中脂肪球的粒径分布于0.2~1.0 µm之间,存在明显脂肪球分布的不均匀性,这使其对近红外光有较强的散射作用[29],由于MSC法和SNV法能够消除颗粒大小及均匀性变化对光谱的影响,所以经MSC法和SNV法处理后可有效消除散射作用引起的干扰。相反,2nd法在去除背景干扰、提高灵敏度的同时放大了噪声,降低了信噪比,使得模型的预测能力下降,导致RMSEP升高了91.9%。可见,预处理方法的选择对蛋白质检测结果有较大影响,其中MSC和SNV是较为理想的预处理方法。
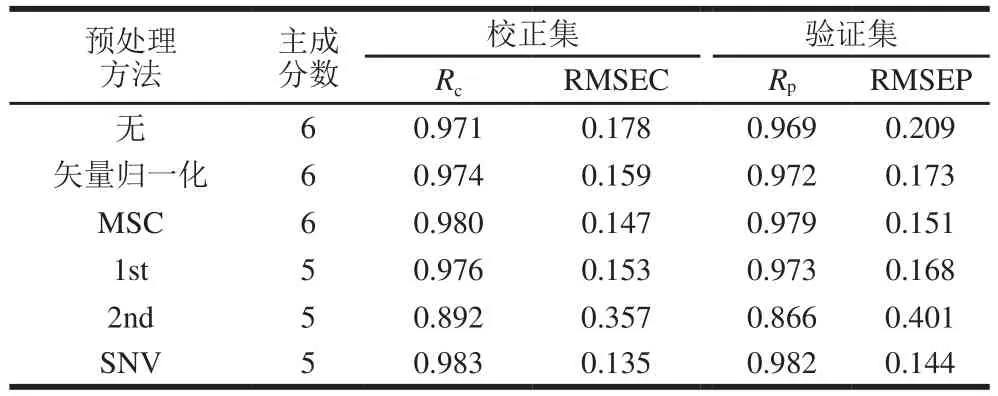
表2 预处理方法的比较Table 2 Comparison of results obtained with different preprocessing methods
2.2.2 波段选择的影响
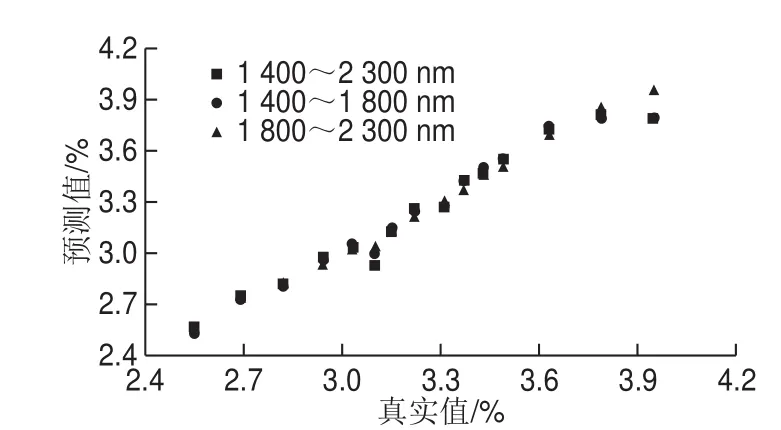
图3 不同波段牛乳中蛋白质含量的预测Fig. 3 Effect of different wavelength regions on prediction of protein concentration in milk
选择与待测组分相关的特征波段,既能降低计算量、提高模型的预测能力和稳健性,又能避免光谱数据间相关性导致的过拟合现象。本实验在1 400~2 300 nm波长范围内研究不同波段对蛋白质含量检测的影响。如图3所示,3 个波段1 400~2 300、1 400~1 800 nm和1 800~2 300 nm的RMSEP分别为0.146、0.142和0.125,其中1 800~2 300 nm波段的预测结果明显好于其他波段,这与Tsenkova等[30]的研究结果基本一致,可见1 800~2 300 nm波段内含有较强的蛋白质含量信息。虽然1 400~1 800 nm波段也含有蛋白质相关的光谱信息,但是由于存在脂肪较强的散射作用和水的强吸收作用,加之牛乳中水的含量(>87%)远大于蛋白质,使得此波段存在复杂的背景干扰。因此,1 800~2 300 nm是蛋白质理想的检测波段。
2.2.3 建模方法比较
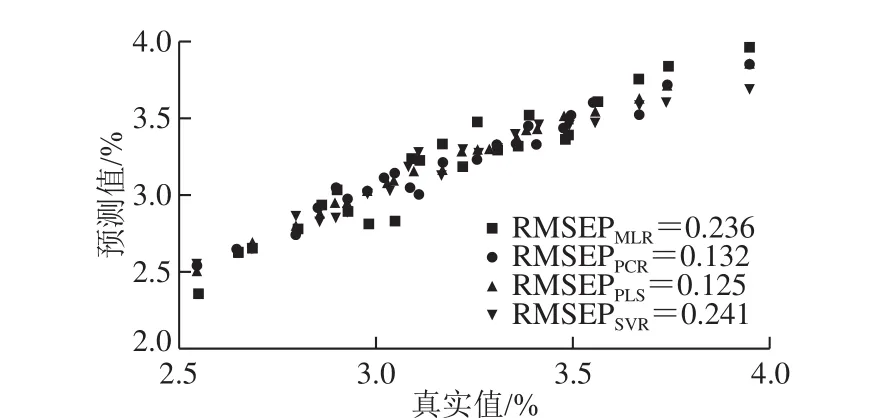
图4 不同建模方法牛乳中蛋白质含量的预测结果Fig. 4 Effect of different modeling methods on prediction of protein concentration in milk
采用MLR、PCR、PLS和SVR对同一牛乳样本进行定量分析,结果如图4所示。4 种建模方法中线性校正方法明显好于非线性,且线性建模方法中PLS法和PCR法对蛋白质的预测性能均优于MLR法。这可能是因为1 800~2 300 nm范围内自变量(光谱数据)与被测目标(蛋白质含量)间不仅有较强的线性关系,同时光谱数据内部存在多重共线性的现象,使得MLR建模方法失效。而PLS和PCR法能够对光谱数据进行分解和筛选,剔除多重相关信息和无解释意义信息的干扰;SVR法作为非线性定量校正方法,其原理是通过升维方式使原样本空间中的非线性问题转化为特征空间中的线性关系进行建模,但是对于样本空间中线性问题使用SVR法会增加计算的复杂性,甚至引起“维数灾难”,导致模型的预测精度降低。由图4可知,PLS法和PCR法建立的蛋白质校正模型的预测结果相差不大,RMSEP分别为0.125和0.132。因此,PLS法和PCR法均适合作为蛋白质检测的建模方法。
2.3 正交试验结果
在实际应用中,由于近红外吸收光弱且易受外界干扰,光谱中夹杂许多无用信息(包括噪声、背景等),为提高信噪比、建立稳定的校正模型,需要对光谱数据进行波段、预处理和建模方法的优选组合。本实验选取预处理方法、检测波段和建模方法3 个因素进行正交试验,以F值作为评价指标,F值越大表明模型的性能越好。经方差分析,以上3 个因素对模型的预测精度存在一定交互作用的影响。由表3可知,影响蛋白质模型预测精度的主次因素为建模方法>检测波段>预处理方法;根据极值分析结果,得到各因素的较好水平组合为A2B3C1,即建模方法为PCR法、检测波段为1 800~2 300 nm、预处理方法为MSC法,这与单因素试验结果并不完全一致,且A2B3C1没有出现于正交试验设计方案中,这可能与预处理方法、建模方法2 个因素间的交互作用影响有关。为了确定最佳建模条件,分别在A2B3C1、单因素试验以及正交设计试验中最优建模条件下对未知样品进行检测,结果表明A2B3C1条件下的预测效果最好,其R2、RMSEP分别为0.993、0.106。可见,经正交试验设计确定的建模条件能够进一步提高蛋白质模型的准确度,也表明正交设计能够有效优化复杂背景下的近红外建模条件。
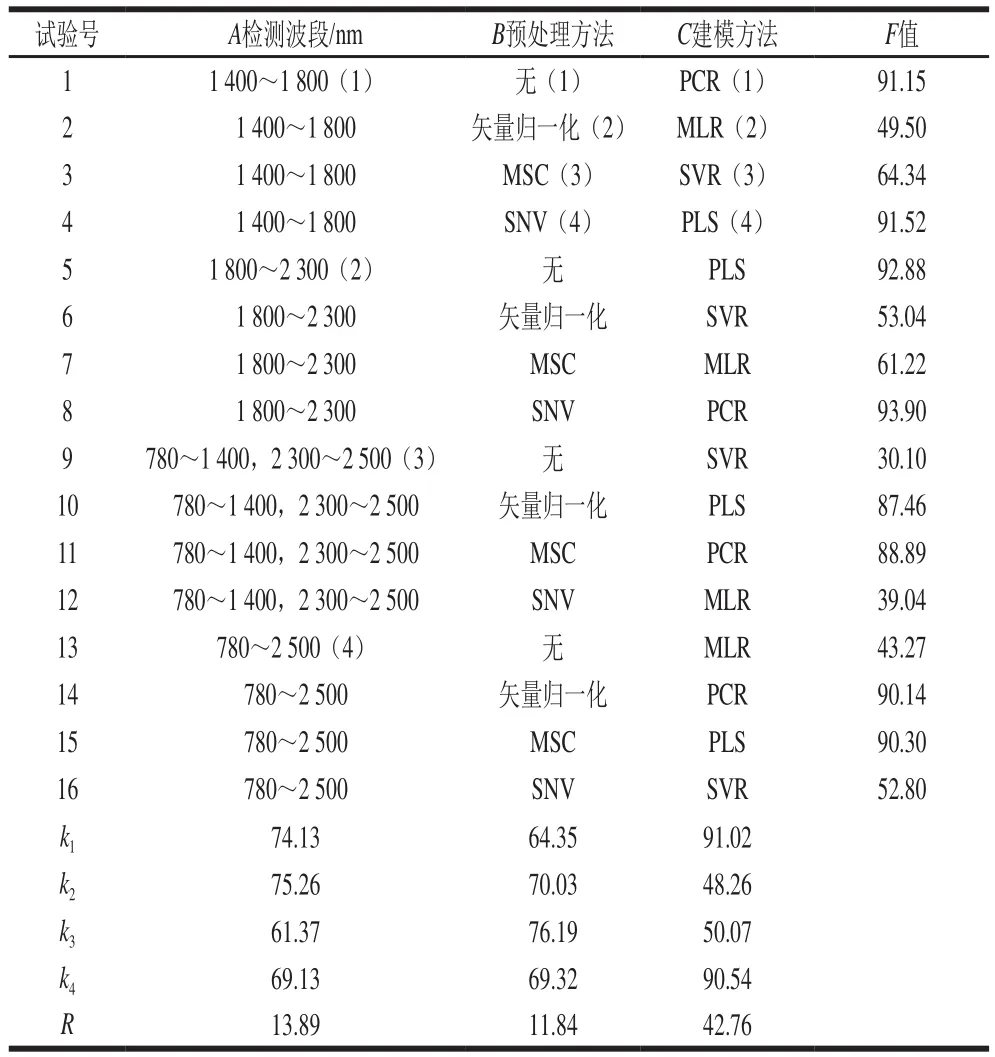
表3 正交试验设计与结果Table 3 Orthogonal array design with results
3 结 论
建模条件的选择直接影响近红外光谱分析结果。本实验以复杂背景条件下的蛋白质为研究对象,采用近红外光谱技术对牛乳中蛋白质含量进行检测,通过正交试验设计优化近红外建模条件,即预处理方法、检测波段和建模方法。结果表明,与传统方法对比,该方法不仅避免了各建模条件存在交互作用的影响,覆盖了主要因素的各种组合,而且以较少实验次数得到了预测准确度较高的分析模型,为近红外蛋白质含量建模条件优选提供了一条有效的途径。
