利用RNA-Seq发掘玉米叶片形态建成相关的调控基因
2020-02-27郭书磊鲁晓民齐建双魏良明张新韩小花岳润清王振华铁双贵陈彦惠
郭书磊,鲁晓民,齐建双,魏良明,张新,韩小花,岳润清,王振华,铁双贵,陈彦惠
利用RNA-Seq发掘玉米叶片形态建成相关的调控基因
郭书磊1,2,鲁晓民1,齐建双1,魏良明1,张新1,韩小花1,岳润清1,王振华1,铁双贵1,陈彦惠2
(1河南省农业科学院粮食作物研究所/河南省玉米生物学重点实验室,郑州 450002;2河南农业大学农学院,郑州 450046)
【】叶片宽度和长度等叶形特性是决定植株形态,进而影响种植密度的重要农艺性状,通过转录组测序技术筛选并挖掘玉米叶片形态建成相关的代谢路径及调控基因,为深入认识叶片发育的分子机理和鉴定叶宽、叶长候选基因奠定基础。以极端窄叶自交系NL409和宽叶自交系WB665为材料,利用RNA-Seq技术鉴定7叶期第七片叶近基部的差异表达基因(DEGs),通过生物信息学分析,筛选与叶片发育密切相关的代谢通路,利用qRT-PCR验证不同激素路径叶形相关基因的表达结果,并结合启动子区域的序列差异挖掘叶形功能基因。分析对照(WB665)和样品(NL409)高通量测序结果,在叶宽形成关键部位共筛选出5 199个DEGs,其中,2 264(43.55%)个基因表达上调,2 935(56.45%)个基因下调表达,下调基因明显多于上调基因;GO功能富集分析表明,差异基因主要富集在细胞膜相关的细胞组分中,涉及代谢过程和细胞响应刺激;KEGG富集分析表明,差异基因主要参与到核糖体、植物激素信号转导、苯丙烷类代谢、乙醛酸和二羧酸代谢等过程,其中核糖体、植物激素信号转导、鞘脂类代谢下调表达基因较多的路径与叶片发育密切相关。核糖体路径富集到多个PRS(PRESSED FLOWER)基因,分析发现()可能在调控窄叶发育过程中发挥重要作用。鞘脂代谢路径富集的基因几乎全部下调表达,引起抑制叶片发育的AP1(APETALA1)类和MAPK(Mitogen-Activated Protein Kinase)类基因上调,以及促进叶片发育的()下调,与窄叶发育受抑制的表型一致。植物激素信号转导路径富集到的油菜素内酯(BR)响应基因和赤霉素(GA)代谢基因下调,细胞分裂素(CTK)和大部分生长素(Auxin)响应基因上调,与窄叶中DELLA蛋白基因上调表达,抑制GA并促进CTK基因表达的作用模式一致。通过qRT-PCR对18个叶片发育相关基因进行分析,结果表明,其表达趋势与转录组结果一致,分析发现BR相关的、Auxin相关的、e以及TCP类转录因子/等基因与窄叶的形成密切相关。明确了一些与玉米叶片发育密切相关的代谢路径,还发现植物激素间的动态平衡对叶片发育有着重要影响,尤其是生长素与油菜素内酯、细胞分裂素与赤霉素之间的相互作用对调控叶片形态可能发挥重要作用。
玉米;叶宽;叶长;RNA-Seq;形态建成;调控基因
0 引言
【研究意义】玉米作为增产潜力巨大的粮食作物,对于保障粮食安全,促进农业发展具有重要作用。近年来,中国玉米的种植密度维持在4.8—6.77万株/hm2,与美国8.5—10.95万株/hm2的种植密度存在显著差距,提高玉米的耐密植特性,成为实现中国玉米高产突破的重要措施[1-2]。叶片是玉米进行光合作用和抗逆作用的主要营养器官,是耐密植理想株型的关键组成因素,对于植株干物质的积累及抗逆性有非常重要的影响。对于大面积密植的农作物,高密度条件下容易出现避荫综合症(shade avoidance syndrome,SAS),迫使群体内个体将能量重新分配,用于叶片和植株伸长生长,从而获得更多的阳光,尽管避荫反应对单个植株的适应与生存是有利的,但是由于避荫反应会削弱植株叶片和储藏器官的发育[3],对提高大面积种植的作物产量是不利的。高密度条件下,叶片宽大的群体冠层间通风透气性差,降低植株的抗病性,加剧病虫害的发生,导致生物量和籽粒产量降低。玉米耐密植株型的合理叶宽、叶长有利于改良理想株型,减少植株的避荫反应,使个体间竞争最小化,对提高种植密度具有重要的促进作用。叶宽、叶长等叶形特性是决定植株形态进而影响种植密度的重要农艺性状,因此,选育适宜高密度种植的合理叶宽、叶长玉米,是提高玉米产量的重要技术手段之一。虽然有大量研究揭示了玉米叶发育及形态建成的遗传基础,但是鲜有关于叶宽、叶长QTL克隆及调控网络的研究结果[4],利用转录组(RNA-seq)筛选与叶宽、叶长发育相关的调控基因,并发掘其可能的调控路径,对叶宽、叶长主效基因研究及耐密株型设计育种具有重要意义。【前人研究进展】叶片的发育起始于茎顶端分生组织(shoot apical meristem,SAM)周边的初始细胞,主要包括叶原基的形成和叶极性轴向的建立[5]。叶原基是由顶端分生组织中心“干细胞”在生长素诱导下形成成簇分布的外缘叶原初生细胞;叶原基形成后在自身遗传机制和环境因子作用下,建立基-顶轴(叶的基部-顶端)、腹-背轴(茎的近轴面-远轴面)、中-边轴(叶的主脉-边缘)3个极性轴向,这些过程中的发育异常将直接或间接影响叶片的发育和形态建成。玉米叶片的发育和形态建成是由多基因调控的复杂过程,主要由遗传因素控制,同时受环境影响,叶宽的遗传相比其他叶形性状受环境影响较小,而且其具有易观测的线性形态,更有利于研究叶片发育过程的形态变化和细胞学特征,是研究叶宽最为理想的模式植物[4]。关于拟南芥、水稻、金鱼草等植物叶片发育的研究,取得了一系列重要进展,深入解析了叶片发育的调控机理,而玉米叶宽、叶长的研究多为遗传分析及QTL定位的报道,只有少数文献关于叶发育基因、、、、、/、、、/和遗传机理的分析[6-14],揭示了叶宽、叶长发育形成的部分分子机理。高通量转录组测序技术,不仅能够检测全基因组所有基因的转录信息,还可以获得特定时期某一生物学过程显著富集的基因,以及特定组织或细胞中的多种转录本和优势表达基因,对研究特定组织的生物学功能具有重要的指导意义,利用转录组发掘玉米叶片发育相关基因,为叶片遗传机理的解析提供了新途径[15-16]。【本研究切入点】关于叶宽、叶长等叶形主效基因克隆的研究相对有限,已有研究仅揭示了叶形发育的部分分子机理,叶宽、叶长候选基因及其调控路径有待进一步解析。【拟解决的关键问题】本研究通过RNA-seq测序探讨叶宽差异极显著自交系的转录表达特性,以期获得与叶形发育相关的关键基因及转录调控网络,更好地理解玉米叶片发育的分子机理,增加对窄叶形成机制的认识,进而为叶形候选基因的克隆及耐密分子育种提供参考。
1 材料与方法
1.1 植物材料
2017年夏极端窄叶自交系NL409(样品)和宽叶自交系WB665(对照)种植于河南省农业科学院现代农业科技示范基地,在田间正常生长至7叶期时(对生的叶耳包裹着新鲜的幼茎即将打开),取第7片叶近基部叶片(近基部1/3叶长处,切取2.0 cm),为了消除个体间差异,将相同基因型的材料3株混合取样,每个材料取3次生物学重复,样品经液氮速冻后-80℃保存,用于RNA提取。极端窄叶自交系NL409是欧洲杂交种连续自交选育而来,宽叶自交系WB665是昌7-2改良系,两者亲缘关系较远。
1.2 RNA样品的提取及转录组测序
委托北京安诺优达基因科技有限公司完成RNA提取、文库构建和转录组测序。首先,采用酚/氯仿法提取Total RNA,通过NanoDrop 2000微量分光光度计、Agilent 2100 Bioanalyzer和Agilent RNA 6000 Nano Kit检测RNA浓度、纯度和完整性;其次,检测合格样品利用Oligo(dT)富集mRNA,并通过Fragmentation Buffer使其短片段化,以短片段mRNA合成双链cDNA,纯化后进行末端修复、加碱基A、加测序接头处理;最后进行PCR扩增完成测序文库制备。采用Illumina NextSeq 550AR平台进行双末端测序,并对获得的原始序列(raw reads)进行过滤,去除含接头、低质量(Q<30)、含N比例大于5%的序列,得到高质量的Clean Reads用于后续分析。
1.3 基因表达数据分析
利用TopHat(v2.0.12)软件,将6个样本过滤后的Clean Reads比对到B73基因组(AGPv3 版本),通过FPKM(Fragments per Kilobase per Millon Mapped Fragments)对基因表达量进行标准化,以FPKM>1为表达标准,随后根据极端窄叶与宽叶的比较,用DEseq(|log2FC(fold change)|≥1和-value/FDR<0.05)确定差异表达基因(differentially expressed genes,DEGs)[17]。利用Uniprot、Swissprot、COG、NR、GO和KEGG等数据库对DEGs进行功能注释,通过GO富集分析显示DEGs显著富集的功能分类[18],用KEGG分析DEGs主要参与的代谢途径和信号通路(kyoto encyclopedia of genes and genomes,KEGG)。
1.4 实时荧光定量PCR(qRT-PCR)分析
选择与叶片发育相关的18个差异表达基因进行qRT-PCR验证。将转录组测序用到的RNA,通过反转录试剂盒(GoScript Reverse Transcription Kit购于promega生物技术有限公司)合成cDNA第一链,以其为模板,利用GoTaq qPCR Master Mix Kit(购于promega生物技术有限公司)在Bio-Rad CFX96实时荧光定量仪上进行qRT-PCR。以玉米中的作为内参基因,每个基因的定量分析设置3次生物学重复,3次技术重复,采用相对定量2-ΔΔCt法分析结果[19]。
2 结果
2.1 叶宽表型和RNA-Seq测序质量分析
极端窄叶自交系NL409和宽叶自交系WB665叶宽形态稳定时期(散粉后一周)的叶宽表型(图1),以及第7叶期的叶宽达到极显著差异(表1),在这两个时期,NL409的叶宽、叶长、叶面积均显著小于WB665。6个RNA-Seq文库原始测序过滤后分析结果(表2),各样本生物学重复间的相关系数为0.984—0.995,其中约88%高质量序列被比对到外显子区域,表明测序结果良好,数据可用于进一步分析。
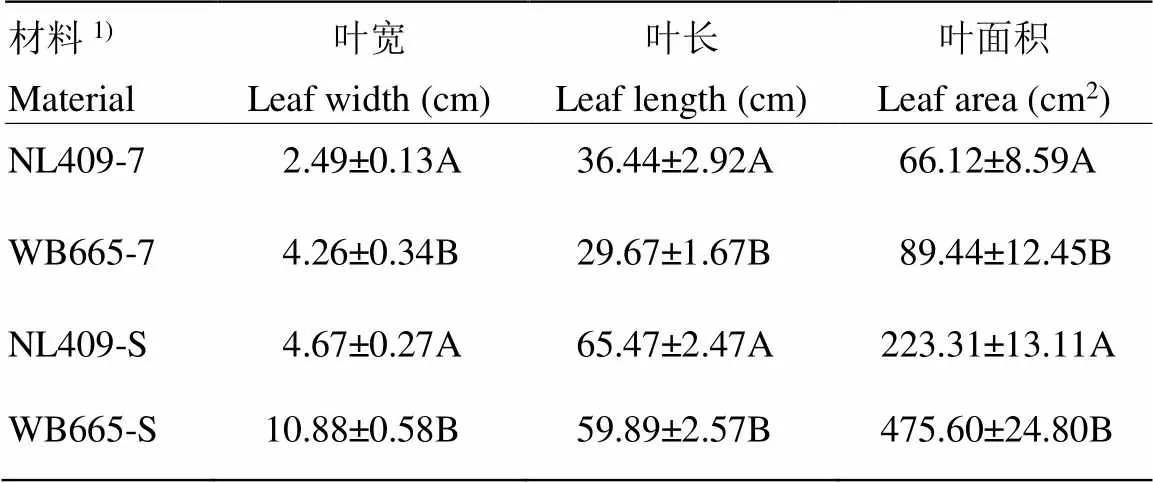
表1 自交系NL409和WB665第7叶期以及稳定时期的叶宽、叶长、叶面积
1)7表示第7叶期,S表示稳定时期。不同大写字母表示在<0.01水平差异显著
1)7 indicate the 7th leaf period, S indicate the stable period. different uppercase letters indicate significant differences at<0.01 levels

图1 自交系NL409(左)和WB665(右)稳定时期的叶片
2.2 基因表达数据分析
选取第7叶期窄叶自交系NL409(T7,样品)和宽叶自交系WB665(C7,对照)叶片发育最宽处进行转录组差异比较分析,发现5 199个差异表达基因,其中2 264(43.55%)个基因上调表达,2 935(56.45%)个基因下调表达。上(下)调1—3倍(fold change)的基因分别占差异表达上(下)调基因总数的29.37%(665)和21.69%(756);上(下)调3—10倍的基因分别占43.86%(993)和29.24%(1 019);上(下)调10—60倍的基因分别占20.76%(470)和12.82%(447);上(下)调60倍以上的基因分别占6.01%(136)和24.29%(713)。从火山图中可以直观地看出对照组与比较样本组之间的表达水平具有显著差异,而且差异60倍以上的下调基因明显多于上调基因(图2)。

表2 样品reads分布情况
1)71、72、73分别代表7叶期的第1、2、3个样品
1)71, 72 and 73 indicate the first, second and third samples of the 7th leaf period respectively
GO功能分析表明,差异表达基因被注释到24个生物过程、22个细胞组分、19个分子功能类别中,差异基因主要富集在细胞膜相关的细胞组分中。其中生物过程显著富集在次生代谢、花粉雌蕊相互作用、花粉识别、防卫反应和细胞识别5个过程(图3);细胞组分分析中差异基因显著富集在细胞外缘、质膜、细胞膜固有成分、细胞膜基本成分、膜部分、共质体、胞间接合和胞间连丝等10个组分类别(图4);分子功能显著富集在单加氧酶活性、氧化还原活性、催化活性、纤维素合成酶活性、ADP结合、碳水化合物结合、铁离子结合、葡糖基转移酶活性等19个类别(图5)。

横坐标为差异表达基因表达倍数变化,纵坐标为表达量变化的统计学显著程度
利用MapMan工具对差异表达基因进行pathway富集分析,差异基因被注释到125条代谢通路中,主要涉及生物合成、能量代谢、信号转导及次生代谢。以-value≤0.05为筛选标准,发现这些基因显著富集到11条代谢路径(表3),其中核糖体路径富集差异基因数目最多达到83个,37个基因表达上调,46个基因下调表达;其次,植物激素信号转导通路富集57个差异基因,24个基因上调,33个基因下调;苯丙烷类代谢路径18个基因上调,15个基因下调;乙醛酸和二羧酸代谢通路15个基因上调,10个基因下调;谷胱甘肽代谢路径13个基因上调,10个基因下调;鞘脂类代谢路径2个基因上调,11个基因下调(表4);油菜素内酯生物合成路径4个基因上调,3个基因下调(表5);类黄酮生物合成路径6个基因上调,2个基因下调;苯并恶唑嗪类生物合成和苯乙烯类、二芳庚酸类、姜酚类生物合成通路也富集到较多差异基因,分别有7个和5个基因下调表达。基于不同代谢通路中基因上下调比例,推断核糖体、植物激素信号转导、鞘脂类代谢这些基因下调表达较多的路径可能与叶片发育,尤其是窄叶的形成密切相关。

Size代表圆点的大小,表示富集到该GO条目基因的数量,q-value表示该GO条目的富集程度,颜色越趋近于红色表示富集程度越高。下同

图4 差异表达基因显著富集的细胞组分

表3 差异表达基因显著富集的代谢通路
植物内源激素广泛参与到叶片发育过程,对叶片形态建成起到一定调节作用。进一步分析植物激素信号转导路径发现,在叶宽形成的关键部位检测到大量参与激素信号响应和转导的差异基因,涉及生长素、油菜素内酯、细胞分裂素、赤霉素、脱落酸、乙烯、茉莉酸等多种激素(图6)。分析不同响应路径(表5和图6)发现,生长素信号转导通路富集到18个差异基因,有12个响应基因表达上调;油菜素内酯信号响应路径富集到6个差异基因,全部下调表达;细胞分裂素响应路径富集的3个基因,表达均上调;赤霉素和水杨酸响应通路分别富集到1个差异基因,表达均上调;脱落酸路径富集到8个基因下调表达,4个上调表达基因;乙烯路径中4个基因上调表达,4个基因下调表达;茉莉酸路径中1个基因上调表达,7个基因下调表达。

表4 鞘脂类代谢路径及其相关基因
FC:表达量差异倍数(NL409/WB665)
FC:Differential multiple of expression (NL409/WB665)

图5 差异表达基因显著富集的分子功能
2.3 荧光定量PCR验证RNA-Seq结果
研究表明多种转录因子参与叶细胞分化及叶片形态建成,如YABBY、MADS类转录因子决定叶原细胞命运向不同方向分化形成不同组织,GRF类调控特定部位细胞的增殖和生长;ARF、SBP、TCP、Myb、HB类调控叶细胞的分化和维管的形成;同时ARF、ARR、GRAS类在叶片发育过程分别参与生长素、细胞分裂素、赤霉素信号的响应和生物合成,影响叶片形态建成。为了进一步验证RNA-Seq结果的准确性,选取与叶片发育密切相关的18个不同的基因进行了qRT-PCR定量分析。对qRT-PCR与RNA-Seq结果进行相关性分析,发现两者的相关系数2=0.962,说明RNA-Seq结果良好。
3 讨论
3.1 叶片发育密切相关的代谢通路
多条路径显著富集到大量与玉米叶片发育相关的基因。拟南芥()和()分别编码不同亚族的核糖体蛋白(PRS),与rRNA形成蛋白复合物促进叶片近轴组织的建立[20-21]。本文核糖体路径显著富集到多个和(电子附表1),并鉴定出玉米()启动子区域存在显著差异(电子附图1),其在窄叶中下调表达的作用模式(图7)与拟南芥、缺失突变导致叶柄变长、叶片变窄的作用机理相似。鞘脂类(Sphingolipid)是细胞膜骨架及脂蛋白的重要组成成分,鞘脂代谢在细胞生长、不同功能细胞分化、细胞内以及细胞间信号传递发挥重要作用,鞘脂合成紊乱含量较低时,能够激活MAPK(mitogen-activated protein kinase)路径和转录因子AP1(APETALA1),进而调控细胞增殖、分化[22-23]。抑制拟南芥花萼叶和茎叶形成,()能够解除的抑制作用,促进叶片发育[24]。本文鞘脂代谢路径富集到的基因大部分下调表达(表4),类和MAPK类基因表达上调(表4),表达下调(表4),与窄叶发育受抑制的表型一致。研究人员分析叶片发育的动态转录组学,不仅发现大量激素信号基因,细胞壁合成前体、纤维素合成酶、蛋白酶体、翻译后修饰基因也被大量检测到[25]。Bolduc等[9]解析调控网络,不仅在叶片和分生组织中发现大量激素合成和响应基因,而且富集到脂类代谢、次生代谢、细胞壁合成和调控RNA转录相关的基因。叶片发育过程不同部位、不同时期的蛋白质组学分析,也富集到大量的尿苷二磷酸-葡糖基/葡聚糖转移酶、细胞壁前体、细胞壁受体激酶、纤维素合成酶和RNA转录相关的基因[26],本文显著富集的路径与前人的研究结果一致。由此可知,上述显著富集的代谢通路(表3)与叶片发育密切相关,这些代谢路径中的基因可能参与叶宽、叶长的形态建成。

Tryptophan metabolism:色氨酸代谢;Auxin:生长素;Ubiquitin mediated proteolysis:泛素介导的蛋白水解;Cell enlargement:细胞增大;Plant growth:植物生长;Zeatin biosynthesis:玉米素的生物合成;Cytokinine:细胞分裂素;Cell division:细胞分裂;Shoot initiation:根的起始;Diterpenoid biosynthesis:二萜类化合物的生物合成;Gibberellin:赤霉素;Stem growth:茎的生长;Induced germination:诱导萌发;Carotenoid biosynthesis:类胡萝卜素的生物合成;Abscisic acid:脱落酸;Stomatal closure:气孔闭合;Seed dormancy:种子休眠;Cysteine and methionine metabolism:半胱氨酸和蛋氨酸代谢;Ethylene:乙烯;Endoplasmic reticulum(ER):内质网;Fruit ripening:果实成熟;Senescence:衰老;Brassinosteroid biosynthesis:油菜素内酯生物合成;Brassinosteroid:油菜素甾醇;Proteasomal degradation:蛋白酶体降解;Cell elongation:细胞伸长;Alpha-Linolenic acid metabolism:α-亚麻酸代谢;Jasmonic acid:茉莉酸;Monoterpenoid biosynthesis类单萜生物合成;Indole alkaloid biosynthesis:吲哚生物碱生物合成;Stress response:应激反应;Phenylalanine metabolism:苯丙氨酸代谢;Salicylic acid:水杨酸;Disease resistance:抗病性。红色表示富集到的差异基因表达上调,绿色表示基因下调Red indicates that DEGs are up-regulated, and green indicates down-regulation of DEGs

表5 植物激素信号转导路径相关基因
FC:表达量差异倍数(NL409/WB665);BR:油菜素内酯Brassinosteroid;CTK:细胞分裂素Cytokinine;GAs:赤霉素信号Gibberellin signaling;GAmrg:赤霉素代谢相关基因Gibberellin metabolism related genes

图7 RNA-Seq与qRT-PCR结果比较
本研究发现油菜素内酯信号转导路径显著富集的基因全部下调(表5),不仅检测到前人已研究表达量降低的BZR类和BRI类基因[9],还发现BR路径中BSK类、TCH类基因也下调表达。生长素信号转导路径富集到的基因大部分上调表达(表5),其中ARF(GRMZM2G078274、GRMZM2G030710和GRMZM5G874163)、AUX-IAA(GRMZM2G104176、GRMZM2G077356、GRMZM2G079957、GRMZM2G074427)和Auxin transporter(GRMZM2G127949)在调控叶片发育过程中发挥着重要作用[9]。此外,高表达时,抑制GA信号及叶基部细胞的生长[25],本文DELLA家族蛋白(GRMZM2G013016)在近基部窄叶中高表达(表5,图6),且赤霉素代谢相关的基因几乎全部下调表达(表5),这与窄叶发育受抑制的结果一致。由此推断油菜素内酯、生长素、赤霉素在叶片发育过程发挥重要作用。
3.2 不同激素途径发掘的叶形重要功能基因
植物器官发育过程中极性建立是器官形态建成的核心。叶片的发育起始于叶原基细胞的分裂增殖,伴随着一系列特定基因的精确调控,叶原基建立多维空间的极性轴向,促使叶原基细胞朝特定的方向分化增殖,并最终影响叶片的形态和大小。
TCP类转录因子通过AUX、BR、GA、CTK等多种激素调控叶片、花发育以及植株形态建成等生长过程。叶片发育过程可调控影响拟南芥叶片及植株形态,而BRI是发挥功能所必需的;此外,多个负向调控叶片边缘区细胞分化增殖,其中、分别负调控、和、影响生长素分布及含量[9,27-28,31]。过量表达TCP II型基因()使西红柿和拟南芥的叶片变小,或表现卷曲叶[27-28],拟南芥的同源基因/(AC205574.3_FG006)在窄叶中上调表达的作用模式(图7,电子附表1)与前人研究结果一致。因此,推测/可能影响多种激素调控玉米叶片发育。
激素BR可影响玉米叶片形态建成。编码一种细胞色素P450蛋白,作用于叶片纵向极性细胞的伸长生长,其功能缺失影响拟南芥叶柄变短,叶片变大变宽[29],拟南芥BR合成相关的P450家族和突变后影响叶细胞变短变少[30-31]。(VIVIPAROUS1)、(VP1/ABI3-LIKE)、(Related to ABI3/VP1)、(Related to ABI3/ VP1-Like)分别编码不同的B3结构DNA结合蛋白,拟南芥缺失突变后影响叶片变小且赤霉素含量降低[32],作为BR受体负向调控拟南芥莲座叶的生长[33],与本文中下调和表达上调(电子附表2),以及赤霉素路径基因下调表达的结果一致(表5)。通过E-box(CACCTG/ CACGTG/CACATG)结合位点激活BR响应和合成基因的表达,维持BR平衡对生长的调节作用[34-35],本文中BR响应基因的启动子区域存在大量该元件(电子附图4—9),说明植物激素BR与叶片发育密切相关。此外,(GRMZM2G143235)和(GRMZM2G344521)在叶宽形成的关键部位上调表达(图7),且位于已定位的叶宽区间内[36],同时启动子区域的序列差异(电子附图2),表明和可能在叶长和叶宽发育过程中发挥着重要作用,由此推测BR通过某种途径影响玉米叶长、叶宽的形成。
生长素合成、分布异常影响叶片形态。玉米与水稻()和()同源,、、和编码一种生长素合成酶,调控叶原基的形成,其功能缺失突变影响叶细胞的分裂和维管束的形成,导致水稻叶片变窄[37-40],与本文在窄叶中下调表达(图7)的作用模式一致。编码一种AGO7型蛋白,是合成ta-siARF所必需的,通过ta-siARF调控的转录,影响玉米叶片腹背面生长素分布,导致突变体叶片变窄卷曲[41]。此外,位于叶宽研究的热点区域[4],而且其启动子区域序列存在显著差异(电子附图3)。由此可知,(GRMZM2G480386)、e(GRMZM5G892991)可能通过影响生长素调控窄叶的形成。
玉米叶片发育过程大量与生长素相关的基因参与其形成。玉米的ChIP分析富集大量生长素AUX-IAA和ARF基因,转录组分析发现生长素基因(、)在玉米叶细胞朝不同功能区生长的过渡区上调表达[9],与本研究中、(图7)表达结果一致。拟南芥受叶原基近轴端特异表达tasiR-ARF的抑制,在近轴端不表达,在远轴端()与、相互作用的同时,影响、和表达上调,促进叶原细胞的分化及远轴组织的形成[42-43];miR165/166抑制HD-ZIP lll类基因、在叶原基远轴端的表达,促进近轴组织的形成[44]。、和(图7)位于已定位的叶宽位点内[4,36]。此外,、、(图7)分别受miR167、miR164、miR166调控并且与叶长、叶宽位点显著相关[36],由此可知(GRMZM2G017709)、(GRMZM5G874163)、(GRMZM2G175827)、(GRMZM2G078274)、(GRMZM2G055585)、(GRMZM2G029692)在叶片发育过程可能发挥特定作用。
植物激素间的的相互作用可调节叶片发育。BR信号路径中的BIN2激酶能够磷酸化ARF2,导致其结合Auxin响应元件的功能丧失,影响拟南芥叶片等器官增大[45-46],同时BR能够诱导BZR1靶基因的表达[47]。与相互作用促进拟南芥下胚轴变长[48]。Auxin能够诱导拟南芥表达,同时通过BR路径促进细胞分裂生长[49]。此外,Auxin还可促进表达,加强BR信号转导,而拟南芥通过影响生长素促进细胞膨大生长[27],ARF能够特异结合Auxin响应元件(TGTCTC)[50],本文BR响应基因启动子富含该元件(电子附图4—电子附图9),而且BR响应基因全部下调,Auxin响应基因上调(表5,图7),这暗示本文中(GRMZM2G030710)、(GRMZM2G078274)(GRMZM2G065635)(GRMZM2G15877)(表5,电子附表1)可能介导Auxin和BR的平衡来调节植物器官以及叶片的发育。
HB类转录因子包含多个KNOX、bHLH和HD-ZIP lll基因。()和属于KNOX家族,在顶端分生组织特定表达,调控细胞分化向膨大生长的过渡,影响叶片多维空间的发育,尤其在基部-末端轴向的形成过程中,与、、相互作用调控叶片发育[9]。KNOX蛋白能够促进细胞分裂素(CTK)合成基因上调,同时抑制GA3和GA20氧化酶合成,促进SAM细胞分裂形成叶原基[9,51-52];提高GA含量可以解除DELLA对叶片发育的抑制作用[52]。与本文中KNOX家族基因(和)(图7)、CTK路径基因(表5)、DELLA基因上调(表5),GA氧化酶基因下调(表5)影响叶片变窄的结果一致。GA可通过DELLA蛋白负向调控CTK响应基因进而影响CTK变化[53]。拟南芥GA信号的抑制因子SPY(SPINDLY)能够正向调控CTK促进植株及叶片的发育[54]。由此可知(GRMZM2G017709)和(GRMZM2G433591)可能通过影响细胞分裂素与赤霉素的平衡参与玉米窄叶的形成。
生长调节因子GRF的互作因子AN3/GIF1作为叶片发育过程的调节开关,主要影响叶细胞横向分裂,分析其调控机理,在叶片基部分裂区富集到大量上调表达的GRF基因,在叶部伸长区仅发现和高量表达,其他GRF基因在叶部伸长区表达量逐渐降低,过量表达和使玉米叶片变小[13-14],与本文中检测到(GRMZM2G041223)和(GRMZM2G018414)在窄叶叶片发育的过渡区域上调表达(图7)的趋势一致,同时和位于叶宽研究的热点区域[4]。(GRMZM2G143235)编码一种TGA结合域的碱性亮氨酸拉链蛋白,主要在SAM和叶片与叶鞘连接处表达,作用于叶基部叶耳、叶舌的形成,影响叶片的发育[55]。ZmARF2(GRMZM2G082836)是ADP核糖基化转录因子,能够调控拟南芥叶面积和株高[56]。由此推断、、、(图7)在玉米叶片发育以及叶宽形成过程中可能发挥重要作用。
4 结论
明确了一些与玉米叶片发育密切相关的代谢路径,发现植物激素间的动态平衡对叶片发育有着重要影响,尤其是Auxin与BR、CKT与GA之间的相互作用对调控叶片形态可能发挥重要作用。
[1] 明博, 谢瑞芝, 侯鹏, 李璐璐, 王克如, 李少昆. 2005—2016年中国玉米种植密度变化分析. 中国农业科学, 2017, 50(11): 1960-1972.
MING B, XIE R Z, HOU P, LI L L, WANG K R, LI S K. Changes of maize planting density in China., 2017, 50(11): 1960-1972. (in Chinese)
[2] 郭书磊, 陈娜娜, 齐建双, 岳润清, 韩小花, 燕树锋, 卢彩霞, 傅晓雷, 郭新海, 铁双贵. 不同密度下玉米倒伏相关性状与产量的研究. 玉米科学, 2018, 26(5): 71-77.
GUO S L, CHEN N N, QI J S, YUE R Q, HAN X H, YAN S F, LU C X, FU X L, GUO X H, TIE S G. Study on the relationship between yield and lodging traits of maize under different planting densities., 2018, 26(5): 71-77. (in Chinese)
[3] YANG C W, XIE F M, JIANG Y P, LI Z, HUANG X, LI L. Phytochrome A negatively regulates the shade avoidance response by increasing auxin/indole acidic acid protein stability., 2018, 44(1): 29-41.
[4] 郭书磊, 张君, 齐建双,岳润清, 韩小花, 燕树锋, 卢彩霞, 傅晓雷, 陈娜娜, 库丽霞, 铁双贵. 玉米叶形相关性状的Meta-QTL及候选基因分析. 植物学报, 2018, 53(4): 487-501.
GUO S L, ZHANG J, QI J S,YUE R Q, HAN X H, YAN S F, LU C X, FU X L, CHEN N N, KU L X, TIE S G. Analysis of meta-quantitative trait loci and their candidate genes related to leaf shape in maize., 2018, 53(4): 487-501. (in Chinese)
[5] 崔晓峰, 黄海. 叶发育的遗传调控机理研究进展. 植物生理学报, 2011, 47(7): 631-640.
CUI X F, HUANG H. Recent progresses from studies of leaf development., 2011, 47(7): 631-640. (in Chinese)
[6] MORENO M A, HARPER L C, KRUEGER R W, DELLAPORTA S L, FREELING M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis., 1997, 11(5): 616-628.
[7] WALSH J, WATERS C A, FREELING M. The maize geneliguleless2 encodes a basic leucine zipper protein involved in the establishment of the leaf blade-sheath boundary., 1998, 12(2): 208-218.
[8] ZHANG X L, MADI S, BORSUK L, NETTLETON D, ELSHIRE R J, BUCKNER B, BUCKNER D J, BECK J, TIMMERMANS M, SCHNABLE P S, SCANLON M J. Laser microdissection of narrow sheath mutant maize uncovers novel gene expression in the shoot apical meristem., 2007, 3(6): 1040-1052.
[9] BOLDUC N, YILMAZ A, MEJIA-GUERRA M K, MOROHASHI K, O'CONNOR D, GROTEWOLD E, HAKE S. Unraveling the KNOTTED1 regulatory network in maize meristems., 2012, 26(15): 1685-1690.
[10] TIMMERMANS M C, SCHULTES N P, JANKOVSKY J P, NELSON T. Leafbladeless1 is required for dorsoventrality of lateral organs in maize., 1998, 125(15): 2813-2823.
[11] DOUGLAS R N, WILEY D, SARKAR A, SPRINGER N, TIMMERMANS MARJA C P, SCANLON M J. Ragged seedling2 Encodes an ARGONAUTE7-like protein required for mediolateral expansion, but not dorsiventrality, of maize leaves., 2010, 22(5): 1441-1451.
[12] Candela H, Johnston R, Gerhold A, FOSTER T, HAKE S. The milkweed pod1 gene encodes a KANADI protein that is required for abaxial/adaxial patterning in maize leaves., 2008, 20(8): 2073-2087.
[13] NELISSEN H, EECKHOUT D, DEMUYNCK K, PERSIAU G, WALTON A, BEL M V, VERVOORT M, CANDAELE J, BLOCK J D, AESAERT S, LIJSEBETTENS M V, GOORMACHTIG S, VANDEPOELE K, LEENE J V, MUSZYNSKI M, GEVAERT K, INZE D, JAEGER G D. Dynamic changes in ANGUSTIFOLIA3 complex composition reveal a growth regulatory mechanism in the maize leaf., 2015, 27(6): 1605-1619.
[14] WU L, ZHANG D, XUE M, QIAN J J, HE Y, WANG S C. Overexpression of the maize GRF10, an endogenous truncated growth-regulating factor protein, leads to reduction in leaf size and plant height., 2014, 56(11): 1053-1063.
[15] 吴庆飞, 秦磊, 董雷, 丁泽红, 李平华, 杜柏娟. 玉米光合突变体hcf136(high chlorophyll fluorescence 136)的转录组分析. 作物学报, 2018, 44(4): 493-504.
WU Q F, QIN L, DONG L, DING Z H, LI P H, DU B J. Transcriptome analysis on a maize photosynthetic mutant hcf136 (high chlorophyll fluorescence 136)., 2018, 44(4): 493-504. (in Chinese)
[16] 邱化荣, 周茜茜, 何晓文, 张宗营, 张世忠, 陈学森, 吴树敬. 基于转录组分析苹果水杨酸特异响应基因MdWRKY40的启动子鉴定. 中国农业科学, 2017, 50(20): 3970-3990.
QIU H R, ZHOU Q Q, HE X W, ZHANG Z Y, ZHANG S Z, CHEN X S, WU S J. Identification of MdWRKY40 promoter specific response to salicylic acid by transcriptome sequencing., 2017, 50(20): 3970-3990. (in Chinese)
[17] TRAPNELL C, WILLAMS B A, PERTEA G, MORTAZAVI A, KWAN G, BAREN M J, SALZBERG S L, WOLD B JHTER L. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation., 2010, 28(5): 511-515.
[18] WANG L K, FENG Z X, WANG X, WANG X W, ZHANG X G. DEGseq: an R package for identifying differentially expressed genes from RNA-seq data., 2009, 26(1): 136-138.
[19] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod., 2001, 25(4): 402-408.
[20] SCHIPPERS J H M, MUELLER-ROEBER B. Ribosomal composition and control of leaf development., 2010, 179(4): 307-315.
[21] YAO Y, LING Q H, WANG H, HUANG H. Ribosomal proteins promote leaf adaxial identity., 2008, 135(7): 1325-1334.
[22] SPIEGEL S, MERRILL Jr A H. Sphingolipid metabolism and cell growth regulation., 1996, 10(12): 1388-1397.
[23] MERRILL Jr A H, SANDHOFF K. Sphingolipids: Metabolism and cell signaling//.Amsterdam, Elsevier Science Press, 2002, 373-407.
[24] MONNIAUX M, MCKIM S M, CARTOLANO M, THEVENON E, PARCY F, MILTANTIS T, HAY A. Conservation vs divergence in LEAFY and APETALA1 functions betweenand., 2017, 216(2): 549-561.
[25] LI P H, PONNALA L, GANDOTRA N, WANG L, SI Y Q, TAUSTA S L, KEBROM T H, PROVART N, PATEL R, MYERS C R, REIDEL E J, TURGEON R, LIU P, SUN Q, NELSON T, Brutnell T P. The developmental dynamics of the maize leaf transcriptome., 2010, 42(12): 1060-1067.
[26] FACETTE M R, SHEN Z X, Björnsdóttir F R, BRIGGS S P, SMITH L G. Parallel proteomic and phosphoproteomic analyses of successive stages of maize leaf development., 2013: 2798-2812.
[27] NICOLAS M, CUBAS P. The role of TCP transcription factors in shaping flower structure, leaf morphology, and plant architecture//:,. Cambridge, MA: Academic Press, 2016: 249-267.
[28] MANASSERO N G U, VIOLA I L, WELCHEN E, GONZALEZ D H. TCP transcription factors: architectures of plant form., 2013, 4(2): 111-127.
[29] KIM G T, TSUKAYA H, UCHIMIYA H. The ROTUNDIFOLIA3 gene ofencodes a new member of the cytochrome P-450 family that is required for the regulated polar elongation of leaf cells., 1998, 12(15): 2381-2391.
[30] FUJIOKA S, LI J, CHOI Y H, SETO H, TAKATSUTO S, NOGUCHI T, WATANABE T, Kuriyama H, YOKOT T, CHORY J, SAKURAI A. Thedeetiolated2 mutant is blocked early in brassinosteroid biosynthesis., 1997, 9(11): 1951-1962.
[31] GUO Z, FUJIOKA S, BLANCAFLOR E B, MIAO S, GOU X P, LI J. TCP1 modulates brassinosteroid biosynthesis by regulating the expression of the key biosynthetic gene DWARF4 in., 2010, 22(4): 1161-1173.
[32] SUZUK M, WANG H H Y, MCCARTY D R. Repression of the LEAFY COTYLEDON 1/B3 regulatory network in plant embryo development by VP1/ABSCISIC ACID INSENSITIVE 3-LIKE B3 genes., 2007, 143(2): 902-911.
[33] HU Y X, WANG Y H, LIU X F, LI J Y.RAV1 is down-regulated by brassinosteroid and may act as a negative regulator during plant development., 2004, 14(1): 8.
[34] YUAN W Y, LUO X, LI Z C, YANG W N, WANG Y Z, LIU R, DU J M, HE Y H. A cis cold memory element and a trans epigenome reader mediate Polycomb silencing of FLC by vernalization in., 2016, 48(12): 1527-1534.
[35] JE B I, PIAO H L, PARK S J,PARK S H, KIM C M, XUAN Y H, PARK S H, HUANG J, CHOI Y D, AN G, WONG H L, FUJIOKA S, KIM M C, SHIMAMOTO K, HAN C. RAV-Like1 maintains brassinosteroid homeostasis via the coordinated activation of BRI1 and biosynthetic genes in rice., 2010, 22(6): 1777-1791.
[36] TIAN F, BRADBURY P J, BROWN P J, HUNG H Y, SUN Q, FLINT-GARCIN S, ROCHEFORD T R, MCMULLEN M D, HOLLAND J B, BUCKLER E S. Genome-wide association study of leaf architecture in the maize nested association mapping population., 2011, 43(2): 159-165.
[37] Ishiwata A, Ozawa M, Nagasaki H, KATO M, NODA Y, YAMAGUCHI T, NOSAKA M, SHIMIZU-SATO S,NAGASAKI A,MAEKAWA M,HIRANO H Y,SATO Y. Two WUSCHEL-related homeobox genes, narrow leaf2 and narrow leaf3, control leaf width in rice., 2013, 54(5): 779-792.
[38] KUBO F C, YASUI Y, KUMAMARU T, SATO Y, HIRANO H Y. Genetic analysis of rice mutants responsible for narrow leaf phenotype and reduced vein number., 2016, 91(4): 235-240.
[39] JIANG D, FANG J J, LOU L, ZHAO J F, YUAN S J, YIN L, SUN W, PENG L X, GUO B T, LI X Y. Characterization of a null allelic mutant of the rice NAL1 gene reveals its role in regulating cell division., 2015, 10(2): e0118169.
[40] FUJINO K, MATSUDA Y, OZAWA K, NISHIMURA T, KOSHIBA T, FRAAIJE M W, SEKIGUCHI H. NARROW LEAF 7 controls leaf shape mediated by auxin in rice., 2008, 279(5): 499-507.
[41] DOUGLAS R N, WILEY D, SARKAR A, SPRINGER N, TIMMERMANSM C P, SCANLON M J.ragged seedling2 encodes an ARGONAUTE7-like protein required for mediolateral expansion, but not dorsiventrality, of maize leaves., 2010: 1441-1451.
[42] DOTTO M C, PETSCH K A, AUKERMAN M J, BEATTY M, HAMMELL M, TIMMERMANS M C P. Genome-wide analysis of leafbladeless1-regulated and phased small RNAs underscores the importance of the TAS3 ta-siRNA pathway to maize development., 2014, 10(12): e1004826.
[43] XIE Y, STRAUB D, EGUEN T, BRANDT R, STAHL M, Jaime F. MARTINEZ-GAECIA J F, WENKEL S. Meta-analysis ofKANADI1 direct target genes identifies a basic growth-promoting module acting upstream of hormonal signaling pathways., 2015, 169(2): 1240-1253.
[44] EMERY J F, FLOYD S K, AIVAREZ J, ESHED Y, HAWKER N P, IZHAKI A, BAUM S F, BOWMAN J L. Radial patterning ofshoots by class III HD-ZIP and KANADI genes., 2003, 13(20): 1768-1774.
[45] OKUSHIMA Y, MITINA I, QUACH H L, THEOLOGIS A. AUXIN RESPONSE FACTOR 2 (ARF2): a pleiotropic developmental regulator., 2005, 43(1): 29-46.
[46] SCHRUFF M C, SPIELMAN M, TIWARI S, ADAMS S, FENBY N, SCOTT R J. The AUXIN RESPONSE FACTOR 2 gene oflinks auxin signalling, cell division, and the size of seeds and other organs., 2006, 133(2): 251-261.
[47] TIAN H Y, LV B S, DING T T, BAI M Y, DING Z J. Auxin-BR interaction regulates plant growth and development., 2018, 8: 2256.
[48] OH E, ZHU J Y, BAI M Y, ARENHART R A, SUN Y, WANG Z Y. Cell elongation is regulated through a central circuit of interacting transcription factors in thehypocotyl., 2014, 3: e03031.
[49] CHUNG Y, MAHARJAN P M, LEE O, FUJIOKA S, JANG S, KIM B, TAKATSUTO S, TSUJIMOTO M, KIM H, CHO S, PARK T, CHO H, HWANG I, CHOE S. Auxin stimulates DWARF4 expression and brassinosteroid biosynthesis in., 2011, 66(4): 564-578.
[50] SAKAMOTO T, MORINAKA Y, INUKAI Y, KITANO H, FUJIOKA S. Auxin signal transcription factor regulates expression of the brassinosteroid receptor gene in rice., 2013, 73(4): 676-688.
[51] JASINSKI S, PIAZZA P, CRAFT J, HAY A, WOOLLEY L, RIEU I, PHILLIPS A, HEDDEN P, TSIANTIS M. KNOX action inis mediated by coordinate regulation of cytokinin and gibberellin activities., 2005, 15(17): 1560-1565.
[52] NELISSEN H, RYMEN B, JIKUMARU Y, DEMUYNCK K, LIJSEBETTENS M V, KAMIYA Y, INZE D, BEEMSTER G T S. A local maximum in gibberellin levels regulates maize leaf growth by spatial control of cell division., 2012, 22(13): 1183-1187.
[53] FONOUNI-FARDE C, KISIALA A, BRAULT M, EMERY R J N, ANOUCK D, FLORIAN F. DELLA1-mediated gibberellin signaling regulates cytokinin-dependent symbiotic nodulation., 2017, 175(4): 1795-1806.
[54] GREENBOIM-WAINBERG Y, MAYMON I, BOROCHOV R, ALVAREZ J, OLSZEWSKI N, ORI N, ESHED Y, WEISS D. Cross talk between gibberellin and cytokinin: theGA response inhibitor SPINDLY plays a positive role in cytokinin signaling., 2005, 17(1): 92-102.
[55] WALSH J, WATERS C A, FREELING M. The maize geneliguleless2 encodes a basic leucine zipper protein involved in the establishment of the leaf blade-sheath boundary., 1998, 12(2): 208-218.
[56] WANG Q L, XUE X J, LI Y L, DONG Y B, ZHANG L, ZHOU Q, DENG F, MA Z Y, QIAO D H, HU C H, REN Y L. A maize ADP-ribosylation factor ZmArf2 increases organ and seed size by promoting cell expansion in., 2016, 156(1): 97-107.
Explore Regulatory Genes Related to Maize Leaf Morphogenesis Using RNA-Seq
GUO ShuLei1,2, LU XiaoMin1, QI JianShuang1, WEI LiangMing1, ZHANG Xin1, HAN XiaoHua1, YUE RunQing1, WANG ZhenHua1, TIE ShuangGui1, CHEN YanHui2
(1Cereal Crops Institute, Henan Academy of Agricultural Sciences/Henan Provincial Key Lab of Maize Biology, Zhengzhou 450002;2College of Agronomy, Henan Agricultural University, Zhengzhou 450046)
【】Leaf shape characteristics are one of important agronomic traits that determine plant morphology and affect planting density. However, the molecular mechanism related to leaf shape remain unknown in maize. Here, transcriptome sequencing technology was used to screen and explore genome-wide analysis of regulatory genes and metabolic pathways involved in leaf morphogenesis. This study will lay the foundation for further understanding the regulator mechanism of leaf development in plant and identifying candidate genes of leaf shapes, such as leaf width and leaf length.【】Extreme narrow-leaf inbred line NL409 and wide-leaf line WB665 were selected as the experimental materials. By RNA-Seq technology, the differentially expressed genes (DEGs) of the seventh leaf base between these two lines were identified during the 7th leaf stage. Furthermore, metabolic pathways closely related to leaf development were also analyzed using a series of bioinformatics analysis. qRT-PCR was used to validate the expression level of DEGs in different hormone pathways, and the further promoter analysis were performed to explore leaf-shape functional genes.【】By analyzing the high-throughput sequencing in WB665 and NL409, a total of 5 199 DEGs were obtained at the primary section of leaf width formation. Of which, 2 264 (43.55%) genes were up-regulated, whereas down-regulated genes were significantly more than up-regulated genes with 2 935 (56.45%) decreased genes. GO enrichment analysis showed that these DEGs were mainly enriched in cell membrane-associated function terms of cellular components, including metabolic process and cell stimulus response. KEGG enrichment analysis showed that these DEGs were mainly involved in ribosome, plant hormone signal transduction, sphingolipid metabolism pathways, phenylpropanoid biosynthesis, glyoxylate and dicarboxylate metabolism and other processes. among which ribosome, plant hormone signal transduction, sphingolipid metabolism pathways with more down-regulated genes were closely related to leaf development. One of PRS (PRESSED FLOWER) family genes, which were enriched in the ribosomal pathway in this study,PRS13 (PFL2) was identified to participate in regulating the development of narrow leaves. The expression pattern of genes enriched in sphingolipid metabolism pathway and its related MAP kinase, AP1-like, and LFY-like were consistent with the result of the inhibited development of narrow leaves. Notably, all of BR (Brassinosteroid) response genes and most of GA (Gibberellin) metabolic genes were down-regulated in plant hormone signal transduction pathway, while the expression level of all the CTK (Cytokinine) response genes and Auxin genes are mostly increased. The action of up-regulated expression of DELLA protein gene affecting the GA and CTK pathways was consistent with the phenotypic result of narrow leaves. Eighteen genes were validated by qRT-PCR. The result showed that the expression trend was consistent with the transcriptome data. Moreover, the BR-related, auxin-related,and TCP-like transcription factorswere identified to be closely associated with the formation of narrow leaves.【】Summarily, this study unveils several metabolic pathways closely related to leaf development in maize, and find the dynamic balance between plant hormones plays an important role in leaf development, especially the interaction between Auxin and BR as well as CTK and GA.
maize (); leaf width; leaf length; RNA-Seq; morphogenesis; regulatory gene
10.3864/j.issn.0578-1752.2020.01.001

2019-05-20;
2019-07-11
中国博士后科学基金(2017M612404)、河南省科技攻关计划项目(182102110122)、河南省农业科学院优秀青年科技基金
郭书磊,E-mail:guosl1309@163.com。通信作者王振华,E-mail:wzh201@126.com。通信作者铁双贵,E-mail:tieshuangg@126.com。通信作者陈彦惠,E-mail:chy9890@163.com
(责任编辑 李莉)
