NaCl处理谷子萌发期种子的转录组学分析
2019-12-19潘教文李臻王庆国管延安李小波戴绍军丁国华刘炜
潘教文,李臻,王庆国,管延安,李小波, ,戴绍军,丁国华,刘炜,3
NaCl处理谷子萌发期种子的转录组学分析
潘教文1,李臻1,王庆国1,管延安2,3,李小波1, 4,戴绍军5,丁国华4,刘炜1,3
(1山东省农业科学院生物技术研究中心/山东省作物遗传改良与生理生态重点实验室,济南 250100;2山东省农业科学院作物研究所/山东省特色作物工程实验室,济南 250100;3山东师范大学生命科学学院,济南 250014;4哈尔滨师范大学生命科学与技术学院,哈尔滨 150025;5上海师范大学生命与环境科学学院/植物种质资源开发协同创新中心,上海 200234)
【】谷子具有抗旱、耐贫瘠的特性,逐渐成为研究禾本科作物的模式作物。本研究以前期筛选并获得的谷子耐盐品种和敏感品种为材料,通过RNA-Seq测序筛选鉴定谷子的盐胁迫响应基因,揭示其响应盐害机制,为培育谷子抗盐新种质提供理论依据。对14个谷子品种进行了萌发期抗盐筛选。谷子不同品种的种子经150 mmol·L-1NaCl处理萌发培养7 d后,分别统计各品种种子的萌发率、根长和芽长,综合各项指标鉴定到豫谷2为耐盐品种、安04为盐敏感品种。以这两个品种盐胁迫前、后萌发期种子为材料,应用RNA-Seq进行转录组测定,分析鉴定盐害下差异表达基因(differentially expression gene,DEG),并对DEG进行GO和KEGG功能富集分析。同时应用qRT-PCR对随机挑选的15个DEG表达模式进行验证,并与RNA-Seq结果进行相关性分析。耐盐品种豫谷2、盐敏感品种安04种子经盐胁迫处理后,分别鉴定出2 786个和4 413个DEG;其中,每个品种在NaCl处理前及处理后分别鉴定出1 470和3 826个DEG。GO和KEGG聚类分析显示,NaCl处理下参与信号转导、抗氧化、有机酸的合成及转运以及次生代谢等相关的DEG在谷子萌发期种子应对盐胁迫响应过程中具有关键性的作用,DEG主要集中在胁迫响应、离子的跨膜转运、氧化还原、次生代谢及有机酸、多胺、苯丙烷的合成等过程。qRT-PCR对15个DEG表达模式的检测结果与RNA-seq结果相关系数为0.8817。其中编码离子通道的基因()、过氧化物酶基因()、黄烷酮-3-羟化酶基因()、MYB转录因子基因等在豫谷2中表达量较高,显示其在谷子萌发期种子响应盐胁迫中可能发挥一定作用。谷子耐盐品种及盐敏感品种的种子对盐胁迫的响应程度具有一定的差异性,且品种对盐害的耐受能力主要与基因对胁迫的响应程度相关,而与DEG的数量相关性不大。
谷子;盐胁迫;萌发期种子;转录组;分子机制
0 引言
【研究意义】盐渍是灌溉农业面临的严峻问题,严重影响了农作物的地理分布和产量。在世界范围内,大约有800×106万hm2的土地受到盐渍的影响,而且每年增长率为1%—2%。盐害可使植物产生渗透胁迫,进一步导致离子毒害,影响种子萌发、植物正常的生长发育和形态建成、光合作用,并产生次生伤害,造成物质代谢紊乱及过氧化物的积累,严重影响了作物的产量及品质[1-4]。谷子(L. Beauv.)又称为粟,去皮后俗称小米,是起源于中国的传统优势作物,也是粮饲兼用作物。其具有抗旱、耐瘠薄、C4高光效、生育期短,适应性广等突出优势[5-6]。不仅在目前旱作生态农业中有重要作用,在国民经济中也具有重要地位。因此,以谷子为材料,鉴定抗盐基因及其功能,研究基因作用机制,对作物的遗传改良和抗盐品种的培育具有重要意义。【前人研究进展】为了有效地应对盐胁迫,植物在进化过程中形成了一系列参与感知渗透胁迫和离子毒害的机制。其中K+离子通道(high-affinity K+channel,HKT)、非选择性阳离子通道(nonselective cation channel,NSCC)等[7]及胞内第二信使Ca2+在植物应对盐胁迫过程中具有重要作用。部分钙结合蛋白、激酶及转录因子等,也参与了胁迫信号的感知及传递[8]。已知谷子对盐碱有一定的耐受性,研究表明在0.3%盐碱以下的土壤,谷子可以较好地生长发育[9]。近年来,随着谷子基因组测序的完成,谷子被认为是研究禾本科作物新的模式作物。在基因组水平上,各种不同的基因家族,以及干旱响应基因、蛋白及非编码RNA等都被鉴定出来[10-14]。【本研究切入点】谷子抗旱性研究开展的较早,部分研究成果已应用到作物育种过程,并取得了较好的经济及社会效益。例如,通过对谷子耐旱品种豫谷1和敏感品种安04转录组学分析,结合干旱相关的QTL,鉴定到20个谷子耐旱的关键基因[13]。通过miRNA测序结合降解组学分析,12个已知的miRNA家族及其56个靶基因被鉴定到,同时,9个新的miRNAs及其26个靶基因也被鉴定到[12]。利用定量蛋白质组学分析也鉴定到一系列的参与谷子干旱响应的蛋白[14]。谷子抗盐研究起步较晚,其对盐胁迫的响应及抗盐机制仍有待深入挖掘。【拟解决的关键问题】通过对不同谷子品种在萌发期进行耐盐筛选,初步获得盐敏感品种安04和耐盐品种豫谷2。本研究以NaCl处理前后这两个品种为材料,利用RNA-seq进行转录组测序,结合数据分析及归纳,进而揭示谷子的耐盐机制。为解析谷子的抗盐机制及培育谷子抗盐品种提供理论依据和基因资源。
1 材料与方法
1.1 试验材料和处理方法
谷子品种济谷11、济谷12、济谷14、济谷15、济8431、鲁谷1号、鲁谷2号、鲁谷6号、鲁谷8号、豫谷2、豫谷11、豫谷14均由山东省农业科学院作物研究所管延安老师课题组馈赠,豫谷1和安04由中国农业科学院作物研究所刁现民老师课题组馈赠。谷子的种子经10%的次氯酸钠消毒后,于含有两层滤纸的9 cm的无菌培养皿中萌发,分别用灭菌的ddH2O和150 mmol·L-1的NaCl处理,每个培养皿放100粒种子,设置3个生物学重复。种子在光照培养箱(白天14 h 30℃/黑暗10 h 25℃)中培养7 d后,统计萌发率、根长和芽长。之后将去掉胚根及胚芽的萌发期种子经液氮速冻,置于-80℃保存备用。
1.2 RNA文库的构建和测序
萌发期种子总RNA用试剂盒(Total RNA Isolation Kit (China))提取。具体步骤参照说明书。用RNA Nano 6000 Assay Kit of Bioanalyzer 2100 system (Agilent Technologies, CA, USA)检测RNA的完整性。分别取3 μg的RNA,用试剂盒(NEBNext UltraTM RNA Library Prep Kit for Illumina (NEB, USA))构建文库。通过Oligo(dT)磁珠富集带有polyA尾的mRNA,随后在NEB Fragmentation Buffer中用二价阳离子将得到的mRNA随机打断。以片段化的mRNA为模板,随机寡核苷酸为引物,在M-MuLV逆转录酶体系中合成cDNA第一条链,随后用RNaseH降解RNA链,并在DNA polymeraseⅠ体系下,以dNTPs为原料合成cDNA第二条链。纯化后的双链cDNA经过末端修复、加A尾并连接测序接头,用AMPure XP beads筛选200 bp左右的cDNA,进行PCR扩增并再次使用AMPure XP beads纯化PCR产物,每个样品3次重复,最终获得文库。
构建完成的文库经Qubit2.0 Fluorometer进行初步定量,随后使用Agilent 2100 bioanalyzer进行检测,并用qRT-PCR对文库有效浓度进行准确定量。
构建成功的文库经Illumina测序,产生150 bp配对末端读数。经对原始数据过滤,获得高质量及可靠的数据。从基因组网站下载参考基因组和基因模型注释文件,使用Hisat2 v2.0.5构建参考基因组的索引,并使用Hisat2 v2.0.5将配对末端过滤后的数据(clean reads)与参考基因组比对。根据基因的长度和比对到该基因的次数来估算每个基因的FPKM值。
利用DESeq2 R package(1.16.1)进行比较组合之间的差异表达分析(每组3次生物学重复)。使用Benjamini&Hochberg方法调整<0.05,结合阈值(|Log2fold change|≥1)筛选每个比对组合的DEG。
通过clusterProfiler R软件实现DEG的GO富集分析,其中修正了基因的长度偏差,考虑具有小于0.05校正值的GO通过DEG显著富集。也利用clusterProfiler RKEGG软件分析KEGG通路中DEG的系统富集情况。具体参考已报道的信息及相关数据库进行基因的富集分析[13-17]。
1.3 qRT-PCR分析
随机选取15个DEG,对它们在NaCl胁迫下豫谷2和安04中的表达模式进行验证。以作为内参,基因特异引物见电子附表1。利用构建文库的RNA反转录成cDNA,使用FastStart Universal SYBR Green (Roche) Master为荧光染料,进行qRT-PCR分析,反应体系为20 μl,SYBR Green Master 10 μl上游引物1 μl,下游引物1 μl,ddH2o反应条件为95℃ 10 min;95℃ 10 s,56℃ 20 s,72℃ 20 s,40个循环。使用2-ΔΔCt法计算基因的相对表达量。数据处理参考Zhang等[18]方法,利用Pearson’s correlation coefficient(PCC)计算2组数据的相关系数。
2 结果
2.1 NaCl处理豫谷2和安04萌发期种子的转录组分析
以150 mmol·L-1NaCl对14个谷子品种的耐盐性进行筛选。表型观察结合根长、芽长和萌发率分析,发现盐胁迫处理7 d后,安04种子的相对萌发率、根长和芽长分别约为对照的20%、30%和31%,而豫谷2种子的相对萌发率、根长和芽长是对照的60%、70%和51%(图1)。综合各项指标,初步鉴定豫谷2为耐盐品种,安04为盐敏感品种(图2-A)。
利用RNA-seq技术,对豫谷2及安04进行转录组测序。将盐处理前后豫谷2样品分别命名为YG2CKS和YG2NTS,将盐处理前后安04的样品分别命名为An04CKS和An04NTS。结果显示,豫谷2盐处理前后(YG2NTS vs YG2CKS)共鉴定到DEG为2 786个,其中上调基因有1 410个,下调基因有1 376个(电子附表2)。安04盐处理前后(An04NTS vs An04CKS)共鉴定到DEG为4 413个,其中上调基因2 241个,下调基因2 172个(电子附表3)。未经盐处理的2个品种间(YG2CKS vs An04CKS)有DEG为1 470个,其中上调基因540个,下调基因930个(电子附表4)。盐处理后2个品种间(YG2NTS vs An04NTS)共有DEG为3 826个,其中上调基因1 521个,下调基因2 305个(图2-B)(电子附表5)。韦恩图分析显示,盐胁迫处理后豫谷2和安04中共同的DEG为1 278个(图2-C),盐胁迫处理前后豫谷2和安04 2个品种间存在882个共有DEG(图2-C)。
2.2 部分DEG表达模式的qRT-PCR分析
qRT-PCR结果显示,15个随机挑取的DEG在NaCl处理后的安04和豫谷2中的表达模式,除存在差异,其他基因变化趋势基本与RNA-seq结果一致,结果间相关系数为0.8817,具有很好的相关性。表明RNA-Seq结果准确、可信。

图1 经150 mmol·L-1 NaCl处理14个谷子品种种子的相对萌发率(A)、相对根长(B)和相对芽长(C)分析
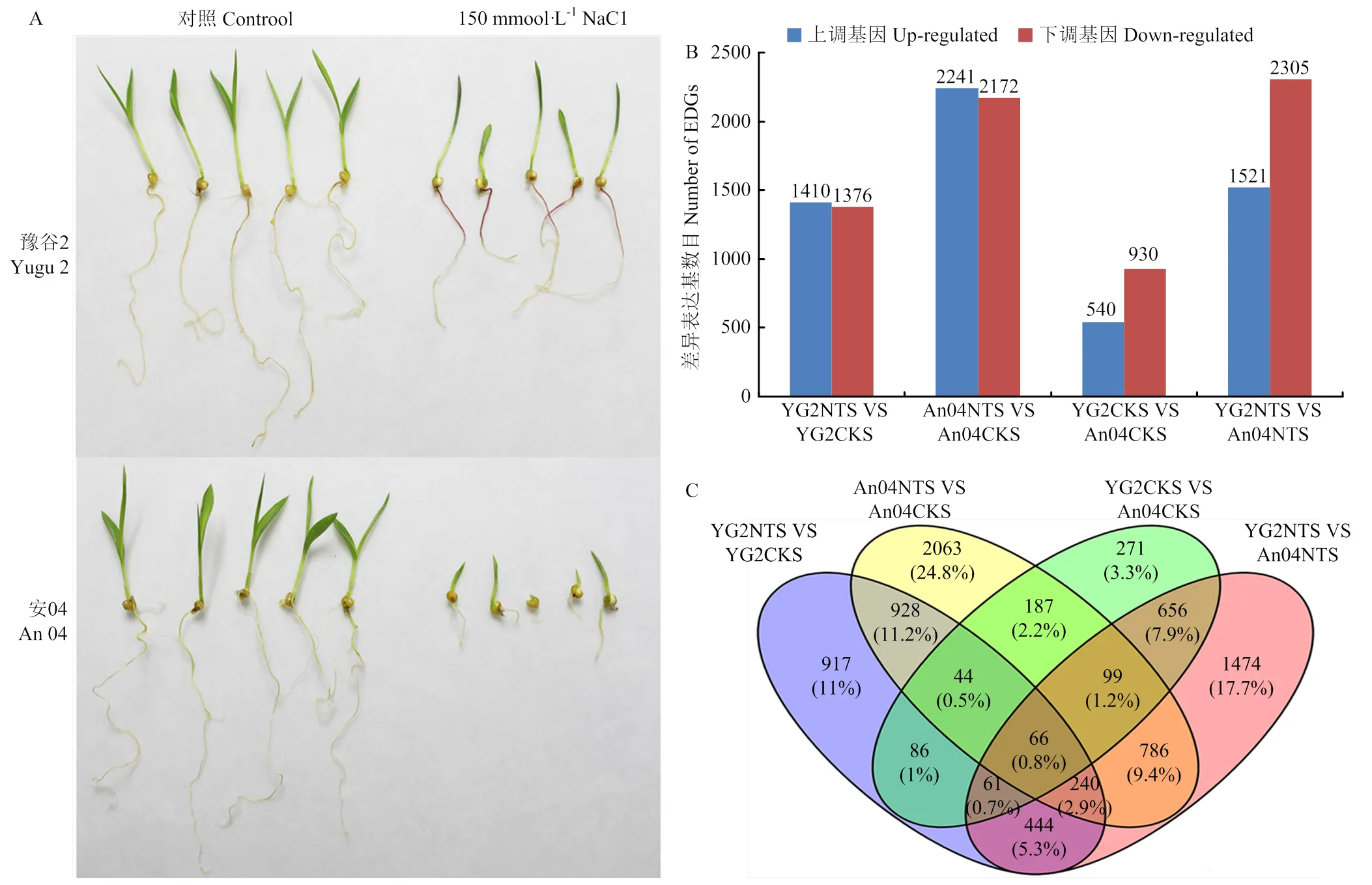
A:150 mmol·L-1 NaCl处理7 d后豫谷2和安04幼苗的表型;B:不同比较组合的DEG统计;C:不同比较组合DEG韦恩图

R值代表RNA-seq和qRT-PCR结果的相关系数 R value indicates the correlation coefficient between data of RNA-seq and qRT-PCR
2.3 DEG功能富集分析
通过对不同比较组鉴定到的DEG进行GO和KEGG代谢途径富集分析。GO富集分析主要分为生物学过程、细胞组分和分子功能3个亚类。DEG主要富集在代谢、细胞学过程、生物调节和刺激响应等生物学过程;DEG主要富集在细胞、细胞膜和细胞器等细胞组分中;DEG主要具有催化、结合和转运的功能(图4)。进一步分析发现,盐胁迫后豫谷2(YG2NTS vs YG2CKS)上调的DEG主要富集在几丁质代谢(GO:0006030)、细胞壁大分子代谢(GO:0044036)、氧化还原(GO:0055114)、多胺代谢(GO:0006595)等过程(电子附表6)。而盐胁迫后安04上调的DEG主要富集在脂肪酸代谢(GO:0006631)、氧化还原(GO:0055114)、有机酸代谢(GO:0006082)等过程(电子附表7)。盐处理后豫谷2下调的DEG主要富集在跨膜运输(GO:0055085)、生物学过程(GO:0008150)、和激素响应(GO:0009725)等过程(电子附表6),而安04下调的DEG主要富集在蛋白磷酸化(GO:0006468)、初级代谢(GO:0044238)和跨膜运输(GO:0055085)等(电子附表7)。鉴于2个品种间的比较能较直接的反应不同品种之间的差异,故对未处理的豫谷2和安04的转录组数据进行了比较(YG2CKS vs An04CKS),结果显示,相对于安04,豫谷2中的基因主要参与各种物质转运过程,如氨基酸跨膜运输(GO:0003333)、离子跨膜转运(GO:0098656)、有机酸跨膜运输(GO:1903825)、和戊糖代谢(GO:0019321)等过程。而安04中的DEG主要富集在细胞碳水化合物生物合成(GO:0034637)(电子附表8)。而盐处理后不同品种间DEG可体现品种对盐胁迫响应的差异,对比显示(YG2NTS vs An04NTS),盐胁迫下豫谷2中上调的DEG主要富集在几丁质代谢(GO:0006030)、胁迫响应(GO:0080134)、离子转运(GO:0006811)和有机酸生物合成(GO:0016053)(电子附表9)。
KEGG富集分析发现,盐胁迫后,豫谷2和安04上调DEG主要富集在精氨酸及脯氨酸代谢、次生代谢物合成、植物激素信号转导、淀粉及糖代谢等途径,显示以上途径可能参与盐胁迫响应(图5、电子附表10和电子附表11)。而下调的DEG主要富集在类苯基丙烷合成、甘油磷脂代谢、氨基酸合成等代谢途径(电子附表10和电子附表11)。品种间的比较显示,盐胁迫处理前(YG2CKS vs An04CKS),即豫谷2中主要富集次生代谢物合成途径,显示该品种本身次生代谢物的合成及含量应高于安04;而盐处理后,各品种上调的DEG显示不同品种对盐胁迫响应情况的不同,其中豫谷2中参与次生代谢、谷胱甘肽代谢及类苯基丙烷合成等途径的DEG明显多于安04,显示这些途径可能参与盐害响应,并对豫谷2抗盐性的形成具有一定作用(电子附表12和电子附表13)。
2.4 部分关键DEG分析
对2个谷子品种盐处理前(YG2CKS vs An04CKS)后(YG2NTS vs An04NTS)DEG分析发现,其主要富集在胁迫相关的GO注释和KEGG途径,可能对谷子抗盐响应及豫谷2品种抗盐性的形成具有重要作用。
盐胁迫处理后有3个编码钙结合蛋白基因(calcium- binding protein,,Seita.3G039700、Seita.8G176000和Seita.4G251700)及3个编码蛋白激酶(CBL-interacting protein kinase,,Seita.3G380200、Seita.9G411700和Seita.8G171900)基因在豫谷2中表达量升高(电子附表14)。编码信号转导相关的类受体蛋白激酶(receptor-like serine/threonine-protein kinase,)基因和促分裂原活化蛋白激酶(mitogen-activated protein kinase kinase kinase,)基因也在豫谷2中具有较高的表达量。显示豫谷2对胁迫信号响应更为迅速。同时,编码离子通道的基因如(potassium transporter、Seita.7G082300和Seita. 5G439500)和(cation transporter,Seita.5G192900和Seita.5G024400)在豫谷2中也表现出明显的盐诱导表达模式(电子附表14)。
盐胁迫导致植物体内活性氧ROS的积累,过量ROS的清除对于植物胁迫抗性非常重要。比对发现,编码活性氧清除酶类和抗氧化蛋白相关的基因如、和硫氧还蛋白基因的表达在豫谷2中高于安04。同时,抗坏血酸-谷胱甘肽循环(AsA-GSH)的组分,如和的表达在盐胁迫后的豫谷2中也有着较高的表达量(电子附表14),显示豫谷2具有强于安04的ROS清除能力。
盐胁迫抑制植物的生长主要通过抑制部分代谢过程及合成一系列具有保护作用的蛋白质和次生代谢物[1]。其中编码多胺合成途径的催化蛋白基因如:精胺合酶(spermine synthase)、多胺氧化酶(polyamine oxidase)、S-腺苷蛋氨酸脱羧酶(S-adenosylmethionine decarboxylase)以及氨基酸合成酶基因如:谷氨酰胺合成酶(glutamine synthetase,)和脯氨酸合成酶(delta-1-pyrroline-5-carboxylate synthase,)在盐胁迫后的2个品种中的表达都明显上调。参与苹果酸代谢的苹果酸合酶(malate synthase,Seita.7G144600)在安04中表达上调,而苹果酸脱氢酶(malate dehydrogenase,Seita.3G137700和Seita.6G251800)在豫谷2中表达上调(电子附表13),对苹果酸的积累具有促进作用。类黄酮是植物体内普遍存在的次生代谢物,在植物的生长发育和胁迫响应过程中起着重要的作用[19]。有5个参与类黄酮代谢的基因在盐胁迫后豫谷2中的表达明显高于安04,特别是麦黄酮合酶(tricin synthase)和黄烷酮加氧酶(flavanone 3-dioxygenase)基因,在未经盐处理的豫谷2中表达就明显高于安04(电子附表13)。有12个编码P450的DEGs在YG2NTS vs An04NTS中表达明显上调,同时也发现有5个P450基因在2个品种中的表达都受到盐胁迫诱导。3-酮酰辅酶A合成酶(3-ketoacyl-CoA synthase,KCS)是长链脂肪酸合成的关键酶,有9个编码的基因在安04中表达明显上调,且盐胁迫前后这些基因的表达均明显高于豫谷2。β-葡萄糖苷酶可以水解纤维素并释放游离的葡萄糖和相应配基,为胚芽生长提供碳源。21个编码β-葡萄糖苷酶在安04中表达明显上调,其中有5个基因(Seita.1G033600、Seita.9G528700、Seita.4G134400、Seita.9G279900和Seita.1G335300)在盐胁迫处理前后的安04中的表达都高于豫谷2(电子附表14),显示安04具有较高的基础代谢水平。

图4 DEG的GO分析

图5 盐胁迫处理后豫谷2和安04上调DEG KEGG富集分析前20个代谢途径的散点图
病程相关蛋白(pathogenesis-related family proteins,PR)是一类高度保守的蛋白家族,主要参与植物抗病反应,在非生物胁迫响应过程中也起着重要的作用[20]。8个PR基因在盐胁迫处理前后的豫谷2中表达都高于安04,5个编码内切几丁质酶(endochitinase A,)基因在盐胁迫后2个品种中的表达都明显上调,且在盐胁迫后豫谷2中的表达明显高于安04。同时8个胚胎晚期发育蛋白基因(late embryogenesis abundant protein,)、6个非特异脂质转运蛋白(non-specific lipid transfer protein,)和2个渗调蛋白(osmotin)的表达在2个品种中都明显上调(电子附表14)。
赤霉素(gibberellin,GA)是植物体内重要的激素,在打破种子休眠,种子萌发及各个生命阶段都发挥重要的作用。植物可通过赤霉素减少的方式使生长减缓从而适应外界环境[21]。GA2氧化酶(gibberellin 2-beta-dioxygenase,GA2ox)是GA降解过程的关键酶,2个编码GA2ox的基因(Seita.4G015700和Seita.5G147400)分别在安04和豫谷2中上调表达。GA20氧化酶(gibberellin 20 oxidase,GA20ox)是GA合成途径最后一步的关键酶,盐处理下2个品种中编码GA20ox 的基因(Seita.5G404900)的表达均上调,但在豫谷2中上调幅度明显低于安04(电子附表14)。参与细胞分裂素的合成的基因(adenylate isopentenyltransferase 3,Seita.J000400)盐胁迫后在安04中的表达明显下调,但在豫谷2中下调并不明显。4个生长素响应基因的表达在胁迫后豫谷2中的表达高于胁迫后安04中的表达(电子附表14)。显示在盐胁迫后耐盐品种还能合成一定水平的细胞分裂素和生长素来维持谷子的生长发育。
盐胁迫下,一系列差异响应的转录因子在豫谷2和安04中被鉴定出来。5个MYB转录因子在NaCl胁迫后表达上调,而这些基因的表达却在安04中被抑制。其他胁迫响应的转录因子,如3个bHLH、5个NAC和12个WRKY转录因子在NaCl处理后的豫谷2中的表达明显高于胁迫后的安04(电子附表14)。显示NaCl胁迫下胁迫相关的转录因子在耐盐品种豫谷2中的响应与敏感品种安04相比更为强烈。
3 讨论
盐渍作为主要的非生物胁迫严重威胁了作物产量和粮食安全,利用现代的各种分子生物学技术阐明作物的耐盐机制,对筛选抗盐品种,培育耐盐新种质具有重要意义。对多种作物耐盐性的研究已有一定报道。研究表明,植物在不同的发育期对盐胁迫的抗性及敏感程度不同。种子萌发期被认为是植物生活周期中最重要和最脆弱的阶段,在棉花、水稻、甜菜和番茄中研究表明,种子的萌发期和幼苗期是植物的盐敏感期,也是进行植物耐盐性筛选的关键期[22-23]。鉴于围绕谷子盐害响应的研究较少,本研究对14个谷子品种萌发期种子耐盐性进行了筛选,鉴定到耐盐品种豫谷2和盐敏感品种安04,并对这两个品种在萌发期的种子进行了转录组分析。本研究对于谷子耐盐品种的筛选提供了鉴定依据,也为下一步揭示谷子的耐盐机制提供了重要线索,为指导农业生产提供了理论依据。
3.1 不同品种间差异表达基因影响并决定品种的特性
作物或品种自身特性在影响其对外界的响应中具有决定作用。已知盐芥()作为拟南芥的一个近缘属,属于盐生植物,极端耐盐[24]。在正常生长条件下,盐芥与拟南芥间就存在较多的差异基因,虽然在盐处理后盐芥与拟南芥中都会有不同的胁迫响应基因的表达发生改变并参与盐害响应,但胁迫响应基因的数量较少[24-25],综上显示不同材料之间基因的差异在决定材料间不同特性中发挥决定性作用。本研究中,对盐处理前后豫谷2及安04中的DEG的GO和KEGG分析,显示正常生长的豫谷2中参与各种物质(如氨基酸,离子,有机酸等)跨膜转运和次生代谢的基因的表达高于安04。经盐胁迫处理后(YG2NTS vs An04NTS),豫谷2中上调表达的基因也主要富集在胁迫响应、离子转运、次生代谢、谷胱甘肽代谢、苯丙烷合成等途径,说明豫谷2中具有一定数量的胁迫诱导基因,且其在盐害下的表达高于安04,表现为胁迫下豫谷2中相应物质的积累更为明显,其在豫谷2抗盐特性中具有一定的作用。鉴于品种间DEG数量较多,多于胁迫下诱导表达的基因数量,结合品种特性的不同,故品种自身的基因在影响并决定品种特性中具有重要作用。
3.2 有机酸参与谷子耐盐性的形成
部分胁迫诱导基因在谷子抗胁迫品质的形成中发挥作用。有机酸在植物初级代谢中起着重要的作用,如三羧酸循环,主要包括苹果酸,柠檬酸和延胡索酸等。这些物质在能量产生、氨基酸的合成、pH的调节、响应生物和非生物胁迫过程中起着关键作用[26]。本研究显示,未经处理的品种间,抗盐品种豫谷2 中特有的基因主要富集在有机酸转运的生物过程,经盐处理后(YG2NTS vs An04NTS),豫谷2中上调的基因同样富集在有机酸的生物合成途径中,苹果酸合酶和苹果酸脱氢酶都受到盐胁迫的诱导。显示有机酸的迅速合成和转运将有助于豫谷2盐胁迫抗性的提高。
3.3 PR基因的高表达调控谷子的耐盐性
PR蛋白主要参与植物的抗病反应,过表达PR蛋白基因能增强植物对病原菌的抗性,其在适应非生物胁迫中也有多重作用[20]。烟草中过表达PR1蛋白能提高其对重金属的耐性[20]。水稻中过表达不但可增强植物对稻瘟病的抗性,而且还可增强其对盐渍和干旱胁迫的抗性[20]。几丁质酶作为PR蛋白,在烟草中过表达哈茨木霉()诱导的几丁质酶能增强转基因材料对生物胁迫和非生物胁迫(盐渍和重金属)的耐性[27]。冬黑麦冷胁迫诱导的和编码具有抗冻活性的几丁质酶,其在大肠杆菌中表达可提高受体菌株的抗冻性[28]。本研究中,显示8个PR蛋白和5个几丁质酶基因在豫谷2中高表达,综合基因特性,推测以上DEG可参与谷子抗盐特性的形成。
3.4 其他
谷子抗盐性的形成还需要多种物质及过程的协同作用。其中细胞色素P450属于单加氧酶超家族,具有广泛的催化活性,可参与各种生物合成及降解途径[29-30]。而植物激素如赤霉素激素,其含量的减少可使植物生长减缓从而适应外界环境[21]。本研究中,鉴定到多个P450基因的表达变化,部分赤霉素降解途径及合成途径中关键酶也发生了变化。同时部分介导植物生长发育和胁迫响应的转录因子的表达也都发生了改变[31],最终影响了盐害下谷子不同品种的抗盐能力。
3.5 谷子萌发期种子对盐胁迫的响应
本研究通过对盐胁迫下谷子抗性的筛选,获得了一个耐盐品种豫谷2和敏感品种安04,初步明确了谷子萌发期种子对盐胁迫响应的作用途径及机制。盐胁迫诱导谷子胞内钙离子浓度的提高,进而其作为第二信使启动一系列的信号转导途径,调控下游基因表达,通过合成、积累一系列的保护性蛋白,如LEA蛋白、PR蛋白、活性氧清除酶类等从而提高谷子的胁迫抗性。同时胁迫下部分初级代谢物(如有机酸,各种氨基酸等)和次生代谢物(如类黄酮,不饱和脂肪酸,多胺等)的积累还可保护细胞免受渗透胁迫及离子毒害,进而提高植物抗性(图6)。耐盐品种豫谷2与敏感品种安04相比,能迅速的感知盐胁迫信号并向下游传递,通过对不同转录因子进行表达调控影响下游胁迫响应基因如、、和活性氧清除酶等基因的表达;有机酸、类黄酮类等代谢物的迅速合成及积累都有助于增强豫谷2的耐盐性。该方面工作为下一步筛选谷子抗盐品种、改良和培育谷子抗盐新种质奠定基础。
4 结论
豫谷2在萌发期的耐盐性明显高于安04。豫谷2中离子跨膜转运、抗氧化、次生代谢、有机酸的合成及转运等相关基因的表达明显高于安04,这些基因的差异表达导致了2个品种的耐盐性差异。

图6 谷子萌发期种子盐胁迫响应的模式图
[1] Deinlein U, Stephan A B, Horie T, Luo W, Xu G, Schroeder J I. Plant salt-tolerance mechanisms., 2014, 19(6): 371-379.
[2] Julkowska M M, Testerink C. Tuning plant signaling and growth to survive salt., 2015, 20(9): 586-594.
[3] Parihar P, Singh S, Singh R, Singh V P, Prasad S M. Effect of salinity stress on plants and its tolerance strategies: a review., 2015, 22(6): 4056-4075.
[4] Acosta-Motos J, Ortuño M, Bernal-Vicente A, Diaz- Vivancos P, Sanchez-Blanco M, Hernandez J. Plant responses to salt stress: Adaptive mechanisms., 2017, 7(1): 18.
[5] Zhang G, Liu X, Quan Z, Cheng S, Xu X, Pan S, Xie M, Zeng P, Yue Z, Wang W, Tao Y, Bian C, Han C, Xia Q, Peng X, Cao R, Yang X, Zhan D, Hu J, Zhang Y, Li H, Li H, Li N, Wang J, Wang C, Wang R, Guo T, Cai Y, Liu C, Xiang H, Shi Q, Huang P, Chen Q, Li Y, Wang J, Zhao Z, Wang J. Genome sequence of foxtail millet () provides insights into grass evolution and biofuel potential., 2012, 30(6): 549-554.
[6] Lata C, Gupta S, Prasad M. Foxtail millet: a model crop for genetic and genomic studies in bioenergy grasses., 2013, 33(3): 328-343.
[7] Yang Y, Guo Y. Elucidating the molecular mechanisms mediating plant salt-stress responses., 2018, 217(2): 523-539.
[8] Zhu J K. Regulation of ion homeostasis under salt stress., 2003, 6(5): 441-445.
[9] Lata C, Sahu P P, Prasad M. Comparative transcriptome analysis of differentially expressed genes in foxtail millet (L.) during dehydration stress., 2010, 393(4): 720-727.
[10] Pandey G, Misra G, Kumari K, Gupta S, Parida S K, Chattopadhyay D, Prasad M. Genome-wide development and use of microsatellite markers for large-scale genotyping applications in foxtail millet [(L.)]., 2013, 20(2): 197-207.
[11] Qi X, Xie S, Liu Y, Yi F, Yu J. Genome-wide annotation of genes and noncoding RNAs of foxtail millet in response to simulated drought stress by deep sequencing., 2013, 83(4-5): 459-473.
[12] Wang Y, Li L, Tang S, Liu J, Zhang H, Zhi H, Jia G Q, Diao Xm. Combined small RNA and degradome sequencing to identify miRNAs and their targets in response to drought in foxtail millet., 2016, 17(1): 57.
[13] Tang S, Li L, Wang Y, Chen Q, Zhang W, Jia G, Zhi H, Zhao B, Diao X M. Genotype-specific physiological and transcriptomic responses to drought stress in(an emerging model for Panicoideae grasses)., 2017, 7(1): 10009.
[14] Pan J, Li Z, Wang Q, Garrell A K, Liu M, Guan Y, Zhou W, Liu W. Comparative proteomic investigation of drought responses in foxtail millet., 2018, 18(1): 315.
[15] Wang J, Zhang Q, Cui F, Hou L, Zhao S, Xia H, Qiu J, Li T, Zhang Y, Wang X, Zhao C.Genome-wide analysis of gene expression provides new insights into cold responses in., 2017, 8(1):713.
[16] Xiong H, Guo H, Xie Y, Zhao L, Gu J, Zhao S, Li J, Liu L. RNAseq analysis reveals pathways and candidate genes associated with salinity tolerance in a spaceflight-induced wheat mutant., 2017, 7(1): 2731.
[17] Wang F, Xu Z, Fan X, Zhou Q, Cao J, Ji G, Jing S, Feng B, Wang T. Transcriptome analysis reveals complex molecular mechanisms underlying UV tolerance of wheat (L.)., 2019, 67(2): 563-577.
[18] Zhang Y, Wang P, Xia H, Zhao C, Hou L, Li C, Gao C, Wang X, Zhao S. Comparative transcriptome analysis of basal and zygote-located tip regions of peanut ovaries provides insight into the mechanism of light regulation in peanut embryo and pod development., 2016, 17(1): 606.
[19] Wang A, Li R, Ren L, Gao X, Zhang Y, Ma Z, Ma D, Luo Y. A comparative metabolomics study of flavonoids in sweet potato with different flesh colors ((L.) Lam)., 2018, 260:124-134.
[20] Wu J, Kim S G, Kang K Y, Kim J G, Park S R, Gupta R, Kim Y H, Wang Y, Kim S T. Overexpression of a pathogenesis-related protein 10 enhances biotic and abiotic stress tolerance in rice., 2016, 32(6): 552-562.
[21] Yamaguchi S. Gibberellin metabolism and its regulation., 2008, 59: 225-251.
[22] 张国伟, 路海玲, 张雷, 陈兵林, 周治国. 棉花萌发期和苗期耐盐性评价及耐盐指标筛选. 应用生态学报, 2011, 22(8): 2045-2053.
ZHANG G W, LU H L, ZHANG L, CHEN B L, ZHOU Z G. Salt tolerance evaluation of cotton () at its germinating and seedling stages and selection of related indices., 2011, 22(8): 2045-2053. (in Chinese)
[23] 於丽华, 王宇光, 孙菲, 杨瑞瑞, 王秋红, 耿贵. 甜菜萌发—幼苗期不同阶段耐盐能力的研究. 中国农学通报, 2017, 33(19): 22-28.
Yu L H, Wang Y G, Sun F, Yang R R, Wang Q H, Geng G. Salt tolerance of sugarbeet: From germination to seedling stage., 2017, 33(19): 22-28. (in Chinese)
[24] Gong Q, Li P, Ma S, Indu Rupassara S, Bohnert H J. Salinity stress adaptation competence in the extremophilein comparison with its relative., 2005, 44(5): 826-839.
[25] Arbona V, Argamasilla R, Gomez-Cadenas A. Common and divergent physiological, hormonal and metabolic responses ofand Thellungiella halophila to water and salt stress., 2010, 167(16): 1342-1350.
[26] Igamberdiev A U, Eprintsev A T.Organic acids: The pools of fixed carbon involved in redox regulation and energy balance in higher plants., 2016, 7: 1042.
[27] Takenaka Y, Nakano S, Tamoi M, Sakuda S, Fukamizo T. Chitinase gene expression in response to environmental stresses in: chitinase inhibitor allosamidin enhances stress tolerance., 2009, 73(5): 1066-1071.
[28] Yeh S, Moffatt B A, Griffith M, Xiong F, Yang D S, Wiseman S B, Sarhan F, Danyluk J, Xue Y Q, Hew C L, Doherty-Kirby A, Lajoie G. Chitinase genes responsive to cold encode antifreeze proteins in winter cereals., 2000, 124(3): 1251-1264.
[29] Jensen K, Moller B L.Plant NADPH-cytochrome P450 oxidoreductases., 2010, 71(2-3): 132-141.
[30] Castorina G, Persico M, Zilio M, Sangiorgio S, Carabelli L, Consonni G. The maize lilliputian1 (lil1) gene, encoding a brassinosteroid cytochrome P450 C-6 oxidase, is involved in plant growth and drought response., 2018, 122(2): 227-238.
[31] Golldack D, Luking I, Yang O. Plant tolerance to drought and salinity: stress regulating transcription factors and their functional significance in the cellular transcriptional network., 2011, 30(8): 1383-1391.
Transcriptomics Analysis of NaCl Response in Foxtail Millet (L.)seeds at germination stage
PAN JiaoWen1, LI Zhen1, WANG QingGuo1, Guan YanAn2, 3, LI XiaoBo1,4, DAI ShaoJun5, DING GuoHua4, LIU Wei1, 3
(1Biotechnology Research Center, Shandong Academy of Agricultural Sciences/Shandong Provincial Key Laboratory of Crop Genetic Improvement, Ecology and Physiology, Jinan 250100;2Crop Research Institute, Shandong Academy of Agricultural Sciences/Shandong engineering laboratory for featured crops, Jinan 250100;3College of Life Sciences, Shandong Normal University, Jinan 250014;4School of Life Sciences and Technology, Harbin Normal University, Harbin 150025;5College of Life and Environmental Sciences, Shanghai Normal University/Development Center of Plant Germplasm Resources, Shanghai 200234)
Foxtail millet (L.) remarkably adapts to adverse ecologies, including drought, barren regions and high soil salinity. These characteristics promote it as a very promising model crop for exploring basic biology processes of plants. Unveil of mechanisms of foxtail millet under salinity is of immense significance.In this research, 14 millet varieties at germination stage were used for resistance screening under 150 mmol·L-1NaCl. The germination rate, root and bud length of each variety were measured after been treated for 7 d, and the salt tolerance variety Yugu 2 and salt susceptible variety An 04 were obtained. These two varieties before and after salt treatment were used for transcriptomes analysis by RNA-seq sequencing. The functional and pathway enrichments of differentially expressed genes (DEGs) were also performed by GO and KEGG analysis. Fifteen DEGs were randomly selected for further qRT-PCR analysis to verify the RNA-seq data.Totally 2 786 and 4 413DEGs were identified in Yugu 2 and An 04, and there were 1 470 and 3 826 DEGs within each cultivar before and after salt treatment, respectively. GO and KEGG enrichment analysis suggested that DEGs were mainly clustered into signaling, antioxidant system, organic acid biosynthesis and transport, and secondary metabolism, and the DEGs participated in response to stress, iontransmembrane transport, redox homeostasis, secondary metabolism, organic acid, polyamine and phenylpropanoid biosynthetic process. The qRT-PCR results showed high correlation coefficient of 0.8817 with the data of RNA-seq. Especially the genes such as cation transporter (), peroxidase (), flavanone 3-dioxygenase () and MYB transcription factors, which displayed higher vary degrees in Yugu2 under salinity may play key roles in salt response process of foxtail millet.There was difference in the response degree of salt tolerance variety and salt sensitive variety of millet under salinity. Moreover, the resistance trait formation and function to salt was mainly depends on the response degree instead of the number of DEGs under stress.
foxtail millet (L.); salt stress; seeds of germination stage; transcriptomics; molecular mechanisms
10.3864/j.issn.0578-1752.2019.22.003

2019-07-19;
2019-09-30
山东省自然科学基金青年基金(ZR2019QC003)、现代农业产业技术体系建设专项资金(CARS-06)、山东省农业科学院重大科技成果培育计划(2015CGPY10)、山东省农业科学院农业科技创新工程(CXGC2018E13)、山东省农业科学院青年英才计划(2016-2018)
潘教文,E-mail:jwpan01@126.com。通信作者刘炜,E-mail:wheiliu@163.com
(责任编辑 李莉)
