寨卡病毒E、NS5蛋白生物信息学分析
2019-12-16邹淑慧李金亮廖莎莎张丽琴刘聪
邹淑慧,李金亮,廖莎莎,张丽琴,刘聪
(赣州市人民医院检验科,江西 赣州 341000)
至2015年5月以来,寨卡病毒 (Zika Virus,ZIKV)在美洲区域的传播不断升级,疫情逐步恶化,甚至有城市化和全球化传播趋势,对国际公共卫生造成严重威胁[1]。2016年2月,我国出现首例输入性寨卡病毒感染,随后国内陆续出现多例ZIKV感染病例,引起我国政府和卫生部门的广泛关注[2]。
寨卡病毒属黄病毒属,为单股正链RNA,长约11kb,分为亚洲型和非洲型[3],在系统发生树上与同为黄病毒属的登革热病毒、Spondweni病毒、日本脑炎病毒、西尼罗病毒非常相近[4]。寨卡病毒基因编码多聚蛋白,包括病毒衣壳蛋白C,膜前体蛋白PrM,包膜蛋白 E和7个非结构蛋白(NS1、NS2A、NS2B、NS3、NS4A、NS4B、NS5)。 E 蛋白是病毒表面的主要蛋白,参与调节结合以及膜融合等方面。非结构蛋白中的NS5是病毒中最大的蛋白,含有RNA-依赖RNA聚合酶和甲基转移酶,负责病毒基因组RNA的合成及加帽[5,6]。本研究将从生物信息的角度分析ZIKV E蛋白和 NS5蛋白序列特征,为进一步挖掘其基因信息,研究其致病机制提供分子依据,从而预防和控制寨卡病毒的感染与流行。
1 材料和方法
1.1 序列来源 所有病毒株E和NS5核苷酸、氨基酸序列均来源于GenBank中已登记的黄病毒属相关参考序列。
1.2 ZIKV E、NS5核苷酸与氨基酸同源性比较 用Clustal X和Bioedit软件对ZIKV不同地理株系及同属成员的E、NS5基因及编码蛋白序列进行比对,计算核苷酸与氨基酸同源性。
1.3 ZIKV E、NS5蛋白二级结构分析 选取中国首例分离的ZIKV基因组序列(登陆号:KU744693.1)为研究对象[2],获取E、NS5基因及氨基酸序列。通过蛋白结构在线预测网站Predict Protein Server(https://www.predictprotein.org/)分析 E、NS5 蛋白的二级结构,对E、NS5蛋白的氨基酸组成以及蛋白序列可能存在的蛋白结合区、α螺旋区、β折叠区以及卷曲结构区域进行分析,同时预测蛋白结构中可能的暴露区域和隐藏区域。
1.4 ZIKV E、NS5蛋白的跨膜区结构分析 利用在线 软 件 TMHMM v2.0 (http://www.cbs.dtu.dk/services/TMHMM/)对E、NS5蛋白氨基酸序列进行跨膜拓扑结构的预测,从而分析跨膜段、膜内段和膜外段的氨基酸序列。
1.5 ZIKV E、NS5蛋白的信号肽分析 应用Anthe-Pro软件分析E、NS5蛋白氨基酸序列中可能存在的潜在信号肽结构。
1.6 ZIKV E、NS5蛋白B细胞抗原表位的预测 运用 Bepipred(http://tools.iedb.org/bcell/)在线服务器程序预测E、NS5蛋白中可能的B细胞线性表位,得分大于0.350(软件默认值)则预测为优势线性B表位。
2 结果
2.1 ZIKV E、NS5核苷酸与氨基酸同源性分析 通过比较ZIKV不同地理株系及同属成员的E、NS5蛋白核苷酸和氨基酸的同源性,结果显示不同地理株的ZIKV E蛋白核苷酸和氨基酸同源性分别为87.1%~100%和94.2%~100%,跟同黄病毒属其他成员比较,发现与Spondweni病毒更接近,氨基酸同源性最高达72.8%(表1)。不同地理株的ZIKV NS5蛋白核苷酸和氨基酸同源性分别为88%~99.9%和95.5%~100%,跟同黄病毒属其他成员比较,也是与Spondweni病毒更接近,氨基酸同源性最高达77.5%(表2)。
2.2 ZIKV E、NS5蛋白二级结构 E蛋白编码504个氨基酸,可能的蛋白结合位点有14个,主要位于 1~13、55~161、232~394aa 区段;α 螺旋区主要集中在 406~418、436~501aa区段,占 15.28%;β 折叠片主要在 2~73、91~143、165~216、236~401aa 区段,占33.93%;无规则卷曲占50.79%;此外,在蛋白中还可能包含两个跨膜结构区域 (461~479aa、483~501aa)。整个蛋白暴露区域和隐藏区域均匀相间分布,见图1。NS5蛋白编码903个氨基酸,可能的蛋白结合位点有22个,主要位于6~119、247~418、518~744、895~899aa 区段;多聚核苷酸结合位点有两个,位于741aa和856aa;α螺旋区主要集中在 2 ~67、158 ~288、350 ~444、545 ~575、611 ~627、753~888aa区段,占36.88%;β折叠片主要在74~146、578~606、702~743aa 区段,占 11.52%;无规则卷曲占51.60%;整个蛋白暴露区域和隐藏区域均匀相间分布,见图1。
2.3 ZIKV E、NS5蛋白的跨膜区结构 TMHMM预测E蛋白有两个跨膜区域,分别为455~477aa、484~503aa,膜外区域为 1~454aa,膜内区域为 478~483aa,见图2。整个NS5蛋白均位于膜外,无跨膜区域,见图3。
2.4 ZIKV E、NS5蛋白的信号肽 E和NS5蛋白均存在潜在信号肽结构,其信号肽断裂位点分别位于 501aa 和 152aa,见图 4、图 5。
2.3 ZIKV E、NS5蛋白的B细胞抗原表位 运用Bepipred法预测B细胞线性表位,结果显示E蛋白平均抗原性指数为0.062,分子内有多个可能的抗原表位区域。其中分值最高的表位区域为317~342aa,分值达1.919(阈值0.350);其次为155~181、66~89、226~238、380~384aa (按分值高低排列),见图6。NS5蛋白平均抗原性指数为0.107,分值最高表位区域为100~114aa,分值达2.297;其次为 360~372、148~159、688~707aa,见图 7。
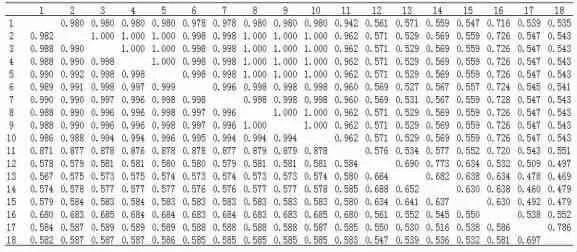
表1 E蛋白核苷酸和氨基酸的同源性比较
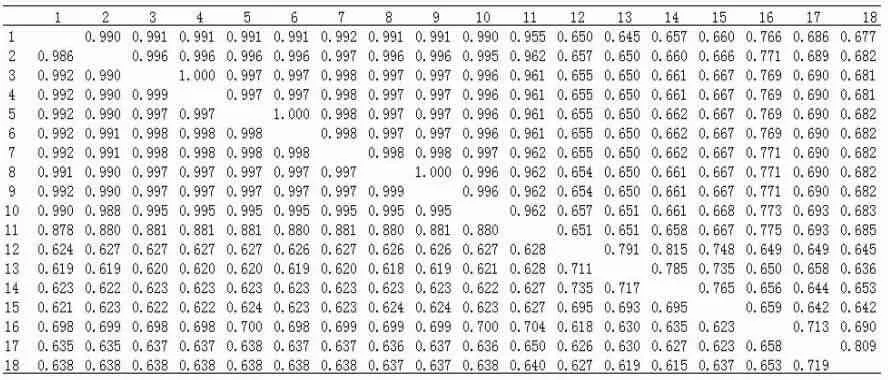
表2 NS5蛋白核苷酸和氨基酸的同源性比较

图1 E蛋白二级结构(1A)及组成成分(1B)、NS5蛋白的二级结构(1C)及组成成分(1D)

图2 E蛋白跨膜区域预测

图3 NS5蛋白跨膜区域预测

图4 E蛋白信号肽结构预测
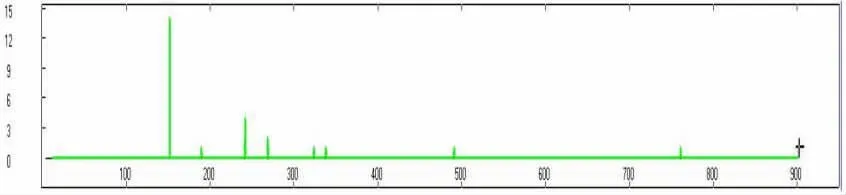
图5 NS5蛋白信号肽结构预测
3 讨论
ZIKV属于黄病毒科黄病毒属的一种虫媒病毒,且是一种人兽共患病毒。寨卡病毒可引起严重的神经系统损害,包括格林巴利综合征(GBS)及新生儿小头畸形,已经成为威胁全球人类健康的潜在因素[7]。目前尚无针对性的预防性疫苗和治疗性药物,因此开发相关疫苗迫在眉睫。
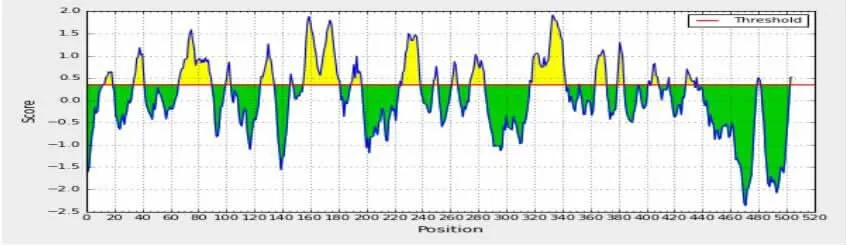
图6 Bepipred工具预测E蛋白的线性B细胞表位
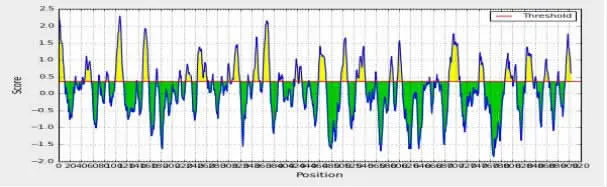
图7 Bepipred工具预测NS5蛋白的线性B细胞表位
在基因组和蛋白质组学时代,基于疫苗开发、单克隆抗体的制备以及快速检测试剂盒的研制,运用生物信息学工具分析蛋白质序列已成为蛋白质研究的重要手段[8]。本文通过生物信息学分析发现不同地理株系的ZIKV E蛋白和NS5蛋白同源性都比较高,提示这两段序列比较保守。跟同黄病毒属其他成员比较,显示与Spondweni病毒关系最近,但同源性也仅有70%多。说明ZIKV虽然与其他黄病毒发病症状相似,但可能已经发展出不同的致病机制。
蛋白质的二级结构对抗原表位影响很大,α螺旋、β折叠的化学键键能较高,成为蛋白质中心的“支架”,很难与合适的抗体嵌合,而β转角和无规则卷曲常位于分子表面,利于抗体结合,成为抗原表位的可能。本研究通过Predict Protein Server分析发现ZIKV E蛋白和NS5蛋白二级结构中无规则卷曲含量最高,分别占50.79%和51.60%,推断这些区域与B细胞表位有关。另外E蛋白和NS5蛋白中还包含有多个可能的蛋白质相互作用位点,提示可能参与多种蛋白或者其他分子之间的相互作用并在ZIKV感染过程中发挥重要作用,可对这些相关功能位点进行深入研究,进一步揭示ZIKV的浸染机制。外膜蛋白通过加强巨噬细胞对抗原的摄取,增强淋巴细胞活化,在免疫反应中表现出重要作用,常作为保护性疫苗候选抗原。跨膜区域预测分析结果显示E蛋白有两个跨膜区域,其中氨基酸1~454位于膜外,而NS5均位于膜外。研究表明抗原表位可能不在这两个跨膜域中[9]。在跨膜蛋白的N端,有一段疏水性氨基酸序列,称为信号肽,新合成的蛋白质通过该信号肽进入正常的分选途径。软件分析表明E蛋白和NS5蛋白含有信号肽,为分泌性蛋白。
抗原优势表位的确定是疫苗研制和临床应用的基础。运用生物信息学方法预测蛋白质的抗原优势表位,并通过体外合成肽段进行实验验证,已广泛应用于蛋白质抗原表位的鉴定[10]。Bepipred方法是结合线性B细胞表位预测的一种经典算法。本研究通过该方法预测发现,ZIKV E蛋白平均抗原性指数为0.062,分子内有多个可能的抗原表位区域, 如:317~342aa、155~181、66~89、226~238、380~384aa。NS5蛋白平均抗原性指数为0.107,可能的表位区域有 100~114aa,、360~372、148~159、688~707aa。对于进一步确认E、NS5蛋白的优势表位信息,需要通过体外试验、动物模型等来进行验证。本研究预测的相关线性表位将为进一步E、NS5蛋白抗原表位的确证及诊疗抗体的开发提供一定的参考依据。
总之,本研究对ZIKV E蛋白和NS5蛋白的生物信息学分析,成功预测到其基本的结构特征和潜在的线性B细胞表位。这将有助于对E、NS5蛋白的深入研究奠定理论基础,为开发ZIKV快速检测的胶体金试剂盒和治疗性抗体提供研究基础。
