抗癌药物基因组学知识表示模型构建
2019-12-03
癌症是严重威胁人民群众健康的重大公共卫生问题。近年来我国癌症发病率、死亡率呈逐年上升趋势,给家庭和社会造成重大经济负担,也是当前社会重大民生“痛点”。党中央、国务院高度重视抗癌新药的注册和审批工作,2018年以来,李克强总理3次主持召开国务院常务会议,对加快抗癌新药的上市等议题做出了重要部署。2018年10月10日,国家医疗保障局宣布将阿扎胞苷、阿法替尼等17种抗癌药物纳入国家医疗保障体系[1]。其中包括12个实体肿瘤药和5个血液肿瘤药,均为临床必需、疗效确切、参保人员需求迫切的肿瘤治疗药品,涉及非小细胞肺癌、肾癌、结直肠癌、黑色素瘤、淋巴瘤等多个癌种。
黑色素瘤已成为世界上发病率增长最快的恶性肿瘤之一,年发病增长率为3%~5%。我国黑色素瘤患者也呈现快速增长的趋势,每年新发病例约2万例。根据个体遗传基因的差异指导合理用药,是精准医疗所要实现的目标之一。发现与药物效用相关的遗传因素和靶标分子,并将其应用于药物设计和临床实践中,对于新药研发、精准施药和提高药物治疗效果有十分重要的意义。在这些新药的研发过程中,药物基因组学(Pharmacogenomics)的研究发挥了重要作用。Gay等人[2]报道了2例肺癌患者在出现NGR1基因融合后使用阿法替尼(40mg qd)治疗,极大地改善了患者咳嗽和气短等症状,是老药新用的一个典型案例;Hida等人[3]的研究表明针对克唑替尼耐药开发出新一代ALK 抑制剂艾乐替尼,能够使非小细胞肺癌恶化或死亡风险降低66%。
在药物基因组学研究中,随着海量数据的累积,越来越需要信息领域的专业人员对规模急速增长和内容纷繁复杂的药物基因组大数据进行收集、整理、建模与挖掘。这些数据能够从不同层次反映药物与基因、药物与疾病系统之间更精细的信息。本文拟从抗癌药物的个性化用药入手,整合抗癌药物领域的异构数据,设计涵盖药物、基因、疾病在内的药物基因组学知识表示模型,并重点将知识图谱的语义类型扩充到药物服用方式、药物使用剂量、药物副作用等个性化用药信息和药物副作用维度。
1 药物基因组学异构数据
从数据组织形式上可将药物基因组学异构数据分为词表、数据库、非结构化文本文档和通用本体库等。
1.1 词表
词表包括医学主题词表(Medical Subject Headings,MeSH)、国际疾病分类(International Classification of Disease,ICD)、美国癌症中心分类词汇汇编(NCI’s thesaurus,NCIt)、医学系统命名法-临床术语(Systematized Nomenclature of Human and Veterinary Medicine-Clinical Term,SNOMED-CT)等。
1.2 数据库
DrugBank:包含药物的基本信息及药理学、药物间相互作用、药物基因组学、相关临床试验、药物毒性、药物靶点的信息;PharmGKB(The Pharmacogenomics Knowledgebase):收集、审编和开放共享具有临床意义的基因-药物关系和基因-表型关系知识[4];RxNorm:临床药物标准命名表,以规范化形式(即活性成分+剂量+剂型以及商品名称)来表示临床药物[5];SIDER(Side Effect Resource):是从药物说明书中和公开的文件中提取的上市药物的不良反应数据库,包括药物适应症、不良反应发生频率、不良反应分类和药物-靶标关系的链接方面的数据;DisGeNET:是整合多种资源的人类疾病-基因-变异的数据库。
1.3 非结构化文本文档
美国食品药品监督管理局(Food and Drug Administration,FDA):美国批准上市的药品及药品使用说明书详细信息;电子病历:以非结构化的文本形式记录患者在医院诊断治疗的全过程,通常包含首页、病程记录、检查检验结果、医嘱、手术记录、护理记录等信息。
1.4 通用本体库
基因本体(Gene Ontology,GO):是对跨物种和跨数据库的基因表达和基因产物属性进行结构化及定义精确的描述,旨在统一各种基因产物数据库的信息表达方式;疾病本体(Disease Ontology,DO):提供一个与人类疾病相关的整合生物医学数据的开源本体,以促进各种疾病及相关健康状况向特定医学代码的映射。
2 药物基因组学知识整合与表示研究现状
知识表示模型的构建可以理解为是一种结构化的有向图集合,其中图的节点代表实体或者概念,边代表实体/概念之间的语义关系,最终组成“实体-关系-实体”三元组[6]。基于上文提到的药物基因组学数据资源,国内外研究者开展了一系列的知识表示模型构建和跨纬度的药物、基因、疾病信息挖掘研究。
在国外,Meng Wang等人[7]利用ICD-9和DrugBank知识,通过构建层次化知识图谱获取电子医学病历中患者、疾病和药物之间的关系,最后实现了安全用药的信息整合与挖掘;Michel Dumontier等人[8]利用PharmGKB数据构建了一个轻量型本体,包含40个核心概念,共涉及药物、表型、基因型、药物治疗多个领域的药物基因组学本体(Pharmacogenomics Ontology,PO);Boyce R D[9]等人利用从美国食品药品监督管理局(FDA)获取的包含药物基因组信息的药物标签提取药物-生物标记物关系,将这些提取的概念及关系标准、完整地表示出来,从而构建药物基因组语义模型。在国内,药物基因组学知识整合的相关工作也逐渐引起关注。弓孟春[10]等人提出了药物基因组学临床部署的总体框架,引导精准医学临床实践的发展方向,构建基于中国人群数据的知识库体系;邢玉华[11]等人对2型糖尿病治疗药物及其相关的基因多态性信息进行了阐述。
通过分析以上研究发现,目前开展的药物知识表示涉及的语义类型通常仅限于药物、基因、疾病,而对药物服用方式、药物使用剂量调整、适用人群、药物副作用等个性化用药信息没有深入探究和描述,能够辅助临床医生、临床药师精准用药的药物基因组学知识表示研究十分有限。因此开展面向精准用药的药物基因组学知识表示模型构建研究具有现实意义。
3 抗癌药物基因组学知识表示模型构建
知识表示模型的构建方法分为自顶向下(top-down)和自底向上(bottom-up)两种[12]。自顶向下构建方法是从高质量的结构化数据中提取本体和模式信息,构建出知识图谱的框架;自底向上构建方法是从实体层出发,提取开放共享数据源中的有效信息,经人工审核后,加入到知识库中。本文采用两种方法结合的构建方式,即先构建知识表示框架,再提取开放数据源中的有效信息,完成知识表示模型的实例填充。
3.1 抗癌药物基因组知识表示模型框架设计
框架设计过程中需明确知识组织的语义类型和表示方式,如以网络的形式表示知识,构建三元组关系。通过对现有的药物基因组知识表示模型的分析,在药物、基因、疾病3种常见语义类型的基础上,扩充抗癌药物基因组学知识表示模型的语义类型,涵盖药物、基因、基因变异、疾病(适应症、疾病发生部位等)、个性化用药(服用方式、服用频率、适用人群、服用剂量、剂量调整等)、不良反应等多个维度。抗癌药物基因组知识表示框架涵盖的类及属性如表1所示。

表1 抗癌药物基因组学知识表示框架涵盖的类及属性
3.2 抗癌药物基因组信息整合与知识抽取
信息整合和知识抽取指通过人工或者自动化技术,从结构化、半结构化或非结构化的开放医学数据中提取出知识表示模型所涉及的基本组成元素,包括实体、属性和关系等,组成有效的三元组形式存入数据库中。下面以黑色素瘤疾病的相关用药为例,进行相关实体、属性及语义关系的抽取。
3.2.1 实体及属性抽取
多数黑色素瘤疾病的发生是由BRAF基因突变引起的。从美国食品药品监督管理局(FDA)公布的药品说明书数据中,通过人工标注的方法获取能够靶向治疗BRAF基因突变引起的黑色素瘤的药物,以及存在药物相互作用关系的药物数据,包括Binimetinib、Cobimetinib、Dabrafenib、Encorafenib、Nivolumab、Trametinib和Vemurafenib。其中,Dabrafenib、Encorafenib和Vemurafenib是BRAF基因突变的靶向药物。从DrugBank和PharmGKB数据库中解析以上7种药物的属性信息,包括药物名称、药物描述、化学式、分子量、商品名等;从RxNorm数据库获取药物及剂量、剂型、用药频率等属性信息。
3.2.2 语义关系定义及抽取
语义关系抽取是知识表示模型构建的重要环节之一。本文语义关系抽取的重点是抽取疾病和药物、疾病和症状、疾病和基因、疾病和疾病、药物和症状、药物和药物等实体间的关联关系,从而为患者和领域专家提供支持。
本文围绕药物、基因、突变、疾病、用药剂量、复用方式、不良反应等制定了15种语义关系。其中一级语义关系10种,二级语义关系5种,并对每一种语义关系进行了详细定义(表2)。通过人工标注的方法,对上文提到的7种药物的药品说明书信息进行标注和语义关系抽取,并将抽取出的语义关系与已经定义的15种语义关系进行概念归并,如“in combination with”=“synergized by”,“recommended dosage”=“routine dosage”。

表2 语义关系定义
对美国食品药品监督管理局公布的药品说明书数据进行实体和语义关系抽取的示例如图1所示。根据实体、属性及语义关系的抽取结果,绘制抗癌药物基因组知识表示框架的示意图(图2)。图2涵盖了药物、基因、突变、疾病、剂量/剂型、人群、不良反应等实体,并构建了实体与实体之间的语义关系。
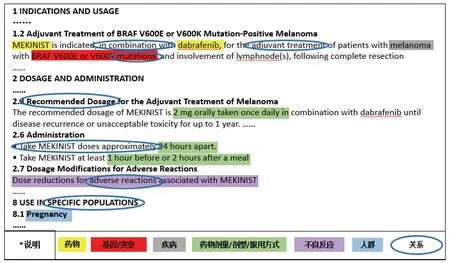
图1 实体及语义关系抽取示例

图2 抗癌药物基因组知识表示示意图
3.3 知识表示模型的实例填充及可视化
将Trametinib等7种药物及相关实体填充到知识表示模型中,最终黑色素瘤相关药物的知识表示模型共包含了136个三元组。其中涉及7种药物及其对应剂量、剂型,1种基因及相关的3种基因变异,14种疾病和30种不良反应。
抗癌药物基因组系知识表示三元组示例如表3所示。

表3 抗癌药物基因组学知识表示三元组示例
使用XML技术,将抽取出的药物、基因、疾病、用药等实体和属性存储于结构化的XML文件中,然后基于Dom4j、XPath等技术对XML文件进行解析,构造相应参数,为可视化分析和展示打好基础。选取开源免费的ECharts可视化图表工具对上述实体及其关系进行可视化展示(图3)。

图3 抗癌药物知识表示模型可视化示例
4 结语
本文通过对异构的药物基因组数据的整合和抽取,构建了以药物为中心,涵盖药物服用方式、药物使用剂量、药物副作用、靶向基因、疾病等多个维度的抗癌药物基因组学知识表示模型,并对其中的语义关系进行了详细定义;同时,在此框架下以黑色素瘤相关药物为实例完成了知识表示模型的填充和可视化展示。抗癌药物基因组学知识表示模型的构建能够发现药物、基因、疾病之间的新知识、新关联,辅助药物信息的关联检索和智能推荐,为临床医生精准用药、联合用药等提供参考依据,为药学科研人员开展新药研发、老药新用等的研究提供理论支持,为癌症患者查询药物知识提供服务支撑。
在此基础上,我们将继续对药物基因组异构数据进行深入探究和分析,比对不同数据资源的元数据项,制定遴选标准,择优整合多维度药物基因组学相关实体,如黑色素瘤相关的症状描述等内容,完善抗癌药物基因组知识表示模型的知识组织体系;同时,开展抗癌药物基因组学知识表示模型的实证研究,将其应用到“中国工程科技知识中心医药卫生知识服务系统”中,从知识表示模型可视化、寻医问药、关联关系查询等角度开展服务,探究模型的可用性和可行的服务模式。
