玉米叶色突变体研究进展
2019-11-25李秦杜何为
李秦 杜何为


摘 要 叶色突变体是研究玉米光合作用和光建成的宝贵材料,对于玉米叶色相关基因的克隆和功能分析,以及提高光合效率和玉米产量方面具有重要意义。简介了elm1淡绿叶突变体、elm2黄绿叶突变体、ygl-1黄绿叶突变体、vyl黄叶突变体、nec-t黄化类病变突变体这五个典型的叶色突变体,以及玉米叶色突变体诱变方法;根据玉米叶色突变体的研究现状及玉米突变体数据库,对23个已知的玉米突变体基因名、基因ID、性状描述及相关图片进行整理总结,并对其基因在染色体上的位置进行了标注。以图片和表格的形式归纳了玉米叶色突变形成机理研究成果。最后,提出问题并做了展望。
关键词 玉米;叶色突变体;基因突变
中图分类号:S513;Q812 文献标志码:A DOI:10.19415/j.cnki.1673-890x.2019.28.005
玉米光合作用的效率直接决定其产量的高低,而高效的光合作用依赖于玉米叶片中叶绿体的正常发育和光合色素的合成与代谢。叶色是叶绿体中各种色素的综合体现,正常生长的玉米叶片中叶绿体发育完整,含量丰富,色素正常合成,通常表现为深绿色。近年來相继发现多种玉米叶色突变材料,本文综述该方面的研究进展。
1 玉米叶色突变体
叶绿体是进行光合作用的场所,叶绿体发育和叶绿素的合成效率可显著影响植物的光合作用[1]。叶色突变体往往与叶绿素合成途径及叶绿体的分化和发育有关[2]。而叶色突变是玉米在种植过程突变频率较高且容易鉴定的突变性状,突变基因往往是直接或者是间接影响到叶绿素的合成、降解,导致叶绿素含量变化,所以叶色突变体也称为叶绿素突变[3-4]。目前,在玉米数据库中(http://www.maizegdb.org)已经报道或分离克隆了200多个与玉米叶色相关基因或QTL位点,其中有白叶突变基因18个、黄绿叶突变基因65个、斑叶突变基因26个、条纹叶突变基因58个,在黄绿叶突变体中致死型的突变有9个[5]。但是其中对绝大多数突变体中的突变位点都是定位在某条染色体或染色体上的某个局部区域,并没有学者进行深入的研究[6]。叶色突变体是研究玉米光合作用和光建成的宝贵材料[7],对于玉米叶色相关基因的克隆和功能分析,以及提高光合效率和玉米产量方面具有重要意义。
1.1 elm1(elongated mesocotyl 1)淡绿叶突变体
elm1突变体是在玉米的紫外线诱变群体中筛选得到的,该突变体的幼苗呈淡绿色,中胚轴伸长明显。研究人员发现elm1突变体光敏色素积累的活性下降。在长日照的生长环境下,elm1叶色突变体比野生型植物开花更早,这表明玉米的光敏色素信号可调节玉米开花时间。尽管elm1突变体光敏色素的光感受器受损,但是elm1突变体可以在田间生长至成熟。而且玉米的elm1突变体成为植物色素介导开花时间的直接证据[8]。玉米elm1基因又被称为ZmHY2(as defined by homology to AtHY2)基因,位于玉米的8号染色体,编码34 kDa的297个氨基酸的蛋白,该蛋白是一种玉米植物色素活性所需的PFB(3E-phytochromobilin)合成酶[9]。序列对比发现,玉米elm1突变体在该基因的第3个内含子的3′剪切点的G突变为A,导致蛋白质翻译的提前终止,从而产生了突变体对光的不敏感性[10]。
1.2 elm2(elongated mesocotyl 2)黄绿叶突变体
玉米elm2突变体是从玉米自交群体中分离的黄绿叶突变体,该突变体叶片呈黄绿色,在连续红色或远红色光照射条件下中胚轴更加细长。幼苗在第一次暴露在光下的同时,开始叶绿体基粒堆积和叶绿素生物合成,与此同时植物开始快速绿化[11],这也被称为光形态的发生[12]。功能性植物色素的生物合成有两种途径,一种是合成光敏色素用于脱辅基蛋白,另一种是合成的发色团[13],其包括血红素加氧酶(Heme oxygenase,HO)。研究人员在elm2突变体中HO1同源基因的cDNA测序中发现31 bp的缺失。在elm2幼苗中,红色和远红光的去黄化反应被破坏,中胚轴明显伸长。在elm2突变体中,内源血红素加氧酶活性显著下降。转基因互补实验进一步证实了黄化玉米HO1基因的功能障碍。此外,观察发现未贴壁的类囊体特异性地堆积在elm2突变体的幼苗期。分析发现,该突变体叶片黄化的原因是HO1基因存在31 bp的缺失导致内源HO活性降低并破坏对红光和远红光的去黄化反应。非贴壁类囊体的特定堆积表明HO1基因调节的叶绿素生物合成是通过协调血红素水平与梯形堆积的调节来实现的。ELM2编码玉米中HO1的同源基因,elm2突变体中非贴壁类囊体的特异性堆积表明叶绿素生物合成受HO1调节,是通过血红素水平的协调与梯形堆积实现的[14]。
1.3 ygl-1(yellow-green leaf-1)黄绿叶突变体
玉米黄绿叶突变体ygl-1是从玉米自交系Ye478和Yuanwu02杂交后自花授粉获得的一种新的叶色突变体,突变体在所有生长阶段呈现黄绿色叶表型。与野生型相比,突变体的叶绿素和类胡萝卜素的含量显著降低,叶绿体的发育严重受阻,光合作用的能力降低,遗传分析显示该突变表型受隐性核基因控制。通过基因图谱的克隆,研究人员获得了突变体ygl-1的基因。叶色黄化的原因是编码cpSRP43蛋白的GRMZM2G007441基因在突变体ygl-1的编码区发生单碱基的缺失,导致叶绿体前体合成异常。并且生理和微观分析表明叶绿体发育受到显著影响。在ygl-1突变体生长六周时,与叶绿体发育相关的一些基因的转录水平显著改变。研究结果直接证明了YGL1基因在玉米叶绿体发育中起着重要作用[15]。ygl-1突变体在水稻中也有发现,该突变体的叶片在生长发育过程中,叶绿素积累速度较野生型慢,随着叶片的成熟,叶绿素含量逐渐增加到接近野生型的水平。由于ygl-1突变体对光系统Ⅰ高效率响应,光系统Ⅱ与野生型无异,使其表现出更高的光合效率。导致该突变的原因是叶绿素合成酶基因YGL1的编码区发生单碱基的突变使叶绿素合成酶催化活性下降,叶绿素积累速率减慢,呈现黄绿叶表型[16]。
光系统的两个主要成分是捕光复合物(LHC)和光反应中心复合物。叶绿素酸酯a经CAO氧化形成叶绿素酸酯b是合成叶绿素b的主要途径,叶绿素b的变化会影响LHCⅡ的含量[39-40]。植物缺乏叶绿素b会导致叶绿体类囊体的显微结构发生变化,基粒减少,基质增多[41]。叶绿素b缺乏突变体对高温及强光均有响应,强光下突变体比野生型具有更强的适应能力[42],叶绿素b缺乏突变体对高温较野生型更敏感,影响植物组织中H2O2的积累水平[43-44]。
4.2 叶绿体的遗传发育
叶绿体普遍存在于绿色植物界,其发育受到核基因调控,同时叶绿体发育及功能状态会形成质体反向信号(Plastid retrograde signaling)传达到细胞核,它们相互协调,相互制约,使得叶绿体得到充分发育[45]。玉米叶色突变体由于核基因发生突变,影响相关蛋白的表达,严重影响叶绿体的正常发育,导致其叶色发生变化。
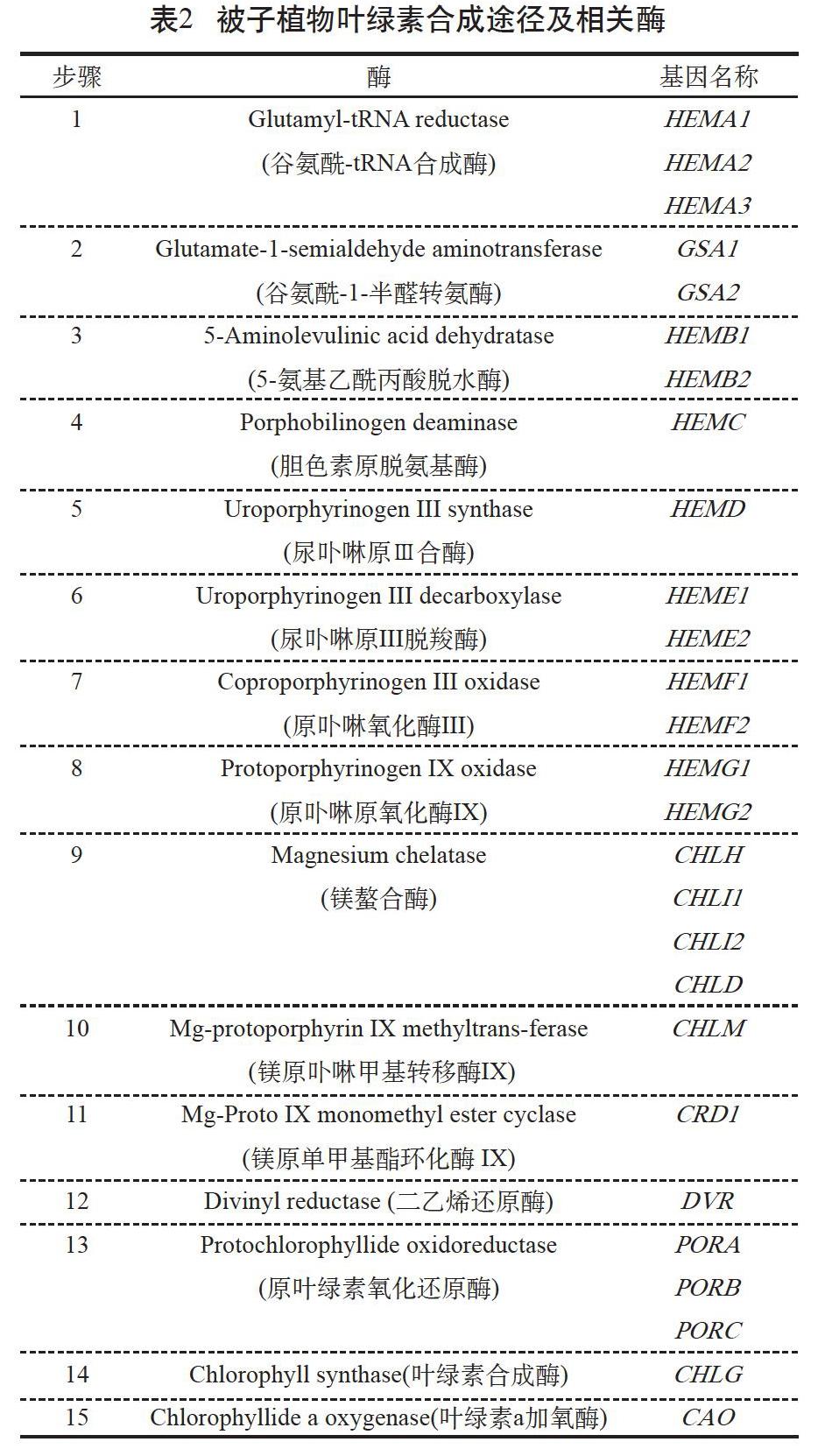
光敏色素相互作用因子家族(PIFs)影响叶绿体的发育和叶绿素的合成,如elm1叶色突变体。Enamul等人发现PIF1可以负调控叶绿素的生物合成,而且PIF1能够特异地与光敏色素A(phyA)和光敏色素B(phyB)的光活化异构体相互作用,调节植物光形态建成阶段叶绿素的合成速率[46]。光敏色素能够在光下结合PIFs启动自身磷酸化进而调节相关基因的转录,如HEMA1、HEMB1、LHCA1[47-48]。PIF1则能够与原叶绿素酸酯氧化还原酶(PORC)的启动子相互作用,影响叶绿素的合成(见图3)[45, 49]。
叶绿体类囊体的发育缺陷同样地会导致玉米叶色的变化。不同植物的类囊体形态各异[50],类囊体的发育受到核基因、转运蛋白的控制,同时囊泡的聚集与增殖是形成基粒片层的关键[51]。该过程有许多酶及蛋白因子的催化和驱动,任何一个功能的缺失都会导致叶绿体发育不良,如elm2、ygl-1、csr1叶色突变体。
此外,叶绿体DNA是否表达还要受到叶绿体中DNA的拷贝数的影响。玉米编码预测的细胞器DNA聚合酶基因white2发生不同区域的突变会引起质体DNA拷贝数呈现梯度降低,导致玉米幼苗产生从白化到浅绿的梯度表型[6]。
5 问题与展望
雖然目前已经发现许多玉米叶色突变体材料,部分网站也对其做了相关性状和基因的统计,但是大多数叶色性状的相关基因还没被找到。突变体材料叶色形成机理和途径还未得到充分了解和深入研究。在玉米品种的改良过程中,其光合作用效率会直接影响产量,玉米叶色突变体材料对研究光合作用具有非常重要的科研价值和应用价值,未来对玉米叶色突变基因的研究还有很大空间。加大对玉米叶色突变基因的研究投入,积极探索其作用机理和调控机制,对提高玉米光合效率具有重要意义。
参考文献:
[1] Alawady A, Reski R, Yaronskaya E, et al. Cloning and expression of the tobacco CHLM sequence encoding Mg protoporphyrin IX methyltransferase and its interaction with Mg chelatase. Plant Mol. Biol. 57(5),679–691.
[2] 陆斌.一个新的水稻缺绿突变体的遗传分析和精细定位[D].扬州:扬州大学,2007.
[3] 杨光明.玉米产业在农业生产中的重要作用及展望[J].农业与技术,2017,37(8):14.
[4] 何冰,刘玲珑,张文伟,等.植物叶色突变体[J].植物生理学报,2006,42(1):1-9.
[5] 韩帅,王立静,钟世宜,等.一个新的玉米叶色突变体的遗传分析及基因定位[J].玉米科学,2012,20(3):26-28.
[6] 彭雷.玉米黄化突变体eti1的鉴定、遗传分析及突变基因初步克隆[D].郑州:河南大学,2015.
[7] 钟世宜,魏海忠,王红红,等.玉米白化突变体As-81647的鉴定及基因定位[J].山东农业科学,2013,45(10):12-15.
[8] Sawers RJH, Linley PJ, Farmer PR, et al. Elongated mesocotyl1, a phytochrome-deficient mutant of maize[J]. Plant Physiology, 2002, 130(1): 155-163.
[9] 杨泽峰,张恩盈,徐暑晖,等.玉米ELM1基因的序列变异及与株型和穗部相关性状的关联分析[J].科技导报,2014,32(35):78-84.
[10] Sawers RJH, Linley PJ, Gutierrez-Marcos JF, et al. The elm1 (ZmHy2) gene of maize encodes a phytochromobilin synthase[J]. Plant Physiology, 2004, 136(1): 2771-2781.
[11] Green BR, Durnford DG. The chlorophyll-carotenoid proteins of oxygenic photosynthesis[J]. Annu Rev Plant Physiol Plant Mol Biol, 1996, 47(1): 685-714.
[12] Davis SJ. The heme-oxygenase family required for phytochrome chromophore biosynthesis is necessary for proper photomorphogenesis in higher plants[J]. Plant Physiology, 2001, 126(2): 656-669.
[13] Sheehan MJ. Structure and expression of maize phytochrome family homeologs[J]. Genetics, 2004, 167(3): 1395-1405.
[14] Dianyi S, Xu Z, Liang L, et al. Chlorophyll deficiency in the maize elongated mesocotyl2 mutant is caused by a defective heme oxygenase and delaying grana stacking[J]. PLoS ONE, 2013, 8(11): e80107.
[15] Guan H, Xu X, He C, et al. Fine mapping and candidate gene analysis of the leaf-color gene ygl-1 in maize[J]. PLoS ONE, 2016, 11(4): e0153962.
[16] 吳自明.水稻黄绿叶基因ygl1的图位克隆及功能分析[D].南京:南京农业大学,2007.
[17] Xing A, Williams ME, Bourett TM, et al. A pair of homoeolog ClpP5 genes underlies a virescent yellow-like mutant and its modifier in maize[J]. The Plant Journal, 2014, 79(2): 14.
[18] Dowell RD, Ryan O, Jansen A, et al. Genotype to phenotype: a complex problem[J]. Science, 2010, 328(5977): 469-469.
[19] Gibson G, Dworkin I. Uncovering cryptic genetic variation[J]. Nature Reviews Genetics, 2004, 5(9): 681-690.
[20] Nadeau, Joseph H. Modifier genes in mice and humans[J]. Nature Reviews Genetics, 2001, 2(3): 165-174.
[21] Suzuki T, Kashiwagi A, Mori K, et al. History dependent effects on phenotypic expression of a newly emerged gene[J]. Biosystems, 2004, 77(1-3): 137-141.
[22] Holding DR, Hunter B G, Chung T, et al. Genetic analysis of opaque2 modifier loci in quality protein maize[J]. Theoretical & Applied Genetics, 2008, 117(2): 157-170.
[23] 钟世宜.三个玉米叶色突变体的鉴定和遗传分析[D].泰安:山东农业大学,2013.
[24] May BP, Liu H, Vollbrecht E, et al. Maize-targeted mutagenesis: A knockout resource for maize[J]. Proceedings of the National Academy of Sciences, 2003, 100(20): 11541-11546.
[25] 杨伟峰.Mutator转座子介导的玉米叶色突变体侧翼序列的克隆及遗传分析[D].保定:河北农业大学,2012.
[26] Motohashi R, Ito T, Kobayashi M, et al. Functional analysis of the 37 kDa inner envelope membrane polypeptide in chloroplast biogenesis using a Ds-tagged Arabidopsis pale-green mutant [J]. Plant J., 2003, 34(5): 719-731.
[27] Beale SI. Green genes gleaned[J]. Trends in Plant Science, 2005, 10(7): 309-312.
[28] Yuan M, Zhao YQ, Zhang ZW, et al. Light regulates transcription of chlorophyll biosynthetic genes during chloroplast biogenesis[J]. Critical Reviews in Plant Sciences, 2017: 1-20.
[29] Tanaka R, Ito H, Tanaka A. Chapter 4 regulation and functions of the chlorophyll cycle[M]. Springer Netherlands, 2010.
[30] Tanaka R, Tanaka A. Chlorophyll cycle regulates the construction and destruction of the light-harvesting complexes[J]. Biochimica et Biophysica Acta, 2011, 1807(8): 968-976.
[31] Czarnecki O, Pfannschmidt T, Grimm B. An Arabidopsis GluTR binding protein mediates spatial separation of 5-aminolevulinic acid synthesis in chloroplasts[J]. Plant Cell, 2013, 23(2): 4476-4491.
[32] Papenbrock J, Susanna Gr?fe, Kruse E, et al. Mg-chelatase of tobacco: identification of a ChlD cDNA sequence encoding a third subunit, analysis of the interaction of the three subunits with the yeast two hybrid system, and reconstitution of the enzyme activity by co-expression of recombinant CHLD, CHLH and CHLI[J]. The Plant Journal, 1997, 12(5): 10.
[33] Adhikari ND, Froehlich JE, Strand DD, et al. GUN4-porphyrin complexes bind the ChlH/GUN5 subunit of Mg-chelatase and promote chlorophyll biosynthesis in Arabidopsis[J]. Plant Cell, 2011, 23(4): 1449-1467.
[34] Adhikari ND, Orler R, Chory J, et al. Porphyrins promote the association of genomes uncoupled 4 and a Mg-chelatase subunit with chloroplast membranes[J]. Journal of Biological Chemistry, 2009, 284(37): 24783-24796.
[35] Noritoshi I, Keisuke K, Takatoshi K, et al. Phytochrome b mediates the regulation of chlorophyll biosynthesis through transcriptional regulation of ChlH and GUN4 in Rice Seedlings[J]. PLoS ONE, 2015, 10(8): e0135408.
[36] Czarnecki O, Grimm B. Post-translational control of tetrapyrrole biosynthesis in plants,algae, and cyanobacteria[J]. Journal of Experimental Botany, 2012, 63(4): 1675-1687.
[37] Tanaka R, Tanaka A. Tetrapyrrole biosynthesis in higher plants[J]. Annual Review of Plant Biology, 2007, 58(1): 321-346.
[38] Yuan M, Zhao YQ, Zhang ZW, et al. Light regulates transcription of chlorophyll biosynthetic genes during chloroplast biogenesis[J]. Critical Reviews in Plant Sciences, 2017, 36(1): 35-54.
[39] 戴新賓,曹树青,许晓,等.低叶绿素b高产水稻突变体及其光合特性的研究[J].植物学报,2000(12):1289-1294.
[40] Biswal AK, Pattanayak GK, Leelavathi S, et al. Modulation of chlorophyll b biosynthesis and photosynthesis by overexpression of chlorophyllide a oxygenase (CAO) in Tobacco[C]. International Conference on Photosynthesis, 2010.
[41] 赵云,王茂林,李江,等.幼叶黄化油菜(Brassica napus L.)突变体Cr3529叶绿体超微结构观察[J].四川大学学报(自然科学版),2003,40(5):974-977.
[42] Havaux M, Tardy F. Thermostability and photostability of photosystem II in leaves of the chlorina-f2 barley mutant deficient in light-harvesting chlorophyll a/b protein complexes[J]. Plant Physiology, 1997, 113(3): 913-923.
[43] 林植芳,彭长连,徐信兰,等.两个新的水稻缺叶绿素b突变体光合作用的热稳定性[J].中国科学,2004,34(5):395-401.
[44] 郭春爱.低叶绿素b对水稻光合机构及其热稳定性的影响[D].南京:南京农业大学,2007.
[45] 李保珠,赵孝亮,彭雷.植物叶绿体发育及调控研究进展[J].植物学报,2014,49(3):337-345.
[46] Huq E. Phytochrome-interacting factor 1 is a critical bHLH regulator of chlorophyll biosynthesis[J]. Science, 2004, 305(5692): 1937-1941.
[47] Tang W, Wang W, Chen D, et al. Transposase-Derived Proteins FHY3/FAR1 Interact with Phytochrome-interacting factor1 to Regulate Chlorophyll Biosynthesis by Modulating HEMB1 during Deetiolation in Arabidopsis[J]. The Plant Cell, 2012, 24(5): 1984-2000.
[48] Goslings D, Meskauskiene R, Kim C, et al. Concurrent interactions of heme and FLU with Glu tRNA reductase (HEMA1), the target of metabolic feedback inhibition of tetrapyrrole biosynthesis, in dark and light grown Arabidopsis plants[J]. Plant Journal, 2010, 40(6): 957-967.
[49] Moon J, Zhu L, Shen H, et al. PIF1 directly and indirectly regulates chlorophyll biosynthesis to optimize the greening process in Arabidopsis[J]. Proceedings of the National Academy of Sciences, 2008, 105(27): 9433-9438.
[50] Austin JR, Staehelin LA. Three-dimensional architecture of grana and stroma thylakoids of higher plants as determined by electron tomography[J]. Plant Physiology, 2011, 155(4): 1601-1611.
[51] 李保珠,趙孝亮,彭雷.植物叶绿体发育及调控研究进展[J].植物学报,2014,49(3):337-345.
(责任编辑:丁志祥)
