陆地棉扩展蛋白基因的鉴定与特征分析
2019-11-19张奇艳雷忠萍宋银海江波贺道华
张奇艳,雷忠萍,宋银,海江波,贺道华
陆地棉扩展蛋白基因的鉴定与特征分析
张奇艳1,雷忠萍2,宋银1,海江波1,贺道华1
(1西北农林科技大学农学院,陕西杨凌 712100;2西北农林科技大学生命科学学院,陕西杨凌 712100)
【】扩展蛋白(Expansin)是细胞壁的重要组成部分,在植物的生长发育及逆境胁迫应答等方面均发挥着重要作用。基于全基因组水平系统鉴定陆地棉Expansin基因家族,并通过生物信息学及表达模式分析,为揭示扩展蛋白基因在棉花生长发育中的功能及后续利用奠定基础。利用BLAST和HMMER在陆地棉基因组中搜索并鉴定扩展蛋白基因家族成员;利用ClustalW、MEGA、MCScanX、Prot Param、MEME、SignalP、Euk-mPLoc、FancyGene和DnaSP等软件对其基因序列和蛋白序列进行生物信息学分析。通过RNA-seq数据分析扩展蛋白基因的表达模式和部分同源基因间表达差异,利用qRT-PCR验证部分扩展蛋白基因的表达谱。陆地棉基因组中含有46个EXPA基因、8个EXPB基因、6个EXLA基因和12个EXLB基因,合计72个Expansin基因;四倍体陆地棉中扩展蛋白成员的数量几乎是二倍体棉种(亚洲棉与雷蒙德氏棉)的2倍。除GhA02和GhD06 2个染色体外,其余各染色体上均分布有数目不等的扩展蛋白基因(2—4个),具有部分同源关系的染色体GhA08和GhD08分别有5个和8个扩展蛋白基因。系统发育树显示,各亚家族成员聚集成群,并且大部分的末端分支均由来源于3个物种的4个(亚)基因组的4个基因组成,如EXPA亚家族的///等等,4个基因之间具有同线性关系。亚细胞定位发现陆地棉所有的扩展蛋白均位于细胞外。基因结构分析显示,扩展蛋白基因由3—5个外显子组成,外显子-内含子结构在进化上高度保守且与氨基酸序列的多样性一致,且在外显子上存在密码子偏好性。RNA-seq数据显示,不同基因在不同时空条件下存在特异性表达,如和相比其他基因在纤维10 DPA和20 DPA中的表达量很高;在不同的组织(如子叶、新叶、老叶、苞叶)中,具有较高的表达量。部分同源基因之间具有不同的表达模式,显示它们之间功能的异化与互补。qRT-PCR结果与RNA-seq数据基本吻合,如和在纤维发育的伸长阶段高量表达。和在3DPA的胚珠中表达活跃。陆地棉基因组中含72个扩展蛋白基因,其在DNA水平和氨基酸水平具有一致的结构多样性和进化保守性,在转录水平具有各异的表达模式,显示出家族内成员间功能上的异化与互补。
陆地棉;扩展蛋白;基因家族;生物信息学;基因表达
0 引言
【研究意义】棉花是一种重要的纤维作物。棉纤维是由胚珠外表皮的单个细胞发育而成,是高度伸长、增厚、没有分支的单细胞表皮毛,与拟南芥表皮毛的发生机制相似。细胞壁对棉纤维的品质起决定作用,是改良棉花品质的重要途径之一。棉纤维的伸长与细胞壁松弛联系密切,扩展蛋白能够打断纤维素微丝间的氢键,从而使细胞壁延展疏松[1]。研究表明,扩展蛋白在植物的生长发育与逆境胁迫应答等方面具有重要作用,对棉花的遗传改良具有潜在价值。【前人研究进展】1992年,Mc Queen-Mason等[2]从黄瓜下胚轴细胞壁中分离纯化得到2个分子量为29和30 kD能诱导细胞壁恢复酸生长的蛋白。这种蛋白粗提液可在体外诱导热失活细胞壁的重新伸展,但伸展过程受到pH和金属离子等因素的影响。研究发现,这种现象是由细胞壁上的一类细胞壁疏松蛋白介导的,因而将其命名为扩展蛋白(Expansin)[3]。扩展蛋白能够打断细胞壁多糖之间的非共价键,从而使细胞壁聚合物发生膨压诱导的蠕动,对细胞壁起到疏松作用,使细胞壁的柔韧性增加而松弛[4-5]。随后,人们相继从水稻[6]、玉米[7]、拟南芥[6]、大豆[8]、番茄[9]、葡萄[10]等100多个物种的细胞壁中也鉴定出扩展蛋白,推测扩展蛋白广泛存在于双子叶植物和单子叶植物中。研究表明,扩展蛋白能影响植物的生长发育,诸如促进组织细胞的生长[11]、种子发育[12]、根毛起始和根系生长[13-14]、叶和茎的发育[15-19]、花粉管伸长[20-21]、促进叶柄脱落[16]、果实成熟[22-23]和降低番茄的裂果率[24]等。另外,在植物抗逆性方面,扩展蛋白也发挥着重要作用,如抗旱性[25-26]、耐盐性[27-28]、抗高温[29]和抗病性[30]等。棉花为人类提供了一种优良的天然纤维,其发育过程包含5个彼此重叠的时期,依次为起始期(initiation)、伸长期(elongation)、初生壁向次生壁发育的转换期(transition)、次生壁合成期(secondary wall biosynthesis)和脱水成熟期(maturation)[31]。研究表明,扩展蛋白在转录水平上与棉纤维的伸长联系紧密[32-33],许多扩展蛋白基因在纤维细胞的伸长中高度表达[34-35]。Xu等[36]发现在棉花中过表达可以显著增加棉铃数量,单株棉花纤维产量可增加40%,对纤维品质和棉株生长没有不利的影响。Li等[37]发现(来自At亚基因组,是Dt亚基因组中的部分同源基因)在次生壁合成和代谢中发挥重要作用。Bajwa等[38]发现,转棉株的纤维长度和马克隆值均有显著提高。以上研究结果均表明扩展蛋白在棉花中具有重要的作用。【本研究切入点】2012年,Wang等[39]和Paterson等[40]分别完成了棉花D基因组雷蒙德氏棉()的全基因组测序工作;2014年,Li等[41]完成了棉花A基因组亚洲棉()石系亚1号的全基因组测序及组装工作,绘制出的亚洲棉基因组约为1 694 Mb。在上述成果的基础上,2015年,Li等[42]和Zhang等[43]分别独立完成了异源四倍体陆地棉()TM-1的全基因组测序及组装。随后,Chen(https://www.cottongen.org/species/Gossypium_ hirsutum/jgi-AD1_genome_v1.1)、Wang等[44]和Hu等[45]分别对棉属的基因组序列图谱进行了更新和完善,为在陆地棉全基因组范围内系统进行扩展蛋白基因家族的鉴定、进化和功能分析提供了条件。【拟解决的关键问题】本研究基于陆地棉及相关二倍体棉种的基因组测序的序列信息,对棉花扩展蛋白基因家族进行筛选鉴定,并从DNA水平(基因外显子-内含子结构、密码子偏好性、同线性)、RNA水平(基于RNA-seq和qRT-PCR的基因表达模式、部分同源基因间表达丰度差异)和氨基酸水平(信号肽、模体结构、理化性质及亚细胞定位)对其进化及功能进行分析,为进一步研究扩展蛋白在棉花(纤维)生长发育等生物学过程中的功能奠定基础。
1 材料与方法
1.1 棉花中扩展蛋白家族成员的鉴定
陆地棉基因组数据下载于http://mascotton.njau.edu.cn[43],亚洲棉基因组数据下载于http://cgp.genomics. org.cn[41]。拟南芥和水稻扩展蛋白家族成员序列信息下载于Daniel Cosgrove实验室建立的网站http://www.personal.psu.edu/。扩展蛋白成员的搜索步骤参见雷忠萍等[46]。以拟南芥和水稻扩展蛋白基因的序列为query,利用BLAST(ftp://ncbi.nlm.nih.gov/blast/ executables/)软件,在陆地棉及亚洲棉基因组序列中检索扩展蛋白基因。然后,利用HMMER(http://hmmer. janelia.org/)软件鉴定上述准扩展蛋白成员是否同时含有pfam01357(Pollen_allerg_1)和pfam03330(DPBB_1)2个保守性的特征结构域,HMMsearch的E值设置为<1E-05。
1.2 棉属扩展蛋白成员系统发育树的构建及基因命名
利用软件ClustalW对所鉴定的棉花、拟南芥和水稻的扩展蛋白氨基酸序列进行多序列比对分析;利用软件MEGA v5.0,采用Neighbor-Joining法构建扩展蛋白氨基酸序列的系统发育树。参照拟南芥及水稻的扩展蛋白的亚家族分类,将棉花的扩展蛋白家族成员归类到EXPA、EXPB、EXLA和EXLB 4个亚家族中,根据陆地棉、亚洲棉各扩展蛋白基因与雷蒙德氏棉Expansin基因之间的同线性关系、系统发育树、及其在染色体上的位置顺序等信息对各基因进行命名。
1.3 同线性及基因家族成员间进化关系分析
通过BLAST比对,将所获得的各扩展蛋白基因定位到相应的染色体上。利用MCScanX软件分析各扩展蛋白基因之间的同线性关系。利用Circos软件图形化展示扩展蛋白基因在染色体上的物理位置及基因间的同线性关系。
1.4 理化性质、信号肽、模体结构及亚细胞定位分析
利用在线软件Prot Param(http://web.expasy.org/ protparam/)获取各扩展蛋白的理论等电点(theoretical)、分子量(molecular weight)、蛋白不稳定指数(instability index)、脂溶指数(aliphatic index)和总平均疏水性(grand average of hydropathy,GRAVY)。利用软件MEME(http://memesuite.org/tools/meme),通过命令“meme GoExpansinPepSequences.txt -protein- oc.-nostatus-time 18000-maxsize 200000-mod zoops -nmotifs 20-minw 6-maxw 25”对扩展蛋白基因家族成员进行模体(基序,motif)结构分析。利用SignalP(http://www.cbs.dtu.dk/services/SignalP)分析信号肽。利用在线软件Euk-mPLoc 2.0(http://www.csbio.sjtu. edu.cn/bioinf/euk-multi-2/#)对各扩展蛋白进行亚细胞定位分析。
1.5 基因结构和密码子偏好性分析
通过对各扩展蛋白基因的CDS序列与DNA序列进行BLAST比对,利用http://bio.ieo.eu/fancygene绘制基因外显子-内含子结构图。利用软件DnaSP 5.10对陆地棉中扩展蛋白基因密码子偏好性(Codon usage bias)进行统计。
1.6 扩展蛋白基因的表达特征分析
1.6.1 转录组(RNA-seq)数据分析 为了获得扩展蛋白基因在不同时空条件的表达特征,参考He等[47]的方法,从NCBI SRA数据库下载2组深度测序(RNA-seq)数据:SRP059947和SRP017168。利用Tophat将RNA-seq的短读序列mapping到陆地棉基因组上,利用cufflinks函数估算扩展蛋白基因在不同时空上的表达水平(fragments per kilobase of exon modelper million reads mapped,FPKM值),通过cuffdiff函数筛出在不同时空之间存在表达差异的基因。根据RNA-seq数据的分析结果,挑选13个扩展蛋白基因进行实时荧光定量PCR分析,以便进一步验证其表达特征。
1.6.2 实时荧光定量PCR(quantitative real-time PCR,qRT-PCR) 于陆地棉材料N874系的花铃期,取-3、-1、0、3、5、10和15 DPA(days post-anthesis)的棉铃分离出胚珠,取5、10、15和20 DPA的棉铃分离出纤维。利用总RNA提取试剂盒(天根,北京)从棉花胚珠和纤维中提取RNA,用NanoDrop 2000和1%的琼脂糖凝胶电泳检测RNA的含量和质量。以RNA为模板反转录合成cDNA。qRT-PCR引物下载于西南大学(http://biodb.swu.edu.cn/qprimerdb),采用UBQ7(AF024716)作为内参基因(表1)。用Applied Biosystems QuantStudio 7 Flex实时PCR系统(Applied Biosystems,USA)进行qRT-PCR。反应程序:94℃ 3 min;94℃ 45 s,52℃ 45 s,72℃ 45 s,40个循环。每个qRT-PCR样品设置3个重复。使用2-△△CT法计算相对表达量。

表1 实时荧光定量PCR引物
2 结果
2.1 扩展蛋白家族成员的鉴定
根据拟南芥和水稻扩展蛋白的基因序列(作为query),通过BLAST比对搜索过程,初步鉴定出棉属准扩展蛋白家族成员。然后通过HMMER检测pfam01357和pfam03330 2个结构域存在与否,最终确认为扩展蛋白。在陆地棉中,共鉴定出72个扩展蛋白家族成员(表2);同时,在二倍体物种亚洲棉中,鉴定出39个扩展蛋白基因,与在雷蒙德氏棉中鉴定出的扩展蛋白数量一致[46]。其中,同为双子叶植物的拟南芥、杨树和大豆中,拟南芥和杨树的扩展蛋白基因数量与二倍体棉相近,大豆的扩展蛋白数量则与四倍体棉相近(表3)。基于本研究所鉴定的棉花、拟南芥和水稻扩展蛋白的系统发育树分析,将陆地棉和亚洲棉扩展蛋白划分为4个亚家族(EXPA、EXPB、EXLA和EXLB)。陆地棉的72个扩展蛋白成员中,46个属于EXPA亚家族,8个属于EXPB亚家族,6个属于EXLA亚家族,12个属于EXLB亚家族。亚洲棉39个扩展蛋白成员的亚家族分布与雷蒙德氏棉类似,EXPA、EXPB、EXLA和EXLB 4个亚家族的成员数分别为26、4、3和6个。数据显示,四倍体陆地棉中扩展蛋白成员的数量几乎是二倍体棉种(亚洲棉与雷蒙德氏棉)的2倍(四倍体陆地棉中EXPA亚家族的成员数量(46)比二倍体的数量(26)的2倍少),说明二倍体经过远缘杂交和基因组加倍形成四倍体后,扩展蛋白家族内的成员得到继承,仅亚家族EXPA中的个别成员发生了丢失。在各个亚家族内,根据各基因在染色体上的位置顺序,对陆地棉扩展蛋白基因进行了命名(表2)。
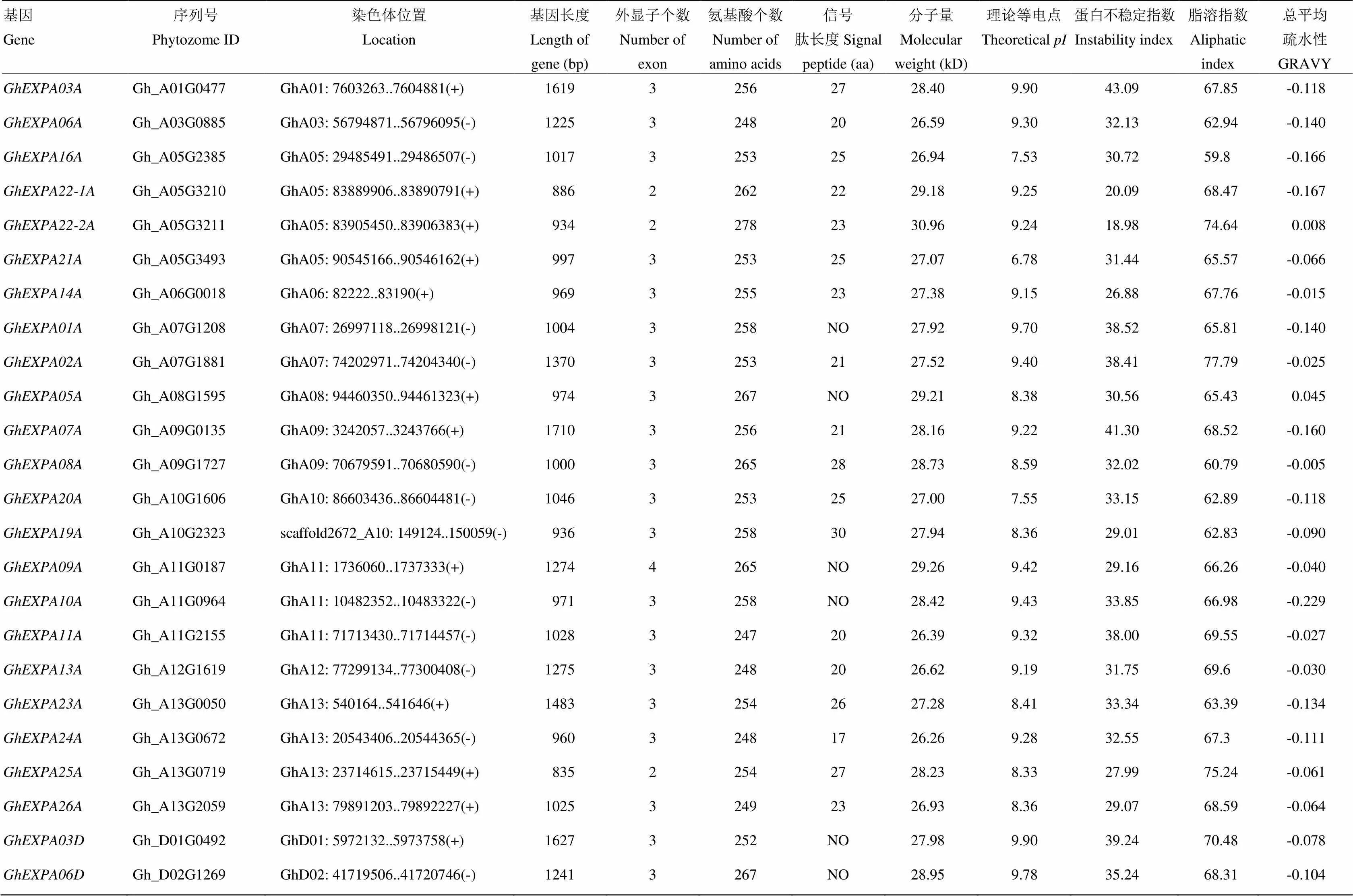
表2 陆地棉扩展蛋白基因家族成员的鉴定

续表2 Continued table 2
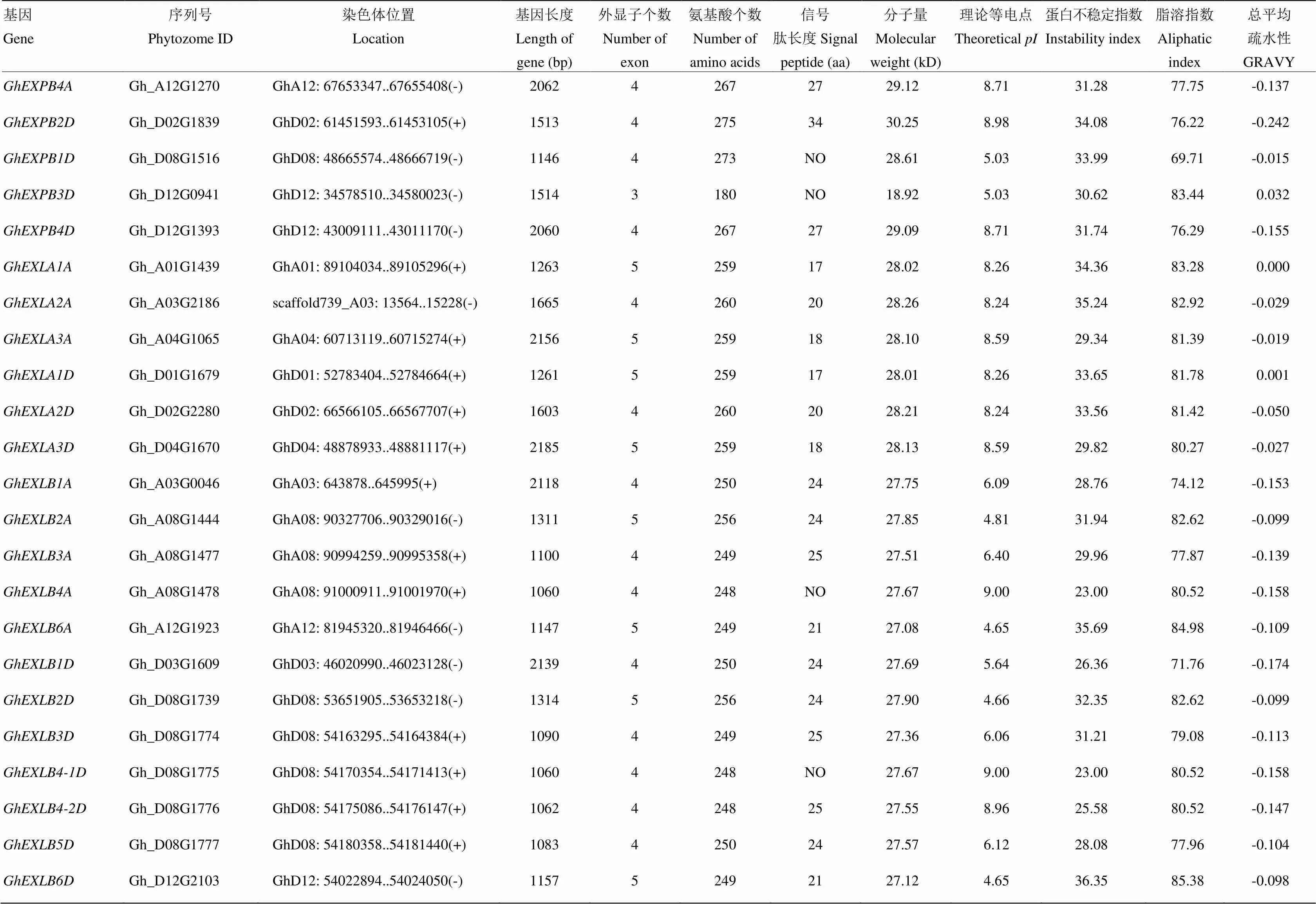
续表2 Continued table 2

表3 不同植物扩展蛋白及其各亚家族内基因数目
鉴定结果显示,扩展蛋白在陆地棉染色体上呈现不均匀分布,在大多染色体上有2—4个扩展蛋白基因。具有部分同源关系(homeologous)的GhA08和GhD08 2个染色体具有较多的扩展蛋白基因,分别为5和8个。染色体GhA02和GhD06上没有扩展蛋白基因。有的基因家族成员在染色体上呈现成簇(cluster)存在的现象,尤其是同一亚家族的成员,如GhD08染色体上的///形成串联重复(tandem duplication)。/、/分别形成了2个小的串联重复。
2.2 系统发育树的构建与同线性分析
陆地棉、亚洲棉和雷蒙德氏棉扩展蛋白的系统发育树显示,亚家族EXLA与EXLB的遗传距离较近,EXPA与其余3个亚家族的相似性较小,表明了各亚家族间的亲缘(起源)关系及进化关系(图1)。亚家族内的各成员聚集在同一分支中(聚集成群),拥有各自的进化枝。大部分的末端分支由来源于3个物种的4个(亚)基因组的4个基因组成,如:EXPA亚家族的///,EXLB亚家族的///等。4个基因分别来自A基因组、At亚基因组、Dt亚基因组和D基因组,表明扩展蛋白的氨基酸序列在物种间/(亚)基因组间具有平行的相似性,四倍体陆地棉的2个亚基因组(At、Dt)与对应的二倍体基因组(A、D)有较近的亲缘关系。
利用软件MCScanX对棉属扩展蛋白在(亚)基因组间的同线性进行分析,结果显示,系统发育树中来源于A、D、At、Dt基因组/亚基因组的4个末端分支基因多具有同线性关系(图1系统发育树中绿色椭圆表示具有同线性关系),相互之间属于垂直同源基因(orthologous),陆地棉的2个亚基因组At与Dt在二倍体物种中也分别存在同线性同源区段。深入分析进一步表明,各基因组内部扩展蛋白之间存在一定的平行同源关系(paralogous),如图1系统发育树中紫色椭圆所示,显示了各基因组内部扩展蛋白基因的扩张(数量增加)历程,即大规模的染色体片段重复(segmental duplication)导致了基因家族的扩张,由共同祖先基因组中的15—17个[5]扩张到陆地棉中的72个。
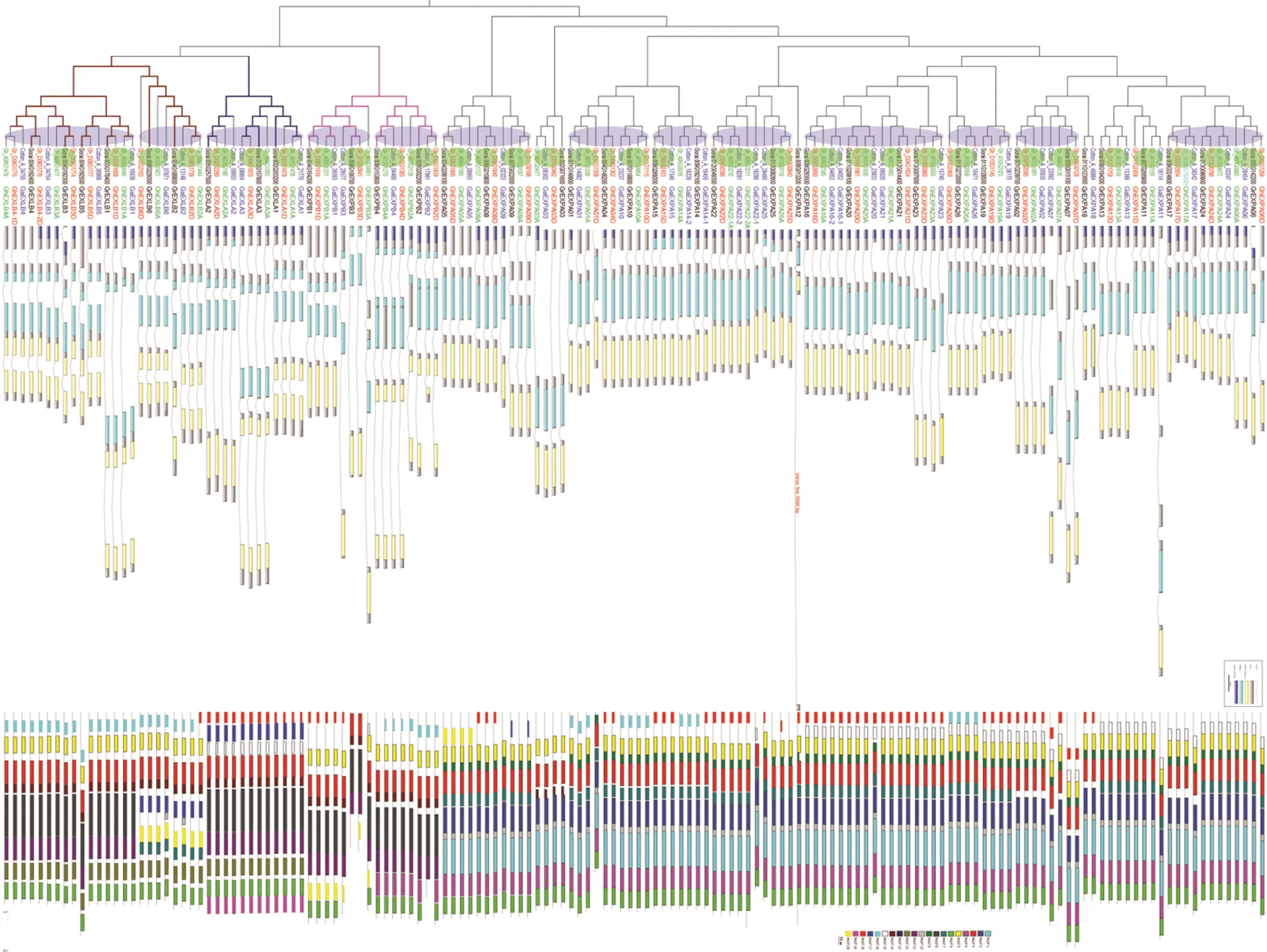
图1 棉属扩展蛋白的系统发育树、外显子-内含子结构和氨基酸模体结构
四倍体陆地棉的2个亚基因组At与Dt间的同线性分析(图2)显示,除个别基因()外,大多数扩展蛋白基因均位于同线性染色体区段上,说明AA与DD杂交加倍进化形成AtAtDtDt的陆地棉后,2个亚基因组At与Dt仍保持较高的相似性,并且遗传了其二倍体祖先基因组的染色体倍增的特点。值得注意的是,这种继承并不是完全的,扩展蛋白基因在染色体结构上发生了微小变化。

图2 陆地棉扩展蛋白基因的染色体位置及亚基因组间的同线性关系
2.3 理化性质、信号肽、模体结构及亚细胞定位分析
陆地棉72个扩展蛋白的氨基酸序列长度大多在247—278(除180()、207()和303()外),分子量大多在26.39—30.96 kD(除18.92()、22.54()和33.80()外),平均值为27.76 kD。等电点在4.65—9.90,平均值为8.18,75%的扩展蛋白的等电点大于8.0,即大多数陆地棉扩展蛋白偏碱性。除个别成员外,扩展蛋白不稳定指数在18.98—43.09,多数蛋白稳定性较好。脂溶指数在58.78—85.38,平均值达到71.39,其中有14个扩展蛋白的脂溶指数超过80,属于嗜热型蛋白。脂溶指数较高使得扩展蛋白可以较好地适应各种环境。总平均疏水性(GRAVY)在-0.247—0.045,均属于亲疏性相当的两性蛋白(大于0表示疏水性,小于0表示亲水性,介于±0.5之间为两性蛋白)[49](表3)。利用软件SignalP分析扩展蛋白信号肽,结果显示72个扩展蛋白中55个含有长度不等(17—34 aa)的信号肽。亚家族EXPB的信号肽长度均值为28.5 aa;亚家族EXLA的信号肽长度均值为18.3 aa,显著地(< 0.01)低于其他3个亚家族。
利用软件MEME对棉属3个物种的扩展蛋白氨基酸序列的保守基序(motif)进行分析(图3),获得20个(A—T)可靠的基序(e<1.20E-118)。其中EXPA亚家族的羧基端保守基序组合均为BJADF,而氨基端基序组合的种类则多达15种,该亚家族的氨基酸基序P、Q、R和T是不相容(兼容)的,其基序组合与系统发育树的呈现结果一致,均能说明基因的进化关系。EXPB亚家族的基序组合大多为PECNHKDF和RECNHKTF(除例外)。EXLA亚家族的基序组合保守程度非常高,组合形式都为RQOCNHKLFS。EXLB亚家族的基序组合为PECNQTGLF和PECNHKLF(除和例外)。值得注意的是,EXPA亚家族含有不同于其他亚家族的模体I,EXLA亚家族含有不同于其他亚家族的模体S,该模体构成表明,各亚家族扩展蛋白在某些代谢途径中可能具有特殊功能。
通过软件Euk-mPLoc 2.0进行亚细胞定位,结果表明,陆地棉72个扩展蛋白也全部定位于细胞外。

图3 棉属扩展蛋白家族氨基酸序列模体(基序)
2.4 基因结构组成和密码子偏好性分析
为了在DNA水平进一步了解陆地棉扩展蛋白,对基因的“外显子-内含子”结构进行分析。陆地棉扩展蛋白基因的DNA长度在829—2 185 bp(除外),其基因结构图(图1)表明,同一亚家族的扩展蛋白基因在“外显子-内含子”结构上非常相似。除个别基因(和)的内含子较长外,系统发育树中聚类在一起的基因的外显子数目、长度基本一致,内含子长度也很接近。表明扩展蛋白基因结构是相对较保守的,在进化上具有一定的保守性,且与氨基酸序列的多样性保持一致。
密码子的偏好性分析既可以为基因表达选择适合的表达系统,又能为密码子的改造而改变基因表达量提供依据。生物的遗传密码中,除色氨酸和甲硫氨酸只有一个密码子外,其余氨基酸都有一个以上的简并密码子。简并密码子中,某一密码子相对同义密码子使用频率单值超过60%或者超过该组同义密码子平均占有频率的1.5倍的密码子即为高频密码子。对陆地棉72个扩展蛋白基因家族成员的18 455个密码子进行统计分析(表4),数据显示,UUG、CCU、GCU、AGA、AGG为高频密码子;UCG、CCG、ACG、GCG、CGG等第2、3位碱基为C和G的密码子为稀有密码子。4个亚家族之间的比较发现,AGG是4个亚家族共有的高频密码子,CGG是共有的低频密码子;EXPA和EXLA 2个亚家族具有较多相似的偏好性密码子。
2.5 基于转录组(RNA-seq)数据的Expansin基因表达模式
基因的表达与植物的生长发育密切相关,基于转录组(RNA-seq)数据的时空表达模式可以为探究扩展蛋白基因的功能提供线索。RNA-seq数据SRP059947中样品包括子叶、根、茎、叶(新叶、老叶)、苞叶、花萼、花冠、雌蕊雄蕊、胚珠(0、10、20、30和40 DPA)、纤维(10和20 DPA)共16个组织器官或发育阶段(图4)。RNA-seq分析表明,在不同组织、不同发育时期扩展蛋白基因的表达量存在较大差异,如:和在10和20 DPA纤维中的表达量很高,和在30和40 DPA胚珠中高量表达,和分别在10和20 DPA的胚珠中活跃表达,在20 DPA胚珠表达量特别高。在10和20 DPA的胚珠及10和20 DPA的纤维中均高量表达,表明对纤维的伸长(elongation (i.e., primary cell wall synthesis, 3–17 DPA))和纤维次生壁的合成阶段(secondary cell walls synthesis (17–23 DPA))有重要贡献。在根中,、、和表达量很高。在茎中,、和特高量表达;另外,和在茎中也有较高表达水平。在新叶中,、、及的表达量很高。在老叶中,、和高量表达。在子叶(cotyledon)中,、、及的表达量很高。值得注意的是,同时在子叶、新叶、老叶、苞叶(bracts)中均有很高的表达量,推测该基因可能对叶片组织的发育起到重要作用。在苞叶和花萼(calyxs)中,、有很高的表达量。在花冠(corollas)和雌雄蕊(gynoecium and androecium)中,、、、、、和具有较高的表达量。这些数据显示,不同的Expansin基因,在不同的时空条件下发挥功能,Expansin基因家族的成员在功能上既各司其职又相互补充。

表4 陆地棉扩展蛋白基因密码子的相对使用频率
aa:氨基酸amino acid;Cod:密码子codon;Freq:频率frequency
2.6 基于qRT-PCR的Expansin基因表达谱分析
基于转录组数据(RNA-seq)的表达模式分析初步提供了各基因在不同时空条件下的表达特征。对于至少在纤维和胚珠的发育的某个阶段表达丰度很高的基因,为了进一步明确其特异表达情况,本研究挑选13个扩展蛋白基因,通过qRT-PCR进行表达模式的验证(图5)。结果表明,qRT-PCR和RNA-seq均显示Expansin基因在各种组织/阶段中差异表达。和在纤维发育的伸长阶段表现出高表达,其中,在15 DPA表达量最高,有着同样的表达趋势。同时,的表达在整个纤维细胞起始和伸长过程中逐渐增加。和在纤维发育的10 DPA表达量较高。和在3 DPA的胚珠发育期间高度表达,在胚珠发育的10和15 DPA超高量表达,在胚珠发育的15 DPA高量表达,而在胚珠发育的3—10 DPA表达量都很高。而和主要在10—15 DPA胚珠和15—20 DPA纤维中表达。总的来说,除了少数基因外,RNA-seq数据和qRT-PCR结果基本一致,进一步显示Expansin基因在胚珠/纤维生长和发育中的潜在功能。
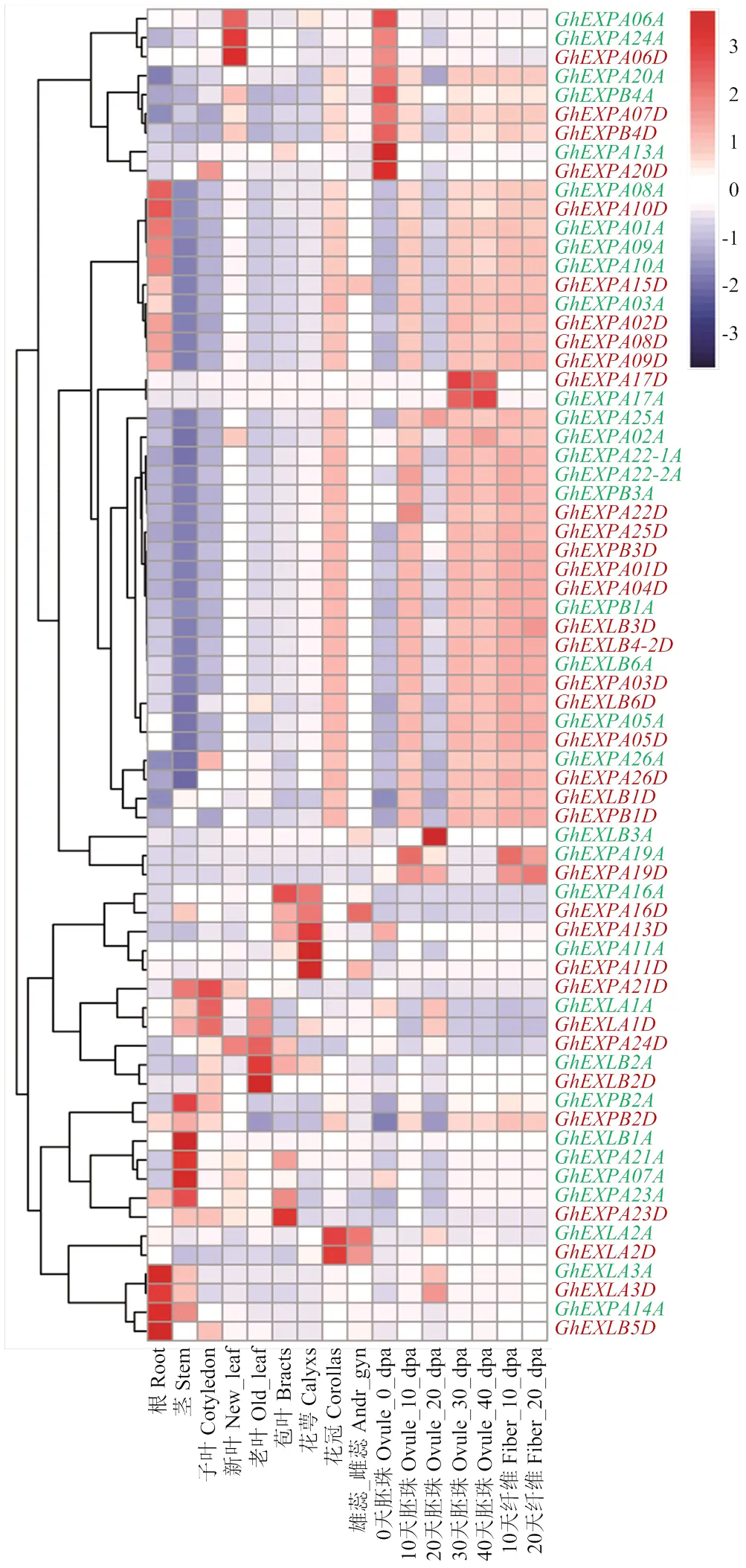
图4 不同组织及不同时期扩展蛋白基因表达分析

2.7 部分同源(homoeologous)基因间的表达丰度比较
异源四倍体的陆地棉,其基因组中每个基因都具有一个部分同源(homoeologous)基因。具有部分同源关系的基因对(pair)从同一祖先进化而来,经过了漫长的物种进化过程,基因的功能是否发生变化、表达模式是否相同,通过对RNA-seq数据(SRP017168和SRP059947)的分析,对陆地棉中具有同线性的直系同源基因的表达量进行比较,二项分布(binomial distribution)统计分析显示,具有部分同源关系的一对基因在相同的时空条件下,其表达模式具有差异。和、和、和、和、和、和这6个部分同源基因对(pair),各基因对之间在不同的时空条件下存在表达差异,At和Dt2个亚基因组的表达丰度不一致。如:幼嫩叶片中,的表达量(位于Dt亚基因)>的表达量(位于At亚基因);在花萼中则相反,的表达量<的表达量。同时也存在一些部分同源基因对,如基因和,在部分时空条件下表达丰度均很低,但在茎、新叶、花冠中,基因的表达量显著低于的表达量。这些分析结果显示,异源四倍体陆地棉的部分同源基因对所发挥的作用可能存在差异,或是在进化过程中发生了功能异化(divergence)。
3 讨论
3.1 扩展蛋白基因家族的成员鉴定及进化分析
在植物界,扩展蛋白基因家族包含4个亚家族:EXPA、EXPB、EXLA和EXLB。本研究在陆地棉全基因组水平上对Expansin基因家族进行鉴定与分析,以氨基酸序列中同时含有Pollen_allerg_1和DPPB_1这两个保守性特征结构域为标准,在陆地棉基因组中共鉴定出72个扩展蛋白基因。将陆地棉与拟南芥、水稻的扩展蛋白基因一起进行系统发育树分析,结果显示,陆地棉中4个亚家族EXPA、EXPB、EXLA、EXLB的基因分别有46、8、6和12个。
陆地棉是由二倍体的祖先物种经过远缘杂交和染色体加倍进化而来,而二倍体的祖先物种的现代种是亚洲棉和雷蒙德氏棉。因此,为了研究陆地棉中Expansin基因家族的进化特征,本研究参阅了雷蒙德氏棉中Expansin基因鉴定的结果[46],同时在亚洲棉中进行了Expansin基因的鉴定(图1的系统发育树)。与雷蒙德氏棉和亚洲棉相比,陆地棉扩展蛋白各亚家族的基因数量几乎是二倍体棉种的2倍(EXPA亚家族46﹕26;EXPB亚家族8﹕4;EXLA亚家族6﹕3;EXLB亚家族12﹕6),但陆地棉扩展蛋白基因的数量(72个)少于二倍体雷蒙德氏棉和亚洲棉的扩展蛋白数量之和(39+39=78),可能原因是在四倍体形成后的进化过程中,染色体片段的复制(segmental duplication)和重排(chromosomal rearrangement)导致这些(6个)基因因异位(ectopy)而被淘汰或丢失(gene loss)。其他物种中的研究表明,双子叶植物中EXPA亚家族比EXPB亚家族的扩展蛋白数量多[48],表2显示陆地棉中EXPA与EXPB亚家族分别占总扩展蛋白数量的63.9%和11.1%,亚洲棉和雷蒙德氏棉EXPA与EXPB类扩展蛋白分别占总扩展蛋白的66.7%和10.3%,与前人研究结果一致。图1还显示,各亚家族内的亲缘关系相近的基因在基因的“外显子-内含子”结构及氨基酸序列模体(motif)组成等方面具有一定的相似性,表明在Expansin的进化方面,DNA水平的多样性与蛋白水平的多样性是基本吻合的。系统发育树的拓扑结构(topology)也和拟南芥、水稻等植物中的结果一致,这些是亚家族划分的有利依据。利用软件Euk-mPLoc 2.0对Expansin家族基因进行了亚细胞定位,结果显示所有的Expansin蛋白均定位于细胞外,推测它们均位于细胞壁中,可能与Expansin蛋白在棉属中所起的作用有关。
植物的共同祖先(last common ancestor)中,4个亚家族EXPA、EXPB、EXLA、EXLB基因的数量分别是10—12、2、1、2[5]。本研究发现,陆地棉扩展蛋白家族获得了极大的扩张。大规模的基因组重复(large-scale genomic duplication)、平行同源(paralogous)基因及部分同源(homoeologous)基因间功能多样化(diversification)是生物进化的动力之一[47,55],也是陆地棉扩展蛋白家族极大地扩张(contributed to the expansion of thefamily)的原因。利用软件MCScanX进行的同线性分析显示,陆地棉中除了3个串联重复(tandem duplications)区段(/;/;///)外,其余均为片段重复(dispersed segmental duplications)。陆地棉进化过程中的多次基因组/染色体片段重复(duplicated (syntenic) blocks)及微量的丢失(gene loss)使得Expansin基因家族成员扩张到72个。
3.2 Expansin家族基因的表达与功能
棉花纤维、胚珠等各种组织的生长发育与基因的表达丰度密切相关,Expansin基因在这些不同的生命活动阶段可能发挥着重要作用。Orford等[34]克隆了扩展蛋白基因,该基因在纤维发育的伸长阶段特异表达;Ruan等[56]研究表明在纤维伸长阶段可观察到扩展蛋白基因的高量表达;Li等[37]发现在纤维细胞伸长期间显著上调表达,表明在纤维发育中具有重要的功能。Bajwa等[38]通过农杆菌介导的转化,在棉花中过量表达可以改善纤维品质。同样,Indrais等[57]通过RT-PCR和Northern印迹发现特异的扩展蛋白对纤维伸长具有贡献作用。本研究中,RNA-seq数据(SRP017168、SRP059947)分析结果同样表明(图4),各扩展蛋白基因在纤维和胚珠的不同发育时期特异表达。如和相比其他基因在纤维中的表达量很高;和在胚珠30和40 DPA高量表达;在胚珠20 DPA表达量特别高。基于RNA-seq数据的分析结果,对于那些在纤维和胚珠的某个时期高量表达的基因,选择了13个基因进行qRT-PCR定量验证,旨在分析Expansin基因对纤维发育过程中的影响和作用,大多数基因的表达模式与RNA-seq数据分析的结果一致。这些特定时空条件下活跃表达的基因,也许是棉花种质创新的重要候选基因。当然,少数基因的基于qRT-PCR的表达模式与RNA-seq数据分析结果存在差异,可能是因为RNA-seq测序样本与本研究中qRT-PCR棉花材料的不同所致。
4 结论
陆地棉基因组中含有72个扩展蛋白基因,包括EXPA、EXPB、EXLA和EXLB 4个亚家族的46、8、6和12个基因。在DNA水平和氨基酸水平上具有进化的多样性与保守性。多数基因具有表达时空特异性。
[1] SMART L B, VOJDANI F, MAESHIMA M, WILKINS T A. Genes involved in osmoregulation during turgor-driven cell expansion of developing cotton fibers are differentially regulated., 1998, 116(4): 1539-1549.
[2] MCQUEEN-MASON S, DURACHKO D M, COSGROVE D J. Two endogenous proteins that induce cell wall extension in plants., 1992, 4(4): 1425-1433.
[3] COSGROVE D J, LI Z C. Role of expansin in cell enlargement of oat coleoptiles (analysis of developmental gradients and photocontrol)., 1993, 103(4): 1321-1328.
[4] COSGROVE D J. Loosening of plant cell walls by expansions.,2000, 407(6802): 321-326.
[5] COSGROVE D J. Growth of the plant cell wall.,2005, 6(11): 850-861.
[6] SAMPEDRO J, COSGROVE D J. The expansin superfamily., 2005, 6(12): 242.
[7] ZHANG W,YAN HW,CHEN WJ,LIU JY,JIANG CP,JIANG HY,ZHU SW,CHENG BJ. Genome-wide identification and characterization of maize expansin genes expressedin endosperm., 2014, 289(6): 1061-1074.
[8] ZHU Y, WU N, SONG W, YIN G, QIN Y, YAN Y, HU Y. Soybean () expansin gene superfamily origins: segmental and tandem duplication events followed by divergent selection among subfamilies., 2014, 14(1): 1-19.
[9] LU Y E, LIU L F, WANG X, HAN Z H, OUYANG B, ZHANG J H, LI H X. Genome-wide identification and expression analysis of the expansin gene family in tomato., 2016, 291(2): 597-608.
[10] DAL SANTO S, VANNOZZI A, TORNIELLI G B, FASOLI M, VENTURINI L, PEZZOTTI M, ZENONI S. Genome-wide analysis of the expansin gene superfamily reveals grapevine-specific structural and functional characteristics.,2013, 8(4): e62206.
[11] CHOI D, LEE Y, CHO H T, KENDE H. Regulation of expansin gene expression affects growth and development in transgenic rice plants., 2003, 5(6): 1386-1398.
[12] YAN A,WU M J,YAN L M,HU R,ALI I,GAN Y B. AtEXP2 is involved in seed germination and abiotic stress response in., 2014, 9(1): e85208.
[13] CHO H T, COSGROVE D J. Regulation of root hair initiation and expansin gene expression in Arabidopsis., 2002, 14(12): 3237-3253.
[14] LEE D K, AHN J H, SONG S K, CHOI Y D. Expression of an expansin gene is correlated with root elongation in soybean., 2003, 131(3): 985-997.
[15] SHIN J H, JEONG D H, PARK M C, AN G. Characterization and transcriptional expression of the α-expansin gene family in rice., 2005, 20(2): 210-218.
[16] CHO H T, COSGROVE D J. Altered expression of expansin modulates leaf growth and pedicel abscission in., 2000, 97(17): 9783-9788.
[17] KELLER E, COSGROVE D J. Expansins in growing tomato leaves., 1995, 8(6): 795-802.
[18] MADOKA G M, MELLEROWICZ E J, ABE H, SCHRADER J, WINZÉLL A, STERKY F, BLOMQVIST K, MCQUEEN-MASON S, TEERI T T, SUNDBERG B. Expansins abundant in secondary xylem belong to subgroup a of the α-expansin gene family., 2004, 135(3): 1552-1564.
[19] 王桂凤, 施季森. 杉木木材形成过程中差异表达基因的鉴定与功能分析[D]. 南京: 南京林业大学, 2008.
WANG G F, SHI J S, Identification and functional analysis of differentially expressed genes in differentiating xylem of Chinese fir () by suppression subtractive hybridization[D]. Nanjing: Nanjing Forestry University. (in Chinese)
[20] COSGROVE D J. New genes and new biological roles for expansins., 2000, 3(1): 73-78.
[21] COSGROVE D J, BEDINGER P, DURACHKO D M. Group I allergens of grass pollen as cell wall-loosening agents., 1997, 94(12): 6559-6564.
[22] ROSE J K, BENNETT A B. Cooperative disassembly of the cellulose-xyoglucan network of plant cell wall: Parallels between cell expansion and fruit ripening., 1999, 4(5): 176-183.
[23] FIGUEROA C R, PIMENTEL P, DOTTO M C, CIVELLO P M, MARTÍNEZ G A, HERRERA R, MOYA-LEÓN M A. Expression of five expansin genes during softening offruit: Effect of auxin treatment., 2009, 53(1): 51-57.
[24] JIANG F L, LOPEZ A, JEON S, DE FREITAS, SERGIO TONETTO, YU Q H, WU Z, JOHN M. LABAVITCH J M, TIAN S K, POWELL, MITCHAM E. Disassembly of the fruit cell wall by the ripening- associated polygalacturonase and expansin influences tomato cracking., 2019, 6(17).
[25] WU Y J, THORNE E T, SHARP R E, COSGROVE D J. Modification of expansin transcript levels in the maize primary root at low water potentials., 2001, 126(4): 1471-1479.
[26] YANG L, ZHENG B, MAO C, QI X, LIU F, WU P. Analysis of transcripts that are differentially expressed in three sectors of the rice root system under water deficit., 2004, 272(4): 433-442.
[27] VESELOV D S, SABIRZHANOVA I B, SABIRZHANOV B E, CHEMERIS A V. Changes in expansin gene expression, IAA content, and extension growth of leaf cells in maize plants subjected to salinity., 2008, 55(1): 101-106.
[28] PITANN B, ZÖRB C, MÜHLING K H. Comparative proteome analysis of maize (L.) expansins under salinity., 2009, 172(1): 75-77.
[29] XU J C, TIAN J, BELANGER F C, HUANG B. Identification and characterization of an expansin geneassociated with heat tolerance in C3 Agrostis grass species., 2007, 58(13): 3789-3796.
[30] DING X H, CAO Y L, HUANG L L, ZHAO J, XU C G, LI X H, WANG S P. Activation of the indole-3-acetic acid-amido synthetase GH3-8 suppresses expansin expression and promotes salicylate- and jasmonate-independentbasal immunity in rice., 2008, 20(1): 228-240.
[31] HAIGLER C H, BETANCUR L, STIFF M R, TUTTLE J R. Cotton fiber: a powerful single-cell model for cell wall and cellulose research., 2012, 3(104): 1-7.
[32] HARMER S, ORFORD S, TIMMIS J. Characterisation of six a-expansin genes in(upland cotton)., 2002, 268(1): 1-9.
[33] 琚铭, 王海棠, 王立科, 李飞飞, 吴慎杰, 朱华玉, 张天真, 郭旺珍. 棉纤维发育相关基因时空表达与纤维品质的关联分析. 作物学报, 2009, 35(7): 1217-1228.
JU M, WANG H T, WANG L K, LI F F, WU S J, ZHU H Y, ZHANG T Z, GUO W Z. Associated analysis between temporal and spatial expression of fiber development genes and fiber quality., 2009, 35(7): 1217-1228. (in Chinese)
[34] ORFORD S J, TIMMIS J N. Specific expression of an expansin gene during elongation of cotton fibers., 1998, 1398(3): 342-346.
[35] HARMER S E, ORFORD S J, TIMMIS J N. Characterization of six alpha-expansin genes in(upland cotton)., 2002, 268(1): 1-9.
[36] XU B, GOU J Y, LI F G, SHANGGUAN X X, ZHAO B, YANG C Q, WANG L J. A cotton BURP domain protein interacts with alpha- expansin and their co-expression promotes plant growth and fruit production., 2013, 6(3): 945-958.
[37] LI Y, TU L L, PETTOLINO F A, JI S M, HAO J, YUAN D J, DENG F L, WANG Q, LLEWWLLYN D J, ZHANG X L. GbEXPATR, a species-specific expansin, enhances cotton fiber elongation through cell wall restructuring., 2016, 14(3): 951-963.
[38] BAJWA K S, SHAHID A A, RAO A Q, BASHIR A, AFTAB A, HUSNAIN T. Stable transformation and expression offiber expansin gene to improve fiber length and micron aire value in cotton., 2015, 10(3389): 838.
[39] WANG K B, WANG Z W, LI F G, YE W W, WANG J Y, SONG G L, YUE Z, CONG L, SHANG H H, SHILIN ZHU, ZOU C S, LI Q, YUAN Y L, LU C R, WEI H L, GOU C Y, ZHENG Z Q, YIN Y, ZHANG X Y, LIU K, WANG B, SONG C, SHI N,KOHEL R J, PERCY R G, YU J Z, ZHU Y X, WANG J, YU S X. The draft genome of a diploid cotton., 2012, 44(10): 1098-1103.
[40] PATERSON A H, WENDEL J F, GUNDLACH H, GUO H, JENKINS J, JIN D, LLEWELLYN D, SHOWMAKER K C, SHU S, UDALL J, YOO M J, BYERS R, CHEN W, DORON-FAIGENBOIM A, DUKE M V, GONG L, GRIMWOOD J, GROVER C, GRUPP K, HU G, LEE T H, LI J, LIN L, LIU T, MARLER B S, PAGE J T, ROBERTS A W, ROMANEL E, SANDERS W S, SZADKOWSKI E, TAN X, TANG H, XU C, WANG J, WANG Z, ZHANG D, ZHANG L, ASHRAFI H, BEDON F, BOWERS J E, BRUBAKER C L, CHEE P W, DAS S, GINGLE A R, HAIGLER C H, HARKER D, HOFFMANN L V, HOVAV R, JONES D C, LEMKE C, MANSOOR S, RAHMAN M U, RAINVILLE L N, RAMBANI A, REDDY U K, RONG J K, SARANGA Y, SCHEFFLER B E, SCHEFFLER J A, STELLY D M, TRIPLETT B A, VAN DEYNZE A, VASLIN M F, WAGHMARE V N, WALFORD S A, WRIGHT R J, ZAKI E A, ZHANG T, DENNIS E S, MAYER K F, PETERSON D G, ROKHSAR D S, WANG X, SCHMUTZ J. Repeated polyploidization ofgenomes and the evolution of spinnable cotton fibers., 2012, 492(7429): 423-427.
[41] LI F G, FAN G Y, WANG K B, SUN F M, YUAN Y L, SONG G L, LI Q, MA Z Y, LU C R, ZOU C S, CHEN W B, LIANG X M, SHANG H H, LIU W Q, SHI C C, XIAO G H, GOU C Y, YE W W, XU X, ZHANG X Y, WEI H L, LI Z F, ZHANG G Y, WANG J Y, LIU K, KOHEL R J, PERCY R G, YU J Z, ZHU Y X, WANG J, YU S X. Genome sequence of the cultivated cotton., 2014,(6): 567-572.
[42] LI F G, FAN G Y, LU C R, XIAO G H, ZOU C S, KOHEL R J, MA Z, SHANG H H, MA X F, WU J Y, LIANG X M, HUANG G, PERCY R G, LIU K, YANG W H, CHEN W B, DU X M, SHI C C, YUAN Y Y, YE W W, LIU X, ZHANG X Y, LIU W Q, WEI H L, WEI S J, HUANG G D, ZHANG X L, ZHU S J, ZHANG H, SUN F M, WANG X F, LIANG J, WANG J H, HE Q, HUANG L H, WANG J, CUI J J, SONG G L, WANG K B, XU X, YU J Z, ZHU Y X, YU S X. Genome sequence of cultivated upland cotton (TM-1) provides insights into genome evolution., 2015, 33(5): 524-530.
[43] ZHANG T Z,HU Y , JIANG W K,FANG L, GUAN X Y,CHEN J D,ZHANG J B,SASKI C A,SCHEFFLER B E,STELLY D M,HULSE-KEMP A M,WAN Q,LIU B L,LIU C X,WANG S,PAN M Q,WANG Y K, WANG A W,YE W X, CHANG L J,ZHANG W P, SONG Q X, KIRKBRIDE R C, CHEN X Y,DENNIS E,LLEWELLYN D J,PETERSON D G, THAXTON P,JONES D C,WANG Q,XU X Y, ZHANG H,WU H T, ZHOU L,MEI G F,CHEN S Q,TIAN Y, XIANG D, LI X H,DING J,ZUO Q Y,TAO L N, LIU Y C,LI J, LIN Y,HUI Y Y, CAO Z S,CAI C P,ZHU X F, JIANG Z,ZHOU B L,GUO W Z,LI R Q,CHEN Z J. Sequencing of allotetraploid cotton (L. acc. TM-1) provides a resource for fiber improvement., 2015, 33(5): 531-537.
[44] WANG M J, TU L L, YUAN D J, ZHU D, SHEN C, LI J Y, LIU F Y, PEI L L, WANG P C, ZHAO G N, YE Z X, HUANG H, YAN F L, MA Y Z, ZHANG L, LIU M, YOU J Q, YANG Y C, LIU Z P, HUANG F, LI B Q, QIU P, ZHANG Q H, ZHU L F, JIN S X, YANG X Y, MIN L, LI G L, CHEN L L, ZHENG H K, LINDSEY K, LIN Z X, UDALL J A, ZHANG X L. Reference genome sequences of two cultivated allotetraploid cottons,and., 2019, 51(2): 224-229.
[45] HU Y, CHEN J D, FANG L, ZHANG Z Y, MA W, NIU Y C, JU L Z, DENG J Q, ZHAO T, LIAN J M, BARUCH K, FANG D, LIU X, RUAN Y L, RAHMAN M, HAN J L,WANG K, WANG Q, WU H T, MEI G F, ZANG Y H, HAN Z G, XU C Y, SHEN W J, YANG D F, SI Z F, DAI F, ZOU L F, HUANG F, BAI Y L, ZHANG Y G, BRODT A, BEN-HAMO H, ZHU X F, ZHOU B L, GUAN X Y, ZHU S J, CHEN X Y, ZHANG T Z.andgenomes provide insights into the origin and evolution of allotetraploid cotton., 2019, 51: 739-748.
[46] 雷忠萍, 贺道华, 海江波, 邢宏宜, 赵俊兴, 程雪妮. 雷蒙德氏棉扩展蛋白基因家族的鉴定和特征分析. 华北农学报, 2016, 31(6): 44-55.
LEI Z P, HE D H, HAI J B, XING H X, ZHAO J X, CHENG X N. Genome-wide identification and characterization of expansin gene family in., 2016, 31(6): 44-55. (in Chinese)
[47] HE D H, LEI Z P, TANG B S, XING H Y, ZHAO J X, JING Y L. Identification and analysis of the TIFY gene family in., 2015, 14(3): 10119-10138.
[48] 郝西, 理向阳, 腊贵晓, 代丹丹, 杨铁钢. 黄瓜扩展蛋白基因家族的鉴定与生物信息学分析. 分子植物育种, 2015, 13(10): 2280-2289.
HAO X, LI X Y, LA G X, DAI D D, YANG T G. Identification and bioinformatic analysis of the expansin gene family in cucumber., 2015, 13(10): 2280-2289. (in Chinese)
[49] 付海辉, 辛培尧, 许玉兰, 刘岩, 韦援教, 董娇, 曹有龙, 周军. 几种经济植物UFGT基因的生物信息学分析. 基因组学与应用生物学, 2010, 30(1): 92-102.
FU H H, XIN P R, XU Y L, LIU Y, WEI Y J, DONG J, CAO Y L, ZHOU J. Bioinformatics analysis of UFGT gene from several economic plants., 2010, 30(1): 92-102. (in Chinese)
[50] KRISHNAMURTHY P, HONG J K, KIM J A, JEONG M J, LEE Y H, LEE S I. Genome-wide analysis of the expansin gene superfamily reveals-specific evolutionary dynamics upon whole genome triplication., 2015, 290(2): 521-530.
[51] 李昊阳, 施杨, 丁亚娜, 徐吉臣. 杨树扩展蛋白基因家族的生物信息学分析. 北京林业大学学报, 2014, 36(2): 59-67.
LI H Y, SHI Y, DING Y N, XU J C. Bioinformatics analysis of expansin gene family in poplar genome., 2014, 36(2): 59-67. (in Chinese)
[52] ZHANG S Z, XU R R, GAO Z, CHEN C T, JIANG Z S, SHU H R. A genome-wide analysis of the expansin genes in., 2014, 289(2): 225-236.
[53] ZHU Y, WU N N, SONG W L, YIN G J, QIN Y J, YAN Y M, HU Y K. Soybean () expansin gene superfamily origins: segmental and tandem duplication events followed by divergent selection among subfamilies.ogy, 2014, 14(1): 1-19.
[54] CAREY R E, COSGROVE D J. Portrait of the expansin superfamily in: comparisons with angiosperm expansins., 2007, 99(6): 1131-1141.
[55] CHOTHIA C, GOUGH J, VOGEL C, TEICHMANN S A. Evolution of the protein repertoire., 2003, 300(5626): 1701-1703.
[56] RUAN Y L, LLEWELLYN D J, FURBANK R T. The control of single-celled cotton fiber elongation by developmental lyreversible gating of plasmodesmata and coordinated expression of sucrose and K+transporters and expansin., 2001, 13(1): 47-60.
[57] INDRAIS E, CHEEMA H M N, SAMAD A, BASHIR A. Temporal expression analysis and cloning of cotton () fiber genes., 2011, 13(1): 89-94.
Identification and Characterization of the Expansin Gene Family in upland cotton ()
ZHANG QiYan1, LEI ZhongPing2, SONG Yin1, HAI JiangBo1, HE DaoHua1
(1College of Agronomy, Northwest A&F University, Yangling 712100, Shaanxi;2College of Life Sciences, Northwest A&F University, Yangling 712100, Shaanxi)
【】 Expansins are a group of non-enzymatic proteins found in the plant cell wall, with important roles in plant growth, development, biotic and abiotic stress responses. To date, no systematic study on the molecular characterization, phylogeny and expression profiling of the upland cotton Expansin gene family has yet been conducted. In this study, a genome-wide identification, characterization and expression analysis of the Expansin gene family in upland cotton was performed. 【】 The members of the Expansin gene family in the upland cotton genome were identified by using the bioinformatics tools BLAST and HMMER, and were further analysed by using a combination of the bioinformatics softwares, such as ClustalW, MEGA, MCScanX, Prot Param, MEME, SignalP, Euk-mPLoc, Fancy Gene and DnaSP. The spatiotemporal expression patterns of the upland cotton Expansin gene family, and the differential expression of some Expansin homoeologs during the different stages of growth were determined by publicly available RNA-seq data. The expression patterns of some candidate Expansin genes were further validated by qRT-PCR. 【】 In the allotetraploid upland cotton, 72 expainsin-coding genes are identified, which is approximately twice as many as in the two diploid cotton species (and), and these expansin-coding genes are grouped into four subfamilies: 46 α-expansins (EXPAs), 8 β-expansins (EXPBs), 6 Expansin-like As (EXLAs), and 12 Expansin-like Bs (EXLBs). Except the two chromosomes GhA02 and GhD06, Expansin-coding genes are unevenly distributed across the other chromosomes ranging from 2 to 4, while the chromosomes GhA08 and GhD08 harbors 5 genes and 8 genes, respectively. Phylogenetic tree reveals that the members of the same subfamily are clustered together. In most cases, four Expansin members from the four (sub-)genomes of three cotton species (,and) tends to cluster together within a given clade, for example, EXPA subfamily memberswhich are located on collinear blocks are clustered into a clade. The computational prediction tool shows that all the Expansin proteins are predicted to be extracellular. The exon-intron structure analysis reveals that the upland cotton Expansin-coding genes typically consist of 3-5 exons interrupted by multiple introns, share an evolutionarily conserved exon-intron structure (consistent with the diversity of amino acid sequences), and have codon usage bias. RNA-seq data shows that different Expansin-coding genes are expressed in a stage- and tissue-specific manner during the developmental stages. For example, transcripts forandare highly abundant in the fire 10 days post anthesis (DPA) and 20 DPA when compared with other Expansin-coding genes.is highly expressed in few tissues, including cotyledons, new leaves, old leaves and bracts. Homoeologous genes exhibits different expression profiles, indicating the functional divergence and complementation. The qRT-PCR results are consistent with the RNA-seq data with the same trends for the expression of each Expansin-coding gene. For instance,andare highly expressed during the fiber elongation stage.andare highly expressed in the ovule at 3 DPA.【】The upland cotton genome contains 72 Expansin-coding genes which encode protein exhibiting the same structural diversity and evolutionary conservation as the coding DNA sequences of expansins, and which display diverse and dynamic expression patterns, implying functional conservation and divergence among the members of cotton Expansin genes.
; Expansin; gene family; bioinformatics; gene expression
2019-04-29;
2019-06-18
国家重点研发计划(2018YFD0100300)、现代农业产业技术体系建设专项资金(CARS-15-44)、全省种质资源保护利用(20171010000004)
张奇艳,E-mail:qiyanzhang318@163.com。雷忠萍,E-mail:zhpinglei@nwafu.edu.cn。张奇艳和雷忠萍为同等贡献作者。通信作者贺道华,Tel:029-87081285;E-mail:daohuahe@nwafu.edu.cn
10.3864/j.issn.0578-1752.2019.21.001
(责任编辑 李莉)
