线粒体代谢介导的表观遗传改变与衰老研究
2019-11-07潘云枫王演怡陈静雯范怡梅
潘云枫,王演怡,陈静雯,范怡梅
综述
线粒体代谢介导的表观遗传改变与衰老研究
潘云枫,王演怡,陈静雯,范怡梅
南京大学医学院,南京 210093
线粒体是细胞物质代谢与能量代谢的中心,在多种生理和病理过程中扮演着重要角色。表观遗传修饰是一种独立于DNA序列并在建立与维持特定基因表达谱中发挥主要作用的遗传调控模式。近年来的研究表明,线粒体能量代谢通过中间产物,介导线粒体–核信号的传递,调节染色质的表观修饰状态,进而影响基因表达。线粒体代谢紊乱可以诱导表观遗传重编程,进而启动衰老表型及退行性疾病的发生。本文综述了线粒体代谢与染色质表观遗传修饰关系的研究进展,探讨了线粒体应激在染色质重组中发挥的作用,展望了其在认知功能障碍等衰老相关性疾病研究中的前景。
线粒体代谢;DNA甲基化;组蛋白修饰;衰老;UPRmt
表观遗传修饰是一种独立于DNA序列并在建立与维持特定基因表达谱中发挥主要作用的遗传调控模式。DNA甲基化和组蛋白乙酰化及甲基化是表观遗传修饰的主要形式。线粒体呼吸链氧化磷酸化过程的某些中间分子(如S-腺苷蛋氨酸(S-adenosylmethionine, SAM)、α-酮戊二酸(α-Ketoglutaric acid, α-KG)、乙酰辅酶A(acetyl coenzyme A, acetyl-CoA)及氧化型的烟酰胺腺嘌呤二核苷酸(Nicotinamide adenine dinucleotide, NAD+)等),是参与表观遗传修饰酶活性调控的辅酶因子。而伴随衰老的线粒体能量代谢障碍将导致细胞氧化磷酸化呼吸链相关分子水平异常,进而影响基因组表观修饰状态,并改变相关基因表达[1,2]。这一发现为线粒体在相关病理过程中的调节作用提供了新见解。
本文在对线粒体代谢和表观遗传修饰简要介绍的基础上,对近年来关于线粒体代谢介导染色质表观遗传修饰的相关研究进行综述,对线粒体应激在染色质重组中发挥的作用展开讨论,并对该领域的研究方向进行展望。
1 线粒体代谢异常在衰老过程中发挥重要作用
线粒体是一种起源于10亿年前被真核细胞吞噬的内共生细菌α-proteobacteria的细胞器,通过氧化磷酸化(oxidative phosphorylation, OXPHOS)为宿主提供了高效产生ATP的优势。作为细胞合成和分解代谢反应的中心,线粒体在基本代谢物和辅助因子(如脂肪酸、氨基酸、铁硫簇)的产生过程中也具有重要作用。此外,线粒体也是细胞中许多生理过程的中心信号平台,例如激活细胞凋亡、调节钙稳态、影响细胞增殖等。基于这些不同的功能,维持线粒体内稳态对细胞和组织的健康至关重要。线粒体功能受损可有多种表现,如过量的活性氧自由基(reacitve oxygen species, ROS)产生、线粒体增殖动力学异常、生物能量代谢失衡、AMP依赖的蛋白激酶(Adenosine 5'-monophosphate (AMP)-activated protein kinase, AMPK)活化、线粒体NAD+水平降低,最终会诱导细胞凋亡[3]。
三羧酸循环(tricarboxylic acid cycle, TCA),也被称为柠檬酸循环或Krebs循环,是线粒体的重要代谢反应。三羧酸循环中间代谢物不能被线粒体内膜渗透,因此存在3种线粒体载体,以帮助这些代谢物通过线粒体内膜外排和导入:(1) α-KG载体,将内部的α-KG和琥珀酸盐交换成外部苹果酸盐,介导α-KG从线粒体到细胞质的流出;(2)柠檬酸盐载体(citrate carrier, CIC),从线粒体运输柠檬酸和异柠檬酸;(3)二羧基酸盐载体(dicarboxylate carrier, DIC),通过线粒体内膜转运琥珀酸盐和草酰乙酸酯。这些载体在三羧酸循环中起着至关重要的作用,它们将三羧酸循环中积累的中间体转运到胞浆中,从而影响α-KG依赖的双加氧酶(2-oxoglutarate-dependent dioxygenases, 2-OGDO)的调控。研究表明,2-OGDO参与染色质表观遗传相关调控,因而三羧酸循环的中间产物是强有力的表观遗传调控因子,这为线粒体的调控功能提供了新的见解[4]。
线粒体也通过电子传递链(electron transport chain, ETC)中的蛋白质复合体为细胞发挥功能提供重要的辅助因子和代谢物质。ETC复合体I将还原型烟酰胺腺嘌呤二核苷酸(Nicotinamide adenine dinucleotide, NADH)氧化为NAD+。许多酶促反应需要NAD+,例如三羧酸循环、DNA修复和蛋白质乙酰化等。
此外,氧化磷酸化与线粒体电子传递链相偶联,涉及电子从NADH和还原型黄素腺嘌呤二核苷酸(Flavin adenine dinucleotide, FADH2)向分子氧的转移,是一种从ADP生成ATP的途径。氧化磷酸化的干扰对细胞产生众多影响,例如:(1) ATP生成减少;(2)活性氧生成增加;(3)促进线粒体NADH和FADH2的积累。线粒体NADH和ROS水平的升高抑制了三羧酸循环酶的活性,从而触发了不同三羧酸循环中间体的积累,这一过程可以改变2-OGDO的活性,干扰神经元的功能[5]。
衰老过程中线粒体形态也会发生明显变化,如小线粒体碎片数量增加、含有空泡和缩短的线粒体嵴的大线粒体体积增大等。这些变化提示衰老过程中线粒体动力学调控发生紊乱。一些研究表明,线粒体动力学在能量代谢中是一项重要的调节机制,因此任何干扰都可能引发代谢紊乱。例如,诱导线粒体分裂使得线粒体呼吸减少,寿命缩短。而在细胞衰老中观察到的线粒体的融合和伸长可以增加抗凋亡能力[6]。随着年龄的增长,包含功能失调线粒体的衰老细胞的出现加剧了衰老过程。此外,有研究发现,在阿尔兹海默症(Alzheimer’s disease, AD)发病过程中,线粒体中淀粉样β肽以及磷酸化和碎裂的tau蛋白积聚的过程触发了线粒体分裂并扰乱了三羧酸循环[5]。氧化应激、缺氧(通过干扰氧化磷酸化)和钙失衡可以通过损害线粒体的稳态进而影响细胞的能量代谢。充分的证据证明衰老以及衰老相关的疾病与增加的氧化压力以及线粒体能量代谢的变化相关[7,8]。线粒体功能障碍激起细胞衰老,最终导致年龄相关的疾病,例如神经退行性疾病(如帕金森病(Parkinson’s disease,PD)及AD等)、心血管疾病、糖尿病及癌症[9,10]。
2 表观遗传修饰异常影响功能基因表达引发衰老
表观遗传调控是独立于DNA序列的基因表达改变,主要包括DNA甲基化、组蛋白乙酰化及甲基化等形式,在建立与维持细胞特定基因表达谱的过程中发挥重要作用。衰老是生命发展的最终阶段,涉及复杂的遗传背景和环境因素的相互作用以及多通路的调控作用。作为功能基因表达调控的关键机制,近年来表观遗传修饰异常在衰老发生、发展中的作用越来越受到关注[11,12]。研究发现,表观遗传机制调节学习与记忆的形成并可能诱发衰老中的认知障碍[5,13,14]。表观修饰改变造成的基因表达异常,引起了与诸如AD、Huntington病(Huntington’s disease, HD)、Rett综合征类精神疾病等神经障碍以及衰老相关的认知缺失。下面将分别对组蛋白乙酰化、组蛋白甲基化、DNA甲基化等表观修饰在衰老相关疾病进程中的作用进行逐一阐述。
2.1 组蛋白乙酰化
组蛋白乙酰化是染色质构型重塑中最为经典的形式,由组蛋白乙酰化转移酶(histone acetyltransferases, HATs)和组蛋白去乙酰化转移酶(histone deace tylases, HDACs)进行动态调控。HDACs包含4类:第Ⅰ、Ⅱ、及Ⅳ类HDACs依赖于金属离子,通过水解作用从组蛋白上移去乙酰基团;第Ⅲ类HDACs包括Sirtuins家族蛋白(silent information regulators, Sirtuins/ SIRTs),依靠NAD+以断裂赖氨酸与乙酰基团之间的键,导致了独特的代谢物O-乙酰-ADP-核糖的产生。
组蛋白乙酰化及其相应修饰酶、去修饰酶的变化在衰老及相关疾病的发生发展过程中起到了重要作用。例如,海马区组蛋白乙酰化导致了一些记忆相关基因表达的增加,从而控制记忆形成。在多种认知障碍疾病的研究中发现,组蛋白低乙酰化还伴随着记忆缺失[15~17]。研究表明,组蛋白乙酰化的降低在AD的记忆损伤中具有关键作用,如AD患者颞叶的H3K18 及 H3K23等位点的组蛋白乙酰化水平显著低于年龄匹配的对照[18]。HDACs活性改变与包括神经退行性疾病、心血管疾病以及癌症等人类疾病的发生与演进相关。HDACs对于学习记忆形成具有相反的作用,过度表达HDAC2的神经元降低了树突棘密度以及突触可塑性,抑制了记忆形成。而HDAC2耗损增加了突触数目,有利于记忆构建[19]。相比于年龄匹配的对照,AD患者海马CA1区和内嗅皮层中的HDAC2会明显增加,许多HDAC2靶向基因乙酰化水平降低,使其转录受到抑制[16]。进一步实验揭示,HDAC抑制剂可以缓解转基因小鼠的认知缺陷[17]。作为第Ⅲ类 HDACs,Sirtuins是一个高度保守的基因家族,在氧化应激、细胞周期调控、胰岛素分泌以及线粒体代谢的调控中都发挥了重要的作用[20,21]。Sirtuins针对组蛋白H4K16、H3K9、及H3K56的去乙酰化,形成抑制基因表达的染色质构型,与寿命延伸延长相关。而衰老细胞中降低的Sirtuins活性,导致细胞基因组组蛋白乙酰化水平增高,产生转录“噪音”[22]。
2.2 组蛋白甲基化
组蛋白甲基化指组蛋白尾端赖氨酸的甲基化修饰,包括单、双或三甲基化。组蛋白甲基化的状态受到组蛋白甲基化转移酶(histone methyltransferases, HMTs),以及组蛋白赖氨酸去甲基化酶(lysine demethylases, KDMs)的调节。HMTs的酶活性发挥类似于DNA甲基化转移酶(DNA methyltransferases, DNMTs),两者均依赖于SAM作为甲基供体。KDM包括两类,即赖氨酸特异的去甲基化酶1 (lysine- specific demethylase 1, LSD1)及包含JmjC结构域的组蛋白去甲基化酶(jumonji-C (JmjC) domain-containing histone demethylase,JHDMs)。LSD1依赖于氨基氧化机制,完成单及双甲基化的赖氨酸的去甲基化。在哺乳动物细胞,三甲基化的赖氨酸的去甲基化由JHDMs催化。JHDMs具有独特的调控策略以达到精细的底物位点及状态的特异性,是主要的组蛋白去甲基化转移酶。JHDMs类组蛋白去甲基化转移酶是2-OGDO成员,其酶活性需要Fe2+、α-KG及O2参与,以逐步氧化甲基基团为甲醛。组蛋白甲基化标记对于基因表达的调节具有位点特异性,例如H3K9me2、H3K9me3及H3K27me3是抑制性标记,而H3K4me2、H3K4me3及H3K36me3为活化的标记。
在动物衰老过程中,伴随着染色质结构的多种变化。年轻动物的异染色质区保持紧密的结构以维持稳定,抑制转座子元件;中年动物出现基因表达谱的实质性改变;老年动物则展示了转录组的更大程度的破坏,其抑制转座子能力降低,并丢失异染色质区域,显示出衰老相关的异染色质聚焦(senescence-associated heterochromatin foci, SAHFs)[1,23]。以上这些改变均与组蛋白甲基化密切相关。组蛋白H3K9及H3K27甲基化是异染色质形成的关键位点,也是基因表达的抑制性修饰,在维持细胞核染色质3D结构中具有基本作用。研究表明,随着衰老或细胞的衰老,H3K9me3及H3K27me3水平会显著降低[24]。Frost等[25]在AD患者神经元细胞核样本中研究发现,异染色质标记H3K9me2位点大量耗损更甚,相比于年龄匹配的对照,定位于核仁外周染色质中心的AD脑免疫组化显示更加扩散。这些观察提示,AD中重要的异染色质结构丢失显著。进一步研究发现,超过1/3的海马神经元中异染色质沉默的基因在AD样本中活化,包括及等基因。其可能的机制是AD中异染色质的松弛引起基因表达的迁移,恢复至去分化的状态。AD中表达增加的基因为多潜能相关的基因,例如及等。Agger等[26]在小鼠胚胎成纤维细胞中揭示,KDM6的活化可以去除H3K27的三甲基化,诱导p16INK4a基因表达,阻止细胞周期的进展,引起细胞衰老。Han等[27]报道,在衰老的恒河短尾猿(rhesus macaque)大脑中,两种H3K4甲基化转移酶SETD7与DPY30的表达增加,位于压力应答基因处的H3K4me2增加,且水平与基因表达相关。
综上所述,组蛋白作为DNA和细胞其他组分之间的功能界面,其各种修饰可以形成不同的组合,构成了可以被转录复合物识别的组蛋白密码。组蛋白密码可能是研究染色质重塑及其衰老相关机制新的方向。
2.3 DNA甲基化
DNA甲基化主要发生于CpG二联核苷的胞嘧啶,由DNA甲基化转移酶(DNA methyltransferases, DNMT)介导将SAM的甲基转移至胞嘧啶上成为5-甲基胞嘧啶(5-methylcytosine, 5mC)。5mC 的去甲基化由10~11易位(ten-eleven-translocation, TET)羟化酶完成,TET介导5mC至5-羟甲基胞嘧啶(5- hydroxymethylcytosine, 5hmC)的转换[28]。TETs也属于2-OGDO家族,其酶活性同样需要辅酶因子Fe2+、α-KG及O2参与,生成的5-hmc 进一步由碱基切除修复酶去除[29]。
DNA甲基化的作用根据甲基化的部位、时间而有所差异。如DNA启动子CpG甲基化一般与基因表达抑制相关,而编码区上的DNA甲基化可以通过保证mRNA转录的保真度来保护其不受假转录本的影响[30]。
衰老是环境和基因相互作用的结果,相关DNA甲基化具有特异性的时空位点性,包括环境、年龄、甲基化位点和组织分区等。环境危险因素(如UV-B、空气污染、烟雾暴露等)可以导致特定基因区域的低甲基化,进而出现衰老表型[31~33]。年龄对于DNA甲基化水平的影响主要反映在人类胚胎中有着最高水平的5mC,后随着年龄增长而逐渐下降。经典研究认为衰老相关的DNA甲基化水平降低发生于全基因组,然而新的研究揭示,衰老相关的DNA甲基化改变不是发生于全基因组的,而是位于基因组特定位点及区域。例如Raddatz等[34]发现在人的上皮基因组的启动子与增强子区域存在年龄相关的DNA甲基化改变。Fernández等[35]也发现,富含H3K4me1的区域倾向于较早出现低甲基化位点。Lister等[36]揭示在小鼠及人类的大脑皮层存在年龄相关的特定CpG位点甲基化改变。AD患者中,易受影响的颞下回锥体神经元及星形胶质细胞表现出DNA甲基化标记水平的低下[37]。Cho等[38]的研究揭示,在衰老的小胶质细胞中,存在白细胞介素1β (interleukin 1 beta, IL1β) 启动子区域CpG位点低甲基化,促进IL1β等促炎细胞因子释放,介导衰老相关神经炎症的发展。少突胶质细胞也存在着细胞特异性的衰老相关DNA甲基化异常,并且大鼠脊髓少突胶质细胞祖细胞的基因组DNA甲基化水平和DNMTs活性在衰老过程中逐渐降低,导致再髓鞘化能力下降。以上研究提示AD等衰老相关疾病中DNA甲基化异常调控存在组织细胞特异性[39],深入探究不同种类细胞的甲基化特征,对于了解疾病相关基因表达改变的原因,以及病因具有重要作用。
3 线粒体代谢紊乱通过诱导表观遗传重编程启动衰老
线粒体与细胞核之间存在着复杂的调控关系。线粒体蛋白组由大约1500个蛋白质组成,由线粒体和核基因组编码。虽然电子传递链的13种基本蛋白等是由线粒体DNA编码的,但绝大多数线粒体蛋白都是在细胞核中编码的[40]。由于线粒体是能量代谢的重要参与者,其功能的破坏可以通过线粒体-核通路触发应激信号,从而诱导核基因表达的变化,这种信号也被成为线粒体负反馈(retrograde response, RTG)[6]。最新研究表明,三羧酸循环的某些中间产物,如α-KG、琥珀酸、延胡索酸等,可以调节DNA及组蛋白甲基化,以及组蛋白乙酰化等表观修饰的水平,影响基因表达进而产生表型改变,使生物体适应不断变化的环境条件,从而形成一种进化上保守的负反馈信号机制(图1)。
3.1 线粒体代谢与组蛋白乙酰化
线粒体是细胞能量代谢中心,线粒体代谢生成的乙酰辅酶A,可以通过柠檬酸转运系统,肉碱/乙酰肉碱转运系统等转运方式,进入细胞核,继而作为乙酰基供体,在HATs的作用下,参与组蛋白的乙酰化。此外,三羧酸循环的代谢产物柠檬酸盐可刺激胞质中乙酰辅酶A的产生,从而激活HATs,进而刺激组蛋白的乙酰化。丙酮酸脱氢酶复合物(pyruvate dehydrogenase complex, PDHC)也可以从线粒体基质转运到细胞核,继而利用丙酮酸生成乙酰辅酶A,促进乙酰化水平。在异常能量代谢条件下,乙酰辅酶A水平降低,同时NAD+水平升高。NAD+是涉及衰老和长寿的去乙酰化酶Sirtuins的底物,高水平的NAD+将导致Sirtuins的活性增加,组蛋白乙酰化水平降低,部分基因的表达受到抑制[3,6](图1)。Duboff等[41]研究发现,AD相关的异常多肽聚集于线粒体内,影响线粒体动力学,扰乱线粒体能量代谢。由于三羧酸循环被明显抑制,乙酰辅酶A以及NAD+等促进组蛋白乙酰化的中间分子水平明显下降,影响记忆相关基因区域的乙酰化水平,减少其表达,促进了记忆缺失等病理表型的出现。
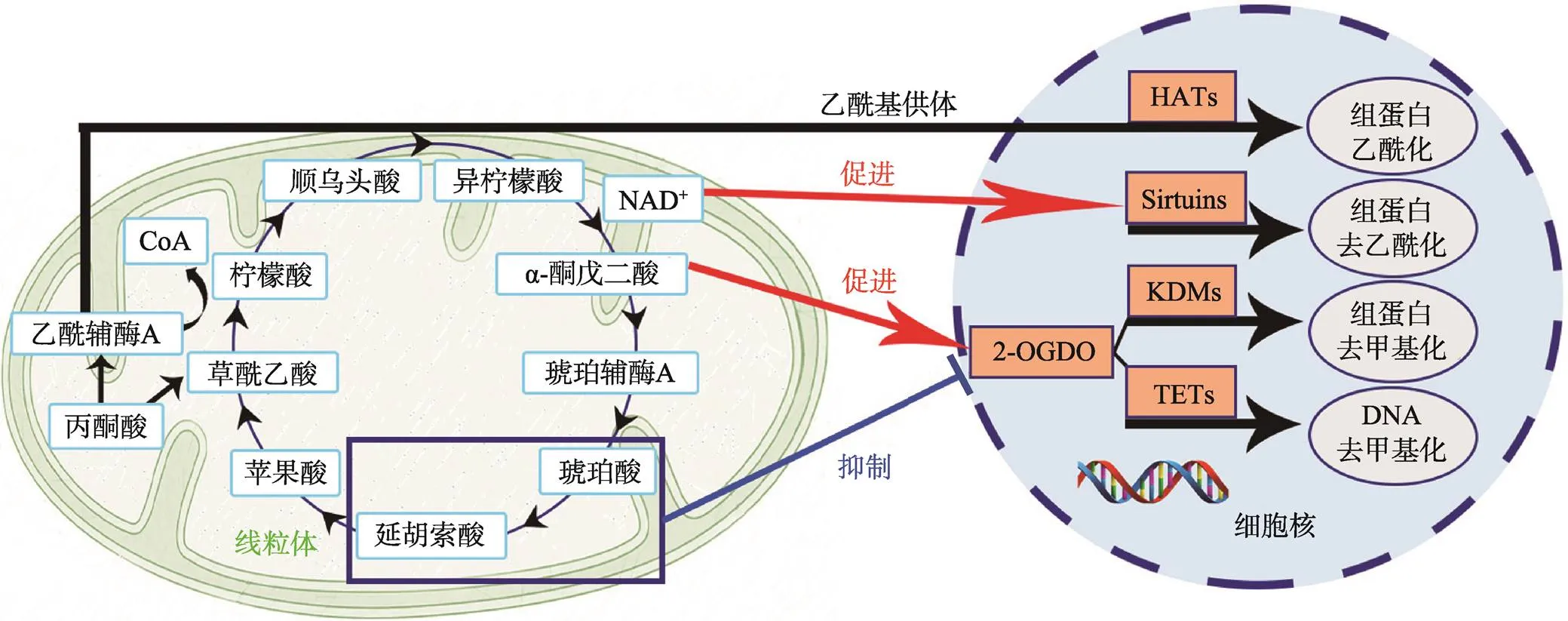
图1 三羧酸循环中间产物对表观修饰的影响
3.2 线粒体代谢与DNA及组蛋白的甲基化
线粒体代谢主要通过调节相关酶的活性和甲基供体水平,影响DNA与组蛋白的甲基化。最新的研究表明DNA去甲基化酶TET和组蛋白赖氨酸去甲基化酶KDM2-7均属于2-OGDO家族。2-OGDO的激活始于α-KG和Fe2+进入它们在酶催化领域的特定位置,随后O2与Fe2+结合,刺激α-KG氧化脱羧成琥珀酸酯和CO2,并引发氧化铁(Fe3+/4+)中间体的形成。随后,这些高价铁氧化羟化酶-底物复合体中的底物分子[42]。同为三羧酸循环中间体的琥珀酸和延胡索酸是2-OGDO的竞争性抑制剂[5](图1)。鉴于α-KG等是三羧酸循环的关键代谢物,这意味着能量代谢在2-OGDO的控制中起中心作用。
线粒体代谢紊乱将影响α-KG水平,继而调节TET和KDM的活性,影响DNA和组蛋白甲基化的水平。Bai等[43]研究发现,二甲双胍通过阻断异柠檬酸脱氢酶1-α-KG-TET1通路,降低Nrf2的羟甲基化水平,消除子宫内膜癌的耐药性。LetOuze等[44]证明琥珀酸脱氢酶(succinatedehydrogenase, SDH)突变引起的癌症与显著的DNA甲基化有关,表明TET被抑制。他们也证实SDH突变体癌组织中琥珀酸盐浓度增加了100倍。此外,研究表明异柠檬酸脱氢酶1和2 (isocitrate dehydrogenase1-2, IDH1-2)的突变可以将异柠檬酸转化为R(-)-2-羟戊二酸,而不是α-KG。Xu等揭示2-羟戊二酸是2-OGDO的竞争性抑制剂,通过抑制TET和KDM,增加人胶质瘤中的DNA及组蛋白甲基化[45]。目前,琥珀酸盐、延胡索酸盐和2-羟戊二酸盐被称为“致癌代谢产物”(oncometabolites),因为它们可以通过抑制2-OGDO的活性使得组织中DNA和组蛋白的甲基化水平增加,影响基因的表达,从而与癌症等退行性疾病的发生密切相关[46]。此外,有研究表明,乙酰辅酶柠檬酸裂解酶(ATP-citrate lyase, ACL)通过乙酰辅酶A依赖的方式促进miR-148a的表达而抑制DNMT1的表达[47],表明乙酰辅酶A通过调节DNMT1的表达而影响DNA甲基化的调控。
已知α-酮戊二酸脱氢酶(α-ketoglutaric acid dehydrogenase, α-KGDH)是α-KG的代谢酶。在衰老及衰老相关疾病患者的细胞中,α-KGDH活性的降低使得α-KG累积增多,从而激活RAS-RAF等信号通路,刺激JHDMs的活性,调节染色质的表观遗传状态,控制基因的表达,进而启动衰老[2,6,7],这意味着三羧酸功能的紊乱可以通过影响JHDMs活性,进而影响组蛋白甲基化以及基因表达。
除了影响相关酶的活性,SAM水平的变化也会影响DNA与组蛋白的甲基化(图2)。SAM是细胞内的通用甲基供体,在各种甲基转移酶的催化下,可以作为DNA、RNA以及组蛋白甲基化的底物。DNA和组蛋白的甲基化水平由SAM的丰度所调控。SAM由甲硫氨酸在甲硫氨酸腺苷转移酶(methionine adenosyltransferas, MAT)的催化下合成。在甲基转移酶作用后,SAM转变成S-腺苷-同型半胱氨酸(S-adenosylhomocysteine, SAH)。SAH可以抑制甲基转移酶活性。Chang等[48]研究发现,在大鼠肝脏中,腺苷和同型半胱氨酸水平的升高,会导致SAH水平升高,抑制SAM依赖的甲基转移酶活性,引起DNA整体甲基化水平降低。同时,口服降糖药二甲双胍通过促进SAM积累,降低SAH水平,从而提高DNA甲基化的水平[49]。由此,甲基转移酶的催化活性由SAM和SAH的比例动态调控,进而潜在地影响DNA和组蛋白的甲基化水平。
此外,在嘌呤的生物合成中,线粒体双功能酶利用亚甲基四氢叶酸合成甲酰四氢叶酸的同时,消耗一碳基团,如甲基(-CH3)、甲酰基(-CHO)、甲烯基(-CH2)、次甲基(-CH)及羟甲基(-CH2OH)等。而丝氨酸的生物合成也需要一碳基团。当线粒体双功能酶不表达时,线粒体生物合成丝氨酸的量增加,多余的丝氨酸会被转运到细胞质中。在细胞质中,丝氨酸的一碳基团通过亚甲基四氢叶酸和四氢叶酸甲酯转变为同型半胱氨酸,生成蛋氨酸。蛋氨酸和ATP反应,最终合成SAM,作为DNA和组蛋白甲基化的甲基供体。线粒体中的丝氨酸合成需要NAD+,在衰老表型的启动和进展过程中,氧化磷酸化被抑制,线粒体NADH/NAD+比值增加,继而导致蛋氨酸和SAM的合成减少,基因组特定区域甲基化水平降低[50]。
以上研究揭示线粒体代谢异常通过调节相关酶活性和甲基供体水平等途径,影响DNA与组蛋白的甲基化,调节基因表达,引发衰老表型。
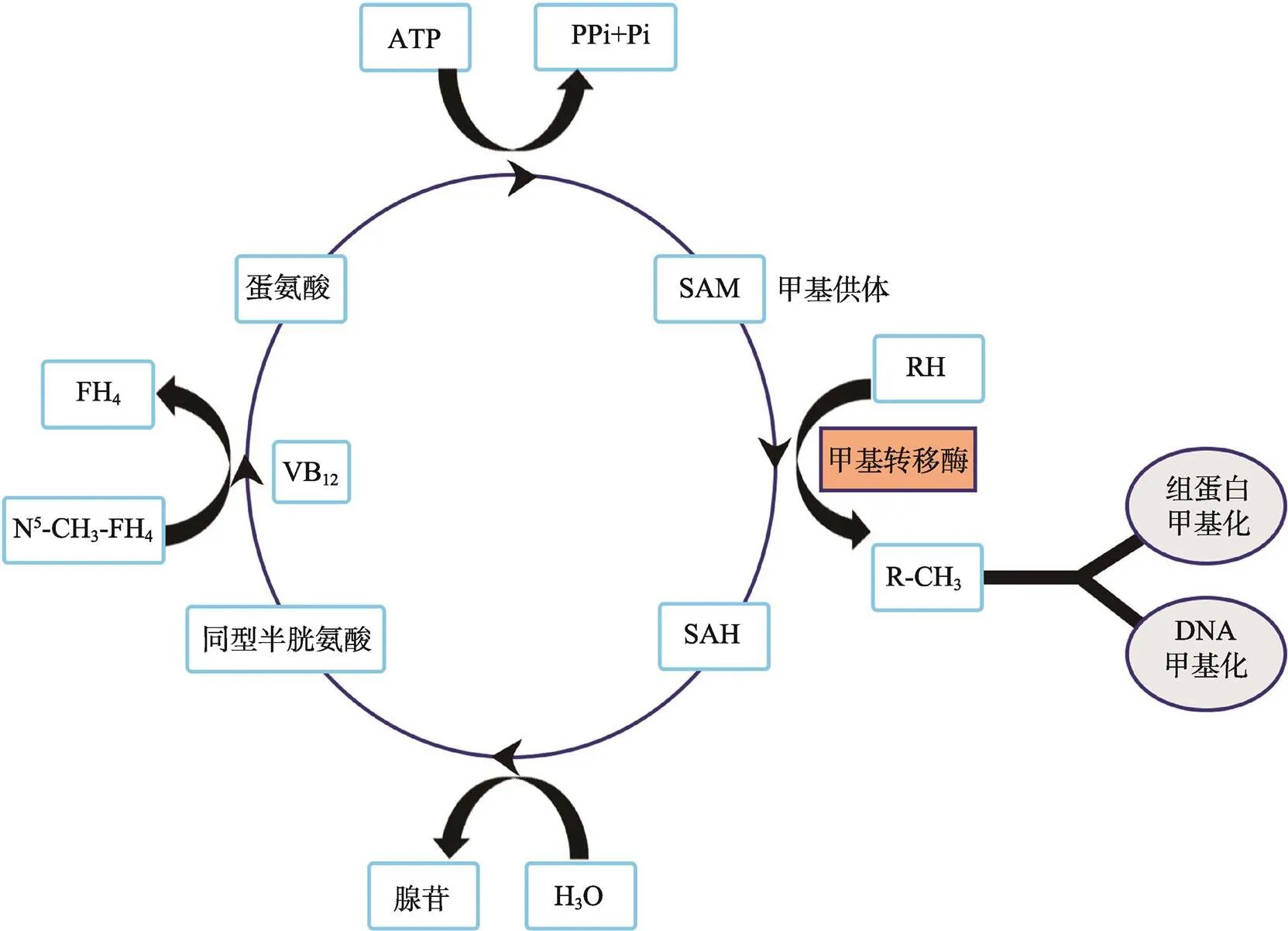
图2 蛋氨酸循环对表观修饰的调控
3.3 线粒体应激导致染色质重组和UPRmt
线粒体功能是细胞中众多通路的核心,因此线粒体功能障碍是从神经退行性疾病到癌症等多种疾病的基础。当线粒体感知压力后,压力可以经由多种不同信号从线粒体传递到胞质及细胞核,从而启动线粒体非折叠蛋白应答(mitochondrial unfolded protein response, UPRmt)。UPRmt是指各种原因引起错误折叠及未折叠蛋白质在线粒体基质中积蓄,通过线粒体-细胞核间的信号传导以及多种转录因子(如ATFS-1和ATF5等)的作用,致使应激蛋白转录增加、其他蛋白翻译减少、蛋白质降解增多的系列反应。线粒体应激蛋白包括分子伴侣(如热休克蛋白(heat shock protein, HSP)等)和蛋白酶LONP1 (lon peptidase1, mitochondrial, LONP1)等。这些蛋白质具有促进错误折叠及未折叠蛋白质的正确折叠等功能,有助于增强细胞对损伤的抵抗力,促进细胞存活[51]。UPRmt包括启动线粒体蛋白稳态的基因转录,以及受损线粒体的恢复。
最初的UPRmt研究是针对线虫()的,显示UPRmt由转录因子ATFS-1调节[52,53]。ATFS-1同时含有线粒体定位序列(mitochondrial targeting sequence, MTS)和细胞核定位序列(nuclear localization sequence, NLS),是介导线粒体-核信号传导的关键分子[54]。线粒体稳态时,ATFS-1被高效地转运至线粒体基质中,然后被LONP1酶解;在线粒体应激状态下,线粒体转运蛋白(如TIM-23[53]等)转运ATFS-1的能力受损,导致ATFS-1在胞质中聚集。此时,ATFS-1含有的NLS发挥作用,使其进入细胞核。
相似的转录应答在哺乳动物中也被揭示。线粒体应激时,氨基酸水平下降、ROS增加、核糖体滞留从而激活GCN2、PERK及HRI等激酶,使翻译起始因子elF2α磷酸化。磷酸化的elF2α减少蛋白质合成,但促进5¢-UTR带有多个上游开放阅读框(upstream open reading frame, uORF)的mRNA序列的转录。由于编码和的5'UTR中都存在多个uORFs,因此及的表达量增加。Fiorese等[55~57]最新研究发现,线粒体压力时,哺乳动物bZIP转录因子ATF5转向定位于细胞核并上调UPRmt相关伴侣蛋白及蛋白酶,以及诸如及等抗凋亡基因表达,激活UPRmt。除了转录适应外,线粒体在发育过程中的应激还会引起长期的染色质变化,这些表观遗传修饰有助于激活线虫和哺乳动物中维持“年轻”状态的UPRmt[58]。生物体通过上调分子伴侣、蛋白酶等UPRmt基因来应对线粒体应激,这种对代谢功能障碍的早期反应将维持一生[59]。具体来说,线粒体应激导致组蛋白H3K9的二甲基化,由甲基转移酶和核因子介导。这一变化导致染色质的整体沉默,但在与UPRmt激活相关的区域打开染色质,以利于DVE-1和ATFS-1结合启动子[60]。此外,需要两种组蛋白赖氨酸去甲基化酶即Jumonji家族蛋白jmjd-1.2和jmjd-3.1以激活UPRmt和线粒体应激,介导线虫的长寿。这些蛋白在哺乳动物中的同源物分别为PHF8和JMJD3,与UPRmt相关基因的甲基化状态、mRNA和蛋白表达呈正相关[61]。因此,发育期的线粒体应激通过特定的表观遗传修饰,允许基因选择性表达并延长寿命。但是,若损伤持续加重,上调的UPRmt相关基因表达不足以维持线粒体功能,持续改变的表观修饰导致细胞基因组不稳定,进而引发细胞凋亡及机体衰老[62]。
4 结语与展望
衰老表型的发展存在细胞氧化应激、线粒体应答及失调,同时伴随着基因组表观修饰改变的过程。线粒体能量代谢障碍导致细胞氧化磷酸化和呼吸链中间分子(如SAM、α-KG、乙酰CoA及NAD+等)水平异常,从而调控相关表观遗传修饰酶活性,影响基因组表观修饰状态,并改变相关基因表达。损伤初期,线粒体未折叠蛋白应答等保护机制启动,增加伴侣分子、蛋白酶等UPRmt相关基因表达,以维持线粒体稳态;随着损伤程度的加重,保护性机制过度激活导致基因组不稳定,提高衰老相关基因表达,最终引发细胞凋亡及机体衰老。因此,线粒体代谢障碍介导基因表达的表观调控异常是衰老表型启动和进展的重要原因(图3)。
组蛋白修饰与DNA甲基化是表观遗传的重要组成部分,与染色质结构密切相关,并影响基因的表达。表观遗传修饰在神经退行性疾病、心血管疾病以及癌症等衰老相关疾病中发挥重要作用[63,64]。由于表观遗传修饰及其所介导的染色质状态具有可塑性,不仅可以作为监测和评估衰老的标志,也可以成为抗衰老治疗的潜在靶点。一些特定的化学物质可以作为药物通过影响组蛋白修饰的特异性酶的状态,维持组蛋白状态的稳定性以延长寿命或改善与年龄相关的表型和疾病。但是这些治疗策略的针对性和有效性还有待提高。随着线粒体代谢在表观遗传修饰中的调控作用不断被提出并证实,人们对衰老过程中线粒体的调节作用越来越关注。线粒体失稳介导基因表达的表观调控异常是衰老表型启动并进展的重要原因,这为AD等衰老相关疾病的研究及治疗提出了新的思路。
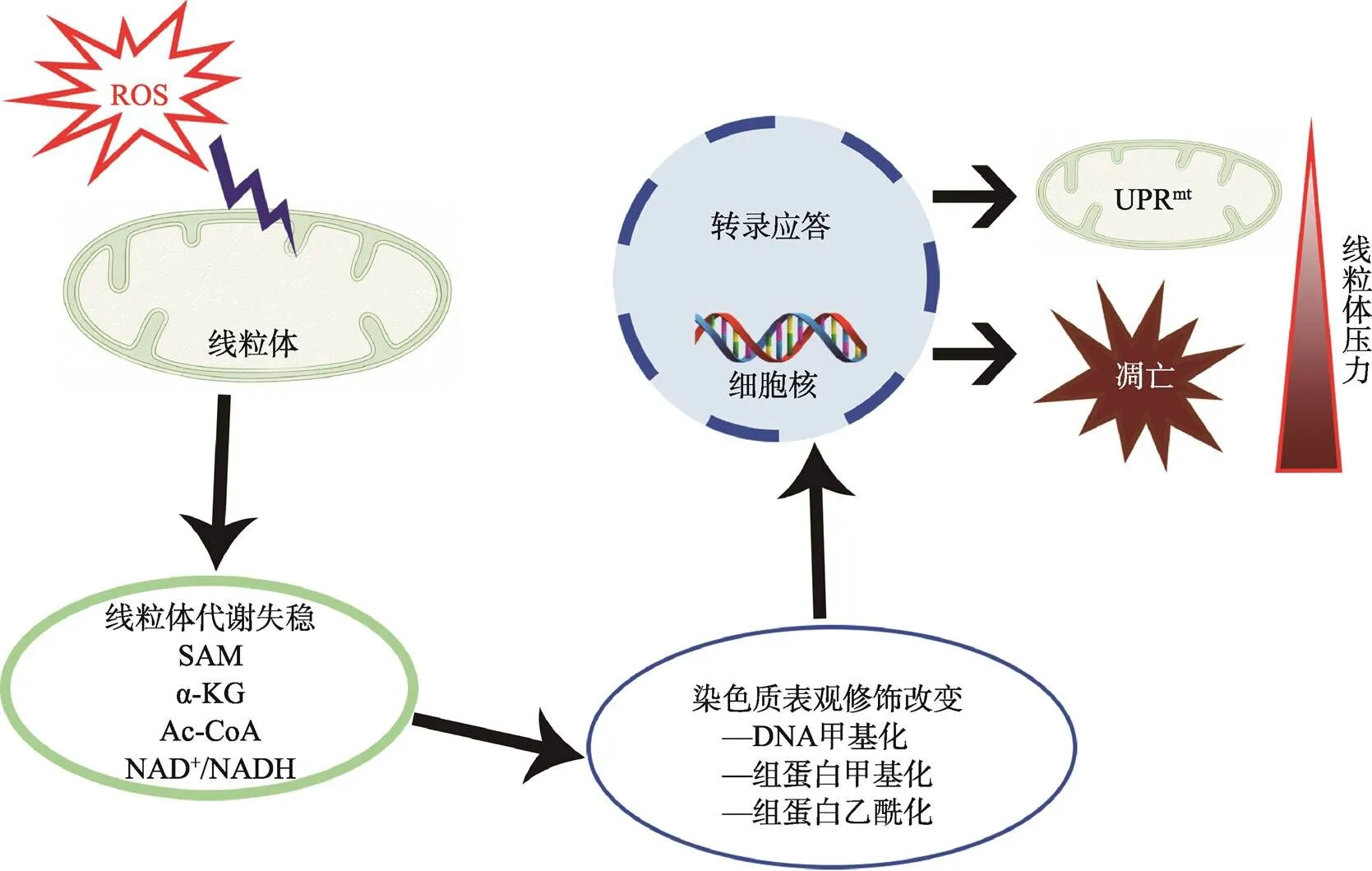
图3 线粒体代谢障碍介导基因表达的表观调控异常引发衰老的机制
目前开展的实验多数针对染色质整体的表观遗传修饰水平,对于线粒体代谢改变导致表观遗传修饰改变的具体区域尚未阐明,仍需要系统地针对特定基因开展研究。此外,在AD模型中神经元DNA甲基化水平的研究中,研究者发现不同个体神经元发生的表观遗传修饰是差异化的,同一个体的不同细胞之间表观修饰变化也是随机的,其具体机制和原因尚未阐明[5]。
线粒体自身也存在表观遗传修饰,但由于线粒体DNA并不被组蛋白包裹,因此线粒体DNA的表观遗传修饰主要是胞嘧啶甲基化/羟甲基化,且与细胞类型、分化状态、年龄以及疾病状态相关[65~74]。关于线粒体表观遗传修饰与衰老之间关系还知之甚少。研究发现在AD、帕金森氏病和肌萎缩侧索硬化患者的动物模型中,大脑部分区域细胞和循环血细胞中线粒体调节区(D环区域)的甲基化水平降低[75],提示线粒体DNA的异常表观修饰与神经退行性疾病相关。可以推测线粒体代谢除了在上述作为逆向线粒体应答的核基因表观遗传模式的调节中发挥作用,对于线粒体自身的表观遗传调节也将产生影响。因而从mtDNA表观遗传角度探讨线粒体功能失调有助于理解线粒体疾病[76]。
线粒体代谢异常除了影响表观修饰,还存在独立于表观修饰的其他途径影响到AD或者衰老的发生与发展。例如研究人员在2岁的小鼠模型中,观察到心肌细胞内线粒体膜上的钙离子通道活性降低,导致心肌收缩力下降[77]。这表明线粒体异常可以通过影响离子通道进而加剧衰老的发生与发展。
衰老及衰老相关疾病的发生发展过程中,往往伴随着细胞内线粒体代谢的异常,并通过表观遗传修饰影响基因表达。线粒体代谢调控表观遗传修饰的研究具有深远意义,可以使人们更好地理解能量代谢在表观遗传调控中的作用,并为衰老等的病因学研究提供新的思路。可以想象,通过结合代谢组学和表观基因组技术,将这些异常代谢产物,与目前研究已经明确的衰老相关分子通路结合分析,有望进一步揭示衰老的分子机制,为AD等衰老相关疾病的研究提供新的潜在干预靶标。
[1] Peleg S, Feller C, Ladurner AG, Imhof A. The metabolic impact on histone acetylation and transcription in ageing., 2016, 41(8): 700–711.
[2] Taylor EM, Jones AD, Henagan TM. A review of Mitochondrial-derived fatty acids in epigenetic regulation of obesity and type 2 diabetes., 2014, 2(3): 1–4.
[3] Ziegler DV, Wiley CD, Velarde MC. Mitochondrial effectors of cellular senescence: beyond the free radical theory of aging., 2014, 14(1): 1–7.
[4] Hausinger RP. FeII/alpha-ketoglutarate-dependent hydroxylases and related enzymes., 2004, 39(1): 21–68.
[5] Salminen A, Haapasalo A, Kauppinen A, Kaarniranta K, Soininen H, Hiltunen M. Impaired mitochondrial energy metabolism in Alzheimer's disease: impact on pathogenesis via disturbed epigenetic regulation of chromatin landscape., 2015, 131: 1–20.
[6] Salminen A, Kaarniranta K, Hiltunen M, Kauppinen A. Krebs cycle dysfunction shapes epigenetic landscape of chromatin: novel insights into mitochondrial regulation of aging process., 2014, 26(7): 1598–1603.
[7] Salminen A, Kauppinen A, Hiltunen M, Kaarniranta K. Krebs cycle intermediates regulate DNA and histone methylation: epigenetic impact on the aging process., 2014, 16: 45–65.
[8] Sun N, Youle RJ, Finkel T. The mitochondrial basis of aging., 2016, 61(5): 654–666.
[9] Breuer ME, Koopman WJ, Koene S, Nooteboom M, Rodenburg RJ, Willems PH, Smeitink JA. The role of mitochondrial OXPHOS dysfunction in the development of neurologic diseases., 2013, 51: 27–34.
[10] Chaturvedi RK, Flint Beal M. Mitochondrial diseases of the brain., 2013, 63: 1–29.
[11] Benayoun BA, Pollina EA, Brunet A. Epigenetic regulation of ageing: linking environmental inputs to genomic stability., 2015, 16(10): 593–610.
[12] Mccauley BS, Dang W. Histone methylation and aging: lessons learned from model systems., 2014, 1839(12): 1454–1462.
[13] Guan JS, Xie H, Ding X. The role of epigenetic regulation in learning and memory., 2014, 268: 30–36.
[14] Sen N. Epigenetic regulation of memory by acetylation and methylation of chromatin: implications in neurological disorders, aging, and addiction., 2014, 17(2): 97–110.
[15] Gräff J, Tsai LH. Histone acetylation: molecular mnemonics on the chromatin., 2013, 14(2): 97–111.
[16] Gräff J, Rei D, Guan JS, Wang WY, Seo J, Hennig KM, Nieland TJ, Fass DM, Kao PF, Kahn M, Su SC, Samiei A, Joseph N, Haggarty SJ, Delalle I, Tsai LH. An epigenetic blockade of cognitive functions in the neurodegenerating brain., 2012, 483(7388): 222–226.
[17] Peleg S, Sananbenesi F, Zovoilis A, Burkhardt S, Bahari- Javan S, Agis-Balboa RC, Cota P, Wittnam JL, Gogol- Doering A, Opitz L, Salinas-Riester G, Dettenhofer M, Kang H, Farinelli L, Chen W, Fischer A. Altered histone acetylation is associated with age-dependent memory impairment in mice., 2010, 328(5979): 753–756.
[18] Zhang K, Schrag M, Crofton A, Trivedi R, Vinters H, Kirsch W. Targeted proteomics for quantification of histone acetylation in Alzheimer's disease., 2012, 12(8): 1261–1268.
[19] Bie B, Wu J, Yang H, Xu JJ, Brown DL, Naguib M. Epigenetic suppression of neuroligin 1 underlies amyloid- induced memory deficiency., 2014, 17(2): 223–231.
[20] Kugel S, Mostoslavsky R. Chromatin and beyond: the multitasking roles for SIRT6., 2014, 39(2): 72–81.
[21] Poulose N, Raju R. Sirtuin regulation in aging and injury., 2015, 1852(11): 2442–2455.
[22] Wątroba M, Dudek I, Skoda M, Stangret A, Rzodkiewicz P, Szukiewicz D. Sirtuins, epigenetics and longevity., 2017, 40: 11–19.
[23] Li W, Prazak L, Chatterjee N, Grüninger S, Krug L, Theodorou D, Dubnau J. Activation of transposable elements during aging and neuronal decline in drosophila., 2013, 16(5): 529–531.
[24] O'sullivan RJ, Karlseder J. The great unravelling: chromatin as a modulator of the aging process., 2012, 37(11): 466–476.
[25] Frost B, Hemberg M, Lewis J, Feany MB. Tau promotes neurodegeneration through global chromatin relaxation., 2014, 17(3): 357–366.
[26] Agger K, Cloos PA, Rudkjaer L, Williams K, Andersen G, Christensen J, Helin K. The H3K27me3 demethylase JMJD3 contributes to the activation of the INK4A-ARF locus in response to oncogene- and stress-induced senescence., 2009, 23(10): 1171–1176.
[27] Han Y, Han D, Yan Z, Boyd-Kirkup JD, Green CD, Khaitovich P, Han JD. Stress-associated H3K4 methylation accumulates during postnatal development and aging of rhesus macaque brain., 2012, 11(6): 1055–1064.
[28] Tan L, Shi YG. Tet family proteins and 5-hydroxymethylcytosine in development and disease., 2012, 139(11): 1895–1902.
[29] Wang J, Zhang KX, Lu GZ, Zhao XH. Research progress on 5hmC and TET dioxygenases in neurodevelopment and neurological diseases., 2017, 39(12): 1138–1149.王建, 张凯翔, 芦国珍, 赵湘辉. 5-羟甲基胞嘧啶及其TET氧合酶在神经系统发育和相关疾病中的研究进展. 遗传, 2017, 39(12): 1138–1149.
[30] Neri F, Rapelli S, Krepelova A, Incarnato D, Parlato C, Basile G, Maldotti M, Anselmi F, Oliviero S. Intragenic DNA methylation prevents spurious transcription initiation., 2017, 543(7643): 72–77.
[31] Puttipanyalears C, Subbalekha K, Mutirangura A, Kitkumthorn N. Alu hypomethylation in smoke-exposed epithelia and oral squamous carcinoma., 2013, 14(9): 5495–5501.
[32] De Prins S, Koppen G, Jacobs G, Dons E, Van de Mieroop E, Nelen V, Fierens F, Int Panis L, De Boever P, Cox B, Nawrot TS, Schoeters G. Influence of ambient air pollution on global DNA methylation in healthy adults: a seasonal follow-up., 2013, 59: 418–424.
[33] Wu Z, Li X, Qin H, Zhu X, Xu J, Shi W. Ultraviolet B enhances DNA hypomethylation of CD4+ T cells in systemic lupus erythematosus via inhibiting DNMT1 catalytic activity., 2013, 71(3): 167–173.
[34] Raddatz G, Hagemann S, Aran D, Söhle J, Kulkarni PP, Kaderali L, Hellman A, Winnefeld M, Lyko F. Aging is associated with highly defined epigenetic changes in the human epidermis., 2013, 6(1): 36.
[35] Fernández AF, Bayon GF, Urdinguio RG, Toraño EG, García MG, Carella A, Petrus-Reurer S, Ferrero C, Martinez-Camblor P, Cubillo I, García-Castro J, Delgado- Calle J, Pérez-Campo FM, Riancho JA, Bueno C, Menéndez P, Mentink A, Mareschi K, Claire F, Fagnani C, Medda E, Toccaceli V, Brescianini S, Moran S, Esteller M, Stolzing A, de Boer J, NisticòL, Stazi MA, Fraga MF. H3K4me1 marks DNA regions hypomethylated during aging in human stem and differentiated cells., 2014, 25(1): 27–40.
[36] Lister R, Mukamel EA, Nery JR, Urich M, Puddifoot CA, Johnson ND, Lucero J, Huang Y, Dwork AJ, Schultz MD, Yu M, Tonti-Filippini J, Heyn H, Hu S, Wu JC, Rao A, Esteller M, He C, Haghighi FG, Sejnowski TJ, Behrens MM, Ecker JR. Global epigenomic reconfiguration during mammalian brain development., 2013, 341(6146): 1237905.
[37] Phipps AJ, Vickers JC, Taberlay PC, Woodhouse A. Neurofilament-labeled pyramidal neurons and astrocytes are deficient in DNA methylation marks in Alzheimer's disease., 2016, 45: 30–42.
[38] Cho SH, Chen JA, Sayed F, Ward ME, Gao F, Nguyen TA, Krabbe G, Sohn PD, Lo I, Minami S, Devidze N, Zhou Y, Coppola G, Gan L. SIRT1 deficiency in microglia contributes to cognitive decline in aging and neurodegeneration via epigenetic regulation of IL-1β., 2015, 35(2): 807–818.
[39] Deng Y, Yu G. The effect of DNA methylation on beta- amyloid accumulation in Alzheimer's disease., 36(4): 2014: 295–300.邓钰双, 余刚. 阿尔兹海默病中DNA 甲基化对β-淀粉样蛋白的影响. 遗传, 2014, 36(4): 295–300.
[40] Jovaisaite V, Mouchiroud L, Auwerx J. The mitochondrial unfolded protein response, a conserved stress response pathway with implications in health and disease., 2014, 217(Pt 1): 137–143.
[41] Duboff B, Feany M, Gotz J. Why size matters - balancing mitochondrial dynamics in Alzheimer's disease,36(6) (2013) 325–35.
[42] Salminen A, Kauppinen A, Kaarniranta K. 2-Oxoglutarate- dependent dioxygenases are sensors of energy metabolism, oxygen availability, and iron homeostasis: potential role in the regulation of aging process,,72(20) (2015): 3897–914.
[43] Bai M, Yang L, Liao H, Liang X, Xie B, Xiong J, Tao X, Chen X, Cheng Y, Chen X, Feng Y, Zhang Z, Zheng W. Metformin sensitizes endometrial cancer cells to chemotherapy through IDH1-induced Nrf2 expression via an epigenetic mechanism,, 2018, 37(42): 5666– 5681.
[44] Letouze E, Martinelli C, Loriot C, Burnichon N, Abermil N, Ottolenghi C, Janin M, Menara M, Nguyen AT, Benit P, Buffet A, Marcaillou C, Bertherat J, Amar L, Rustin P, De Reynies A, Gimenez-Roqueplo AP, Favier J. SDH mutations establish a hypermethylator phenotype in paraganglioma,, 2013, 23(6): 739–752.
[45] Xu W, Yang H, Liu Y, Yang Y, Wang P, Kim SH, Ito S, Yang C, Wang P, Xiao MT, Liu LX, Jiang WQ, Liu J, Zhang JY, Wang B, Frye S, Zhang Y, Xu YH, Lei QY, Guan KL, Zhao SM, Xiong Y. Oncometabolite 2-hydroxyglutarate is a competitive inhibitor of α-ketoglutarate-dependent dioxygenases., 2011, 19(1): 17–30.
[46] Yang M, Soga T, Pollard PJ. Oncometabolites: linking altered metabolism with cancer., 2013, 123(9): 3652–3658.
[47] Londoño Gentile T, Lu C, Lodato PM, Tse S, Olejniczak SH, Witze ES, Thompson CB, Wellen KE. DNMT1 Is regulated by ATP-Citrate lyase and maintains methylation patterns during adipocyte differentiation., 2013, 33(19): 3864–3878.
[48] Shyh-Chang N, Locasale JW, Lyssiotis CA, Zheng Y, Teo RY, Ratanasirintrawoot S, Zhang J, Onder T, Unternaehrer JJ, Zhu H, Asara JM, Daley GQ, Cantley LC. Influence of threonine metabolism on S-adenosylmethionine and histone methylation., 2012, 339(6116): 222–226.
[49] Cuyàs E, Fernández-Arroyo S, Verdura S, García RÁ, Stursa J, Werner L, Blanco-González E, Montes-Bayón M, Joven J, Viollet B, Neuzil J, Menendez JA. Metformin regulates global DNA methylation via mitochondrial one-carbon metabolism., 2018, 37(7): 963–970.
[50] Naviaux RK. Mitochondrial control of epigenetics., 2008, 7(8): 1191–1193.
[51] Fiorese CJ, Haynes CM. Integrating the UPRmtinto the mitochondrial maintenance network., 2017, 52(3): 304–313.
[52] Nargund AM, Fiorese CJ, Pellegrino MW, Deng P, Haynes CM. Mitochondrial and nuclear accumulation of the transcription factor ATFS-1 promotes OXPHOS recovery during the UPR(mt)., 2015, 58(1): 123–133.
[53] Nargund AM, Pellegrino MW, Fiorese CJ, Baker BM, Haynes CM. Mitochondrial import efficiency of ATFS-1 regulates mitochondrial UPR activation., 2012, 337(6094): 587–590.
[54] Lin YF, Schulz AM, Pellegrino MW, Lu Y, Shaham S, Haynes CM. Maintenance and propagation of a deleterious mitochondrial genome by the mitochondrial unfolded protein response., 2016, 533(7603): 416–419.
[55] Wu Y, Williams EG, Dubuis S, Mottis A, Jovaisaite V, Houten SM, Argmann CA, Faridi P, Wolski W, Kutalik Z, Zamboni N, Auwerx J, Aebersold R. Multilayered genetic and omics dissection of mitochondrial activity in a mouse reference population., 2014, 158(6): 1415–1430.
[56] Fiorese CJ, Schulz AM, Lin YF, Rosin N, Pellegrino MW, Haynes CM. The transcription factor ATF5 mediates a mammalian mitochondrial UPR., 2016, 26(15): 2037–2043.
[57] Arnould T, Michel S, Renard P. Mitochondria retrograde signaling and the UPR mt: where are we in mammals?, 2015, 16(8): 18224–18251.
[58] Merkwirth C, Jovaisaite V, Durieux J, Matilainen O, Jordan SD, Quiros PM, Steffen KK, Williams EG, Mouchiroud L, Tronnes SU, Murillo V, Wolff SC, Shaw RJ, Auwerx J, Dillin A. Two conserved histone demethylases regulate mitochondrial Stress-Induced longevity., 2016, 165(5): 1209–1223.
[59] Tian Y, Garcia G, Bian Q, Steffen KK, Joe L, Wolff S, Meyer BJ, Dillin A. Mitochondrial stress induces chromatin reorganization to promote longevity and UPR (mt)., 2016, 165(5): 1197–1208.
[60] Xiao M, Yang H, Xu W, Ma S, Lin H, Zhu H, Liu L, Liu Y, Yang C, Xu Y, Zhao S, Ye D, Xiong Y, Guan KL. Inhibition of α-KG-dependent histone and DNA demethylases by fumarate and succinate that are accumulated in mutations of FH and SDH tumor suppressors., 2012, 26(12): 1326–1338.
[61] Fiorese CJ, Haynes CM. Integrating the UPRmt into the mitochondrial maintenance network., 2017, 52(3): 304–313.
[62] Tatar M, Sedivy JM. Mitochondria: masters of epigenetics., 2016, 165(5): 1052–1054.
[63] Gray MW. Mitochondrial evolution., 2012, 4(9): a011403.
[64] Lane N, Martin W. The energetics of genome complexity., 2010, 467(7318): 929–934.
[65] Chestnut BA, Chang Q, Price A, Lesuisse C, Wong M, Martin LJ. Epigenetic regulation of motor neuron cell death through DNA methylation., 2011, 31(46): 16619–16636.
[66] Infantino V, Castegna A, Iacobazzi F, Spera I, Scala I, Andria G, Iacobazzi V. Impairment of methyl cycle affects mitochondrial methyl availability and glutathione level in Down's syndrome., 2010, 102(3): 378– 382.
[67] Shock LS, Thakkar PV, Peterson EJ, Moran RG, Taylor SM. DNA methyltransferase 1, cytosine methylation, and cytosine hydroxymethylation in mammalian mitochondria., 2011, 108(9): 3630–3635.
[68] Dzitoyeva S, Chen H, Manev H. Effect of aging on 5-hydroxymethylcytosine in brain mitochondria., 2012, 33(12): 2881–2891.
[69] Bellizzi D, D'aquila P, Scafone T, Giordano M, Riso V, Riccio A, Passarino G. The control region of mitochondrial DNA shows an unusual CpG and non-CpG methylation pattern., 2013, 20(6): 537–547.
[70] Byun HM, Panni T, Motta V, Hou L, Nordio F, Apostoli P, Bertazzi PA, Baccarelli AA. Effects of airborne pollutants on mitochondrial DNA methylation., 2013, 10: 18.
[71] Manev H, Dzitoyeva S. Progress in mitochondrial epigenetics., 2013, 4(4): 381–389.
[72] Pirola CJ, Gianotti TF, Burgueño AL, Rey-Funes M, Loidl CF, Mallardi P, Martino JS, Castaño GO, Sookoian S. Epigenetic modification of liver mitochondrial DNA is associated with histological severity of nonalcoholic fatty liver disease., 2012, 62(9): 1356–1363.
[73] Feng S, Xiong L, Ji Z, Cheng W, Yang H. Correlation between increased ND2 expression and demethylated displacement loop of mtDNA in colorectal cancer., 2012, 6(1): 125–130.
[74] Ghosh S, Sengupta S, Scaria V. Comparative analysis of human mitochondrial methylomes shows distinct patterns of epigenetic regulation in mitochondria., 2014, 18: 58–62.
[75] Blanch M, Mosquera JL, Ansoleaga B, Ferrer I, Barrachina M. Altered mitochondrial DNA methylation pattern in alzheimer Disease-Related pathology and in Parkinson Disease., 2016, 186(2): 385–397.
[76] D'aquila P, Bellizzi D, Passarino G. Mitochondria in health, aging and diseases: the epigenetic perspective., 2015, 16(5): 569–585.
[77] Jahangir A, Ozcan C, Holmuhamedov EL, Terzic A. Increased calcium vulnerability of senescent cardiac mitochondria: protective role for a mitochondrial potassium channel opener., 2001, 122(10): 1073– 1086.
Mitochondrial metabolism’s effect on epigenetic change and aging
Yunfeng Pan, Yanyi Wang, Jingwen Chen, Yimei Fan
Mitochondrion is the metabolic center and powerhouse of cells producing cellular energy which plays an important role in various physiological and pathophysiological processes. Recent research demonstrates that mitochondrial energy metabolism mediates the transmission of mitochondrial-nuclear signals through intermediate products which regulates epigenetic presentation of the chromatin and thereby affects gene expression. Epigenetic modification, a genetic regulatory model, is independent of DNA sequence and plays a major role in establishing and maintaining a specific gene’s expression profile. Disorders of mitochondrial metabolism can induce epigenetic reprogramming which in turn initiates aging phenotypes and degenerative diseases. This review introduces recent research progress on the relationship between mitochondrial metabolism and chromatin-related epigenetic modification, discusses the role of mitochondrial stress in chromatin recombination, and suggests future research directions and their application in the study of age-related diseases such as cognitive dysfunction.
mitochondrial metabolism; DNA methylation; histone modifications; aging; UPRmt
2019-04-08;
2019-06-08
国家自然科学基金面上项目(编号:81771504)资助[Supported by the National Natural Science Foundation of China (No. 81771504)]
潘云枫,本科生,专业方向:临床医学。E-mail: 161232029@smail.nju.edu.cn
王演怡,本科生,专业方向:口腔医学。E-mail: 161232043@smail.nju.edu.cn
潘云枫和王演怡并列第一作者。
范怡梅,博士,副教授,研究方向:疾病的表观遗传调控机制。E-mail: ymfan@nju.edu.cn
10.16288/j.yczz.19-065
2019/6/11 17:47:37
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190611.1747.003.html
(责任编委: 朱卫国)
