基于棉花转录组测序的SSR分子标记的开发
2019-09-17刘冬梅娄喜艳吴狄
刘冬梅 娄喜艳 吴狄

摘要:通过对棉花转录组进行测序获得的57 695条Unigenes,利用MISA软件进行搜索,得出简单重复序列(simple sequence repeat,简称SSR)的分布情况,共挖掘检索得到1 886个SSR位点,SSR的出现频率为3.27%。所有SSR位点分布于1 759条Unigenes上,发生频率为3.05%,平均每20.8 kb会出现1个SSR位点。有117条Unigenes含有1个以上SSR位点,含有复合型SSR的Unigenes数目为56条,其中三核苷酸基元类型分布最多,SSR数量为 1 582个,占挖掘出总SSR数量的83.88%,而单、二、四、五、六核苷酸重复类型所占的比例较小。最后通过Primer 3程序进行SSR引物设计,得到部分引物序列,可用于棉花遗传多样性分析、分子标记辅助育种以及种质资源的保存等。
关键词:棉花;转录组测序;SSR;分子标记;引物设计
中图分类号: S562.024 文献标志码: A 文章编号:1002-1302(2019)07-0032-03
微卫星DNA,即简单重复序列(simple sequence repeat,简称SSR),通常构成其重复基元的核苷酸个数为1~6个,特点是长度长达几十个核苷酸,为串联重复序列。单一序列的保守性较强,在基因组中,微卫星侧翼序列就有此特性。因此首先通过克隆相应的微卫星侧翼DNA片段,对其数目进行扩增,然后对扩增DNA进行全部测序,再对微卫星的侧翼序列引物进行人工合成,最终实现通过PCR扩增微卫星的目的。自分子领域发展以来,人们最常用的分子标记技术主要有随机扩增多态性DNA(RAPD)、扩增片段长度多态性(AFLP)、序列标签位点(STS)、限制性片段长度多态性(RFLP)、单核苷酸多态性(SNP)、SSR等[1-6]。与其他分子标记相比,SSR标记具有其独特的优点,(1)SSR标记的操作非常简便,无需消耗大量试验器材即可进行,整个过程消耗的时间较其他几种分子标记短,且对于多态性而言,SSR标记所发掘出的多态性较高;(2)SSR标记所开发的位点具有多等位基因的特性,且在分子水平上,其提供的信息量较高;(3)SSR标记识别出的基因位点呈共显性,在整个基因组有着均匀分布,且整个过程无放射、辐射危险。SSR标记适用于DNA指纹图谱的构建[7]、基因定位[8]和遗传多样性分析[9]等方面。高度变异是微卫星中重复基元的显著特性,微卫星数目可呈现出整倍性变异,并且重复基元序列中的序列有可能不完全相同,因而造成多个位点的多态性。
目前关于棉花全基因组SSR分子标记开发的研究报道较少,随着棉花品种数量的逐渐增多,田间表型农艺性状鉴定方法已难以满足快速并准确鉴定品种、质量、亲缘关系等的需求。本研究以陆地棉洞A转录组的测序结果为试验材料,对全转录组SSR位点进行挖掘,利用生物信息学技能和MISA软件,发掘基于陆地棉洞A基因型的SSR分子标记数据,在RNA水平上分析棉花SSR的特点及规律,以期为在分子水平上研究棉花种质资源、鉴定亲缘关系及进行分子辅助育种奠定基础。
1 材料与方法
1.1 棉花转录组数据来源
2014年8月,以陆地棉洞A不育和可育花药为材料,委托深圳华大基因科技有限公司进行转录组测序,获得的全基因序列共包含57 695条Unigenes,以此为材料进行SSR位点的挖掘。
1.2 棉花转录组SSR位点检索
利用SSR位点挖掘软件MISA对陆地棉洞A花药的 57 695 条Unigenes进行SSR位点挖掘,共挖掘出6种不同类别的SSR,分别是单、二、三、四、五、六核苷酸SSR。
1.3 SSR引物挖掘
利用MISA软件进行SSR位点挖掘之前首先将单、二、三、四、五、六核苷酸重复次数的操作参数分别设置为 ≥12 bp、≥6 bp、≥5 bp、≥5 bp、≥4 bp、≥4 bp;其次对长度条件参数进行设置,当2个微卫星可以组合为1个复合微卫星时,这2个微卫星之间的距离必须小于100 bp。
1.4 引物设计
通过Primer 3程序进行SSR引物设计。
2 结果与分析
2.1 棉花转录组序列中SSR位点的数量
通过MISA软件对棉花转录组获得的Unigenes进行挖掘,从表1可以看出,共挖掘出1 886个SSR位点,SSR出现频率(搜索出的SSR数量与搜索序列总数的比值)为3.27%。所有SSR位点分布于1 759条Unigenes上,发生频率(搜索出的含有SSR序列的数量与搜索序列总数的比值)为3.05%,平均每20.8 kb会出现1个SSR位点(搜索出的序列总长度与SSR总数量的比值)。其中含有1个以上SSR位点的Unigenes数量较少,有117条;含复合型SSR的序列数更少,为56条。
2.2 棉花转录组SSR基元频率特征及引物设计
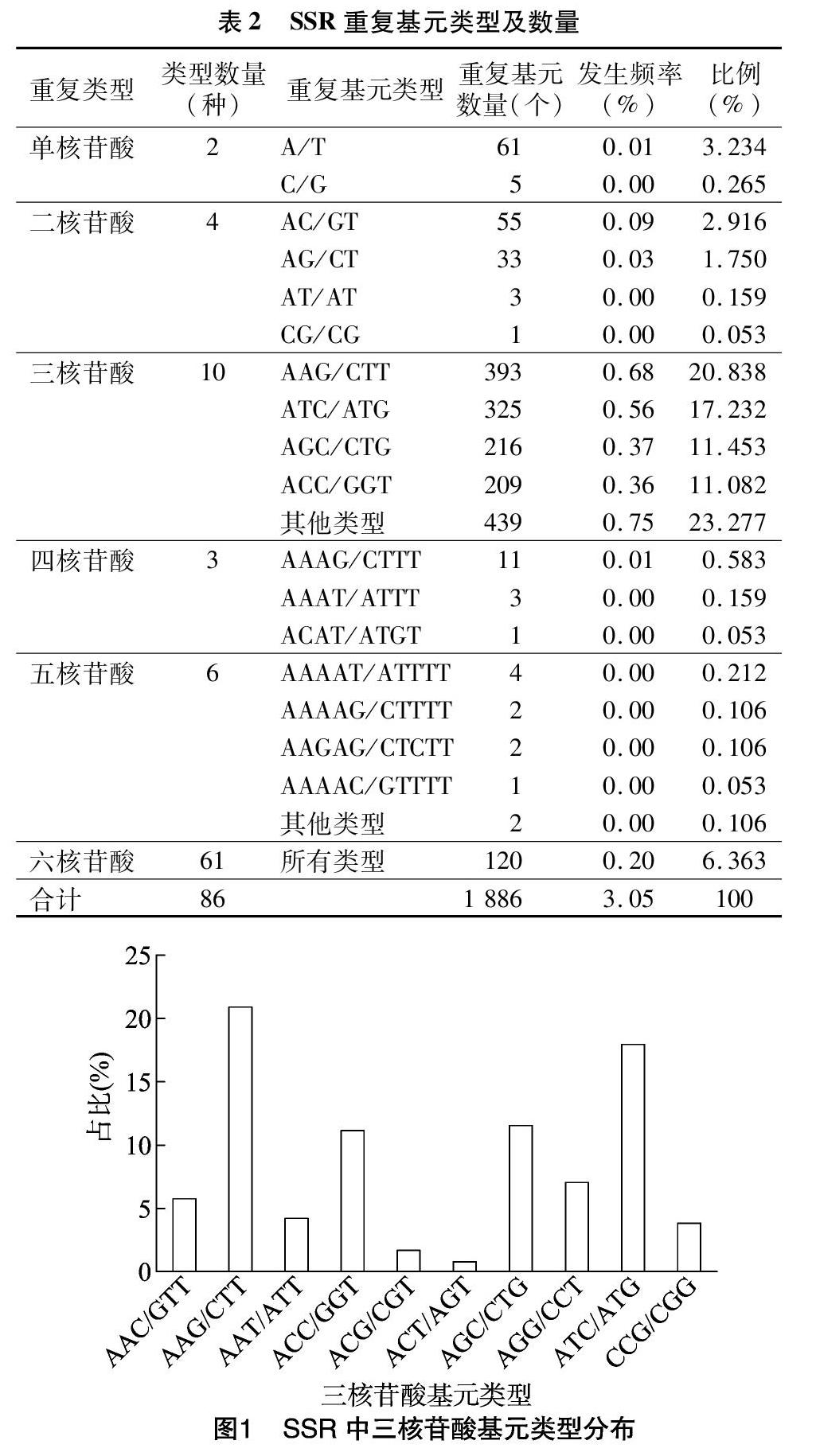
由表2可知,棉花转录组的SSR种类较为丰富,本研究共挖掘到86种重复的SSR类型,且各重复类型之间的数量差别较大,分布极其不平衡。棉花转录组中三核苷酸SSR重复基元类型的分布最多,SSR重复基元数量为1 582个,占挖掘出总SSR数量的83.88%;SSR位点数范围为1~393个,最多的为AAG/CTT,其次是ATC/ATG,数量为 325个,AGC/CTG的数量为216个,ACC/GGT数量为209个。单、二、四、五、六核苷酸重复类型所占的比例较小,SSR重复基元数量分别为66、92、15、11、120个,总比例之和为16.12%。在棉花转录组Unigenes中,重復基元类型最多的是六核苷酸,为61种;其次是三核苷酸重复类型,有10种。从图1可以看出,在三核苷酸重复基元类型中,AAG/CTT的SSR位点数量最多,为393个,占总SSR位点数量的20.84%,发生频率为0.68%;其次是ATC/ATG,占总SSR位点数量的 17.23%;再次是AGC/CTG,占11.45%;ACC/GGT较少,占1108%;其他三核苷酸重复基元类型占总SSR位点的2328%。单、二、四、五、六核苷酸重复类型数量分别为2、4、3、6种,共占重复类型总数的17.44%。通过对棉花转录组57 695条序列进行SSR位点搜索,共搜索得到 1 886 个SSR位点,利用Primer 3引物设计程序所得出的部分SSR位点特异引物见表3。
2.3 棉花转录组序列中SSR基元重复次数分布
SSR位点的多态性是由基元重复次数的变化所决定的,通过棉花SSR位点重复次数分布统计结果(图2)可以得出,棉花转录组基元随着重复次数的增加,其数量和比例逐渐减少。棉花转录组SSR中基元重复次数主要集中在4~7次,占总数的94.91%,其中重复8~10次的SSR位点数量为28个,占总数的1.48%;重复11~14次数的SSR位点为61个(3.23%),重复15~17次的SSR位点有7个(0.37%),其中重复16次的SSR位点有0个。重复次数多于20次为较高重复次数,棉花转录组中没有重复次数高于17次的SSR位点,即无高重复次数的SSR位点,低重复次数(重复1~10次)与一般重复次数(重复11~20次)的SSR位点较多。
3 讨论
本研究利用棉花花药的转录组序列数据,从57 695条序列中成功挖掘检索出1 886个SSR位点, 棉花转录组序列中SSR位点的总发生频率为3.05%,通过对比杜仲(2.90%)[10]、南方红豆杉(2.07%)[11]可以发现,棉花转录组序列中SSR位点的发生频率略高,但略低于红松(4.24%)[12],且明显低于樱桃(15.62%)[13]。棉花转录组SSR标记重复次数最多的是三核苷酸重复基元类型,高级基元重复次数较为微小,且跨度大,可为基元的重复次数与SSR标记的多态性呈正相关提供依据[14]。三核苷酸中的AAG/CTT基元的丰度最高,这与海甘蓝三核苷酸为丰度最高的基元[15]一致,即棉花转录组SSR中三核苷酸SSR在理论上具备更高的多态性,可作为潜在的SSR重复基序进行有关的引物挖掘及开发。由此推测,通过利用棉花转录组得到的SSR引物,同样可以用来进行棉花的遗传多样性分析、分子标记辅助育种以及种质资源的保存等。
参考文献:
[1]涂明月,李 杰,何亚丽,等. 利用RAPD标记鉴定草地早熟禾种质资源的遗传多样性[J]. 草业学报,2017,26(7):71-81.
[2]石 悦,于肖夏,于 卓,等. 高丹草AFLP分子遗传连锁图谱的构建[J]. 西北农林科技大学学报(自然科学版),2017,45(10):7-14.
[3]曹雯梅,刘述忠,杨青华,等. STS标记鉴定河南历史主推小麦品种春化光周期基因及与品种冬春性的相关性[J]. 分子植物育种,2016,14(1):117-124.
[4]王 婵,赵 泓,张丽英,等. 大葱细胞质雄性不育系及保持系线粒体基因组RFLP分析[J]. 农业生物技术学报,2015,23(7):888-893.
[5]匡 猛,王延琴,周大云,等. 基于单拷贝SNP标记的棉花杂交种纯度高通量检测技术[J]. 棉花学报,2016,28(3):227-233.
[6]Fan C J,Liu Q Y,Zeng B S,et al. Development of simple sequence repeat(SSR)markers and genetic diversity analysis in blackwood(Acacia melanoxylon)clones in China[J]. Silvae Genetica,2016,65(1):49-54.
[7]宋海斌,崔喜波,马鸿艳,等. 基于SSR标记的甜瓜品种(系)DNA指纹图谱库的构建[J]. 中国农业科学,2012,45(13):2676-2689.
[8]叶卫军,杨 勇,周 斌,等. 分子标记在绿豆遗传连锁图谱构建和基因定位研究中的应用[J]. 植物遗传资源学报,2017,18(6):1193-1203.
[9]周丽霞,吴 翼,肖 勇. 基于SSR分子标记的油棕遗传多样性分析[J]. 南方农业学报,2017,48(2):216-221.
[10]黄海燕,杜红岩,乌云塔娜,等. 基于杜仲转录组序列的SSR分子标记的开发[J]. 林业科学,2013,49(5):176-181.
[11]李炎林,杨星星,张家银,等. 南方红豆杉转录组SSR挖掘及分子标记的研究[J]. 园艺学报,2014,41(4):735-745.
[12]张 振,张含国,莫 迟,等. 红松转录组SSR分析及EST-SSR标记开发[J]. 林业科学,2015,51(8):114-120.
[13]宗 宇,王 月,朱友银,等. 基于中国樱桃转录组的SSR分子标记开发与鉴定[J]. 园艺学报,2016,43(8):1566-1576.
[14]Gao L F,Tang J F,Li H W,et al. Analysis of microsatellites in major crops assessed by computational and experimental approaches[J]. Molecular Breeding,2003,12(3):245-261.
[15]戚維聪,程计华,黄邦全,等. 基于海甘蓝RNA-Seq序列开发EST-SSR分子标记[J]. 江苏农业学报,2014,30(5):997-1002. 司振书,殷国政,路建彪,等. H9N2亚型AIV双重RT-PCR检测方法的建立[J]. 江苏农业科学,2019,47(7):35-37.
