小麦新种质CH1357抗白粉病遗传分析及染色体定位
2019-09-10乔麟轶郭慧娟张树伟常利芳李东方阎晓涛任永康张晓军畅志坚
陈 芳 乔麟轶 李 锐 刘 成 李 欣 郭慧娟 张树伟 常利芳 李东方 阎晓涛 任永康 张晓军,* 畅志坚,*
小麦新种质CH1357抗白粉病遗传分析及染色体定位
陈 芳1乔麟轶2,3李 锐2刘 成4李 欣2,3郭慧娟2张树伟2常利芳2李东方5阎晓涛2任永康2张晓军2,3,*畅志坚2,3,*
1山西大学生命科学学院, 山西太原 030006;2山西省农业科学院作物科学研究所/ 作物遗传与分子改良山西省重点实验室, 山西太原 030031;3农业部黄土高原作物基因资源与种质创制重点实验室, 山西太原 030031;4山东省农业科学院作物研究所, 山东济南 250100;5新疆生产建设兵团第六师农业科学研究所, 新疆五家渠 831300
白粉病是影响小麦产量和品质的一种主要病害。小偃麦衍生品系CH1357对白粉病具有较好的成株抗性, 苗期对27个菌株表现为免疫或高抗, 是一个高抗白粉病的优异抗源。为了明确其抗白粉病基因在染色体上的位置, 对台长29/CH1357和绵阳11/CH1357的F1、BC1及F2:3家系进行了遗传分析, 并利用分离群体分组分析法(bulked segregant analysis, BSA)将其初步定位。CH1357的白粉病抗性受1对显性核基因控制, 位于染色体5DS, 暂命名为。其侧翼连锁标记为和, 在2个作图群体台长29/CH1357和绵阳11/CH1357中的遗传距离分别为2.0 cM/11.3 cM和1.5 cM/8.9 cM。与5DS染色体上已报道的其他抗白粉病基因抗谱不同, 可能是一个新的抗源。
白粉病抗性; SSR标记; 连锁图谱; 基因定位;
小麦(L., 2= 6= 42, AABBDD)是世界上主要粮食作物之一, 其产量和品质与粮食安全密切相关。由禾本科布氏白粉菌(DC.) f. sp.()侵染引起的白粉病是限制小麦产量和品质的一种主要病害, 在全世界小麦主产区普遍发生, 尤其是在过量使用氮肥和大量种植半矮秆小麦品种的国家盛行[1-2]。据全国农业技术推广服务中心测报, 2018年小麦白粉病发病面积预计高达603万公顷。其中, 对长江中下游、江淮、滇南、陇南、晋南、豫北、鲁中南高产灌区等麦区生产危害尤为严重(http://www.natesc. org.cn/sites/cb/)。挖掘抗病基因进行小麦遗传改良, 培育适应当地的抗病品种是控制病害最有效的措施, 对小麦种业发展和保障我国粮食安全具有重要意义[3]。
迄今为止, 国内外已鉴定出100多个抗小麦白粉病基因或QTL, 其中已被正式命名的基因位点有64个[4]。其中, 一些已被广泛应用于小麦的育种和生产, 如、、和等。然而, 由于小麦白粉菌具有菌株多样、病菌毒性基因变异快等特点, 大多数广泛种植的品种在推广应用3年至5年便开始逐渐丧失抗性[5-6]。例如, 20世纪70年代引进的小麦–黑麦T1BL·1RS易位系上抗白粉病基因的过度利用, 使得其毒性菌株的频率直线上升, 导致1990年小麦白粉病大流行, 对小麦产量造成严重损失[7-8]。因此, 持续挖掘和定位新的抗白粉病基因, 开发可用于分子育种的功能标记, 进行抗病基因的合理布局, 将对小麦品种的抗性改良起到积极的推进作用。
CH1357是八倍体小偃麦小偃7430与普通小麦冀麦26杂交, 再与中8701杂交选育而来的小偃麦渗入系, 其在成株期对白粉病表现出优良抗性, 苗期对多个菌株表现为免疫或高抗, 具有较高的育种应用价值。本研究对CH1357的白粉病抗性进行遗传分析, 并确定CH1357中白粉病抗性基因所在染色体的位置, 旨在为克隆并有效利用该抗源种质提供理论依据。
1 材料与方法
1.1 供试材料与白粉病菌株
白粉病苗期抗病性鉴定材料包括抗病亲本CH1357 (系谱为中8701//冀麦26/小偃7430), 感病亲本为台长29 (TC29)和绵阳11 (MY11), 台长29/CH1357的F1、BC1和483个F2:3家系, 绵阳11/CH1357的F1、BC1和411个F2:3家系。其中2个杂交组合的F2群体作为作图群体, 其F2:3家系用于统计抗病性的分离比, 以确定控制白粉病抗病基因的个数, F1和BC1群体的鉴定结果用于确定抗病基因的显隐性。
、和的载体品种UIka/8*Cc、KM2939和鸟麦用于不同白粉菌株的抗性反应比较。其中, UIka/8*Cc为前苏联品种; KM2939为小麦-冰草远缘杂交获得的新种质, 其系谱为4201/CMH83. 605//FC[16]; 鸟麦为地方品种[17]。
上述遗传群体F1、BC1和F2:3家系的抗病性鉴定在苗期进行, 所用白粉菌株为E09, 由中国农业科学院植物保护研究所周益林研究员惠赠, 本实验室通过活体幼苗保存。32个在我国中、北部小麦种植区采集并分离的白粉病菌株用于研究抗病亲本CH1357对不同菌株的抗性反应(表1), 由中国科学院遗传与发育生物学研究所农业资源研究中心安调过博士提供。
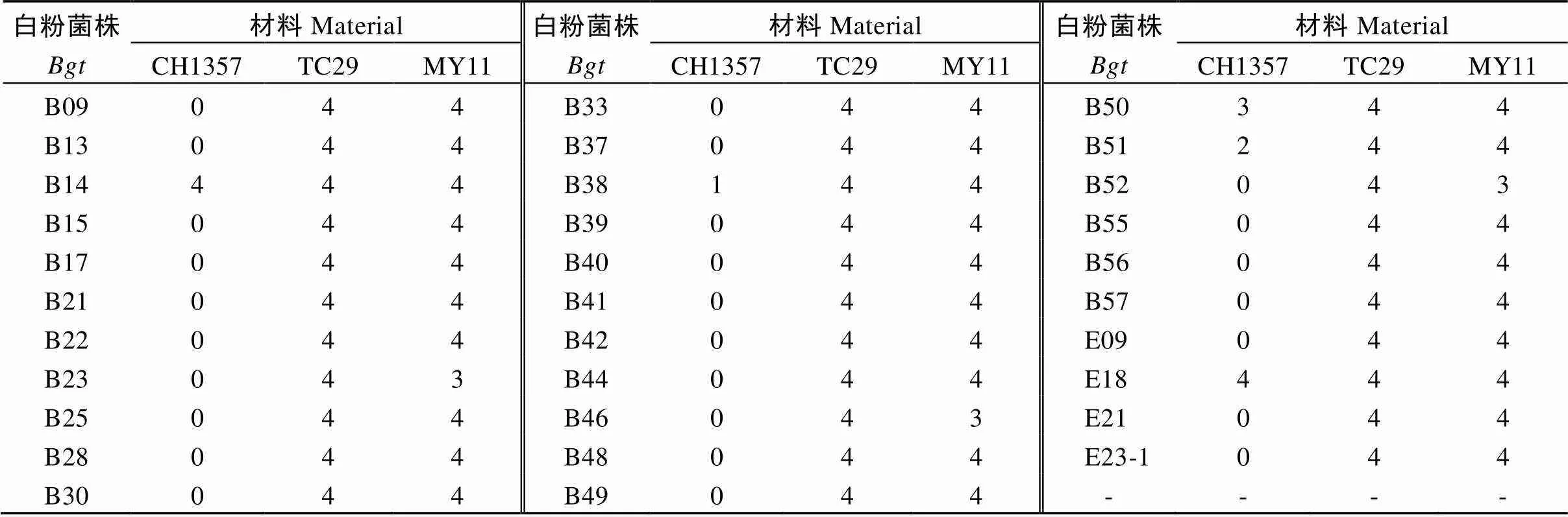
表1 CH1357、台长29 (TC29)和绵阳11 (MY11)苗期对32个白粉菌株的抗性反应
1.2 苗期白粉病抗病性评价
在人工气候室中对供试材料进行苗期白粉病抗病性鉴定。利用75%酒精溶液对种子消毒后种植于72穴育苗盘内, 每穴播5粒种子, 每个育苗盘种植8个感病品种, 台长29作为感病对照, 以确保病害的充分发生。人工气候室的光照强度为125 μmol m–2s–1, 光周期为12 h, 温度控制在16~22℃, 湿度控制在65%~70%。种植约10 d后用喷雾器喷湿幼苗, 将预先繁殖好的新鲜白粉菌采用人工扫抹法对二叶期幼苗接菌, 每材料处理5株苗, 重复鉴定3次。接菌约12 d, 待对照充分发病后, 采用0~4级分级标准[9]调查每株材料的侵染类型(infection type, IT)。IT = 0为免疫; IT = 0;为近免疫; IT = 1为高抗; IT = 2为中抗; IT = 3为中感; IT = 4为高感。调查数据用SPSS软件进行c2检验, 以推断抗病基因的数目及显隐性。
1.3 基因组DNA提取及PCR扩增
三叶期剪取台长29/CH1357和绵阳11/CH1357的F2单株叶片及亲本叶片, 利用CTAB法[10-11]提取基因组DNA。采用分离群体分组分析法(bulked segregant analysis, BSA), 根据抗病调查结果随机选取10株纯合抗病植株(表型为0级和0; 且其F2:3家系植株全部为抗病)和10株纯合感病植株(表型为4级且其F2:3家系植株全部为感病), 分别将其DNA等量混合建立抗病池和感病池。
根据GrainGenes (https://wheat.pw.usda.gov/GG3/)公布的引物序列, 选用分布于小麦21条染色体的767对SSR引物在亲本台长29、绵阳11、CH1357以及抗病池和感病池间筛选多态性。根据筛选结果, 再补充差异标记所在染色体的其他分子标记。筛选的多态性引物进一步利用11个抗病单株和11个感病单株验证, 对多态性的引物利用作图群体进行PCR扩增。
PCR体系15 µL, 含2 µL DNA模板, 1.5 µL 10× buffer (10 mmol L–1Tris-HCl, pH 8.3, 50 mmol L–1KCl, 1.5 mmol L–1MgCl2), 0.3 µL dNTP (0.2 mmol L–1), 2 µL引物(10 µmol L–1)和0.15 µL酶。PCR扩增程序为95℃变性5 min, 95℃变性30 s, 55~62℃ (依引物退火温度而定)复性30 s, 72℃延伸40 s, 35个循环, 72℃延伸10 min, 12℃保存。利用8%非变性聚丙烯酰胺凝胶(Acr与Bis质量比为19∶1)电泳检测PCR扩增产物, 硝酸银染后显色照相并观察结果。
1.4 染色体定位及遗传连锁图谱的构建
根据分子标记检测结果及抗病鉴定结果, 利用Haldane函数计算目标基因与标记之间的遗传距离(cM), 借助MapMaker 3.0软件[12]构建遗传连锁图谱。
2 结果与分析
2.1 CH1357、台长29和绵阳11的白粉病抗性
利用32个白粉菌株分别接种亲本CH1357、台长29和绵阳11的两叶期幼苗, CH1357对E09等27个菌株均表现为免疫(IT = 0), 对B38表现为高抗(IT = 1), 对B51分别表现为中抗(IT = 2), 对B50表现为中感(IT = 3), 对B14和E18表现为高感(IT = 4); 台长29和绵阳11对32个白粉菌株均表现为感白粉病(IT = 3或4)(表1)。
2.2 CH1357的白粉病抗性遗传分析
利用E09对抗病亲本CH1357、感病亲本台长29和绵阳11以及这2个抗感杂交组合的F1、BC1和F2:3家系群体进行苗期抗病性鉴定, 以分析控制CH1357中白粉病抗性基因的数目及显隐性。2个杂交组合的正反交F1植株对E09菌株均表现为免疫或近免疫(IT = 0或0;), 与抗病亲本CH1357的抗病反应类型一致, 说明CH1357中的白粉病抗性受显性核基因控制。经χ2检验2个杂交组合后代BC1植株的抗、感分离比例符合孟德尔单基因遗传(表2)。利用E09菌株对台长29/CH1357的483个F2:3家系进行抗病性鉴定, 其中纯合抗病家系为120个, 抗、感分离的家系为231个, 纯合感病家系为132个, 抗性分离比例符合1∶2∶1 (χ2= 1.509,= 0.470)。利用同一白粉菌株接种CH1357的另一组合绵阳11/CH1357的411个F2:3家系, 其中纯合抗病家系为108个, 发生抗、感分离的家系为208个, 纯合感病家系为95个, 经χ2检验符合孟德尔分离比例1∶2∶1 (χ2= 0.883,= 0.643)(表3)。上述CH1357两个杂交组合后代的表型鉴定结果表明CH1357对白粉菌株E09的抗性由单显性核基因控制, 将其暂命名为。
2.3 PmCH1357的染色体定位
所选的767对SSR引物中, 406对(52.9%)在亲本CH1357和台长29间存在多态性, 进一步在抗、感池间筛选多态性, 并利用抗、感小群体进行验证, 发现标记和在亲本、抗感池以及抗感小群体中均能扩增出一致的多态性条带(图1)。通过小麦数据库GrainGenes (https://wheat.pw.usda. gov/GG3/)及Wheat-urgi (https://wheat-urgi.versailles. inra.fr/),和位于染色体5DS。随后, 又利用5DS上已发表的与抗白粉病基因连锁的标记及其他SSR引物(共91对)进一步筛选, 发现6对引物、、、、[13]和[14]在抗、感亲本和抗、感池间具有多态性。利用上述8对引物对台长29/CH1357的421个F2单株DNA进行扩增, 通过连锁分析, 初步将CH1357的抗白粉病基因定位于5D染色体短臂, 其两侧邻近标记和的遗传距离分别为2.0 cM和11.3 cM (图2-a)。又用上述8对引物在绵阳11、CH1357及其抗、感病池和抗、感病小群体间进行多态性鉴定, 发现这些引物与绵阳11/CH1357中的抗白粉病基因连锁。进一步对绵阳11/CH1357的341个F2单株进行DNA扩增, 通过遗传连锁分析, 将绵阳11/CH1357中的抗白粉病基因定位于染色体5DS, 两侧邻近标记为和, 遗传距离分别为1.5 cM和8.9 cM (图2-b)。
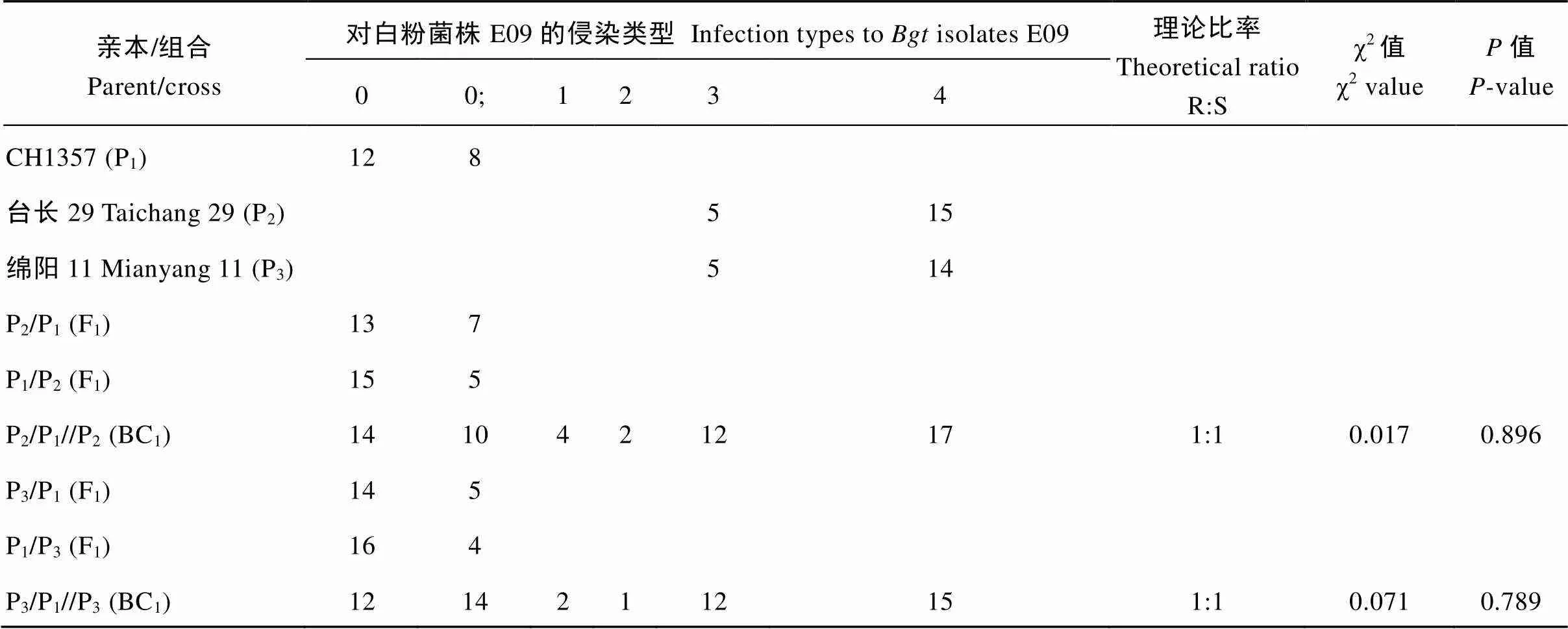
表2 2个作图群体不同世代对白粉菌株E09的苗期抗性和分离比率
IT 0~2为抗病型; IT 3~4为感病型。
IT 0−2 were scored as resistance and IT 3−4 as susceptibility.

表3 2个F2:3作图群体对白粉菌株E09的苗期抗性
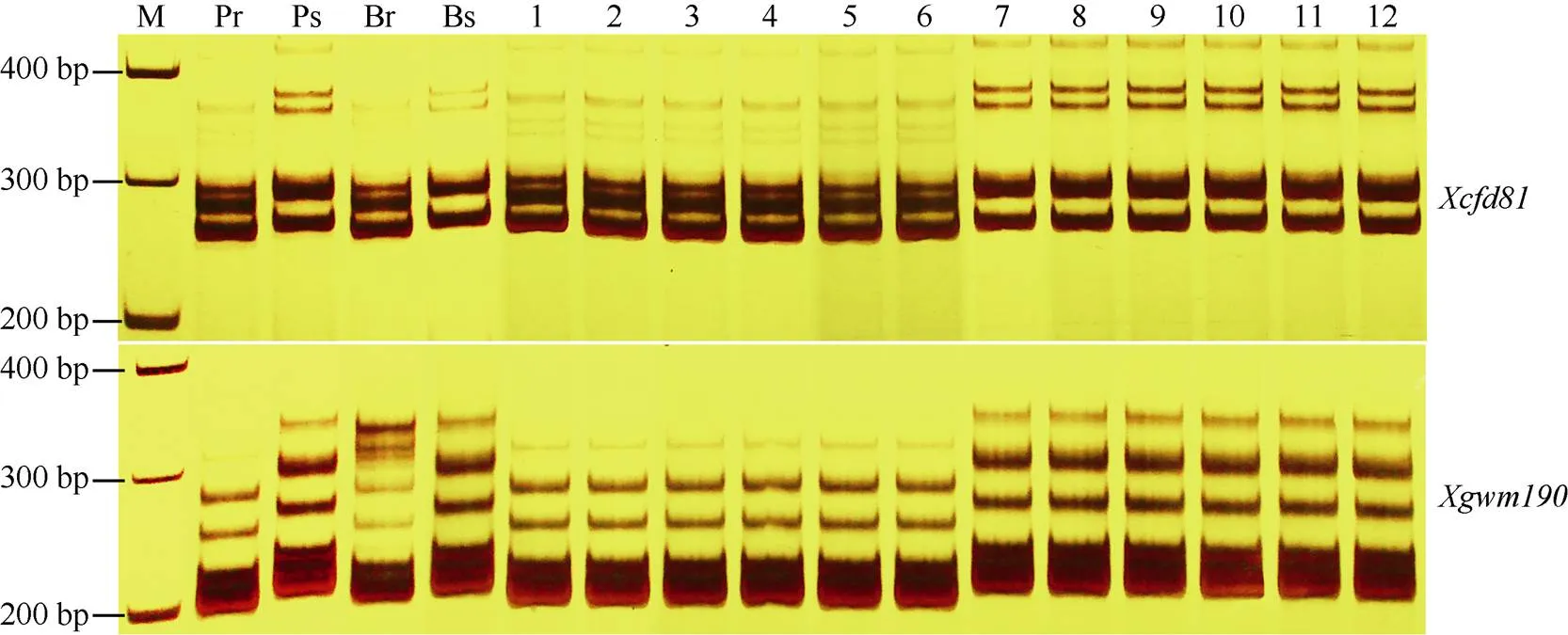
图1 SSR标记Xcfd81和Xgwm190在CH1357 F2群体中的扩增结果
M: marker; Pr: 抗病亲本CH1357; Ps: 感病亲本台长29; Br: 抗病池; Bs: 感病池; 1~6: 纯合抗病株; 7~12: 纯合感病株。
M: marker; Pr: resistant parent CH1357; Ps: susceptible parent Taichang 29; Br: resistant bulk; Bs: susceptible bulk; 1−6: homozygous resistant plants; 7−12: homozygous susceptible plants.
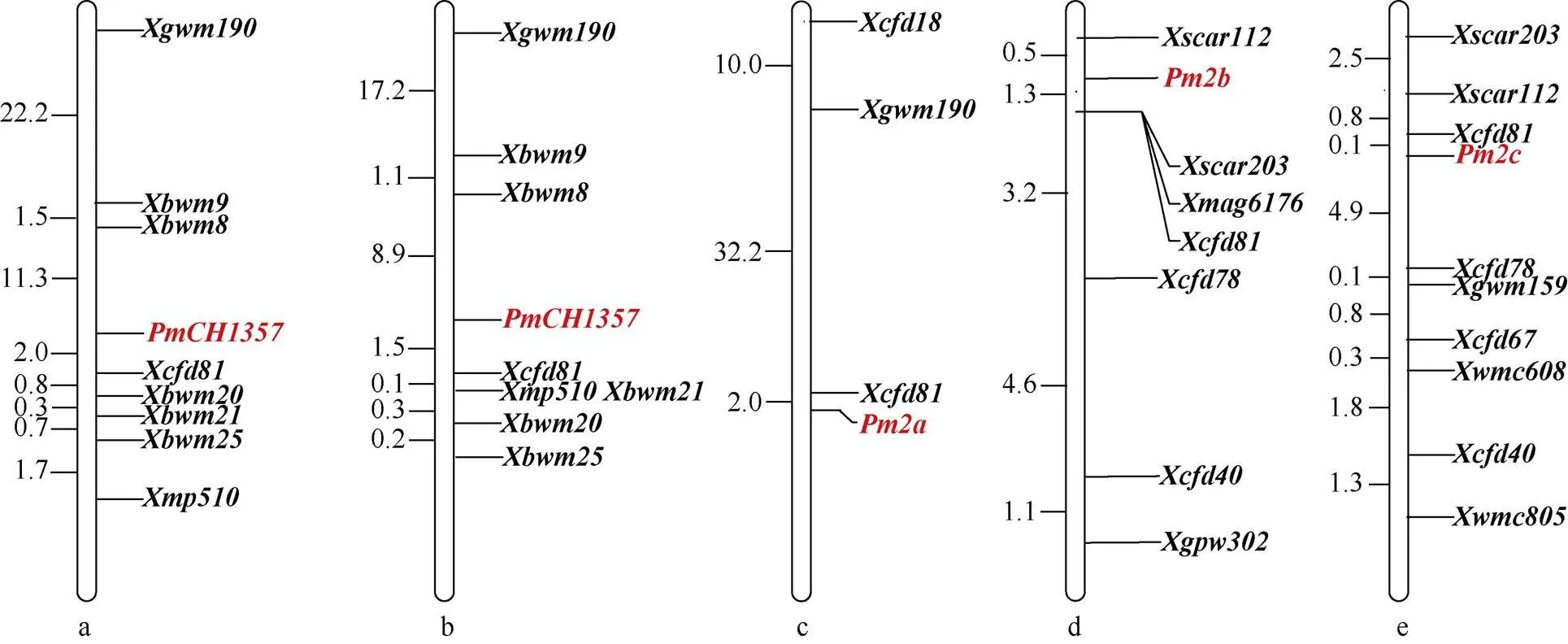
图2 PmCH1357在群体台长29/CH1357 (a)与绵阳11/CH1357 (b)中的遗传定位及其与Pm2位点其他基因的比较
2.4 PmCH1357与染色体5DS上其他抗白粉病基因的比较
利用与连锁的8对引物对、、的载体品种UIka、KM2939、鸟麦进行DNA扩增, 发现5对标记(、、、和)在CH1357和3个抗白粉病基因的载体品种中的扩增条带都一致(图3)。
利用12个白粉菌株对、、和的载体品种进行苗期白粉病抗性鉴定(表4)表明, CH1357对B38、B40、B51分别表现为高抗、免疫和中抗, 而UIka/8*Cc ()对该3个菌株均表现为中感; CH1357对E18、E21分别表现为高感和免疫, 而KM2939 ()对E18、E21的抗性反应分别为高抗和中感; CH1357对B14、E14均表现为高感, 而鸟麦()对B14、E14的抗性反应均为免疫。可见与、和对12个白粉菌株的抗性反应明显不同。
3 讨论
小麦白粉病菌株具有群体大、变异快等特点, 在抗源单一的情况下, 长期广泛种植含单一抗病基因的品种, 一旦出现新的毒性菌株, 会导致抗病基因的抗性丧失, 引起小麦白粉病大流行, 从而对小麦生产造成严重威胁[5-6]。目前虽然定位了大量的小麦主效抗白粉病基因, 但其中许多已对优势白粉病菌株丧失抗性, 可有效用于生产上的抗病基因甚少。因此, 不断挖掘新的抗白粉病基因, 保持抗病基因抗源的多元化至关重要。
利用我国中、北部搜集的32个白粉菌株对CH1357进行苗期白粉病抗性鉴定, CH1357对其中的29个菌株具有抗性(表1)。由此可见, CH1357对我国中、北部的白粉病具有广谱型抗性, 在生产上具有较大的应用潜力。通过对台长29/CH1357和绵阳11/CH1357的2个F2分离群体进行抗病性遗传分析和抗病基因的鉴定, 发现CH1357的白粉病抗性受一对显性核基因控制, 其抗病基因位于5DS染色体上。由于单基因遗传一般对病原菌表现为垂直抗性, 极容易被新的病原菌致病型所克服, 因此CH1357在生产上应用时应当注意, 应尽量避免单独使用, 与其他抗病基因结合使用, 可以有效增加品种的抗病性, 并延长其抗病持久性。
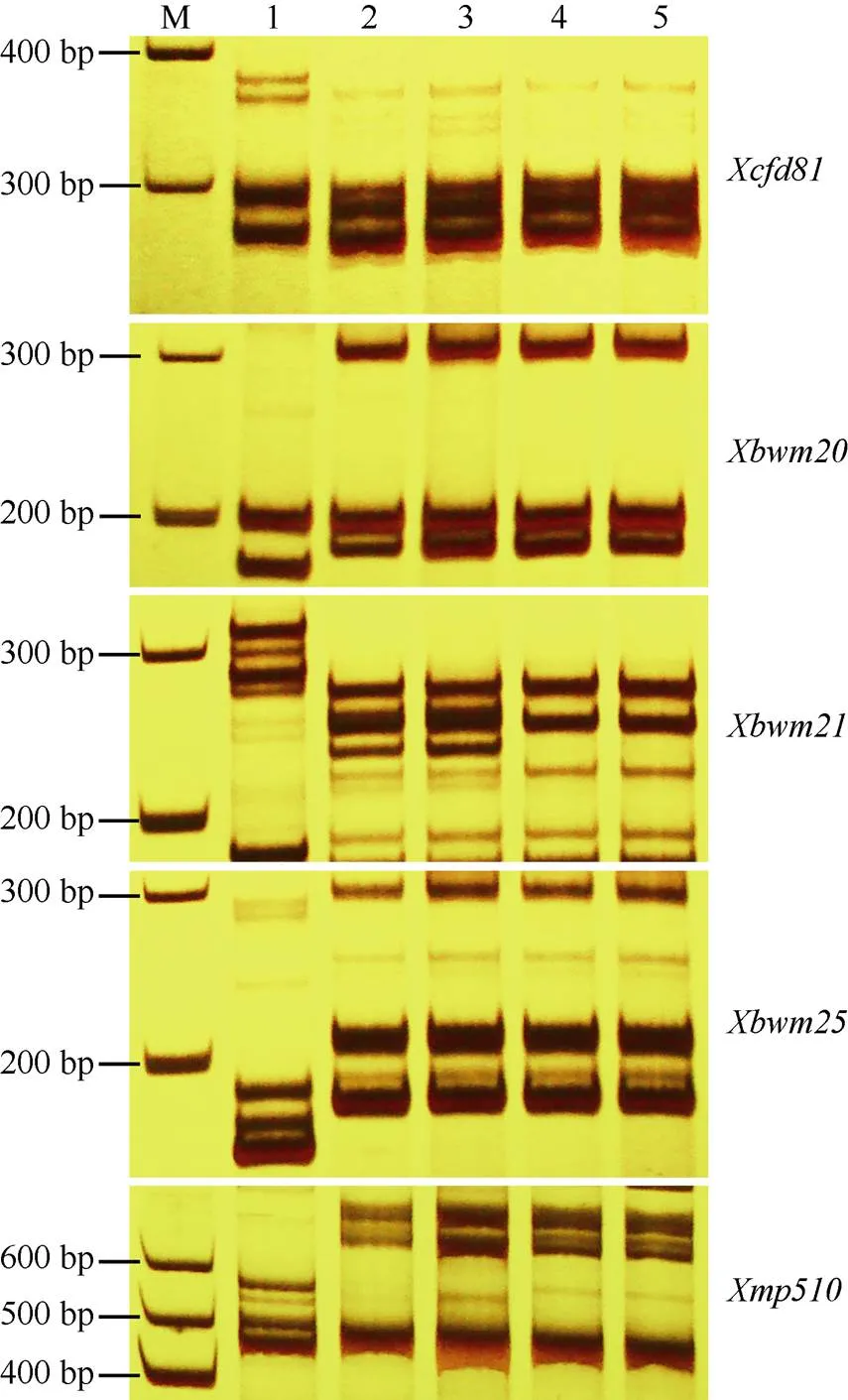
图3 CH1357中连锁标记与染色体5DS上部分已知抗白粉病基因的比较
M: marker; 1: 台长29; 2: CH1357; 3: UIka () ; 4: KM2939 (); 5: 鸟麦()。
M: marker; 1: Taichang 29; 2:CH1357; 3: UIka (); 4: KM2939 (); 5: Niaomai ().
目前, 已在小麦5DS染色体上鉴定出多个抗白粉病基因, 如来源于前苏联品种UIka的[15], 冰草衍生品系KM2939和PB3558的[16]和[13], 小麦地方品种鸟麦的[17], 中国品种良星66、中麦155、汶农14、婴泊700和Subtil的[18]、[19]、[18]、[20]和[21]以及来源于德国品种Tabasco的[22]、法国品种FG-1的[23]和英国品种Brock的等[24]。其中、、、、等可能是的等位基因, 但不是的等位基因, 因为利用15个白粉病菌株对Tabasco´Ulka/8*Cc (Chancellor)杂交的F2群体进行等位性测验发现这2个基因对测验菌株的抗性反应不同[23]。上述报道中的抗白粉病基因都与分子标记紧密连锁(图2), 其中与标记的遗传距离为2.0 cM (图2-c),为1.3 cM (图2-d),为0.1 cM (图2-e),为5.5 cM,为2.8 cM,为3.6 cM,为1.8 cM,为0.9 cM,为0.9 cM,为3.1 cM,为0.3 cM,为0.1 cM, 而在2个遗传群体中与标记的遗传距离分别为2.0 cM和1.5 cM。因此, 推测可能也位于位点或附近。在2个作图群体中连锁标记的顺序和遗传距离不完全相同, 可能是遗传背景差异造成的。
在同一染色体位点存在多个等位基因这一现象并不罕见。小麦白粉病抗性中, 已在[25]、[17]、[26]、[27]、[28]和[29]位点鉴定出多个抗病等位基因。迄今为止, 已在基因座上鉴定出17个功能等位基因。类似于, 在基因座上鉴定出越来越多的等位基因, 丰富了该位点的多样性。与是否具有等位关系, 尚需经过等位性测试进行证明。Sanchez- Martin等[30]利用突变体测序的方法获得了位点上的1个功能基因, 根据该基因序列设计引物扩增本研究中的抗病材料, 结果显示二者序列并无差异。而与、和对不同白粉菌株的抗病反应存在明显差异(表4), 推测与5DS上的其他抗白粉病基因不同, 可能是一个新的抗源。其连锁的分子标记, 可用于分子标记辅助选择(MAS), 提高育种效率。
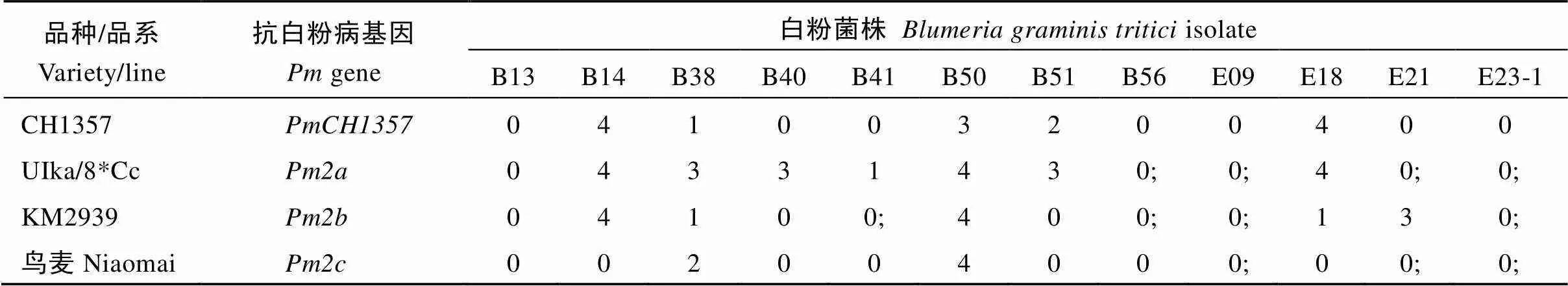
表4 CH1357与染色体5DS上已知抗白粉病基因对12个白粉菌株的抗性反应比较
4 结论
小麦种质CH1357对我国不同地区采集的32个白粉菌株中的27个表现为免疫或近免疫, 可作为我国黄淮及北部冬麦区的白粉病优质抗源, 其白粉病抗性受1对显性核基因控制, 暂命名为, 并将其抗病基因初步定位于小麦5DS染色体, 两侧连锁标记和的遗传距离分别为2.0 cM/11.3 cM和1.5 cM/8.9 cM, 推断与染色体5DS上的其他抗白粉病基因不同, 可能是一个新的抗源。
[1] Johnson J W, Baenziger P S, Yamazaki W T, Smith R T. Effects of powdery mildew on yield and quality of isogenic lines of ‘Chancellor’ wheat., 1979, 19: 349–352.
[2] Bennett A G A. Resistance to powdery mildew in wheat: a review of its use in agriculture and breeding programmes., 1984, 33: 279–300.
[3] Line R F, Chen X M. Successes in breeding for and managing durable resistance to wheat rusts., 1995, 79: 1254– 1255.
[4] Zhang D Y, Zhu K Y, Dong L L, Liang Y, Li G Q, Fang T L, Guo G H, Wu Q H, Xie J Z, Chen Y X, Lu P, Li M M, Zhang H Z, Wang Z Z, Zhang Y, Sun Q X, Liu Z Y. Wheat powdery mildew resistance genederived from wild emmer (var.) is tightly linked in repulsion with stripe rust resistance gene., 2019. doi: 10.1016/j.cj.2019. 03.003.
[5] 杨作民, 唐伯让, 沈克全, 夏先春. 小麦抗病育种的战略问题: 小麦对锈病和白粉病第二线抗源的建立和利用. 作物学报, 1994, 20: 385–394.Yang Z M, Tang B R, Shen K Q, Xia X C. Strategic issues in wheat disease resistance breeding: Establishment and utilization of second-line resistance sources of wheat rust and powdery mildew., 1994, 20: 385–394 (in Chinese with English abstract).
[6] Huang X Q, Hsam S L K, Zeller F J. Identification of powdery mildew resistance genes in common wheat (L.). IX. cultivars, land races and breeding lines grown in China., 1997, 116: 233–238.
[7] 周阳, 何中虎, 张改生, 夏兰琴, 陈新民, 高永超, 井赵斌, 于广军. 1BL/1RS易位系在我国小麦育种中的应用. 作物学报, 2004, 30: 531–535. Zhou Y, He Z H, Zhang G S, Xia L Q, Chen X M, Gao Y C, Jing Z B, Yu G J. Utilization of 1BL/1RS translocation in wheat breeding in China., 2004, 30: 531–535 (in Chinese with English abstract).
[8] Zeller F J, Hsam S L. Chromosomal location of a gene suppressing powdery mildew resistance genesandin common wheat (L. em. Thell.)., 1996, 93: 38–40.
[9] 盛宝钦. 用反应型记载小麦苗期白粉病. 植物保护, 1988, 14(1): 49. Sheng B Q. Using infection type records the wheat powdery mildew at seedling stage., 1988, 14(1): 49 (in Chinese with English abstract).
[10] Murray M G, Thompson W F. Rapid isolation of high molecular weight plant DNA., 1980, 8: 4321–4325.
[11] 陈昆松, 李方, 徐昌杰, 张上隆, 傅承新. 改良CTAB法用于多年生植物组织基因组DNA的大量提取. 遗传, 2004, 26: 529–531. Chen K S, Li F, Xu C J, Zhang S L, Fu C X. An efficient macro-method of genomic DNA isolation fromleaves.(Beijing), 2004, 26: 529–531 (in Chinese with English abstract).
[12] Lander E S, Green P, Abrahamson J, Barlow A, Daly M J, Lincoln S E, Newberg L A. MAPMAKER: an interactive computer package for constructing primary genetic linkage maps of experimental and natural populations., 1987, 1: 174–181.
[13] Lu Y Q, Wu X Y, Yao M M, Zhang J P, Liu W H, Yang X M, Li X Q, Du J, Gao A N, Li L H. Genetic mapping of a putative-derived powdery mildew resistance gene by a combination of bulked segregant analysis and single nucleotide polymorphism array., 2015, 35: 96.
[14] 付必胜, 刘颖, 张巧凤, 吴小有, 高海东, 蔡士宾, 戴廷波, 吴纪中. 与小麦抗白粉病基因紧密连锁分子标记的开发. 作物学报, 2017, 43: 307–312. Fu B S, Liu Y, Zhang Q F, Wu X Y, Gao H D, Cai S B, Dai T B, Wu J Z. Development of markers closely linked with wheat powdery mildew resistance gene., 2017, 43: 307–312 (in Chinese with English abstract).
[15] McIntosh R A, Baker E P. Cytogenetical studies in wheat: IV. Chromosome location and linkage studies involving thelocus for powdery mildew resistance., 1970, 19: 71–77.
[16] Ma P T, Xu H X, Xu Y F, Li L H, Qie Y M, Luo Q L, Zhang X T, Li X Q, Zhou Y L, An D G. Molecular mapping of a new powdery mildew resistance genein Chinese breeding line KM2939., 2015, 128: 613–622.
[17] Xu H X, Yi Y J, Ma P T, Qie Y M, Fu X Y, Xu Y F, Zhang X T, An D G. Molecular tagging of a new broad-spectrum powdery mildew resistance allelein Chinese wheat landrace Niaomai., 2015, 128: 2077–2084.
[18] Sun Y L, Zou J W, Sun H G, Song W, Wang X M, Li H J.and: new alleles offor resistance to powdery mildew in the Chinese winter wheat cultivars Liangxing 66 and Wennong 14., 2015, 99: 1118–1124.
[19] Sun H G, Song W, Sun Y L, Chen X M, Liu J J, Zou J W, Wang X M, Zhou Y F, Lin X H, Li H J. Resistance of ‘Zhongmai 155’ wheat to powdery mildew: effectiveness and detection of the resistance gene., 2015, 55: 1017–1025.
[20] Ma P T, Zhang H X, Xu H X, Xu Y F, Cao Y W, Zhang X T, An D G. The geneconfers broad-spectrum powdery mildew resistance in the multi-allelicchromosome region of the Chinese wheat cultivar YingBo 700., 2015, 35: 124.
[21] Jin Y, Xu H X, Ma P T, Fu X Y, Song L P, Xu Y F, Zhang X T, An D G. Characterization of a newallele associated with broad-spectrum powdery mildew resistance in wheat line Subtil., 2018, 8: 475.
[22] Gao H D, Zhu F F, Jiang Y J, Wu J Z, Yan W, Zhang Q F, Jacobi A, Cai S B. Genetic analysis and molecular mapping of a new powdery mildew resistant genein common wheat., 2012, 125: 967–973.
[23] Ma P T, Xu H X, Li L H, Zhang H X, Han G H, Xu Y F, Fu X Y, Zhang X T, An D G. Characterization of a newallele conferring powdery mildew resistance in the wheat germplasm line FG-1., 2016, 7: 546.
[24] 李根桥, 房体麟, 朱婕, 高亮亮, 李闪, 解超杰, 杨作民, 孙其信, 刘志勇. 普通小麦品种Brock抗白粉病基因分子标记定位. 作物学报, 2009, 35: 1613–1619. Li G Q, Fang T L, Zhu J, Gao L L, Li S, Xie C J, Yang Z M, Sun Q X, Liu Z Y. Molecular identification of a powdery mildew resistance gene from common wheat cultivar Brock., 2009, 35: 1613–1619 (in Chinese with English abstract).
[25] Ch S, Hsam S L, Hartl L, Zeller F J, Mohler V. Powdery mildew resistance genein cultivar Virest is a member of the complexlocus in common wheat (L.em Thell.)., 2003, 106: 1420–1424.
[26] Sourdille P, Robe P, Tixier M H, Doussinault G, Pavoine M T, Bernard M. Location of, a powdery mildew resistance allele in wheat, by using a monosomic analysis and by identifying associated molecular markers., 1999, 110: 193–198.
[27] Schmolke M, Mohler V, Hartl L, Zeller F J, Hsam S L K. A new powdery mildew resistance allele at thewheat locus transferred from einkorn ()., 2012, 29: 449–456.
[28] Huang X Q, Wang L X, Xu M X, Röder M. Microsatellite mapping of the powdery mildew resistance genein common wheat (L.)., 2003, 106: 858–865.
[29] Xue F, Wang C Y, Li C, Duan X Y, Zhou Y L, Zhao N J, Wang Y J, Ji W Q. Molecular mapping of a powdery mildew resistance gene in common wheat landrace Baihulu and its allelism with., 2012, 125: 1425–1432.
[30] Sánchez-Martín J, Steuernagel B, Ghosh S, Herren G, Hurni S, Adamski N, Vrána J, Kubaláková M, Krattinger S G, Wicker T, Doležel J, Keller B, Wulff BB. Rapid gene isolation in barley and wheat by mutant chromosome sequencing., 2016, 17: 221.
Genetic analysis and chromosomal localization of powdery mildew resistance gene in wheat germplasm CH1357
CHEN Fang1, QIAO Lin-Yi2,3, LI Rui2, LIU Cheng4, LI Xin2,3, GUO Hui-Juan2, ZHANG Shu-Wei2, CHANG Li-Fang2, LI Dong-Fang5, YAN Xiao-Tao2, REN Yong-Kang2, ZHANG Xiao-Jun2,3,*, and CHANG Zhi-Jian2,3,*
1College of Life Science, Shanxi University, Taiyuan 030006, Shanxi, China;2Institute of Crop Science, Shanxi Academy of Agricultural Sciences / Shanxi Key Laboratory of Crop Genetics and Molecular Improvement, Taiyuan 030006, Shanxi, China;3Key Laboratory of Crop Gene Resources and Germplasm Enhancement on Loess Plateau of Ministry of Agriculture, Taiyuan 030006, Shanxi, China;4Crop Research Institute, Shandong Academy of Agricultural Sciences, Jinan 250100, Shandong, China;5Sixth Agricultural Division Agricultural Science Research Institute of Xinjiang Production and Construction Crops, Wujiaqu 831300, Xinjiang, China
Powdery mildew is a serious disease affecting yield and quality of wheat. Wheat-introgression line CH1357 is highly resistant tof. sp.() at the adult plant stage and immune or highly resistant to 27isolates at the seedling stage. Two mapping populations (F1, BC1, and F2:3) derived from Taichang 29/CH1357 and Mianyang 11/CH1357 were used to map the powdery mildew resistance gene by the bulked segregant analysis. The powdery mildew resistance in CH1357 was controlled by a single dominant gene, temporarily named. This gene was located on the short arm of chromosome 5D and linked to SSR markersand, with genetic distances of 2.0 cM and 11.3 cM in the Taichang 29/CH1357 population and 1.5 cM and 8.9 cM in the Mianyang 11/CH1357 population, respectively.differs from othergenes reported on chromosome 5DS in resistance spectrum, which may be a new source of resistance.
resistance to powdery mildew; SSR markers; linkage map; gene mapping;
本研究由国家重点研发计划项目(2017YFD0100600), 山西省农业科学院项目(YGG17123, YCX2018D2YS01), 山东省自然科学基金项目(ZR2017MC004), 山西省重点研发计划项目(201703D211007, 201803D221018-5, 201803D421020)和山西省重点科技创新平台(201605D151002)资助。
This study was supported by the National Key Research and Development Program of China (2017YFD0100600), Shanxi Academy of Agricultural Sciences (YGG17123, YCX2018D2YS01), the National Natural Science Foundation of Shandong Province (ZR2017MC004), the Key Research and Development Program of Shanxi Province (201703D211007, 201803D221018-5, 201803D421020), and the Key Scientific and Technological Innovation Platform (201605D151002).
张晓军, E-mail: zxjemail@163.com; 畅志坚, E-mail: wrczj@126.com, Tel: 0351-7130805
E-mail: chenfang8857@126.com
2019-01-18;
2019-05-12;
2019-05-21.
10.3724/SP.J.1006.2019.91011
URL: http://kns.cnki.net/kcms/detail/11.1809.s.20190517.1713.009.html
