韩国赤芝全基因组重测序分析*
2019-08-31朱风丽徐晓兰陈体强石林春缪晓青
朱风丽,徐晓兰,陈体强,石林春,缪晓青**,兰 进
(1.福建农林大学食品科学学院 福州 350002;2.福建农林大学蜂学学院 福州 350002;4.中国医3.学福科建学省院农北业京科协学和院医食学用院菌药研用究植所物 研福究州所3 5北0 0京1 4 ;1 00193)
灵芝(Ganoderma)也被称为“不朽的蘑菇”、“传统中药的象征”,是著名的药用真菌之一。《中华人民共和国药典》(2015 年版)中收录赤芝(Ganoderma lucidum)和紫芝(G.sinense)为灵芝的药材来源。灵芝能够产生大量的生物活性物质,现有400多种化合物被鉴定,主要是三萜和多糖类物质。灵芝种类较多,栽培种主要为赤芝[1]。近年来,由于野生灵芝资源的破坏较为严重,使得具有重要药用价值的野生灵芝种质资源濒临灭绝。我国灵芝的主栽品种部分来自韩国和日本等地,由于与引进品种产地的气候环境相差较大,菌种的长时间栽培导致品种衰退,出现产量下降、品质降低、抗病虫及杂菌能力较差等缺点。因此应大力推动自主选育的适于我国生长的优良品种[2]。目前选育技术方法传统、育种周期长、遗传改良效率偏低。2011年,赤芝CGMCC5.0026 基因组测序的完成推进了赤芝基因组学的研究进程[3],使赤芝在基因组范围内进行遗传分析成为可能,为分子标记辅助育种提供技术手段,这为赤芝新品种的选育打下基础。
近年来,随着测序技术的发展及许多物种基因组测序的不断完成,重测序技术也得到了广范的应用,利用全基因组重测序技术可以在全基因组范围内挖掘单核苷酸多态性(SNP)、插入缺失标记(InDel)、结构变异(SV)和基因拷贝数变异(CNV),并广泛应用于变异检测、遗传图谱构建、性状定位和群体进化研究等[4]。Shin 等[5]将韩牛重测序数据分别与UMD 3.1 和Btau 4.6.1两个牛的参考基因组进行比对,分别发现了46 301和28 613 个SNP,这些SNP 是韩牛特有的可能与产奶机制、多汁性以及黄色的牛毛有关,同时发现了大多数的SNP 位于启动子区域,这表明SNP 调控了与性状相关基因的表达。Zhang 等人[6]通过全基因组重测序技术,将芥菜中与花色相关的基因BjPC2 定位到了染色体B04 上。Varshney 等人[7]将包含选育品系、地方品种、野生种等在内的292种木豆进行基因组重测序,在基因组上发现了几个可能与驯化、育种相关的区域。并通过基因组关联分析,确定了一些后选基因与农艺重要性状之间的相关性。Lee 等人[8]将起源于韩国的早花大豆突变株系进行全基因组重测序共筛选出了30个与开花相关的SNPs和25个Indels。
为了研究韩国赤芝菌株的基因组序列情况,应用二代测序的方法对韩国赤芝菌株进行了全基因组重测序。本文将韩国赤芝基因组重测序数据与赤芝CGMCC5.0026(参考基因组)进行比较以揭示其在SNP、InDel、SV存在的变异,旨在发现韩国赤芝菌株特有的序列特征,为研究不同来源的菌株之间的差异提供数据。并且初步探索了与菌丝生长速度和灵芝三萜合成相关的基因变异,为赤芝的分子标记育种及完善药用真菌模式体系提供基础。
1 材料与方法
1.1 材料
赤芝CGMCC5.0026[3]保存于本实验室,韩国赤芝(韩芝)由中国医学科学院药用植物研究所兰进教授馈赠。两种菌株均为二倍体。
1.2 方法
1.2.1 赤芝菌丝的生物量
将2 个赤芝菌株在28℃恒温培养箱中进行活化,将活化好的菌株转接至PDA培养基中,当菌丝生长至直径为5-6 cm 时,用打孔器在菌丝边缘处打孔,并将10个直径为1 cm的菌丝块转接至PDA液体培养基中,28℃,180 rpm黑暗下培养8 d,然后用超纯水冲洗至洗脱液为无色。45℃烘干至恒重。每个实验平行3次。
1.2.2 灵芝三萜含量的测定
菌丝的PDA 液体培养方法同“1.2.1”项下。将清洗干净的菌丝液氮研磨成细粉后烘干至恒重。根据实验室前期得到的提取灵芝三萜的条件,采用香草醛-高氯酸显色法测定灵芝三萜含量。称取0.05 g左右干燥的灵芝菌丝细粉,加入3 mL 80%的乙醇浸提4 h,然后在温度为60℃下超声提取20 min。将提取液定容至10 mL,取1 mL 水浴挥干后加入0.2 mL 5%的香草醛-冰醋酸和0.8 mL的高氯酸溶液,70℃水浴15 min,冷却至室温,最后加入4 mL 的冰醋酸,摇匀后在560 nm 处测定吸光度值。
1.2.3 韩芝的全基因组重测序
用植物基因组DNA 提取试剂盒(天根生化科技(北京)有限公司)进行DNA的提取。将提取的质量较好的DNA 送至北京百迈客生物科技有限公司进行全基因组重测序。首先对提取的基因组DNA进行检测,合格后用超声波法将DNA 片段化,然后将片段化的DNA 进行片段纯化、末端修复、3'端加A、连接测序接头,然后用琼脂糖凝胶电泳进行片段大小选择,进行PCR 扩增以形成测序文库,建好的文库先进行文库质检,质检合格的文库用Xten进行测序。对测序得到的原始reads(双端序列)进行质量评估,并过滤得到Clean Reads,用于后续生物信息学分析。用bwa 软件[9]将Clean Reads 与参考基因组序列进行比对,基于比对结果进行SNP、Small InDel、SV 的检测和注释,并对DNA水平差异基因进行挖掘与注释分析。
1.2.4 韩芝与赤芝CGMCC5.0026 基因组间变异的检测与注释
根据韩芝的Clean Reads 在赤芝CGMCC5.0026 基因组[3]上的定位结果,用GATK 软件工具包[10]进行SNP和Small InDel 的检测,其中SNP 的检测是根据Clean Reads 在参考基因组的定位结果,使用Picard(http://sourceforge.net/projects/picard/.(Picard))进 行 去 重 复(Mark Duplicates)、GATK 进 行 局 部 重 比 对(Local Realignment)、碱基质量值校正(Base Recalibration)等预处理,以保证检测得到的SNP准确性,再使用GATK进行SNP的检测,过滤,并得到最终的SNP位点集。并用SnpEff 软件[11]进行SNP 和Small InDel 变异的注释与预测。使用breakDancer 检测SV变异,并对SV进行注释。
1.2.5 DNA水平变异基因的挖掘与功能注释
寻找韩国赤芝与赤芝CGMCC5.0026 间发生的非同义突变SNP、CDS区发生的InDel与SV的基因,将这些变异基因通过BLAST[12]与NR[13]、SwissProt[13]、GO[14]、COG[15]、KEGG[16]功能数据库进行比对,并对这些基因的功能进行注释。
1.2.6 RT-PCR检测
将“1.2.1”中培养的菌丝使用TranZol UP(北京全式金)提取RNA,然后用TaKaRa PrimeScript TM RT reagent Kit with gDNA Eraser Kit(购自TaKaRa)反转录成cDNA。根据灵芝全基因组测序的结果,找出本实验中注释到的9 个与生长相关的转录因子的CDS 序列,使用Primer premier 5 软件设计引物,引物序列(表1),以灵芝菌丝cDNA 为模板,用SYBR®Premix Ex Taq TM Ⅱ(Tli RNaseH Plus)(购自TakaRa)进行PCR。反应程序为:94℃预变性30 s,94℃变性5 s,60℃退火30 s,共40个循环。荧光定量用甘油醛-3-磷酸脱氢酶(GPD)作为内参基因,基因的相对表达量用2-ΔΔCT计算。
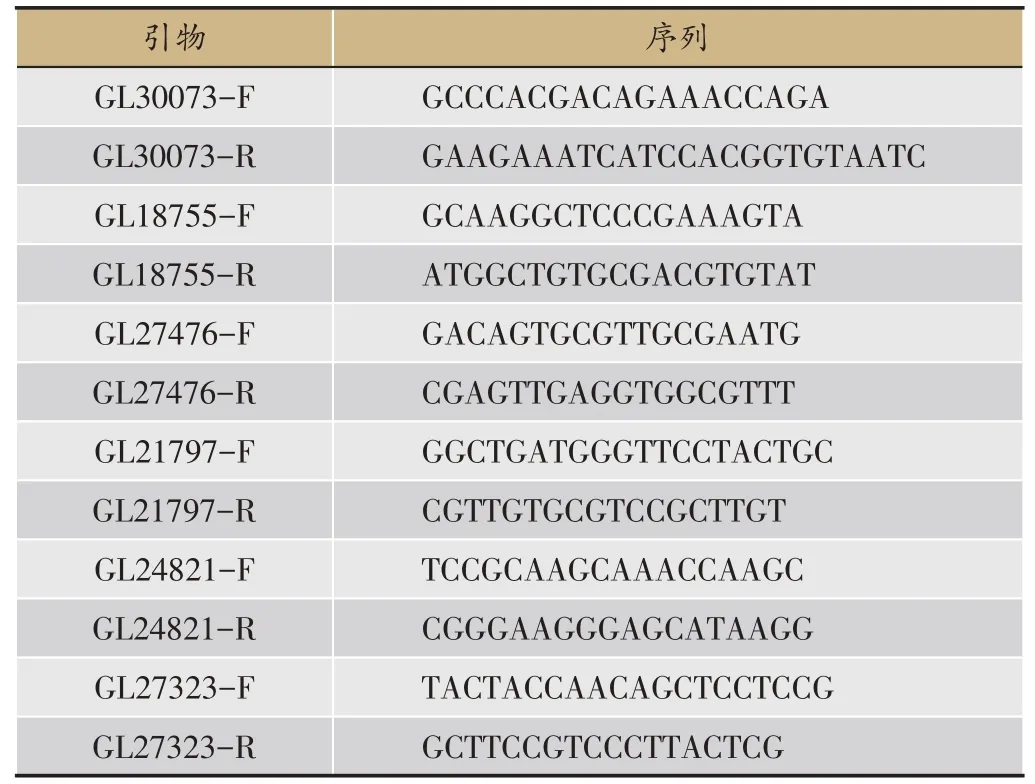
表1 RT-PCR引物序列
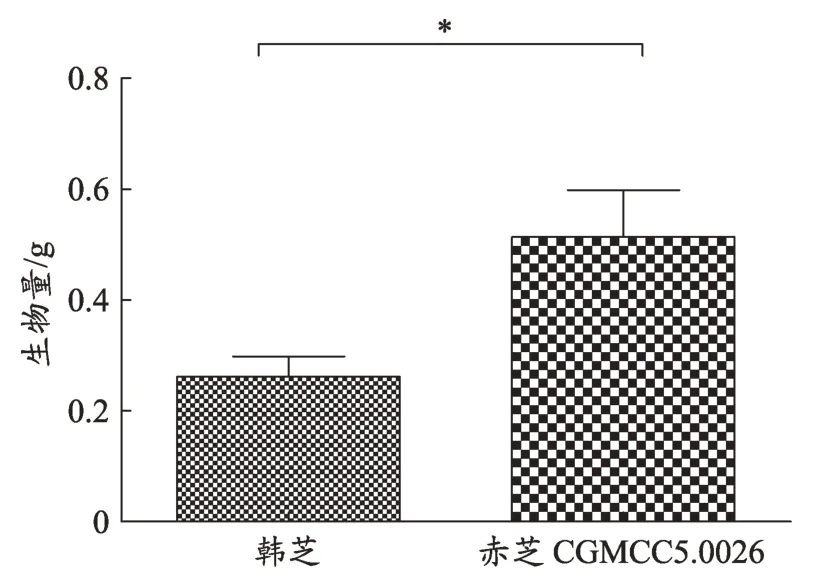
图1 两种菌丝的生物量
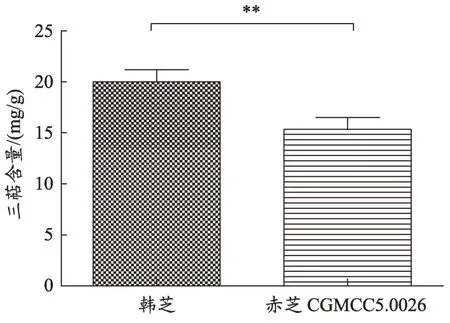
图2 两种菌丝的三萜含量
1.2.7 数据处理
数据分析使用SPSS17.0,显著性分析采用单因素方差分析和多重比较。P <0.05为差异性显著,P <0.01为差异性极显著。
2 结果与分析
2.1 两种赤芝菌株的菌丝生物量
液体摇培8 天后,两种赤芝的菌丝干重(图1)所示,赤芝CGMCC5.0026 的菌丝干重为0.51 g 是韩芝的1.97倍,并在统计学上表现为显著性差异。
2.2 三萜含量的比较
对赤芝CGMCC5.0026 和韩芝菌丝的三萜含量进行比较,结果表明(图2),韩芝菌丝的三萜含量为20.00 mg·g-1是赤芝CGMCC5.0026的1.30倍,在统计学表现为极显著差异。
2.3 韩芝的全基因组重测序
对重测序的Q30、GC 含量、与参考基因组的比对率、平均覆盖深度进行统计,其统计结果(表2)。得到的reads 数目有7 679 704 个,将重测序结果与赤芝CGMCC5.0026基因组比较,发现与参考基因组的匹配率为87.79%,测序深度为44倍。
2.4 韩芝SNP的检测与注释
韩芝的SNP 检测结果表明,总共检测到的SNP 数量为291212 个,其中SNP 的Ti/Tv(转换/颠换)的比值为2.81,其中杂合类型的SNP比例为62.87%。SNP变异的基因数量为13 914,占灵芝预测总基因的86.35%。对SNP的检测结果进行注释,发现,SNP的变异主要发生在CDS区(37.04%),在CDS区中主要的突变类型为同义编码突变(58.57%)和非同义编码突变(40.53%)(表3)。
2.5 韩芝的Small InDel检测与注释
根据样品的Clean Reads 在参考基因组上的定位结果,检测韩芝与参考基因组间在CDS区的Small InDel变异,其变异总数为9 458个(表4)。根据检测得到的Small InDel 位点在参考基因组上的位置信息,对比参考基因组的基因、CDS 位置等信息,对InDel 位点进行功能或类型的分类,结果(表5)所示。从表中可以看出,InDel 变异多发生在基因的上游(37.31%)、下游(33.43%)区域和CDS 区域(12.71%),在CDS 区中,发生的变异大多为移码突变(50.58%)。
2.6 韩芝的SV的检测与注释
使用breakDancer 对SV 进行检测的结果表明,韩芝的结构变异总数为3925,其中,变异最多的为缺失类型变异,其数量为1437(表6)。根据检测到的SV变异在参考基因组上的位置信息,对比参考基因组的基
因、CDS位置等信息,对韩芝SV变异进行位置的注释,并对缺失(DEL)、插入(INS)、反转(INV)三种类型的结构变异进行注释,其结果(表7)所示,从表中可以看出,SV的变异主要发生在外显子区域。

表2 赤芝5.644 测序数据统计
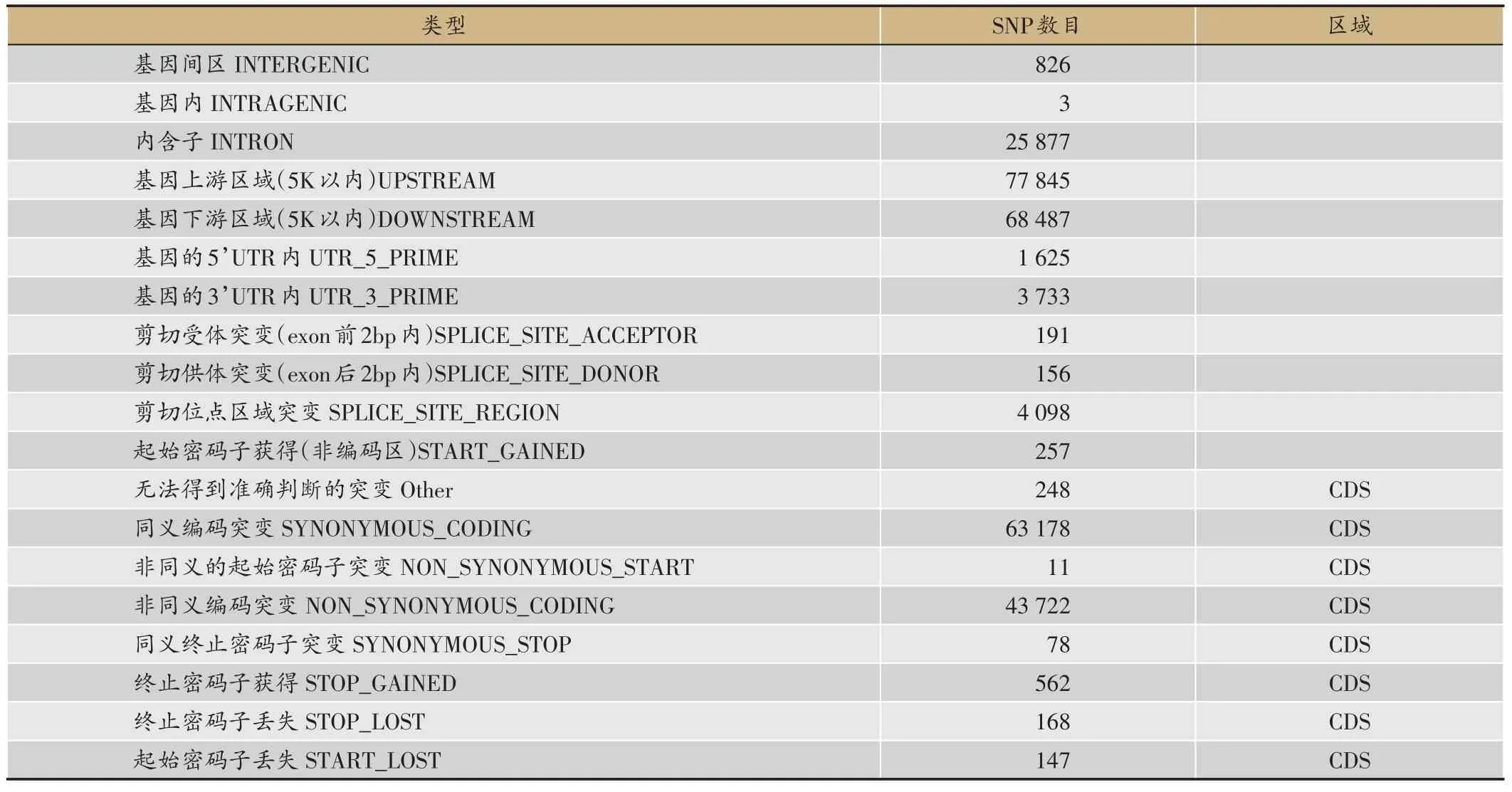
表3 SNP注释结果

表4 Small Indels的CDS区统计
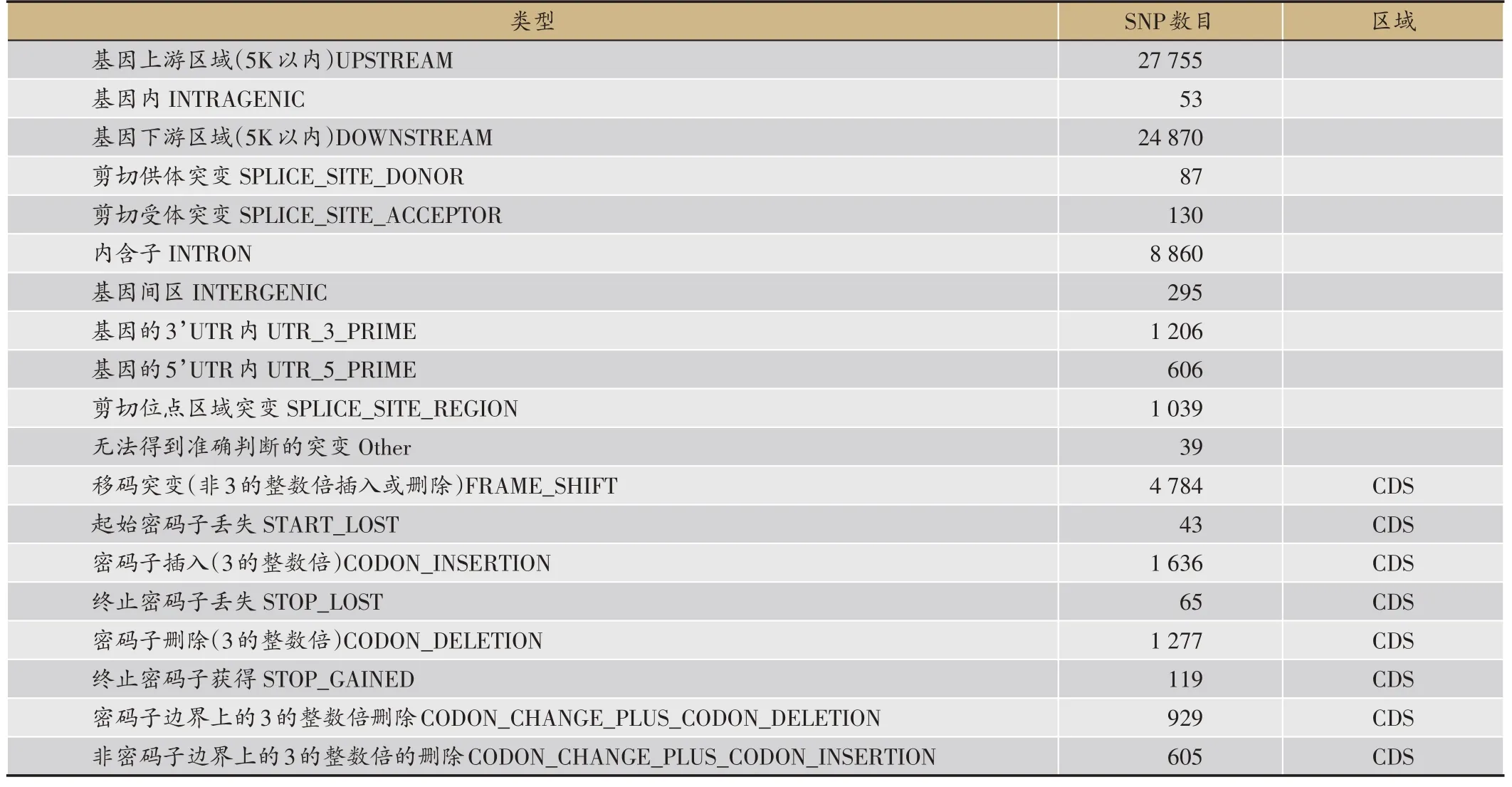
表5 Small Indel注释结果

表6 SV统计
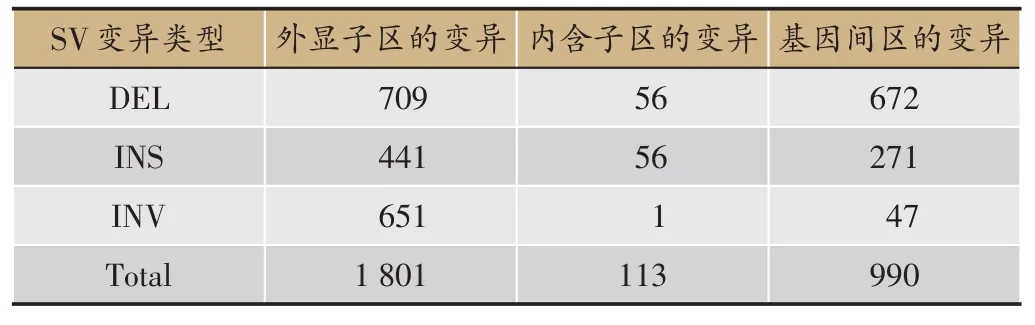
表7 SV注释
2.7 韩芝DNA水平变异的分析
在CDS区发生的变异可能会引起基因功能的变化,检测到韩芝与参考基因组间发生的非同义突变SNP、CDS 区发生的InDel 与SV 的基因数量分别为10 607、4 774、1 428。共有9 649个变异基因得到了注释,COG共注释到了4061个变异基因,其中存在较多变异基因的功能类为氨基酸运输和代谢(361 个)、次生代谢物的生物合成、运输和分解代谢(352个)、碳水化合物的运输和代谢(315 个)(图3)。将得到的9649 个变异基因进行KEGG 分析,这些变异基因主要注释到的代谢通路为碳代谢(79 个)、氨基酸的生物合成(72 个)和RNA转运(70个)(图4)。
这些变异基因中有86个为转录因子,目前对于灵芝中转录因子的研究,分析与灵芝菌丝生长及灵芝三萜含量相关的转录因子GlSwi6[17]和GlPacC[18]发现,两个基因CDS 区发生的变异类型分别为密码子删除和非同义编码突变。除此之外,从这些变异的转录因子中还发现了4个可能与韩芝菌丝生长相关的候选基因PacC/Rim101、Zap1、Spt8 和Bfr2。这 些 转 录 因 子 的CDS区均发生了非同义编码突变(表8)。
注释为细胞色素P450 的变异基因有195 个,与已报道的[3]和羊毛甾醇合酶(LSS)高度相关的78 个CYP基因相比,有70 个相同的基因,其中有12 个CYP512和1个CYP5144,这13个基因在CDS区均发生了变异,变异类型为非同义编码突变、移码突变、终止密码子获得、密码子改变+密码子删除及密码子(3 的整数倍)插入(表9)。对基因GL15605 的变异进行图示,该基因DNA 序列2186核苷酸处发生单碱基突变(图5 A),使得氨基酸从精氨酸转变为赖氨酸(图5 B)。
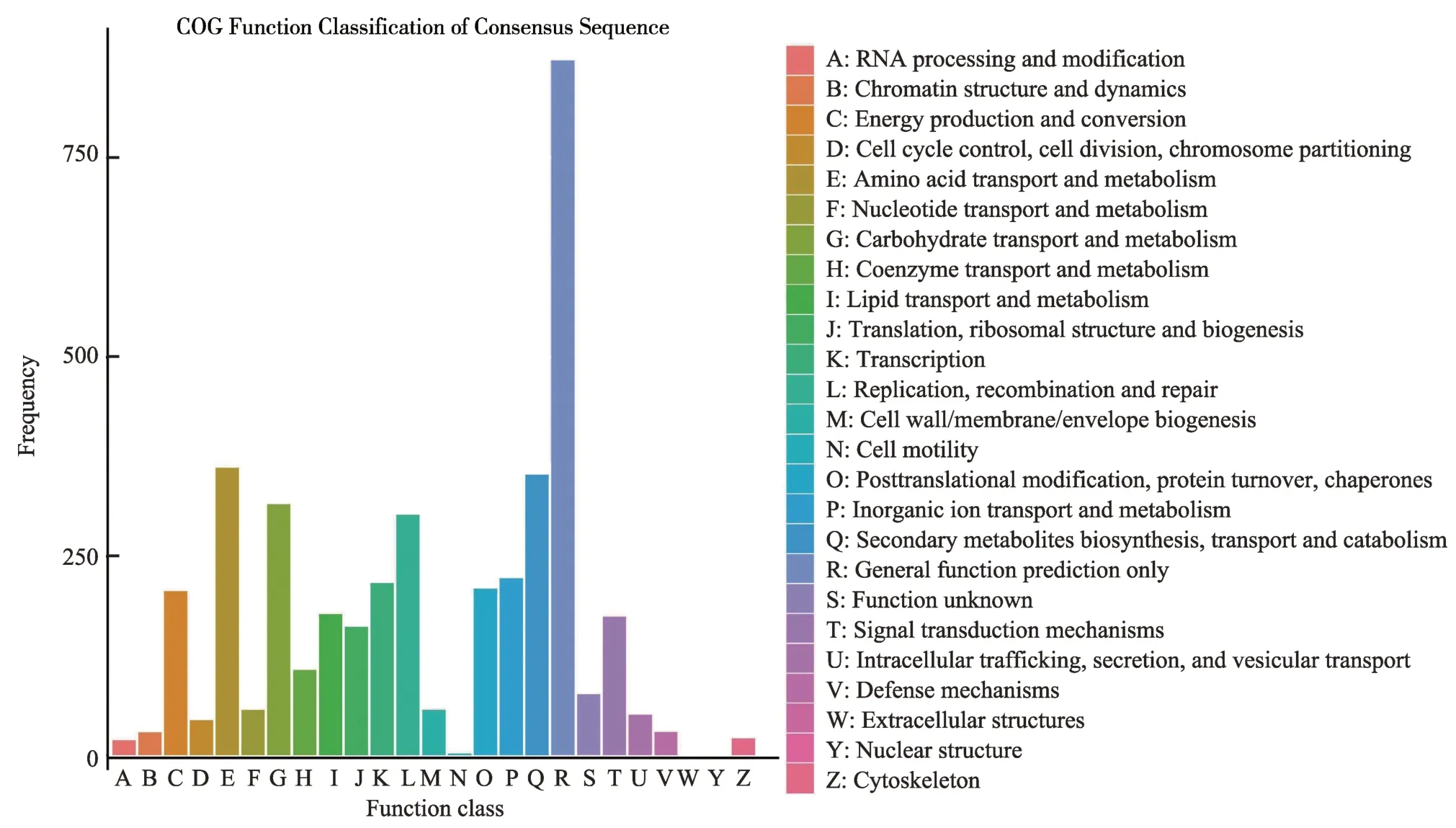
图3 变异基因COG注释图

图4 变异基因的KEGG注释图
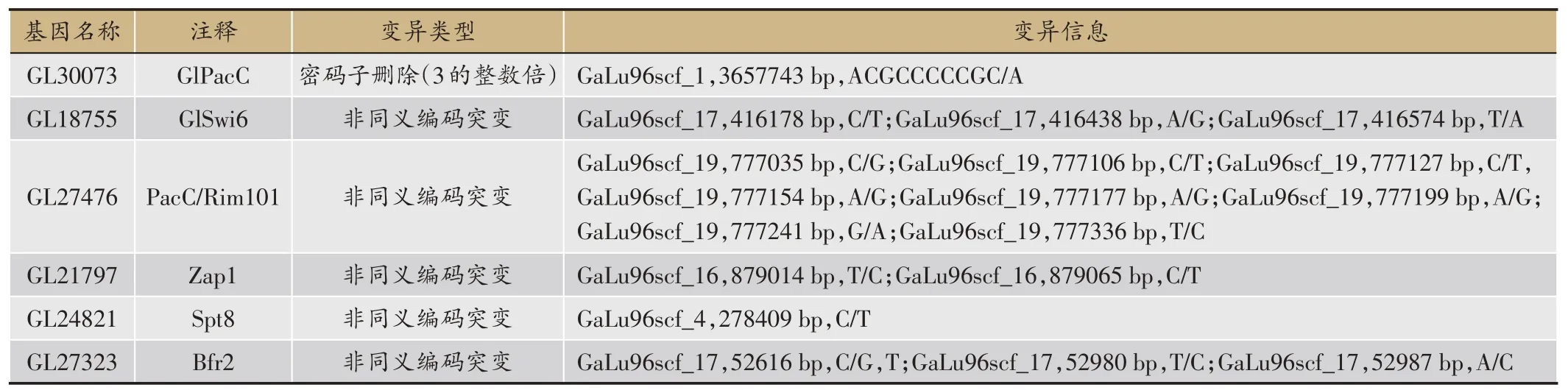
表8 与生长相关的转录因子的变异
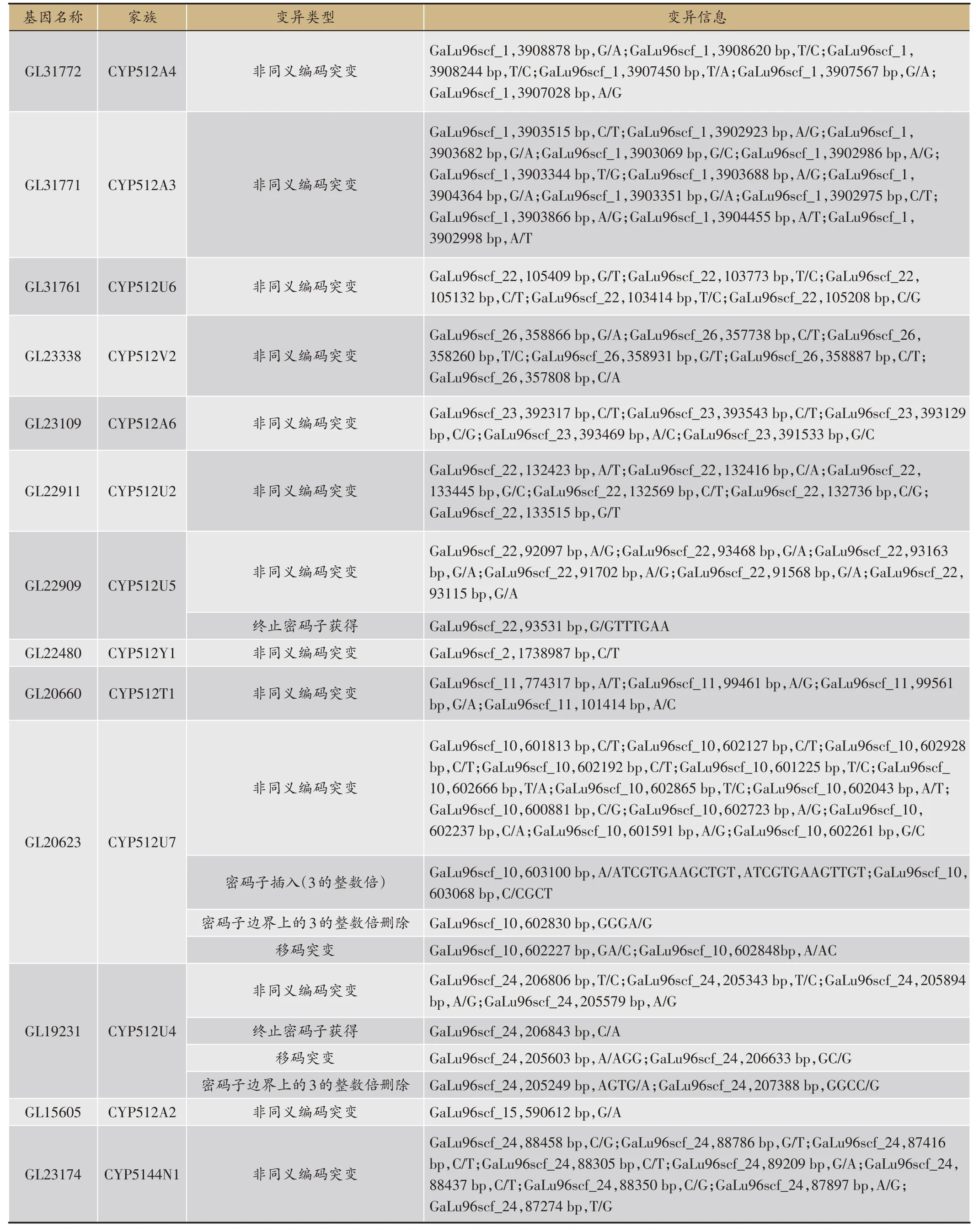
表9 细胞色素P512和P5144家族的变异
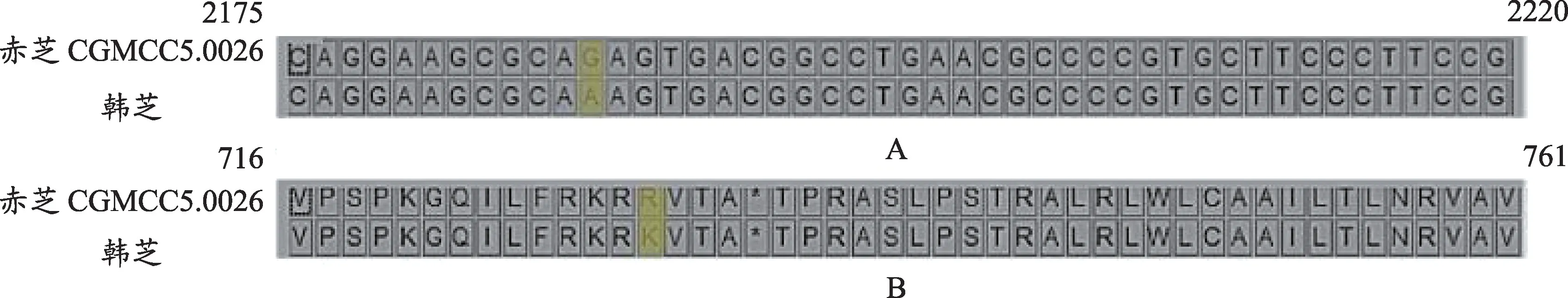
图5 基因GL15605序列比对
2.8 对注释的与生长相关的转录因子的RT-PCR检测结果
对注释的与生长相关的6 个转录因子进行RTPCR分析,结果表明(图6),韩芝的基因表达量高于赤芝CGMCC5.0026,注释为PacC/Rim101(GL27476)和Zap1(GL21797)的基因,在两种菌丝中的差异具有统计学意义。其它4个基因的表达量差异不显著。
3 讨论
随着越来越多生物的基因组被测序,其基因组重测序技术已成为分子育种研究的重要手段。本文对韩芝进行基因组重测序,得到了大量的变异基因,在COG注释中,这些变异基因主要注释到了氨基酸的运输和代谢,次生代谢物的生物合成、运输和分解代谢,碳水化合物的运输和代谢。
变异基因中,注释到的转录因子共有86 个,主要的类型为bZIP、C2H2、CBF、HTH、MADS、TFII_related和一些真菌特有的转录因子。其中,C2H2类型转录因子PacC 的缺失对真菌菌丝的生长和分生孢子的产量具有一定的抑制作用[19,20]。赤芝转录因子GlPacC 沉默,对菌丝生长速度、子实体发育和灵芝酸合成具有抑制作用。韩芝中该基因CDS 区发生了密码子删除(3的整数倍),CGCCCCCGC碱基缺失。在韩芝中还发现了另一个pH响应转录因子PacC/Rim101(GL27476)在CDS 区也发生了非同义编码突变。此外,Swi6 转录因子属于APSES 家族,是真菌特有的一个蛋白家族[21]。有研究表明,赤芝中转录因子GlSwi6基因沉默菌株降低了菌丝生长速度,增加了菌丝分支,并且抑制了原基和子实体的形成,降低了灵芝酸的含量[17],本研究发现韩芝中该基因CDS 区发生了非同义编码突变。除此之外,还发现了一些与生长相关的转录因子Zap1、Spt8和Bfr2在CDS区也发生了非同义编码突变。有研究表明,酵母菌在锌受限型培养基中生长受到限制,这与Zap1 转录因子的靶基因有关[22]。SPT 包括SPT3、SPT7、SPT8 和SPT20,是SAGA 复合体的重要成分蛋白。禾谷镰刀菌中,与野生型相比,Fgspt3 和Fgspt8缺失突变株的菌丝生长明显受到抑制,并且不能产生分生孢子,同时与孢子形成相关的基因FgFlbC和FgRen1的表达量也下调[23]。Bfr2基因对细胞生长周期具有调控作用[24]。菌丝生长受很多基因和通路的调控,调控过程比较复杂。转录因子在结合区域产生的SNP可能会影响转录因子的结合特性,影响转录因子在等位基因上的结合强度,从而影响SNP 所在基因的表达情况[25]。在研究中我们发现,赤芝CGMCC5.0026 的生物量高于韩芝。对灵芝中注释的与生长相关的6个转录因子,通过RT-PCR 检测发现赤芝CGMCC5.0026 的表达量均低于韩芝,由于在RT-PCR中使用甘油醛-3-磷酸脱氢酶(GPD)作为内参基因,该内参基因在不同灵芝中的表达量有所差异,本实验中发现韩芝中的GPD表达量(Cq值为24.5)要高于赤芝CGMCC5.0026(Cq值为22.17),因此在计算基因的相对表达时,韩芝中基因的表达量要高于赤芝CGMCC5.0026。这6个转录因子的变异是否会参与灵芝菌丝的生物量以及如何参与菌丝生长的调控还需要后续实验的进一步验证。
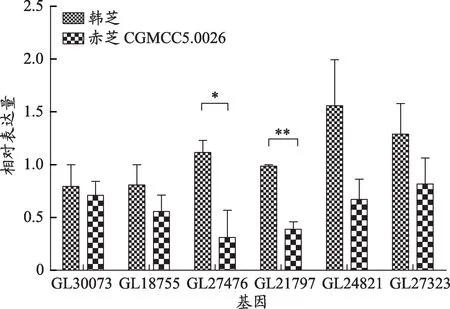
图6 不同菌丝的基因相对表达量
肌动蛋白骨架在真菌细胞极性生长、产孢、应激和维持细胞形态等方面发挥重要作用。本文中,韩芝中部分肌动蛋白相关蛋白CDS 区发生了非同义编码突变、移码突变。肌动蛋白相关蛋白(ARP5)缺陷型植物表现出植株矮小和细胞大小异常[26],本研究注释到的两个肌动蛋白相关蛋白(ARP5)均发生了非同义编码突变。
此外,本研究还发现,注释为细胞色素P450 的变异基因有195个,这些基因在KOG分类注释中,有135个变异基因参与次生代谢产物的生物合成,转运和分解代谢,4 个变异基因参与了能量的产生和转化。与Chen 等人研究中78 个可能和赤芝三萜合成相关的CYP450相比,发现有70个相同的CYP450,其中,有12个CYP512 和1 个CYP5144,研 究 表 明,在 动 物 中CYP512 和CYP5144 对甾体激素和睾酮等激素的结构具有修饰作用,并且这两个P450家族与羊毛甾醇合酶(LSS)共表达[3],由于细胞色素P450 基因变异较多,因此本实验对不同菌株菌丝的三萜含量进行测定,发现韩芝菌丝三萜含量明显高于赤芝CGMCC5.0026,这说明韩芝中大量细胞色素P450 的变异可能会影响赤芝三萜的含量。这将为细胞色素P450 参与三萜的合成提供新的证据。
赤芝全基因组测序的完成为灵芝成为药用模式真菌奠定了坚实的基础[3]。灵芝生长发育和发酵与栽培的研究为灵芝的一般生物学研究奠定了基础[27]。灵芝的药用成分丰富,目前已分离出400多种活性物质,包括多糖、萜类、甾醇类、生物碱等[28]。灵芝的基因组分析进一步证实了灵芝具有三萜、聚酮、倍半萜等多条次生代谢合成途径[27],目前研究较多的是合成灵芝三萜的MVA 途径,其上游途径中的一些关键酶基因如AACT[29]、HMGS[30]、HMGR[31]、FPS[32]、MVD[33]、LSS[34]和SQS[35]已被克隆及进行基因过表达研究。赤芝基因组测序的完成筛选出600 多个转录调控蛋白和219 个CYP450 编码基因[3]。目前,灵芝遗传转化的研究也进一步开展,如电击法[36]、PEG介导法[37]以及农杆菌介导法[38]。这为灵芝功能基因组学的研究奠定了基础。本研究进行韩芝全基因组重测序,筛选出一些潜在的与菌丝生长相关的转录因子,并且对细胞色素P450对三萜含量的影响具有指导意义,这对完善灵芝模式真菌研究体系提供了基础。
综上所述,本研究通过全基因组重测序筛选了韩国赤芝与中国赤芝间变异的转录因子和细胞色素P450,并对韩芝菌丝生长和三萜合成相关基因的变异进行初步挖掘和分析,对揭示赤芝菌丝生长和三萜合成的遗传机制具有重要意义,而这些变异基因的具体功能还有待进一步验证。
