长江中下游及云贵高原芦苇居群的遗传变异特征
2019-08-22胡思文何汝嘉周淇王树人黄睿曹新益丁浩曾巾赵大勇
胡思文 ,何汝嘉 ,周淇 ,王树人 ,黄睿 ,曹新益 ,丁浩 ,曾巾,赵大勇 *
1. 河海大学/水文水资源与水利工程科学国家重点实验室/河海大学全球变化与水循环国际联合实验室,江苏 南京210098;2. 湖泊与环境国家重点实验室/中国科学院南京地理与湖泊研究所,江苏 南京 210008;3. 河海大学水利学科专业实验教学中心,江苏 南京210098
芦苇(Phragmites australis),禾本科芦苇属多年生草本植物,分布广泛,在湿地生态系统中承担重要的生态功能。作为湿地生态系统中最常见的优势种之一,芦苇具有净化水体的作用,也是水体生态系统中物流和能流的物质基础,其较高的生产力可以被湖泊湿地中初级消费者利用,芦苇的茎秆和地下茎可以防止沉积物流失和河岸腐蚀,能够维持生态系统多样性和环境稳定性(岑璐瑶等,2019;Saltonstall et al.,2016)。近年来,我国的一些芦苇湿地出现了种群退化现象,当前,保护我国芦苇的遗传资源,探讨不同生态型芦苇的遗传差异应当成为研究热点。
不同生境中的芦苇在长期进化、适应特异生境的过程中,产生了可遗传的性状差异,并随繁衍演替形成了种间丰富的生态型(杨允菲等,1998)。由于不同地理区域芦苇种群所处的环境差异较大,互有明显界限,在水深、盐度、水分、土壤等环境因子的作用下,芦苇的基径、株高、穗长等性状会发生显著变化。例如,新疆芦苇在不同生境适应的过程中形成的多种生态型,在形态及营养结构器官上有着显著差异(刘明智等,2011)。黄河三角洲的芦苇根据生境的盐分差异被分为4种生态型:淡水沼泽芦苇、咸水沼泽芦苇、低盐草甸芦苇和高盐草甸芦苇(赵可夫等,1998)。河西走廊地区不同生境的芦苇根据形态、生理生化等特征被分为水生芦苇、轻度盐化草甸芦苇、重度盐化草甸芦苇及沙丘芦苇4个生态型(任东涛等,1992)。任东涛等(1994)结合前人数据及其实验结果,采用主分量分析及模糊聚类的方法指出划分芦苇生态型的综合指标体系包括形态、生态、遗传代谢及抗逆性生理等4个方面。
表型可塑性和遗传变异是植物适应异质性生境的重要策略,因此,同种植物的不同居群可根据外部形态与生理特征进行生态型的基本划分,但仅着眼于表观差异可能会低估或高估生态型的变异程度,形态相似的种群极容易被忽略(庄瑶等,2010)。Theunissen提出:植物不同生态型的基因差异是自然选择下遗传变异的结果(Theunissen,1997)。可见遗传变异是不同生态型形成的基础,随着分子技术的发展,分子标记能够为生态型的识别提供更为可靠、稳定的参考指标,越来越多的研究使用分子技术对芦苇生态型进行更为细致的划分。张淑萍(2001)以形态特征和遗传变异为基础,通过聚类分析将黄河下游典型湿地的芦苇种群分为盐生芦苇、淡水芦苇和巨型芦苇。林文芳等(2007)使用ISSR和RAPD技术对河西走廊4种生态型芦苇的遗传相似度进行分析,进一步证实了4种生态型芦苇由水生生境经盐渍生境向沙丘生境逐渐演化的趋势特点。目前关于芦苇的研究已经有了良好的生态基础,主要集中在芦苇基础理论的研究、芦苇病虫害的防治、芦苇在环境保护中的应用以及芦苇的开发利用(宋金枝等,2018)。但关于不同芦苇居群遗传差异的研究区域多集中于小空间尺度,如:黄河三角洲、河西走廊等盐度梯度显著的地区,或生境差异显著的芦苇居群,如:旱生芦苇和水生芦苇,以及形态差异显著的芦苇居群等。针对不同地理区域的淡水湖泊芦苇的相关研究还比较少,且一些根据形态及地域生境差异划分的生态型并没有遗传分化方面的证据。
叶绿体基因组(chloroplast DNA,cpDNA),是仅次于核基因组的第二大基因组,广泛存在于植物叶绿体内(Zhou et al.,2014)。作为植物中一个独立的细胞器,叶绿体拥有一套完整的基因组,基因含量较为保守、易于测序,且 cpDNA的一些特定非编码区由于序列结构特点,极易发生突变(李健仔等,2002)。因此,cpDNA对于种间类群系统发育的研究有重要意义。rpl16基因属于cpDNA非编码区,具有高度的AT重复茎环变异和序列长度变异,相较于编码区的基因片段进化速率更高,可为低分类群间的系统发育研究提供更多的信息。
空间上广泛分离的生态型可以显示出由基因确定的不同特征,并且局限于它们发生的地理区域,因为跨度较大的地理区域易形成具有高度异质性的生境,需要种群内基因型多样化以提供足够的变异类型来适应特定的自然条件,因此,遗传变异是芦苇适应异质环境的重要方法(颜忠诚,2001)。我国湖滨带芦苇居群广布的淡水湖泊集中分布于长江中下游和云贵高原,且这两个地理区域经度和纬度跨度都比较大(在年平均气温、降水、日照等气候特征上有一定差异,具有一定的地理隔离),芦苇居群易形成大幅度的生境差异,种群间遗传多样性可能较高,进而形成不同的生态型,因此,地理区域选取长江中下游和云贵高原,在淡水湖泊的芦苇研究中具有一定的代表性。本研究以不同地理区域为划分指标,通过分析芦苇叶绿体基因组中的rpl16基因序列探讨地理区域跨度较大的长江中下游和云贵高原共19个湖滨带芦苇居群的亲缘关系、遗传多样性和遗传结构,并分析了不同湖泊芦苇的形态差异与遗传差异的异同,以期在分子水平为不同地理区域芦苇生态型的划分提供遗传分化方面的证据。
1 材料与方法
1.1 样品采集
本实验所有芦苇样品于2018年7-8月采自长江中下游平原淡水湖泊和云贵高原淡水湖泊中的规模较大的典型天然湖泊,长江中下游选取:鄱阳湖(Poyang Lake)、石臼湖(Shijiu Lake)、巢湖(Chaohu Lake)、长荡湖(Changdang Lake)、淀山湖(Dianshan Lake)、滆湖(Gehu Lake)、高邮湖(Gaoyou Lake)、洪泽湖(Hongze Lake)、升金湖(Shengjin Lake)、太湖梅梁湾(Taihu Meiliang Bay)和阳澄湖(Yangcheng Lake),共12个芦苇居群,32个个体;云贵高原选取:滇池(Dianchi Lake)、抚仙湖(Fuxian Lake)、毛板桥水库(Maobanqiao Reservoir)、青山嘴水库(Qingshanzui Reservoir)和阳宗海(Yangzong Lake),共7个芦苇种群,23个个体。每个湖泊一个采样点,其中巢湖、抚仙湖、滇池是比较重要的淡水湖泊,故设置了两个采样点。
样点的样地框定在10 m×10 m的只有芦苇、或者芦苇为绝对优势植物种的方形样地,且芦苇密度每平方米大于5株,每个采样点按照常规采样方式选取生长状态健康的数株芦苇,用10 m卷尺量取记录每株芦苇基径、株高、最大叶长、最大叶宽、中部节长和节数作为表型数据,每株芦苇采集的叶片置于封口袋中,并于4 ℃保存运输,芦苇叶样品带回实验室后保存于-20 ℃。
本研究所用样品的信息编号及采样点分布图分别见表1和图1。
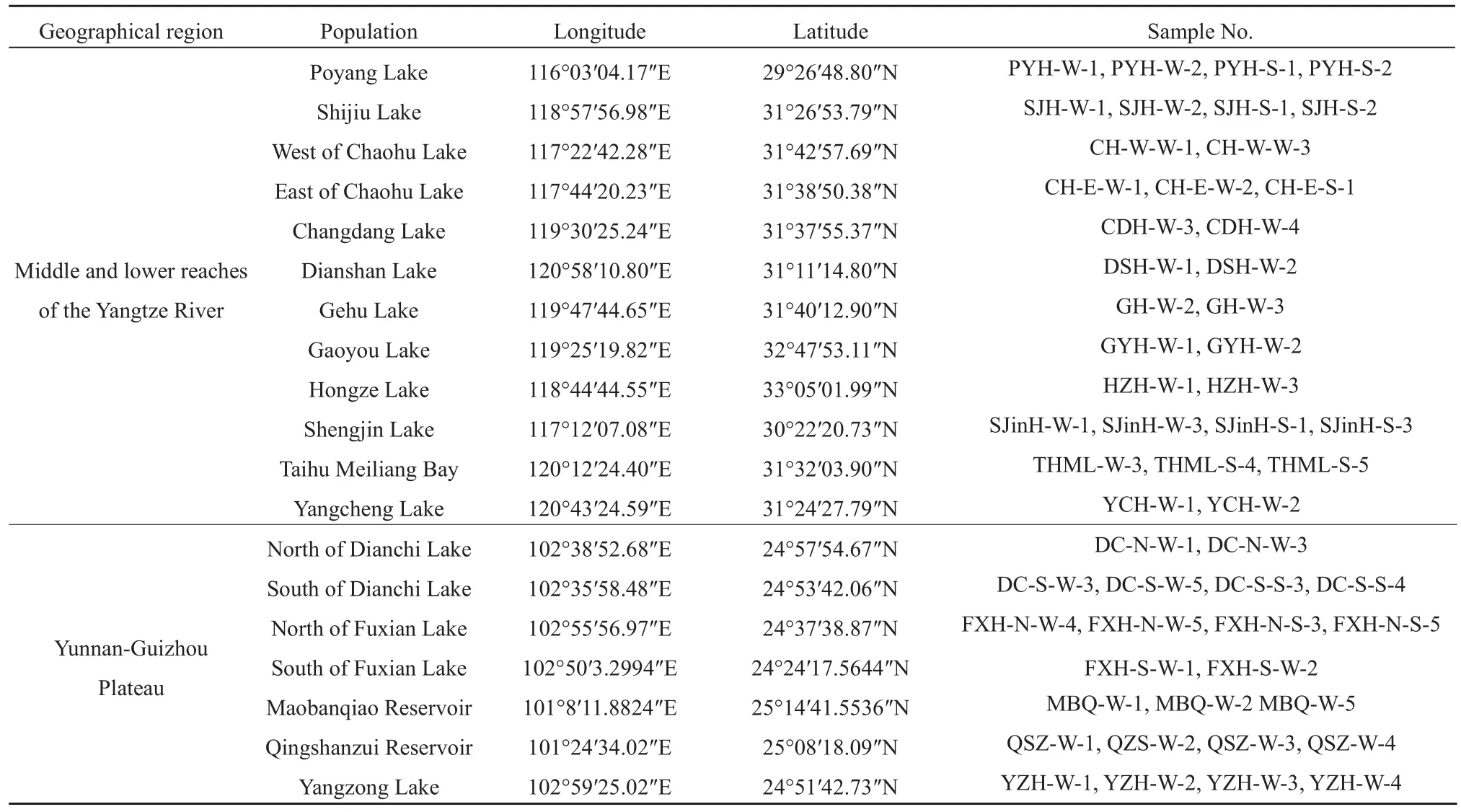
表1 样品来源信息Table 1 Information of the sample source
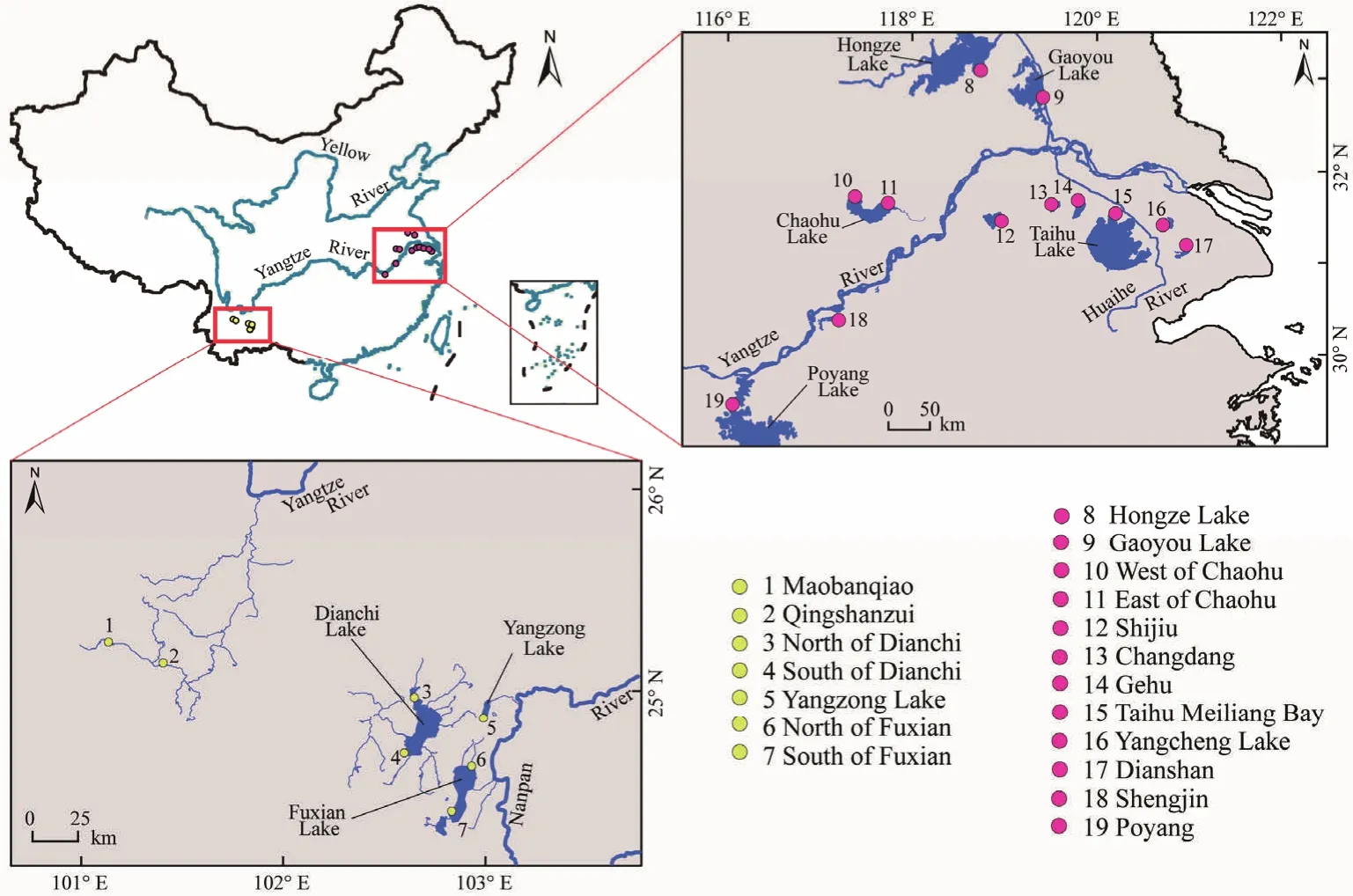
图1 长江中下游及云贵高原芦苇样品分布图Fig. 1 Distribution of sampling sites in the middle and lower reaches of the Yangtze River and the Yunnan-Guizhou Plateau
1.2 样品DNA提取及PCR扩增
所有芦苇样品的总DNA采用HP Plant DNA Kit(D2485-01)试剂盒提取,提取前先用液氮充分研磨样品,提取后于-20 ℃冰箱内保存,采用引物rpl16-F71(GCTATGCTTAGTGTGTGACTCGTTG)和 rpl16-R1661(CGTACCCATATTTTTCCACCA CGAC)扩增rpl16序列(龚晓洁,2009)。所有样品rpl16基因的PCR反应均采用Ex Taq DNA聚合酶进行扩增,体系为25 μL。包括:0.25 μL TaKaRa Ex Taq (5 U·μL-1),2.5 μL 10·Buffer,4 μL dNTP Mixture,上下游引物(10 μmol·L-1)各 0.5 μL,0.8%琼脂糖电泳检测无降解的DNA模板1 μL,用ddH2O补充至25 μL。扩增程序为:94 ℃预变性4 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,共35个循环,最后72 ℃延伸5 min。1.5%琼脂糖电泳检测扩增产物,根据Marker确认产物片段大小,取明亮单一的目的片段条带交由生物公司(南京金斯瑞生物科技有限公司)进行DNA测序和拼接。
1.3 数据分析
对测序获得的序列在NCBI上进行Blast比对,确定为目的序列,并将序列上传至Genbank获取登录号。使用 Clustalx 1.8软件进行多序列的比对排列,采用MEGA 7进行居群间遗传距离分析,并采用最大简约法构建基于rpl16基因序列的系统发育树,用启发式搜索最简约树,Gaps/missing做完全缺失状态处理,自展法(Bootstrap-method)检验系统发育树进化枝的可靠程度,参数选择Tree-Bisection-Regrafting (TBR)法,共循环1000次重复。利用DNAsp 6.0软件计算居群总的核苷酸多样性 Pi、单倍型多样性 Hd以及单倍型统计(王成树等,2002)。通过Arlequin软件的AMOVA程序进行不同地理居群间和居群内的遗传变异组成分析,及不同地理居群间分化系数 Fst,并根据 Fst计算基因流 Nm=0.25(1-Fst)·Fst-1(张晓芸等,2018)。对不同湖泊芦苇样品的形态指标使用SPSS软件进行单因素方差分析。
2 结果与讨论
2.1 rpl16基因序列分析
本研究获得了 55个芦苇样品的 rpl16基因序列,包含了内含子和两翼外显子以及部分编码区,得到的长江中下游芦苇样品的Genbank登录号为:MK648345-MK648367,云贵高原芦苇样品的Genbank登录号为:MK648373-MK648395。样品的rpl16基因序列片段的G+C含量平均为31.65%,明显低于 A+T的平均含量:68.35%,这与一些文献中非编码区的A+T含量较高结果一致(李毳,2016;王宁等,1999)。经DNAsp 6.0检测,序列对齐后空白/缺失位点79个,保守位点1110个,变异位点4个,简约信息位点3个。通过序列比对发现,存在以碱基颠换为主的遗传变异类型,同时还有一些位点存在碱基的缺失和插入。如:与长江中下游所有芦苇样品相比,云贵高原的所有芦苇样品均在第293-299位点发生缺失突变,且在第413和第421位点发生了颠换突变;毛板桥水库和青山嘴水库的芦苇样品在第340个位点发生颠换突变;淀山湖的一株芦苇样品在第777位点插入碱基T。同一湖泊采集的淹水和非淹水生境中的芦苇样品的rpl16序列无差异,可见地理隔离是造成不同芦苇居群遗传差异的主要原因,因此在后续的分析中不再将水淹条件作为考虑指标之一。
2.2 聚类分析
序列比对后,利用MEGA 7软件基于rpl16基因序列重建了(MP)最简约系统发育树,并进行了不同地理区域芦苇居群间的遗传距离分析。
2.2.1 系统发育分析
序列比对后,利用MEGA 7基于rpl16基因序列重建最简约系统发育树(MP),分支上的数字表示自展支持率。由图2可以看出,长江中下游的样品被归类为一个置信度为91%的大支,与云贵高原的芦苇样品区分开,其中鄱阳湖和滆湖样品在支上的分布较分散,说明这两个居群的芦苇样品间具有较高的分散性,而太湖梅梁湾、升金湖、洪泽湖、高邮湖、淀山湖、长荡湖、巢湖和和石臼湖的芦苇样品均聚类在一起,说明这些居群样品间的分散度较小。云贵高原的芦苇样品中,毛板桥和青山嘴水库的芦苇样品被归类为一个置信度为 68%的单独亚支,结合序列碱基差异比对,推测与序列个别位点的突变有关。滇池、抚仙湖及阳宗海的每株芦苇样品各聚为一类的置信度达到 100%,且相同居群间样品的分布均较为分散,可能是这3个地方的芦苇进化时间较长,具有更高的遗传可塑性,相对来说比较独立所致,属于同一地理区域的毛板桥和青山嘴水库的芦苇虽然单独形成一个分支,但与其亲缘关系仍较近,长江中下游的芦苇样品与其亲缘关系较远,可能是因为不同的地理位置形成了不同的生存环境,使芦苇产生了基因水平的变异,并随着居群的繁衍保留,从而形成了不同的地理生态型。总的来说,不同地理区域之间的芦苇样品在进化时间和系统发育上存在一定差异,以地理区域为区分指标,可将长江中下游和云贵高原的芦苇居群分为两个单系。
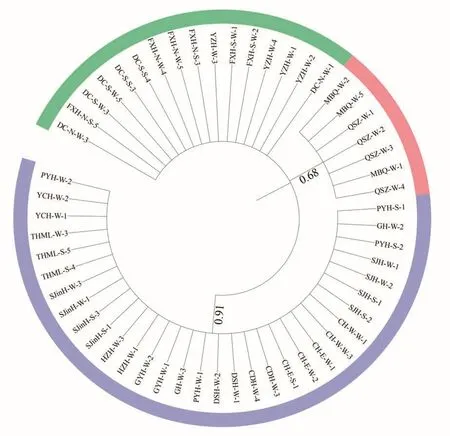
图2 基于rpl16 序列重建的最简约系统发育树Fig. 2 Maximum parsimony tree reconstruction based on rpl16 sequence
2.2.2 遗传距离分析
遗传距离是用来衡量物种间或同一物种不同群体间遗传分化的指标,可用来描述群体的遗传结构和居群间的差异,一般认为群体分化时间越短,遗传距离越小(黄椰林,2002;李欣,2008)。利用 MEGA 7计算本研究样品居群间的遗传距离。长江中下游各居群间以及云贵高原的滇池、抚仙湖、阳宗海两两之间的遗传距离较小,说明其有共同的祖先来源,且遗传跨度较小,不再在表2中列出。
由表2可以看出,19个湖滨带芦苇居群间的遗传距离在0.0009-0.0035之间,其中毛板桥居群与长江中下游各居群间的遗传差异最大,相对遗传距离为0.0034-0.0035;其次是青山嘴居群与长江中下游各居群间的相对遗传距离:0.0026-0.0027。云贵高原的滇池居群、抚仙湖居群、阳宗海居群和毛板桥居群与青山嘴居群之间的遗传差异最小,相对遗传距离为0.0009。除毛板桥居群和青山嘴居群外,云贵高原各居群与长江中下游各居群间的遗传距离均为 0.0017-0.0018。这些结果符合 Koppitz(1999)对全球芦苇研究得出的结论:地理距离增加,克隆间的遗传距离增加。本研究中毛板桥水库和淀山湖芦苇居群的地理距离最大,约为:2031 km。总的分析来说:同一地理生态型居群间的芦苇遗传跨度较小,没有明显的遗传距离差异,不同地理区域居群间存在一定的差异,但差异不大。Nei et al.(1979)通过研究得出:同一物种在地理居群间遗传距离变动在0-0.05之间,亚种间的遗传距离在 0.02-0.2之间,种间遗传距离变动在 0.013-0.052之间。因此,本研究中不同地理区域居群间芦苇的变异仍在亚种到种间的变化范围内。
2.3 遗传多样性与遗传结构分析
2.3.1 遗传多样性分析
物种的遗传多样性是指种内个体之间或一个群体内不同遗传变异的总和。大量理论和实验研究表明,生物居群中遗传变异的大小与其进化速率成正比,因此,遗传多样性可以揭示居群的进化历史(沈浩等,2001)。本研究利用DNAsp 6.0软件从居群的核苷酸多样性(Pi)和单倍型多态性(Hd)分析 19个湖滨带芦苇居群的遗传多样性。结果表明:19个湖滨带芦苇居群共计4种单倍型,除云贵高原毛板桥居群的一株芦苇单独为一单倍型外,其余单倍型分类与聚类结果一致,不同地理居群间无共享单倍型,这符合常见的种群分布规律,即:不同大小的多个种群分布在不同区域,分隔开来的种群会独立进化,突变产生新的单倍型,且新的单倍型在其他种群中不存在(徐刚标,2011)。Saltonstall(2003)、Lynch et al.(2002)研究得出:芦苇每个叶绿体单倍型代表一种母系遗传的谱系,可见不同的地理区域的芦苇已经形成了不同的基因型。居群总核苷酸多样性 Pi=0.00113,平均核苷酸差异数k=1.254,居群总单倍型多态性Hd=0.575,从不同地理居群来看,长江中下游居群核苷酸多样性和单倍型多态性均为 0,云贵高原居群核苷酸多样性Pi=0.00047,单倍型多态性Hd=0.466,遗传多样性水平较高,可能与其起源较早有关,积累了更多的变异,这一点与聚类分析结果一致。总的分析,不同地理区域的群体遗传多样性水平相差较大。
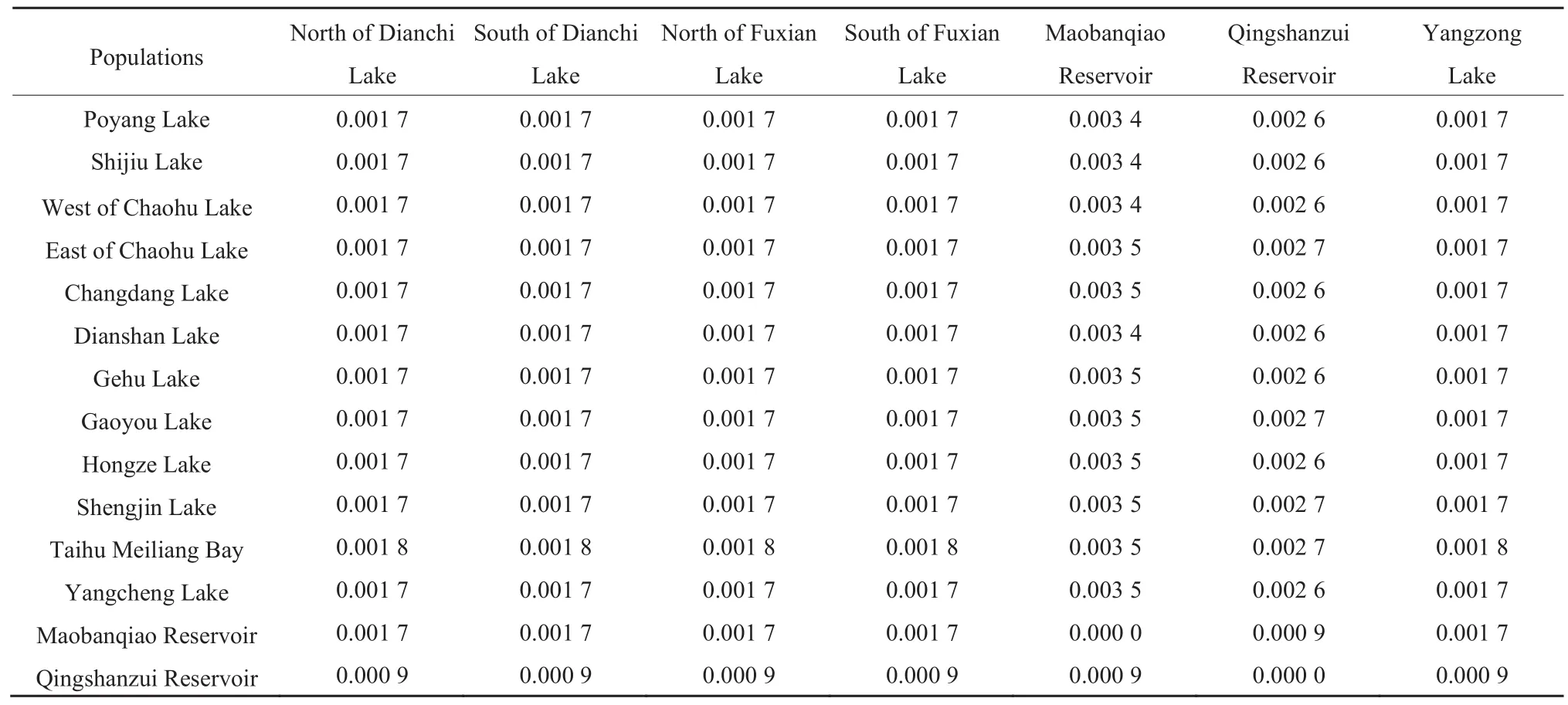
表2 不同芦苇居群间的遗传距离Table 2 Genetic distance between different Phragmites australis populations
2.3.2 遗传结构分析
物种的遗传结构是指居群间和居群内的遗传变异在空间和时间上的分布特征和格局,是研究植物适应性、物种形成过程及其进化机制的重要基础(何薇,2014)。将19个湖滨带芦苇居群根据地理位置分为长江中下游和云贵高原两类,进行分子方差分析(AMOVE),由表3可得:53.54%的遗传变异来自于不同地理居群间,46.46%的遗传变异来自于同一地理居群间,计算遗传差异指数Fst,居群总遗传差异指数Fst=0.53537,长江中下游芦苇居群的遗传差异指数Fst=0.53724,基因流Nm=0.21534,云贵高原芦苇居群的遗传差异指数Fst=0.53278,基因流 Nm=0.21923,两个地理区域居群间的基因流Nm均小于1,说明不同的地理格局会对种系的遗传分化产生一定的影响,不同地理种群之间存在一定分歧,根据Buso et al.(2010)的研究结果:Nm小于1,说明居群间的遗传分化较强烈。
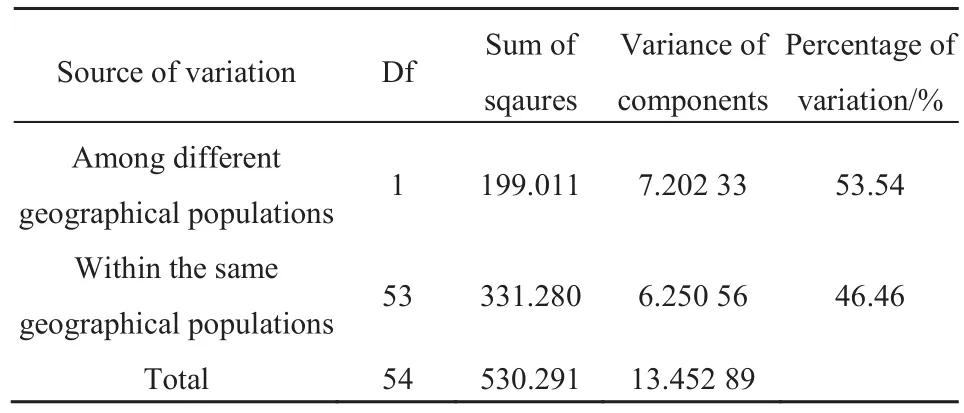
表3 不同芦苇居群间的分子方差分析(AMOVE)Table 3 Analysis of molecular variance (AMOVE) between different Phragmites australis populations
2.4 形态差异与遗传差异的异同
芦苇的形态作为其表型特征,具有一定的变异性和稳定性,受到来自基因和环境两方面的影响,由环境导致的形态变异称之为“可塑性”,而基因差异也会导致形态差异。在采集芦苇样品时记录了每株芦苇的基径、株高、最大叶长、最大叶宽、中部节长和节数作为表型数据,由于每一个湖泊采样点的芦苇分布都比较均匀,认为每个湖泊的芦苇所处的环境变量一致,故取每个湖泊芦苇样品表型数据的平均值来探讨形态差异与遗传差异的异同。表4列出了不同湖泊芦苇样品的表型数据平均值,并对不同湖泊间芦苇的形态变异程度使用SPSS软件进行单因素方差分析。
根据不同湖泊芦苇样品的表型数据平均值及单因素方差分析,不同湖泊芦苇在6个形态指标上差异显著,差异程度依次是:最大叶宽>最大叶长>节数>株高>基径>中部节长。最大叶宽的差异主要体现在洪泽湖、滇池和青山嘴水库的芦苇样品的最大叶宽明显小于其他湖泊,同时洪泽湖和滇池的芦苇样品的基径也明显小于其他湖泊;最大叶长的差异表现为阳澄湖和抚仙湖的最大叶长显著高于其他湖泊;淀山湖和太湖的芦苇样品的节数明显多于其他湖泊;淀山湖和毛板桥水库的芦苇样品株高较高;不同湖泊芦苇样品的中部节长也存在差异。总的来说,不同地理区域和同一地理区域的芦苇存在均一定的形态差异,结合遗传差异分析,本研究中芦苇的的形态变异有一定的遗传基础,且在形态变异上“可塑性”的影响更大,但仅根据形态差异来划分生态型具有片面性,“可塑性”和遗传基础对形态变异的贡献比率也有待进一步研究。
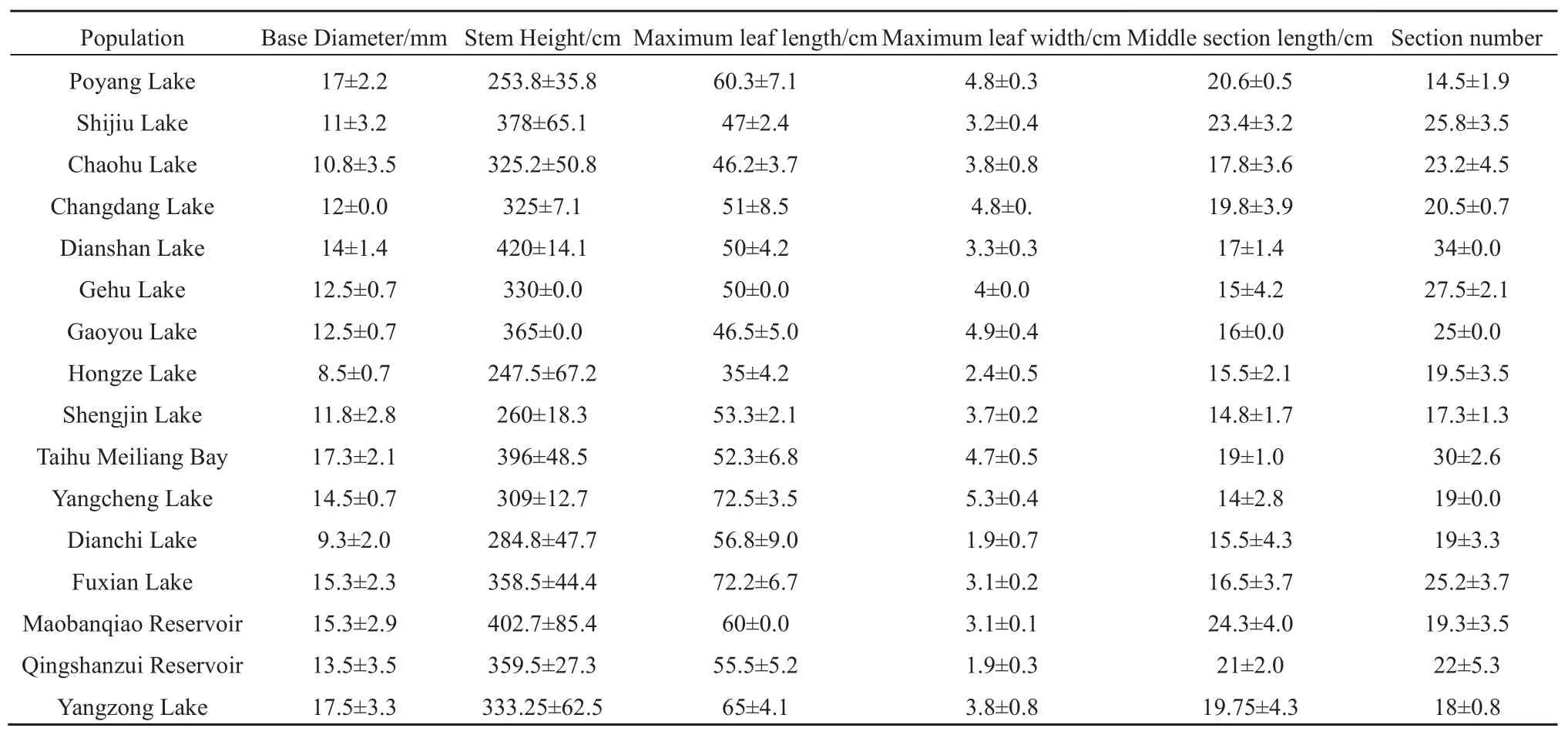
表4 不同湖泊芦苇样品的表型数据平均值Table 4 Mean value of phenotypic data of Phragmites australis samples collected from different lakes
种群间的遗传变异反映的是物种对不同生境的适应情况,种群的遗传多样性越高,其对不同生境的适应力越强,利用分子技术可以从微观角度探索不同生境中的芦苇遗传分化的程度及进化关系。龚晓洁(2009)利用rpl16等基因证实了河西走廊4种生态型芦苇的划分,并从遗传多样性角度分析了4种生态型芦苇的进化关系。何薇(2014)通过rpl16基因对中国各个省的樱桃居群进行了遗传多样性和遗传结构分析,讨论了不同省樱桃居群间的亲缘关系。张俪文等(2018)结合了遗传变异和生境盐度分析得出黄河三角洲滨海地区芦苇群体的遗传多样性与生境盐度具有正相关关系。邱天(2018)从芦苇的表型变异结合分子生态学的角度分析了小空间尺度上芦苇的遗传变异与表观遗传变异有显著相关性。本研究利用分子技术,选取地理区域为我国淡水湖泊较为集中的长江中下游和云贵高原,提取采集芦苇样品的总DNA,经PCR扩增后测序得到cpDNA非编码区的rpl16基因序列,并基于rpl16序列分析发现不同地理区域的芦苇居群在序列碱基、亲缘关系、遗传距离和遗传结构均存在一定差异,证明了两种地理生态型芦苇具有遗传上的差异,且地理隔离可能是引起差异的主要原因。
3 结论
(1)本研究得到的55个芦苇样品的rpl16基因序列,遗传变异类型主要是碱基的缺失和颠换,且长江中下游芦苇居群基因水平上的变异程度较云贵高原的低。
(2)芦苇居群的亲缘关系和地理距离成负相关,长江中下游和云贵高原的芦苇居群在发育树上形成不同分支,且滇池、抚仙湖及阳宗海的芦苇是较为古老的居群,居群间的遗传距离在 0.0017-0.0035之间,和地理距离成正相关。
(3)不同地理区间的群落遗传多样性水平差异明显,云贵高原居芦苇居群的核苷酸多样性Pi=0.00047,单倍型多态性Hd=0.466,显著高于长江中下游Pi=0,Hd=0。分子方差分析(AMOVE)显示:53.54%的遗传变异来自于不同地理居群间,高于同一地理居群间的遗传变异46.46%。两个地理区域居群间的基因流Nm均小于1,居群间的遗传分化较强烈。
(4)不同湖泊芦苇在形态指标上差异显著,差异程度依次是:最大叶宽>最大叶长>节数>株高>基径>中部节长,结合遗传差异分析本研究中芦苇的的形态变异有一定的遗传基础,且在形态变异上“可塑性”的影响更大,但仅根据形态差异来划分生态型具有片面性,“可塑性”和遗传基础对形态变异的贡献比率也有待进一步研究。
