大白菜ENT基因家族的鉴定与生物信息学分析
2019-08-21庞文玉王安杨宝谊
庞文玉 王安 杨宝谊


摘要:核苷转运蛋白(nucleoside transporter,ENT)是一类具有跨膜运输核苷和核苷类似物的功能蛋白。为深入研究大白菜ENT基因家族的功能,利用生物信息学方法对大白菜基因组中的ENT基因家族成员进行鉴定,并对其基因组信息、蛋白生理生化特征、基因结构、保守结构域、系统进化树等方面进行了研究。结果表明,在大白菜基因组中共鉴定到12个ENT基因,通过系统发育树分析,可以将12个ENT基因分为2类;对大白菜ENT蛋白氨基酸序列多重比对和蛋白跨膜域的分析表明,BrENT蛋白存在11个跨膜域,为跨膜蛋白;通过MEME软件对大白菜ENT蛋白motif的预测还发现了14个比较保守的motif。染色体分析表明,ENT基因在大白菜10条染色体上不均匀分布,而且存在明显的串联重复现象,推测串联复制是ENT基因家族基因扩增的主要方式,研究结果为在基因组范围内研究ENT基因的功能奠定了基础。
关键词:大白菜;核苷转运蛋白;基因家族;生物信息学
中图分类号: S634.101 文献标志码: A 文章编号:1002-1302(2019)12-0052-05
核苷转运蛋白是一类具有跨膜运输核苷和核苷类似物的功能蛋白,被转运的核苷在核苷酸合成代谢的补救途径中发挥重要作用。核苷转运蛋白可分为扩散型的核苷转运蛋白(equilibrative nucleoside transporter,ENT)和富集型的核苷转运蛋白(concentrative nucleoside transported,CNT)2种类型。其中ENT在生物体内的分布非常广泛,ENT家族被克隆的首个成员是hENT1,其cDNA是1996年从人胎盘中克隆得到的[1]。目前,在哺乳动物、细菌、酵母、寄生虫和植物中均发现并鉴定到了ENT[2]。由于核苷转运蛋白与癌症、神经和心血管病的治疗,寄生虫病的防治以及抗病毒、人体保健等方面密切相关,因此在动物中得到深入而广泛的研究[3-4]。
植物核苷轉运蛋白的研究还仅限于模式植物拟南芥和水稻中。李剑首先从拟南芥幼苗中克隆到核苷转运蛋白基因AtENT1,并对其表达模式进行了研究[5]。结果表明AtENT1在拟南芥根、茎、叶、花序轴和花等器官中均有较强表达,且生殖生长阶段表达水平高于营养生长阶段[5]。随后对核苷转运功能的研究表明,AtENT1具有转运腺苷和尿苷的能力,首次证实了植物中核苷转运蛋白的转运功能[6]。Li等又报道了AtENT3的转运活性,并对AtENT1、AtENT2、AtENT3、AtENT4、AtENT6、AtENT7和AtENT8进行了研究与分析[7]。Wormit等利用酵母细胞研究了AtENT4、AtENT6、AtENT7和AtENT8的生化功能,发现AtENT4、AtENT6和AtENT7具有广泛的底物选择性,既能转运嘌呤类核苷,也能转运嘧啶类核苷[8]。对水稻核苷转运蛋白的研究则表明OsENT2也具有核苷转运蛋白的功能[9]。另有研究表明,植物核苷转运蛋白还具有转运细胞分裂素的能力[9-12],这是继生长素转运蛋白PIN家族后发现的第2类植物激素转运蛋白。对植物核苷转运蛋白基因表达模式的研究表明,核苷转运蛋白基因呈现广谱表达性[9-11,13-15],但是关于植物核苷转运蛋白生物学功能的研究依然比较匮乏,仅有报道表明拟南芥AtENT1通过核苷酸代谢途径影响花粉萌发与营养生长过程[13]。
核苷酸的挽救途径是植物适应外界环境的一条重要代谢途径,在植物体内广泛存在,并参与了多种生理过程,如胚成熟和萌发,体外器官发生,打破休眠,果实成熟,贮藏器官发育与发芽,叶片衰老,植物组织培养中细胞的分裂、分化和调节以及植物对外界环境的反映等,对植物的生长发育具有重要意义[16]。因此,通过对植物ENT的研究,可为改善作物抗逆性、提高营养利用效率和促进养分的合理分配提供参考依据。
大白菜是我国北方乃至东南亚地区的重要蔬菜作物,南北各地均有栽培,也是我国种植面积最大的蔬菜作物。目前关于大白菜ENT(BrENT)的研究几乎是一片空白。大白菜(栽培品种Chiifu-401-42)全基因组测序的完成使得对大白菜中相关基因家族的鉴定和功能分析成为可能。对BrENTs的研究有助于深入认识BrENTs家族成员,了解BrENTs在种子萌发、叶片衰老、营养物质分配和再利用以及抗逆性等过程中的作用,因此,开展BrENTs的研究具有重要意义。本研究利用生物信息学方法对大白菜ENT基因家族成员进行了鉴定和基因组注释,并分析了其基因结构和保守域,以期为进一步研究BrENTs功能奠定基础。
1 材料与方法
1.1 大白菜ENT基因家族成员的鉴定
首先,利用拟南芥ENT蛋白的氨基酸序列在大白菜数据库(http://brassicadb.org/brad/)中进行Blast P比对筛选候选蛋白。利用搜索到的ENT蛋白的氨基酸序列对大白菜基因组数据库进行二次Blast P比对搜索。最后,利用Pfam数据库、SMART数据库和NCBI的保守域数据库分析候选蛋白的保守域以验证ENT蛋白鉴定的准确性。
1.2 大白菜ENT基因的基因组信息和染色体定位
通过大白菜基因组数据库获得ENT基因的序列和基因组注释信息,并根据ENT基因在染色体上的位置和染色体的长度将ENT基因定位到对应染色体上。
1.3 大白菜ENT蛋白的生理生化分析
通过Compute pI/Mwsoftware (http://www.expasy.ch/ tools/pi_tool.html)预测ENT蛋白的分子量和等电点[17]。通过TargetP软件(http://www.cbs.dtu.dk/services/TargetP)进行亚细胞定位分析[18]。
1.4 大白菜ENT基因的基因结构、保守域和系统进化树分析
GSDS网站(http://gsds1.cbi.pku.edu.cn/)[19]用来绘制ENT基因的外显子-内含子结构图。DNAman软件用于对ENT蛋白氨基酸序列进行多重比对和跨膜域的预测。MEME网站(http://meme.sdsc.edu/meme/meme.html)[20]用来预测ENT蛋白氨基酸序列中的保守motif。利用MEGA 5.0软件,使用邻接法(neighbor-joining method)构建系统进化树,采用泊松校正,成对删除和1 000次重复等建树参数[21]。
2 结果与分析
2.1 大白菜ENT基因家族的鉴定和注释
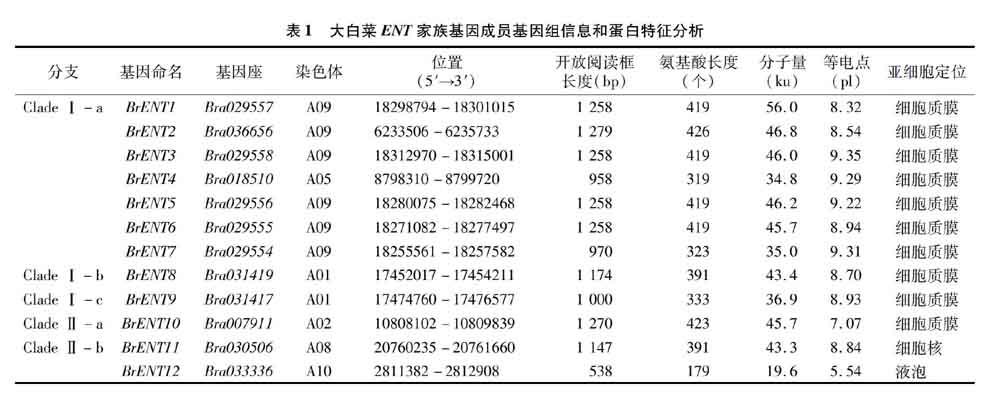
根据拟南芥ENT蛋白的氨基酸序列,对大白菜数据库进行Blast P搜索,最终在大白菜基因组中鉴定到12个ENT基因家族成员。如表1所示,这12个ENT基因长度在538~1 279 bp,编码179~426个氨基酸,蛋白分子量在19.6~56.0 ku 之间。其中,BrENT2的开放阅读框和编码的氨基酸序列长度最长,BrENT12的开放阅读框和编码的氨基酸序列长度最短。等电点(pl)在5.54~9.35之间,BrENT12等电点最低,BrENT3等电点最高。根据TargetP软件对ENT蛋白亚细胞定位的预测表明除BrENT11和BrENT12分别定位在细胞核和液泡中,其余绝大多数ENT蛋白定位在细胞质膜上(BrENT1~BrENT10)。
对大白菜ENT基因染色体定位(图1)的研究表明,ENT家族成员在大白菜的10条染色体上呈现明显的不均匀分布,其中9号染色体上有6个ENT基因,1号染色体上有2个ENT基因,2、5、8和10号染色体上均有1个ENT基因,在其他染色体上则没有分布。另外,ENT基因存在明显的串联重复现象,并在染色体上呈现2个串联重复基因簇,其中BrENT8和BrENT9为一对串联重复基因,在1号染色体上形成一个串联重复基因簇,BrENT1、BrENT3、BrENT5、BrENT6和BrENT7则在9号染色体上形成一个较大的串联重复基因簇。
2.2 大白菜ENT基因结构分析
为明确大白菜ENT基因结构,根据大白菜基因组信息获取了每一个ENT基因的基因组序列和CDS序列信息并绘制外显子-内含子结构图(图2),结果表明大白菜ENT基因含有1~7个数量不等的内含子,其中BrENT10和BrENT11均含有1个内含子,BrENT12含有2个内含子,BrENT4含有4个内含子,BrENT7、BrENT8、BrENT9含有6个内含子,而BrENT1、BrENT2、BrENT3、BrENT5、BrENT6则含有7个内含子。另外,通过对基因结构图和基因进化树的比较分析发现基因的亲缘关系越近,其基因结构和内含子数量也越相似。
2.3 大白菜ENT蛋白的氨基酸序列比对和保守域分析
为研究大白菜ENT蛋白的保守结构域,使用DNAman软件对大白菜ENT蛋白的氨基酸序列进行多重比对(图3)。结果表明大白菜ENT蛋白的氨基酸序列比较保守,并含有ENT蛋白家族典型的11个跨膜域(图4)[7]。推测大白菜ENT蛋白是跨膜蛋白,这与上述对大白菜ENT蛋白在细胞质膜上的亚细胞定位结果一致。另外,利用MEME软件对大白菜ENT蛋白保守motif的预测分析,鉴定到了14个比较保守的motif(motif 1~14)(图5)。
2.4 大白菜ENT基因的系统发育分析
为分析大白菜ENT基因的亲缘关系和进化模式,使用MEGA 5.0软件对选取的大白菜及其近缘模式植物拟南芥ENT蛋白的氨基酸序列构建了NJ进化树。结果表明,大白菜和拟南芥ENT基因可以被聚类成兩大分支,Clade Ⅰ和Clade Ⅱ。Clade Ⅰ包含9个BrENT基因,又分为4个亚分支,其中Clade Ⅰ-a是比较大的亚分支,包含BrENT1~BrENT7 7个基因,Clade Ⅰ-b和Clade Ⅰ-c分别含有1个BrENT基因,而Clade Ⅰ-d只包含1个拟南芥ENT基因AtENT2。Clade Ⅱ包含3个BrENT基因,可以进一步分成CladeⅡ-a和Clade Ⅱ-b 2个分支,其中BrENT10位于Clade Ⅱ-a,而BrENT11和BrENT12位于Clade Ⅱ-b(图6)。
3 结论与讨论
本研究根据拟南芥ENT蛋白的氨基酸序列,通过对大白菜数据库进行Blast P搜索,最终在大白菜基因组中鉴定到12个ENT基因家族成员,并对其家族成员进行基因和蛋白特征分析、基因结构分析、保守域分析以及系统发育分析,为后期大白菜ENT基因功能研究提供了基础。
基因扩增是基因组进化最主要的驱动力之一,是产生具有新功能的基因和进化出新物种的主要分子机制之一。基因可以通过多种方式进行扩增,包括全基因组复制、串连复制、片段复制和逆转座复制等。本研究鉴定到12个大白菜ENT基因,而拟南芥基因组中共有8个ENT基因,推测大白菜ENT基因家族出现了基因扩增现象。值得注意的是在这12个大白菜ENT基因中有8个基因分布在2个串联重复基因簇中,以串联重复的形式存在,表明串联复制是ENT基因家族基因扩增的主要方式。BrENT8和BrENT9是一对串联重复基因,其中BrENT9是AtENT7的同源基因,而在拟南芥中没有发现BrENT8的同源基因,推测BrENT9以串连复制的形式发生后,在进化过程中又发生了比较明显的变异,但是其基因分化机制及具体的基因功能有待深入研究。
大白菜和拟南芥分化之前出现过3次全基因组复制事件(α、β、γ复制事件),分化发生之后大白菜基因组又发生了全基因组三倍化事件[22]。由于基因组加倍后,会伴随着发生染色体重排,基因丢失等现象。大白菜ENT基因家族成员数量只有拟南芥的1.5倍,说明伴随着大白菜基因组三倍化事件也产生了大量的基因丢失事件,尤其是在大白菜基因组中没有鉴定到AtENT2的同源基因,其基因丢失具体的发生机制仍有待进一步研究。
參考文献:
[1]Griffiths M,Yao S Y,Abidi F,et al. Molecular cloning and characterization of a nitrobenzylthioinosine-insensitive (ei) equilibrative nucleoside transporter from human placenta[J]. Biochemical Journal,1997,328(3):739-743.
[2]Boswellcasteel R C,Johnson J M,Roeur Z,et al. Expression and purification of human and Saccharomyces cerevisiae equilibrative nucleoside transporters[J]. Protein Expression and Purification,2017,142:68-74.
[3]Huang W Y,Zeng X,Shi Y G,et al. Functional characterization of human equilibrative nucleoside transporter 1[J]. Protein and Cell,2017,8(4):284-295.
[4]Rebba C. Boswell-casteel and franklin a.Hays.equilibrative nucleoside transporters-a review[J]. Nucleosides,Nucleotides and Nucleic Acids,2017,36(1):7-30.
[5]李 剑. 拟南芥核苷转运蛋白AtENT1基因的克隆、表达模式及功能的初步分析[D]. 北京:中国科学院遗传与发育生物学研究所,2001.
[6]Mohlmann T,Mezher Z,Schwerdtfeger G,et al. Characterisation of a concentrative type of adenosine transporter from Arabidopsis thaliana (ENT1,At)[J]. FEBS Letters,2001,509(3):370-374.
[7]Li G Y,Liu K F,Baldwin S A,et al. Equilibrative nucleoside transporters of Arabidopsis thaliana - cDNA cloning,expression pattern,and analysis of transport activities[J]. The Journal of Biological Chemistry,2003,278(37):35732-35742.
[8]Wormit A,Traub M,Florchinger M,et al. Characterization of three novel members of the Arahidopsis thaliana equilibrative nucleoside transporter (ENT) family[J]. Biochemical Journal,2004,383(1):19-26.
[9]Hirose N,Makita N,Yamaya T,et al. Functional characterization and expression analysis of a gene,OsENT2,encoding an equilibrative nucleoside transporter in rice suggest a function in cytokinin transport[J]. Plant Physiology,2005,138(1):196-206.
[10]Sun J,Hirose N,Wang X,et al. Arabidopsis SOI33/AtENT8 gene encodes a putative equilibrative nucleoside transporter that is involved in cytokinin transport in planta[J]. Journal of Integrative Plant Biology,2005,47(5):588-603.
[11]Hirose N,Takei K,Kuroha T,et al. Regulation of cytokinin biosynthesis,compartmentalization and translocation[J]. Journal of Experimental Botany,2008,59(1):75-83.
[12]Kudo T,Kiba T,Sakakibara H. Metabolism and long-distance translocation of cytokinins[J]. Journal of Integrative Plant Biology,2010,52(1):53-60.
[13]Bernard C,Traub M,Kunz H H,et al. Equilibrative nucleoside transporter 1 (ENT1) is critical for pollen germination and vegetative growth in Arabidopsis[J]. Journal of Experimental Botany,2011,62(13):4627-4637.
[14]Cornelius S,Traub M,Bernard C,et al. Nucleoside transport across the plasma membrane mediated by equilibrative nucleoside transporter 3 influences metabolism of Arabidopsis seedlings[J]. Plant Biology,2012,14(5):696-705.
[15]Chen K L,Xu M X,Li G Y,et al. Identification of AtENT3 as the main transporter for uridine uptake in Arabidopsis roots[J]. Cell Research,2006,16(4):377-388.
[16]Girke C,Arutyunova E,Syed M,et al. High yield expression and purification of equilibrative nucleoside transporter 7(ENT7)from Arabidopsis thaliana[J]. Biochimica et Biophysica Acta,2015,1850(9):1921-1929.
[17]Gasteiger E,Gattiker A,Hoogland C,et al. ExPASy:the proteomics server for in-depth protein knowledge and analysis[J]. Nucleic Acids Research,2003,31(13):3784-3788.
[18]Emanuelsson O,Nielsen H,Brunak S,et al. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J]. Journal of Molecular Biology,2000,300(4):1005-1016.
[19]Hu B,Jin J P,Guo A Y,et al. GSDS 2.0:an upgraded gene feature visualization server[J]. Bioinformatics,2015,31(8):1296-1297.
[20]Bailey T L,Williams N,Misleh C,et al. MEME:discovering and analyzing DNA and protein sequence motifs[J]. Nucleic Acids Research,2006,34(Web Server issue):369-373.
[21]Saitou N,Nei M. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution,1987,4(4):406-425.
[22]Lysak M A,Koch M A,Pecinka A,et al. Chromosome triplication found across the tribe Brassiceae[J]. Genome Research,2005,15(4):516-525.魏 磊,常 霞,全彥涛,等. 五加科植物鲨烯合酶核苷酸及其编码氨基酸序列的生物信息学分析[J]. 江苏农业科学,2019,47(12):57-62.
