马铃薯红色薯肉调控基因的精细定位与候选基因分析
2019-08-13许芸梅李玉梅贾玉鑫张春芝李灿辉黄三文祝光涛
许芸梅,李玉梅,3,贾玉鑫,张春芝,李灿辉,黄三文,祝光涛
马铃薯红色薯肉调控基因的精细定位与候选基因分析
许芸梅1,李玉梅1,3,贾玉鑫2,张春芝2,李灿辉1,黄三文2,祝光涛1
(1云南师范大学生命科学学院/马铃薯科学研究院,昆明 650500;2中国农业科学院深圳农业基因组研究所,深圳 518120;3昆明理工大学信息工程与自动化学院,昆明 650500)
【】薯肉颜色是马铃薯重要的农艺性状,它直接影响马铃薯的营养和商品价值,一直是马铃薯遗传研究和育种改良的重要目标。本研究通过对二倍体红色薯肉分离群体的混池分析、基因精细定位和候选基因表达分析,确定调控红色薯肉的候选基因,为下一步基因功能、遗传调控研究及彩色马铃薯的分子育种奠定基础。【】本研究通过向二倍体红色薯肉亲本导入自交不亲和抑制基因获得BC1S1群体,从300个单株中挑选18株红色薯肉和21株黄色薯肉个体提取基因组DNA,分别测序进行混池分析。通过集团分离分析法(bulked segregation analysis,BSA)对基因进行初步定位;在定位区间内开发分子标记,对796份BC1S1植株进行基因型分析,筛选交换单株,并结合表型对基因进行精细定位;借助参考基因组注释信息和qRT-PCR表达量分析确定候选基因。【】本研究通过构建薯肉颜色分离的二倍体BC1S1群体,利用BSA-seq分析把调控薯肉花青素合成的主效位点定位在第10号染色体48.70—52.20 Mb。最终,利用分子标记将该基因定位于51.47—51.85 Mb的377 kb区间内。基于参考基因组注释信息,此区间包括5个基因,其中2个基因注释为MYB类转录因子,结合表达量数据推测这2个基因为候选基因,编号分别为PGSC0003DMG400013966、PGSC0003DMG400013965。【】本研究将调控马铃薯薯肉花青素积累的一个主效位点定位于第10号染色体51.47—51.85 Mb之间,推测PGSC0003DMG400013966和PGSC0003DMG400013965为候选基因。
红色薯肉;花青素代谢;BSA-seq;精细定位;候选基因
0 引言
【研究意义】马铃薯是全球第一大块茎类粮食作物,也是继水稻、小麦、玉米后的第四大重要粮食作物[1]。薯肉颜色是重要的商品性状,直接影响着消费者的选择倾向。花青素赋予马铃薯丰富多彩的颜色,是影响薯肉呈色的关键色素之一。同时,花青素具有较强的抗氧化能力,具有重要的营养价值,因此,彩色马铃薯选育是一重要的育种方向。植物中主要含有六种花青素:天竺葵色素Pelargonidin(Pg)、矢车菊色素Cyanidin(Cy)、飞燕草色素Delphinidin(Dp)、芍药色素Peonidin(Pn)、矮牵牛色素Petunidin(Pt)和锦葵色素Malvidin(Mv)[2]。红色马铃薯块茎中主要含有天竺葵色素、矢车菊色素和芍药花色素等以红色为基础色调的花青素[3]。尽管在许多植物中花青素的合成代谢途径已得到较为详细的研究,在马铃薯中重要的合成基因也已被克隆[4-6],但是调控薯肉花青素积累的基因还没有明确的报道。本研究旨在定位调控薯肉花青素积累的关键基因,为进一步关键基因的克隆,薯肉花青素合成调控机制的解析和彩色马铃薯的育种提供理论基础。【前人研究进展】花青素存在于植物各个组织中,基因家族广泛地参与了调控花青素积累的过程。JIANG等[7]研究发现,花瓣中央产生色斑的山字草与花瓣基部产生色斑的山字草杂交后,后代在两个部位均产生色斑,存在分离。研究发现基因的两个等位基因编码的蛋白存在6个氨基酸的差异,并指出该等位基因的结构差异是造成色素积累部位差异的原因。HUANG等[8]发现,天然紫色柚子中存在一个调控花青素合成的基因(),在其附近发现基因,该基因截短54 bp破坏了R2结构域,CgRuby2是的竞争者,通过与同一个bHLH竞争结合位点来抑制花青素的合成。在马铃薯中,前人最早用四倍体鉴定了3个控制薯皮颜色的基因位点:和,已分别定位于2、11和10号染色体。花青素的生物合成过程由结构基因和调控基因共同作用。结构基因(red)和(purple)分别编码二氢黄酮醇-4-还原酶(DFR)和类黄酮-3’5’-羟化酶(F3’5’H),控制天竺葵素和矮牵牛色素的合成[4-5],使薯皮分别呈现红色和紫色,并且位点对于位点具有上位效应[9-10]。调控基因(developer)控制花青素在薯皮中的特异积累[11],在二倍体中对应基因(inhibitor)。基因编码一个R2R3-MYB转录因子,调控薯皮中基因和的表达,使得花青素在薯皮中积累。早期的研究认为[12],(pigmented flesh)基因控制薯肉花青素合成,其与基因紧密连锁并受到基因的调控。近些年的研究也陆续发现了一些对于薯肉花青素积累具有一定作用的基因和位点[13-14]。【本研究切入点】控制薯肉中的花青素合成的开关基因尚不明确,有待进一步研究。【拟解决的关键问题】以薯肉颜色分离(红/黄)的二倍体BC1S1群体为材料,利用BSA-seq和分子标记筛选的方法对红色薯肉颜色调控基因进行精细定位,借助参考基因组信息与实时荧光定量PCR分析,预测候选基因。
1 材料与方法
1.1 材料
以自交不亲和的红色薯肉二倍体材料CIP 001(P1)为母本,含有自交不亲和抑制基因的白色薯肉二倍体材料E-172(P2)为父本杂交获得F1群体,从F1群体中选择8个植株花粉混合与P1回交获得BC1群体,从此群体中选择一株红色薯肉植株自交获得二倍体BC1S1群体。BC1S1群体薯肉颜色表现为红色与黄色的分离。
1.2 田间试验与表型调查
2016年夏季以CIP 001(P1)作母本,E-172(P2)作父本杂交获得F1群体。2016年冬季以F1群体中8个植株的花粉混合授到P1柱头上回交产生BC1群体。2017年夏季从BC1群体中选取一株红色薯肉植株自交得到BC1S1群体。2017年冬季播种BC1S1群体的种子获得300株幼苗,移栽至花盆中。2018年春季收获薯块并调查薯肉颜色。2018年夏季播种1 000粒BC1S1群体的种子,共定植796株,待薯块充分成熟至冬季收获,对所有单株进行表型鉴定和交换单株基因型分析。薯肉颜色调查方法如下:选取每个单株中较大的薯块,切薯块横切面,判断从周皮往里至内髓区域,若出现红色花青素积累的特征,则归类于红色薯肉,无花青素积累则归类于黄色薯肉(黄色薯肉由类胡萝卜素物质呈色,无花青素积累的薯肉均定义为黄色薯肉)。试验材料种植在云南师范大学马铃薯试验基地。
1.3 BSA-seq分析
首先提取BC1S1群体的亲本(BC1)和BC1S1群体中39个单株(18个红色薯肉单株和21个黄色薯肉单株)的基因组DNA。基因组DNA提取参照刘秀丽等[15]的方法,有所改进。植物基因组DNA的提取借助12个八连排管与U型细胞培养板,实现高通量提取。使用Illumina HiSeqTM2000/MiseqTMPE150测序平台,分别对每个个体进行测序。亲本(BC1)和BC1S1群体的单株分别进行20×和2×深度全基因组重测序。得到测序数据首先区分亲本的两套单倍型,具体为:将亲本重测序的reads比对到DM(v4.03)参考基因组[16]上,提取亲本中杂合的SNP位点构成亲本杂合SNP集,再将后代重测序的reads比对到参考基因组并提取后代每个单株在亲本杂合SNP位置的基因型,得到自交后代纯合的区域,利用个体间纯合区域进行延伸,最终获得亲本的两套单倍型。按照表型将自交后代单株的测序结果混作两个极端池。以亲本的一套染色体基因型作为参照,计算后代两个极端池中目标表型亲本的基因型频率,具体为:使用bwa软件将每个个体比对到DM参考基因组上,然后使用samtools sort,samtools rmdup对比将得到的bam文件排序去重,再使用samtools mpileup和bcftools call命令提取变异位点,使用1 Mb为计算单位、100 kb滑动窗口分别计算两池的SNP-index,并计算两者的差值Δ(SNP-index)。利用R语言对SNP-index和Δ(SNP-index)进行绘图,图形中每个点都代表一个窗口,横坐标是滑动窗口在染色体上的物理位置,纵坐标是SNP对应的SNP-index和Δ(SNP-index)。取全基因组Δ(SNP-index)最高1%窗口中的最小限值作为阈值,对应的基因组区域作为候选区域进行后续分析。
1.4 Indel标记开发
亲本重测序的reads比对到参考基因组上得到bam文件,使用Platypus.py(v0.7.4)对bam文件进行Indel calling,提取杂合Indel(0.3 ≤ Indel-index ≤ 0.7)。依据参考基因组将杂合Indel的位置前后各延伸150 bp,提取序列,使用DesignPrimer对候选基因区域的Indel进行引物设计。
1.5 精细定位
在主效基因位点区域内筛选合适的Indel引物,引物筛选原则如下:扩增产物长度90—250 bp,GC含量40%—60%,退火温度55—60℃,插入缺失片段5—15 bp,引物送至昆明硕擎生物科技有限公司合成。以11个单株基因组DNA(1个亲本、5个黄色薯肉、5个红色薯肉)对引物进行多态性验证,扩增产物通过聚丙烯酰胺凝胶电泳确定带型,判断引物是否可用。所用引物见表1。使用引物对BC1S1群体进行初步筛选得到交换单株,结合基因型与表型进行初步定位。在交换单株的重组区域内进一步增加Indel标记,缩小定位区间,结合基因型与表型进行精细定位。
1.6 候选基因预测
在精细定位区间,根据马铃薯参考基因组的注释信息,并结合花青素代谢途径相关基因,预测控制薯肉花青素合成的候选基因。提取亲本(BC1)薯肉RNA并反转录为cDNA,PCR扩增候选基因的CDS序列,并推导氨基酸序列,获得亲本(BC1)中候选基因的蛋白序列。所用引物见表2。
1.7 候选基因表达量分析
从DM参考基因组中提取候选基因的CDS序列。使用Primer Premier 5,在CDS序列中跨内含子设计qRT-PCR引物。设计引物原则如下:引物长度20—35 bp(不超过38 bp),扩增产物长度100—140 bp,GC含量45%—55%,退火温度58—60℃,上、下游引物的退火温度差值不超过1℃,所用引物见表3。引物经普通PCR验证特异性后使用。内参基因为,基因编号为PGSC0003DMG400018449。
从BC1S1群体中选取3株红色薯肉与3株黄色薯肉的植株,薯块膨大至开始积累色素的时期,取薯肉组织,使用天根多糖多酚植物总RNA提取试剂盒(DP441)分别提取RNA,用TaKaRa PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒(RR047A)对RNA进行反转录,得到cDNA。使用TaKaRa TB GreenTMTMII试剂盒(RR820A)和Applied Biosystems StepOnePlus Real-Time PCR System仪器,对候选基因进行表达量分析。

表2 候选基因验证所用引物

表3 RT-PCR所用引物
2 结果
2.1 BC1S1分离群体薯肉颜色表型鉴定
BC1S1群体共种植796个单株,收获薯块743份,其中红色薯肉454株,黄色薯肉289株,不正常生长、未结薯植株共53株,如图1-A。
2.2 控制薯肉花青素积累的BSA-seq
BSA-seq分析结果如图1-B,发现在第10号染色体上的48.70—52.20 Mb,Δ(SNP-index)大于阈值0.4,区间出现了显著的信号位点,图中箭头指示处为目标区域,整个基因组只有此处一个信号位点,因此将该群体中控制薯肉花青素积累的主效基因位点定位于第10号染色体的48.70—52.20 Mb之间。
2.3 主效基因位点的精细定位
在48.70—52.20 Mb之间,基于BC1的基因组数据分析共得到1 406对Indel标记。根据引物位置选择5对分布较均匀的标记,对BC1S1群体796个单株进行分子标记筛选,在此区间共得到交换单株104株。结合基因型与表型分析发现,在P4与P5之间基因型与表型存在连锁关系,如图2。在P4与P5分子标记之间进一步设计Indel标记,对104株交换单株进行筛选,确定其基因型,结合薯肉表型确定了4株关键交换单株。154号单株的薯肉呈现黄色,Indel标记从P9处为h带型,之后为a带型,另外172号单株的薯肉呈现红色,Indel标记在P9位置为a带型,之后为h带型。以上两个单株的基因型和表型说明候选基因在P9下游;732号与768号单株的薯肉呈现红色,Indel标记从P15处为a带型,之前为h带型,说明候选基因在P15上游。根据4株关键交换单株的重组信息,将区间定位在标记P9—P15之间,物理位置为51 473 988—51 850 970 bp,距离377 kb,其中包括5个基因。
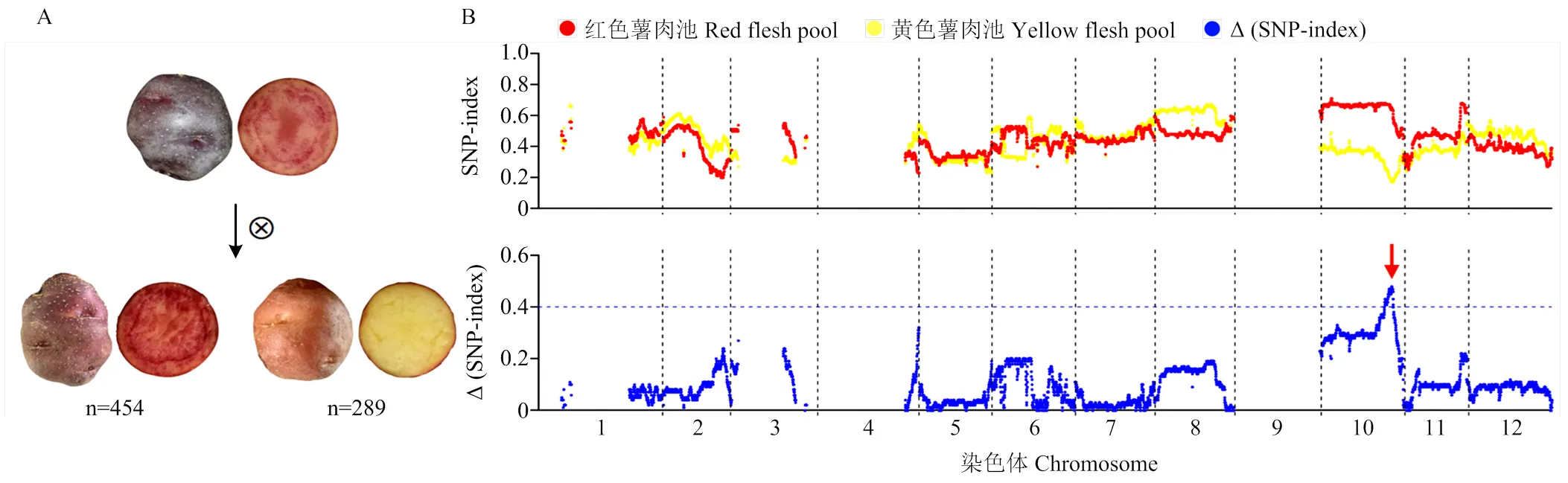
A为BC1S1群体的亲本(BC1)和BC1S1群体典型个体的表型,n表示对应表型的个体数目;B上方为红色薯肉池和黄色薯肉池的SNP-index,下方为Δ(SNP-index)结果,横坐标为染色体的物理位置,纵坐标为SNP对应的SNP-index和Δ(SNP-index)值,红色箭头所指为目标区域

图2 调控薯肉花青素合成主效基因的精细定位
2.4 候选基因的预测分析
结合参考基因组DM的注释信息,在377 kb候选区间内共有5个基因,基因相对位置如图2。其中包含3个未知功能基因和2个MYB类转录因子,如表4。其中PGSC0003DMG400013966注释为MYB转录因子,PGSC0003DMG400013965注释为R2R3-MYB转录因子。前人许多研究表明此类基因功能较为保守,是调控花青素合成的关键基因[17-21]。前人在马铃薯薯块颜色的研究中,证明基因控制薯皮花青素的积累,其编码R2R3-MYB转录因子[11]。为了明确本研究中定位的基因和前人克隆的是否相同,本研究扩增了亲本(BC1)PGSC0003DMG400013965的cDNA序列,比对发现此基因的氨基酸序列和已经发表的()的氨基酸序列完全一致,表明本材料中的PGSC0003DMG400013965就是已经克隆的基因,负责调控薯皮中花青素的积累。

表4 候选区间的基因信息
为了从表达水平对候选基因进行验证,分别提取红色薯肉与黄色薯肉的RNA并反转录为cDNA,采用实时荧光定量PCR检测两个在不同材料薯肉中的表达水平。结果显示(图3),PGSC0003DMG400013966在两份材料薯肉中均表达,在红肉中的表达量显著高于黄肉,暗示PGSC0003DMG400013966参与薯肉花青素积累过程;PGSC0003DMG400013965的表达水平与薯肉颜色也存在明显的相关性,在黄肉中表达量非常低,但在红色材料中PGSC0003DMG400013965的表达水平非常高,且显著高于黄色材料。在前人的研究中,过表达PGSC0003DMG400013965在薯肉中也出现了花青素的积累[11],结合本研究结果,推测PGSC0003DMG400013965或以某种尚不明确的方法参与到薯肉花青素的积累调控。因此,本研究推测此分离群体中PGSC0003DMG400013966与PGSC0003DMG400013965(基因)均调控薯肉中花青素的积累,共同发挥调控作用。
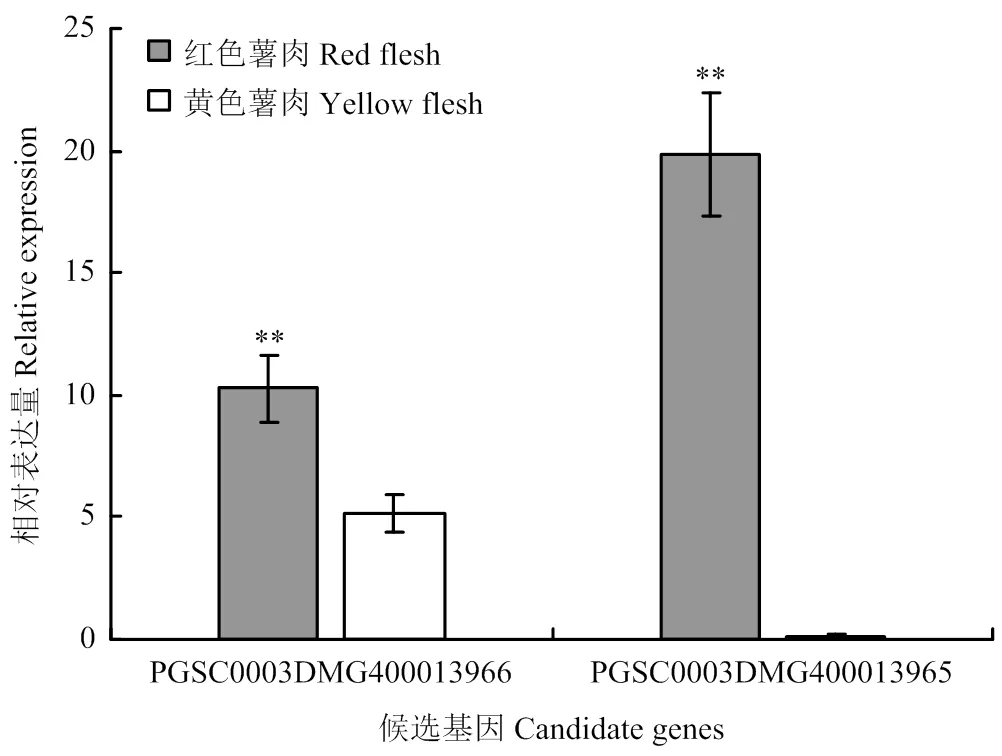
通过qRT-PCR检测候选基因在红色薯肉与黄色薯肉中的表达水平。每种颜色3个生物学重复,取平均值,采用t-test检测显著性,**表示P<0.01
3 讨论
彩色马铃薯具有重要的营养和经济价值,从遗传水平解析薯肉颜色的调控,不仅具有重要的科学价值,同时将有效地加快彩色马铃薯的分子育种进程。花青素是赋予薯肉颜色最重要的色素之一,花青素的生物合成是类黄酮物质合成的一个分支[21]。在植物中花青素的生物合成通路已经解析得比较清楚:合成起始于一个4-香豆酰辅酶A分子和3个丙二酰辅酶A分子,经过多步反应合成无色花色素[22-23],再在花青素合成酶的作用下,形成不同颜色的初级花青素,最后再经过糖基或酰基化修饰,形成有色稳定的花青素[24-26]。马铃薯中花青素合成受到结构基因和调节基因的共同控制。结构基因编码花青素合成过程中的关键酶,如基因和[4-5],而调节基因则负责调控结构基因的组织特异性表达,如基因调控薯皮中基因和的表达,控制薯皮花青素的积累[11,27]。目前结构基因研究的比较清晰,但是很多组织特异的调节基因尚不明确。
关于马铃薯薯肉颜色的遗传调控,前人做了许多研究,但关键调控基因一直未有定论。De Jong[12]的研究认为,(pigmented flesh)控制着薯肉花青素的积累,并且认为的表达受到的调控并与紧密连锁,未指出此基因的物理位置。ENDELMAN等[28]的研究分别定位了两个控制薯肉花青素积累的:基因位于第9号染色体30 812 479—49 453 657 bp之间,候选基因为PGSC0003DMG400012891,在本研究试验群体BSA-seq分析中第9号染色体未出现信号;基因位于第10号染色体53 482 273—56 116 729 bp之间,未提出候选基因,此基因与(51.74 Mb)存在一定连锁,但在其研究中可以同时定位。前人的研究[12]有一定的矛盾之处,本研究对候选基因进行了精细定位,定位区间内有两个MYB类转录因子,PGSC0003DMG400013966(51.72 Mb)和PGSC0003DMG400013965(51.74 Mb)在基因组上相距22 kb,可认为是紧密连锁,且定位区间和Endelman等[28]研究不符。通过表达量分析,证明两个基因在薯肉花青素的合成中均发挥作用,其中PGSC0003DMG400013965为控制薯皮花青素合成的,而PGSC0003DMG400013966是不是前人报道的,以及其基因身份和功能有待进一步的试验验证。
早在20世纪初,研究人员就提出了控制四倍体薯皮颜色的--调控模型[29]。DODDS[9]和LONG[10]等在此基础上提出了二倍体中控制薯皮花青素积累的--模型。Jung等[11]在四倍体中克隆了基因,基本解析了薯皮花青素积累的调控方式。然而薯肉花青素积累的调控方式一直没有得到深入研究。本研究定位的两个基因跟DE JONG[12]提出的-基因互作模型十分吻合。两个基因的身份,基因是否调控表达,以及薯肉中是否存在--这种调控模式目前尚不清楚,后续的基因功能研究将有助于进一步揭示薯肉花青素合成的调控机理,为彩色马铃薯的遗传育种提供更多的理论依据。
4 结论
本研究利用薯肉颜色分离的BC1S1群体,将控制薯肉花青素积累的主效基因定位到马铃薯第10号染色体51 473 988—51 850 970 bp之间,物理距离约为377 kb。结合参考基因组的注释信息、表达量分析和前人的研究结果,证明PGSC0003DMG400013965和PGSC0003DMG400013966基因在调控薯肉花青素的积累中共同起作用。
[1] 李颖, 李广存, 李灿辉, 屈冬玉, 黄三文. 二倍体杂种优势马铃薯育种的展望. 中国马铃薯, 2013, 27(2): 96-99.
LI Y, LI G C, LI C H, QU D Y, HUANG S W. Prospects of diploid hybrid breeding in potato., 2013, 27(2): 96-99. (in Chinese)
[2] LIU Y, TIKUNOV Y, SCHOUTEN R E, MARCELIS L F M, VISSER R G F, BOVY A. Anthocyanin biosynthesis and degradation mechanisms invegetables: A review., 2018, 6: 52.
[3] LACHMAN J, HAMOUZ K. Red and purple coloured potatoes as a significant antioxidant source in human nutrition - A review., 2005, 51(11): 477-482.
[4] ZHANG Y F, CHENG S P, DE JONG D, GRIFFITHS H, HALITSCHKE R, DE JONG W. The potatolocus codes for dihydroflavonol 4-reductase., 2009, 119(5): 931-937.
[5] JUNG C S, GRIFFITHS H M, DE JONG D M, CHENG S P, BODIS M, DE JONG W S. The potatolocus codes for flavonoid 3',5'- hydroxylase., 2005, 110(2): 269-275.
[6] DE JONG W S, DE JONG D M, DE JONG H, KALAZICH J, BODIS M. An allele of dihydroflavonol 4-reductase associated with the ability to produce red anthocyanin pigments in potato (L.)., 2003, 107(8): 1375-1383.
[7] JIANG P, RAUSHER M. Two genetic changes in-regulatory elements caused evolution of petal spot position in., 2018, 4(1): 14-22.
[8] HUANG D, WANG X, TANG Z Z, YUAN Y, XU Y T, HE J X, JIANG X L, PENG S A, LI L, BUTELLI E, DENG X X, XU Q. Subfunctionalization of thegene cluster during the domestication of citrus., 2018, 4(11): 930-941.
[9] DODDS K S, LONG D H. The inheritance of colour in diploid potatoes. I. Types of anthocyanidins and their genetic loci., 1955, 53(1): 136-149.
[10] DODDS K S, LONG D H. The inheritance of colour in diploid potatoes. II. A three-factor linkage group., 1956, 54(1): 27-41.
[11] JUNG C S, GRIFFITHS H M, DE JONG D M, CHENG S P, BODIS M, KIM T S, DE JONG W S. The potato() locus encodes an R2R3 MYB transcription factor that regulates expression of multiple anthocyanin structural genes in tuber skin., 2009, 120(1): 45-57.
[12] DE JONG H. Inheritance of pigmented tuber flesh in cultivated diploid potatoes., 1987, 64(7): 337-343.
[13] ZHANG Y F, JUNG C S, DE JONG W S. Genetic analysis of pigmented tuber flesh in potato., 2009, 119(1): 143-150.
[14] ZHANG H L, YANG B, LIU J, GUO D L, HOU J, CHEN S C, SONG B T, XIE C H. Analysis of structural genes and key transcription factors related to anthocyanin biosynthesis in potato tubers., 2017, 225: 310-316.
[15] 刘秀丽, 吕红, 范小峰. 两种茄科植物基因组提取方法的比较. 陇东学院学报, 2017, 28(1): 53-56.
LIU X L, Lü H, FAN X F. A Comparison on the methods for genome extraction on two plant species of solanaceae., 2017, 28(1): 53-56. (in chinese)
[16] The Potato Genome Sequencing Consortium. Genome sequence and analysis of the tuber crop potato.2011, 475(7355): 189-195.
[17] HUANG W J, KHALDUN A B M, Lü H Y, DU L W, ZHANG C J, WANG Y. Isolation and functional characterization of a R2R3-MYB regulator of the anthocyanin biosynthetic pathway from., 2016, 35(4): 883-894.
[18] PEREZ-DIAZ J R, PEREZ-DIAZ J, MADRID-ESPINOZA J, GONZALEZ-VILLANUEVA E, MORENO Y, RUIZ-LARA S. New member of the R2R3-MYB transcription factors family in grapevine suppresses the anthocyanin accumulation in the flowers of transgenic tobacco., 2016, 90(1/2): 63-76.
[19] LIU F, YANG Y J, GAO J W, MA C L, BI Y P. A comparative transcriptome analysis of a wild purple potato and its red mutant provides insight into the mechanism of anthocyanin transformation., 2018, 13(1): e0191406.
[20] LIU Y H, LIN-WANG K, DENG C, WARRAN B, WANG L, YU B, YANG H Y, WANG J, ESPLEY R V, ZHANG J L, WANG D, ALLAN A C. Comparative transcriptome analysis of white and purple potato to identify genes involved in anthocyanin biosynthesis., 2015, 10(6): e0129148.
[21] HUANG W J, KHALDUN A B M, CHEN J J, ZHANG C J, Lü H Y, YUAN L, WANG Y. A R2R3-MYB transcription factor regulates the flavonol biosynthetic pathway in a traditional Chinese medicinal plant,., 2016, 7: 1089.
[22] KOES R E, QUATTROCCHIO F, MOL J N M. The flavonoid biosynthetic pathway in plants: function and evolution., 1994, 16(2): 123-132.
[23] HOLTON T A, BRUGLIERA F, LESTER D R, TANAKA Y, HYLAND C D, MENTING J G T, LU C Y, FARCY E, STEVENSON T W. Cloning and expression of cytochrome P450 genes controlling flower colour., 1993, 366(6452): 276-279.
[24] HOLTON T A, CORNISH E C. Genetics and biochemistry of anthocyanin biosynthesis., 1995, 7(7): 1071-1083.
[25] BOSS P K, DAVIES C, ROBINSON S P. Expression of anthocyanin biosynthesis pathway genes in red and white grapes., 1996, 32(3): 565-569.
[26] LISTER C E, LANCASTER J E, WALKER J R L. Developmental changes in enzymes of flavonoid biosynthesis in the skins of red and green apple cultivars., 1996, 71(3): 313-320.
[27] DE JONG H. Inheritance of anthocyanin pigmentation in the cultivated potato: a critical review., 1991, 68(9): 585-593.
[28] ENDELMAN J B, JANSKY S H. Genetic mapping with an inbred line-derived F2population in potato., 2016, 129(5): 935-943.
[29] SALAMAN R N. The inheritance of colour and other characters in the potato., 1911, 5(1): 192-193.
Fine Mapping and Candidate Genes Analysis for Regulatory Gene of Anthocyanin Synthesis in Red-Colored Tuber Flesh
XU YunMei1, LI YuMei1,3, JIA YuXin2, ZHANG ChunZhi2, LI CanHui1, HUANG SanWen2, ZHU GuangTao1
(1School of Life Science, Yunnan Normal University/Joint Academy of Potato Sciences, Kunming 650500;2Agricultural Genomics Institute as Shenzhen, Chinese Academy of Agricultural Sciences, Shenzhen 518120;3Faculty of Information Engineering & Automation, Kunming University of Science and Technology, Kunming 650500)
【】 The color of tuber flesh is an important agronomic trait, which directly affects the nutritional and commercial value of potatoes. Accordingly, it is an important goal for potato genetic research and breeding improvement. In this study, we mapped the locus controlling red flesh color by bulked segregant analysis (BSA) and fine mapping, and predicted the candidate genes aided by expression analysis, which provides a foundation for gene function characterization, regulation analysis as well as the molecular breeding of colored potatoes. 【】 We crossed a red flesh diploid clone with thegene donor to produce F1, and the F1was backcrossed with the red flesh clone, and finally generated a BC1S1diploid population. From the BC1S1segregation population with 300 individuals, the genomic DNA of 18 red-colored flesh and 21 yellow-colored flesh individuals were extracted respectively and used for sequencing, and the locus controlling red flesh was preliminarily mapped by BSA-seq. The locus was fined mapped through genotyping and phenotyping of the recombination plants from 796 BC1S1individuals. Based on reference genomic annotation and qRT-PCR expression analysis, candidate genes were predicted.【】 In this study, BC1S1segregating population with red and yellow-colored tuber flesh was constructed. The major locus which regulates related anthocyanin synthesis in potato flesh was mapped on chromosome 10 within a physical distance between 48.70 Mb and 52.20 Mb. Finally, molecular markers were used to locate the gene into a 377 kb genomic interval from 51.47 Mb to 51.85 Mb. There are five annotated genes in this genomic region based on reference genome annotation information, two of them were MYB transcription factors. We predicted that the two genes, PGSC0003DMG400013966 and PGSC0003DMG400013965, were candidate genes controlling red tuber flesh. 【】 In this research, the major locus which regulates accumulation of anthocyanin in potato tuber flesh finally was mapped to chromosome 10 with an interval from 51.47 Mb to 51.85 Mb and predicted the candidate genes, PGSC0003DMG400013966 and PGSC0003DMG400013965.
red-colored flesh; anthocyanin metabolism; BSA-seq; fine mapping; candidate genes
2019-03-04;
2019-05-13
云南省高端人才引进计划(2013HA025)
许芸梅,Tel:18314556263;E-mail:Jaim427@outlook.com。
祝光涛,Tel:13520505348;E-mail:zhuguangtao@caas.cn
(责任编辑 赵伶俐)
