陆海高代回交群体抗黄萎病QTL定位
2019-07-24郭志军赵云雷陈伟王红梅龚海燕桑晓慧崔艳丽赵佩
郭志军,赵云雷,陈伟,王红梅,龚海燕,桑晓慧,崔艳丽,赵佩
(中国农业科学院棉花研究所/棉花生物学国家重点实验室,河南安阳455000)
我国棉花黄萎病是由大丽轮枝菌引起的土传维管束病害,实际生产中很难彻底根治,已成为我国棉花生产中最严重的病害之一,给棉花生产造成了极大损失[1-2]。长期棉花生产实践表明,选育并种植抗病品种是最有效的防治措施。然而,由于陆地棉栽培种抗源匮乏,而且传统育种方法选择效率低、周期长,抗病育种进展较慢。通过连锁分析定位与抗病基因紧密连锁的分子标记,进而用于分子标记辅助选择育种,可有效加快抗病新品种培育进程[3-5]。
目前,国内外开展了大量的棉花抗黄萎病QTL(Quantitative trait loci)定位方面的研究,定位群体也逐渐由陆陆群体和初级群体向陆海群体和高级群体演变,并取得了很大的进展,定位了大量的抗病QTL[6-10]。但直接应用于抗病遗传改良并应用于生产实践的尚未见报道,因此需要进一步发掘和鉴定抗黄萎病QTL,以加快分子标记辅助选择抗病育种进程。本研究通过构建以陆地棉栽培种鲁棉研28为遗传背景的陆海高代回交群体,定位与黄萎病抗性相关的QTL,以期为分子标记辅助选择育种提供标记。
1 材料与方法
1.1 试验材料
选用高抗黄萎病海岛棉品种“海7124”和高感黄萎病陆地棉标准系 “TM-1”配制抗感组合F1,再以陆地棉主栽品种鲁棉研28为轮回亲本进行连续回交、自交,最终分别得到137个BC4F1和BC4F2:3株系。
1.2 试验设计
2013年分别在河南安阳中国农业科学院棉花研究所病圃(以下简称“病圃”)和试验农场东场(以下简称“大田”),对 137 个 BC4F2:3株系进行抗病性鉴定。病圃环境下,设3次重复,单行区,行长8 m,株距0.25 m,0.8 m等行距,随机区组排列。病圃是人工感染的水泥池,长为20 m,宽为2.5 m,所接病原菌为具中等致病力的安阳菌系。每个病池均设抗病对照和感病对照,分别以中植棉2号和冀棉11为抗病和感病对照。大田环境下,设3次重复,两行区,6 m行长,株距0.25 m,0.8 m等行距,随机区组排列,其他管理同大田。
大田环境下于9月12号、9月25号,病圃环境下于8月5号、8月28号,按照全国统一病情分级标准GB/T 22101.5―2009《叶棉花抗病虫性评价技术规范第5部分:黄萎病》[11](详见表1)进行病害调查,计算病情指数和相对(矫正)病情指数,并采用Excel 2010统计分析抗病鉴定结果,并进行正态分布检验。
病情指数=(∑级数×每级的病株数/调查总株数×4)×100;
相对病情指数=实测病指×K.
其中K值是校正系数,用全国统一规定的感病对照病情指数50.0,除以本期鉴定感病对照病指实测值。

表1 棉花黄萎病Ⅴ级划分标准Table 1 Grading stand for cotton Verticillium wilt with five levels
1.3 分子检测
采用改良的CTAB法提取海7124、TM-1和鲁棉研28以及BC4F1群体的DNA[12]。利用2 010对SSR引物进行多态性检测,再利用多态性引物对BC4F1群体进行扩增检验。PCR(Polymerase chain reaction)扩增体系、反应程序和聚丙烯酰胺凝胶电泳参照张军等[13]的方法。
1.4 遗传图谱构建及QTL定位
参考Yu Jing等[14]构建的整合遗传连锁图谱,将在亲本间和群体上均表现出多态性的Sim-ple sequence repeats(SSR)标记直接锚定在染色体上,用Mapchart2.2生成遗传连锁图。
采用WinQTLCart V2.5[15]软件以复合区间作图法检测QTL,设置步长为1 cM(centi Morgan),用似然比值即LR值进行显著性检验,当LOD≥2.5时,则认为存在QTL。QTL命名参照本课题已有的命名方法[16]。
2 结果与分析
2.1 多态性SSR引物筛选以及在BC4F1群体中扩增效果
采用2010对SSR引物进行亲本间多态性筛选,挑选扩增条带清晰而且在海7124和TM-1、鲁棉研28之间具有多态性的引物,最终共筛选出494对多态性SSR引物,多态性比率约为24.6%。继而,采用494对多态性SSR引物对BC4F1群体进行扩增检验,494对多态性引物中仅有243对引物具有群体多态性,占多态性引物的比率约为50%。
2.2 遗传图谱的构建
将243对具有群体多态性的SSR引物与Yu Jing等[14]构建的整合遗传连锁图谱进行比对,直接将标记定位到相应的染色体上,并采用Mapchart2.2软件绘制遗传连锁图(图1)。最终共将216个多态性SSR标记锚定在26条染色体上,另有27个标记未能进入连锁群。每条染色体上多态性标记分布2~25个不等,其中Chr3、Chr10、Chr12、Chr13等四条染色体上标记最少只有2个,第16条染色体上标记最多为25个。采用进入连锁群的216个多态性位点绘制连锁图,图谱可覆盖棉花基因组全长3 880 cM,标记间平均距离15.77 cM。

图1 整合遗传连锁图谱和定位的QTL在图谱上的图示Fig.1 The integrated genetic linkage map and QTL mapped in the linkage map

图1(续)Fig.1 (Continued)
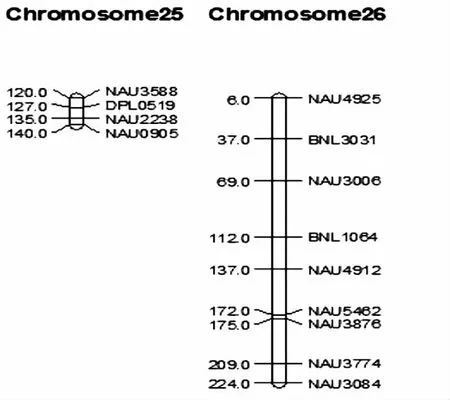
图1(续)Fig.1 (Continued)
2.3 不同试验条件下病情指数变异
棉花黄萎病抗性属数量性状,由微效多基因控制,易受环境影响。数量性状表现型是基因型与环境互作的结果,应服从正态分布。因此,在进行QTL定位前,首先要对群体表型进行正态分布检验。对大田和病圃不同调查时期的BC4F2:3家系病情指数进行正态分布统计,结果表明大田和病圃两个环境下,只有病圃环境下8月28日调查时期下BC4F2家系的峰度略大于1,其余时期的BC4F2家系病指斜度和峰度绝对值均小于1,服从正态分布,调查结果可用于QTL定位(表 2)。
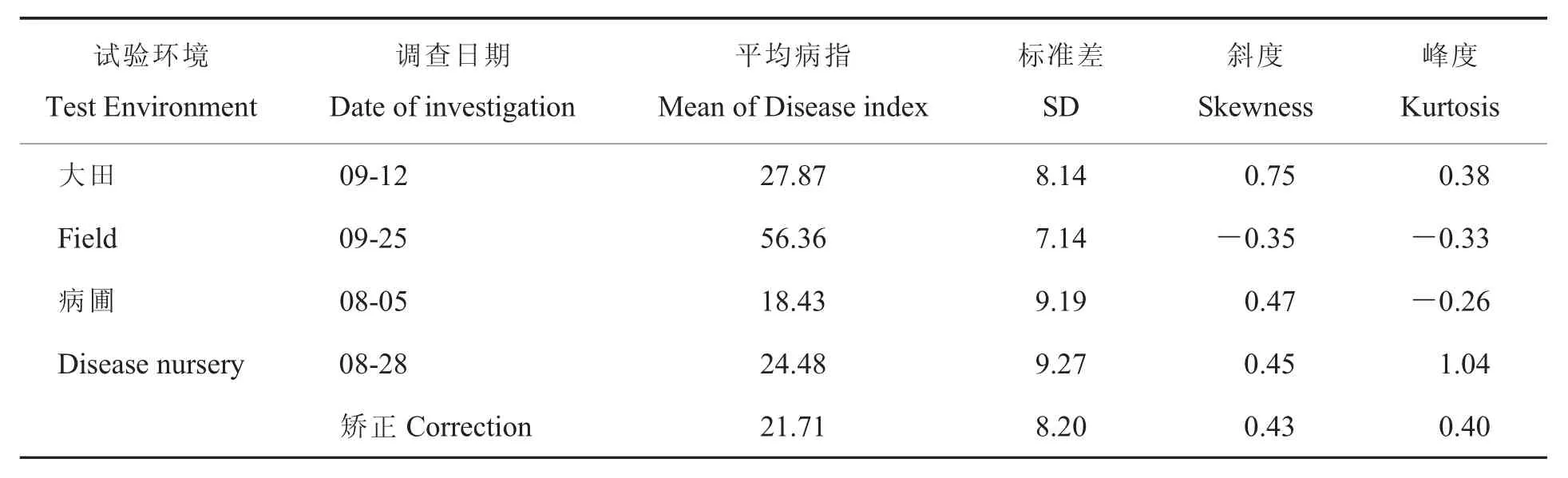
表2 BC4F2家系病指基本统计分析Table 2 The descriptive analysis of the disease index to Verticillium wilt by the BC4F2families
2.4 QTL定位分析
采用WinQTLCart V2.5软件以复合区间作图法定位QTL,共定位到6个与黄萎病抗性相关的 QTL(表 3)。
大田环境下,在9月12日这一时期检测到2个 QTLs即 qnV14-2和 qnV14-3,2个 sQTL均位于第14条染色体上,均与分子标记NAU3312相连锁,贡献率分别为19.98%和14.84%;大田9月25日调查时期未检测到QTL。
病圃环境下,共定位到4个QTLs。其中在8月5日这一时期定位到2个QTLs即qdV1-1和qdV15-1,分别位于Chr1和Chr15染色体上,分别与分子标记NAU5107和NAU3346相连锁,贡献率分别为14.15%和20.26%;病圃8月28日调查时期未检测到QTL。
采用相对病情指数,也定位到2个QTLs即qdV5-1和qdV14-1,分别位于Chr5和Chr14染色体上,分别与分子标记JEPPR0050和NAU3913相连锁,贡献率分别为8.56%和11.11%。
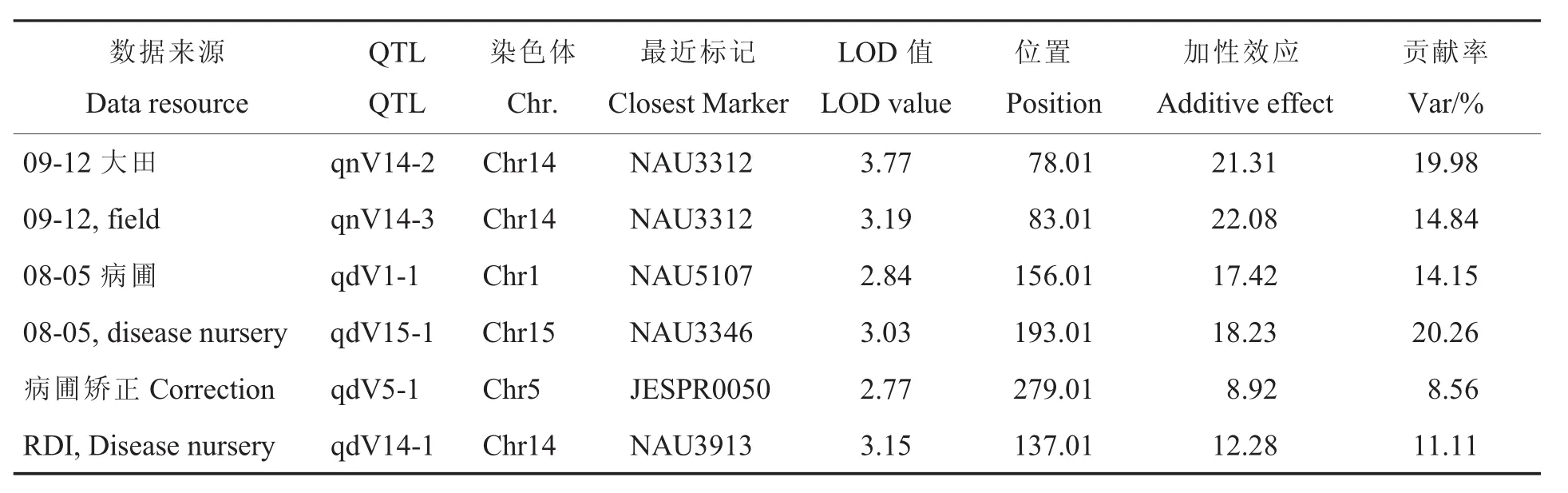
表3 复合区间作图法定位抗黄萎病QTLTable 3 Mapping QTL for Verticillium wilt resistance by composite interval mapping method
3 讨论
3.1 关于作图群体的选择
作图群体不仅是构建遗传连锁图的根本,也是QTL定位的基础,其中构建群体时所选择的亲本及群体的类型是决定群体优劣的重要因素。本研究首先选用高抗黄萎病的海岛棉品种海7124和高感黄萎病的陆地棉标准系TM-1做抗、感亲本,配制杂交组合F1,以确保分离后代的黄萎病抗性表现型具有明显的差异。同时,为了创制能够更好的应用于生产实践的抗病新品系,自F1之后将轮回亲本更换为生产上综合性状优良、大面积推广的陆地棉栽培种鲁棉研28。经4轮回交,BC4F1群体遗传背景大都趋近于鲁棉研28,可有效地削弱QTL与遗传背景的互作效应,可提高定位的精度。但是连续回交,也造成作图时标记偏分离现象严重,前人研究中同样出现[17-19]。偏分离位点的存在势必会对作图的精度造成一定的影响,然而若仅仅是简单的删除这些偏分离位点,将会导致遗传连锁群覆盖率偏低,也会遗漏很多重要的信息。因此采用较高的LOD值或采用比较作图或抽样群体作图是处理偏分离标记的比较好的方法[20]。本研究在采用较高LOD值时能够进入连锁群的标记较少,且大多数标记被定位在同一连锁群上,究其原因,主要在于经连续回交,BC4F1群体所携带的海岛棉片段很少,因此参考Yu Jing等[14]的高密度遗传连锁图来定位QTL。
近年来,陆海渐渗系逐渐被应用到棉花相关性状的QTL定位。戎福喜等[21]采用陆海渐渗系定位了与棉花吐絮期叶绿素含量、荧光参数及相关性状的QTL,共计44个。赵君等[10]采用染色体片段代换系定位了一个抗病主效QTL,位于D4染色体,可解释表型变异64.8%。本研究中,对BC4F1群体进行扩增时发现,BC4F1群体中只渗透有少数的海岛棉染色体片段,最少的为1个,最多的为38个,杂合居多,纯合的海岛棉染色体片段仅有12个,而且其中7个片段聚合在同一份材料中。因此,本群体可为今后开展以陆海渐渗群体或CSSL群体抗病QTL精细定位奠定基础。
3.2 QTL定位
棉花黄萎病是一种土传真菌维管束病害,其发生和发展受环境影响很大,因此,抗病性鉴定准确性至关重要。为提高试验精准度,本研究设置大田和病圃两个环境进行抗病性鉴定。
采用复合区间作图法在两个环境下共检测到6个QTLs,其中大田环境下2个,病圃环境下4个。大田环境下检测到的QTLs相对较少,可能与2013年气温偏高有关,气温偏高在一定程度上会抑制黄萎病害的发生和发展,对鉴定结果有一定的影响。定位到的6个QTLs中除qdV5-1以外,其余5个QTLs贡献率均大于10%。其中,QTL qdV15-1位于第15条染色体上,张保才[22]定位 到 的 QTLs DImean-1-BC2F1、DIf-1-BC1S1 和DI825-1-BC2F1以及王沛政[23]定位到的QTL qVW-Chr.15-1同样位于第15条染色体上。病圃环境下定位到的QTL qdV5-1与张保才[22]定位的DImean-3-BC2F1、DIf-5-BC2F1以 及 杨 昶[24]定 位 的qVV-A5-1RIL592、qVL-A5-1RIL592 和 qVL-A5-1RILBP2和宁志怨[25]定位的qVW-A5-1均位于第5条染色体上。本研究中在大田和病圃两个环境下,在第14条染色体上共检测到3个QTLs分别为 qnV14-2、qnV14-3、 qdV14-1,张保才[24]在第14条染色上检测到 2个 QTLs DIf-4-BC2F1、DIf-3-BC2F1,同时宁志怨[25]也在第 14条染色体(D2)上检测到 QTL qVWD2-1。同时,本研究在病圃条件下,在第1条染色体上新检测到1个QTL qdV1-1。
由于不同的QTL定位研究所采用的材料、标记均有所不同,因此,不同研究之间所共有的标记较少,很难将已定位的QTL与前人相比较。通过与前人相比较,也只能明确本研究所定位到的QTL与前人的在染色体水平上相同。然而,随着生物信息学和比较基因组学的发展及其在玉米相关性状基因比较定位上的成功应用[26-27],为通过参考高密度遗传连锁图谱进行QTL定位和整合提供参考。本研究进行QTL定位时,参考Yu Jing等[14]整合的遗传连锁图,经与Guo等[28]的高密度遗传连锁图谱相比对,虽然标记之间的距离有所不同,但在染色体上的位置和顺序基本一致。因此,本研究所定位的QTL应该是准确可靠的,与之相关联的分子标记可以尝试应用于分子标记辅助选择育种。
4 结论
本研究定位到了6个抗黄萎病QTLs,分别位于6条染色体上,第14条染色体上最多为4个。与6个QTLs相连锁的分子标记NAU3312、NAU5107、NAU3346、JESPR0050、NAU3913 可尝试应用于分子标记辅助选择抗病育种。
