冰菜盐胁迫下的转录组分析
2019-06-11练冬梅赖正锋姚运法林碧珍洪建基
练冬梅, 赖正锋, 姚运法, 林碧珍, 洪建基
冰菜盐胁迫下的转录组分析
练冬梅, 赖正锋, 姚运法, 林碧珍, 洪建基*
(福建省农业科学院亚热带农业研究所,福建 漳州 363005)
为了解冰菜()叶片抗盐相关基因组学,利用Illumina Hi-seq TM2500高通量测序技术研究冰菜叶片在400 mmol L–1NaCl胁迫下转录组基因的差异表达。结果表明,从400 mmol L–1NaCl胁迫和对照的冰菜叶片中共获得13.01 Gb Clean data,Q30碱基均大于90.08%。共获得123个差异表达基因(DEGs),包括73个上调基因,50个下调基因,其中功能注释的基因有96个。根据Unigene库序列进行GO、COG和KEGG注释,筛选出8个与抗盐性相关差异表达基因,植物激素代谢相关基因,脱落酸8ʹ-羟基化酶、吲哚-3-乙酰酸酰胺合成酶和茉莉酮酸酯ZIM结构域蛋白基因均下调表达,生长素响应蛋白、细胞分裂素合酶基因则上调表达,糖代谢相关基因棉子糖合成酶基因上调表达,质膜H+-ATPase基因上调表达,脱水蛋白基因下调表达。这为冰菜耐盐基因组学和分子生物学的研究奠定基础。
冰菜;盐胁迫;叶片;转录组;差异表达基因
土壤的盐渍化已经成为全球性的生态问题,影响农作物生长发育和产量的重要制约因素,因此,研究盐生植物的耐盐机制对防治土壤盐渍化具有重要意义。中国沿海面积广阔,部分地区由于自然和人为因素造成了土壤盐渍化,其中,福建省地处沿海的土壤也受到不同程度的破坏。而园林植物生长缓慢,种类较少,资源贫乏[1],土壤修复慢,因此需要一些耐盐速生植物来维持生态环境的可持续发展。近来,福建地区已成功引进种植冰菜,并受到消费者的青睐。
冰菜(),即冰叶日中花,为番杏科(Aizoaceae)日中花属植物,原产非洲南部和东部。冰菜在20.0%~40.0%海水浓度及低于400 mmolL–1NaCl下均具有极强的耐盐性[2–3]。冰菜含丰富的营养物质,每100.0 g冰菜含有31.0g叶酸、77.0g视黄醇、926.0g-胡萝卜素、0.7 g蛋白质[4]。所以,冰菜作为一种具有较高生态功能和保健功能的蔬菜,值得深入研究和广泛推广种植。当前,国内外对冰菜的相关研究主要集中在栽培技术、抗盐生理生态上[5–7],而对其细胞和分子水平的研究还亟待发展完善。
基于测序技术的进步,近年来植物响应非生物胁迫,尤其是盐胁迫的转录组研究成果层出不穷[8–11]。目前,转录组分析是揭示植物耐盐分子机制的主流方法,可以全面、系统地反映植物的耐盐分子机制[12]。分析盐生植物在盐胁迫下的转录组是研究植物耐盐分子机制和鉴定耐盐相关基因的重要策略之一[13–14]。冰菜的分子遗传学和功能基因组学研究不深,冰菜在盐胁迫下的分子调控机制仍不清楚,关联转录组学是一种鉴定调控目标性状候选基因的新方法。本研究采用Illumina Hiseq 2500高通量测序技术,从分子层面研究和解析盐胁迫处理后冰菜叶片基因的表达变化,以寻找冰菜叶片中耐盐关键基因,为冰菜耐盐基因组学和分子生物学研究提供参考。
1 材料和方法
1.1 材料
冰菜()种子来自于福建省农业科学院亚热带农业研究所。种子经3% H2O2消毒3 min,流水冲洗数次,将种子放置在干净的基质中,置于培养箱中培养至三叶一心,选择生长一致的幼苗种植于花盆中,每盆3株,胁迫组(S)和对照组(Control)各3盆。每2 d浇1/2Hoag- land营养液1次,置于人工气候室中,进行为期2周的预培养。此后,为避免盐冲击,每2 d递增浇含50 mmol L–1NaCl的1/2Hoagland营养液,直到400 mmolL–1,对照仅浇1/2Hoagland营养液,以后仍然每2 d浇1/2Hoagland营养液1次,再培养植物35 d[15],冰菜可采食时,剪下植株相同部位的叶片(尽量去除中上部较新鲜嫩绿的叶片),每处理3个重复,每个处理的所有叶片混合后置于冷冻管中,液氮速冻,存于–80℃超低温冰箱备用。
1.2 叶片总RNA提取、转录组测序和分析
用PureLink Plant RNAReagent Kit试剂盒提取冰菜叶片的总RNA,用分光光度计测定A260和A280值,计算样品浓度,然后用15%的变性聚丙烯酰胺凝胶电泳,检测质量合格后送北京百迈客生物技术有限公司进行转录组测序,并得到转录组数据。运用生物信息学技术和方法,对测序数据进行前期处理、序列比对、基因及基因组的注释、基因表达分析等。
2 结果和分析
2.1 数据组装和分析
对照和胁迫处理冰菜叶片经高通量测序和质量控制,共获得13.01 Gb有效数据(Clean data),其中对照为6.62 Gb,胁迫处理的为6.39 Gb,其Q30碱基均大于90.80%,表明测序结果可靠,可用于后续的分析。从表1可见,总共产生69 734条单基因序列(Unigene)和131 228条转录本序列(Transcipt), N50分别为2 323和2 058 bp,组装完整性较高; 其中长度为200~300 bp的Unigene数量最多,有22 060条,占31.63%,且随着长度的增加,Unigene数量减少。
2.2 Unigene功能注释
为获得Unigene的功能注释信息,通过NR、Swiss-Prot、KEGG、COG、KOG、NOG、GO和Pfam等8个数据库进行注释分析(表2),在69 734条Unigene中,共获得26 360条(37.80%)注释,以上8个数据库分别获得26 079 (37.39%)、16 708 (23.95%)、9 624 (13.80%)、8 029 (11.51%)、14 431 (20.69%)、24 156 (34.64%)、15 700 (22.51%)和17 910条(25.68%)注释。
2.3 差异表达基因分析
通过对照和胁迫处理冰菜叶片差异基因的表达分析,共获得123个差异表达基因(differential expression genes, DEGs),包括73个上调基因,50个下调基因;将DEGs单基因序列分别在8个数据库中进行注释,共获得96条基因功能注释,COG、GO、KEGG、KOG、Pfam、Swiss-Prot、eggNOG和NR分别注释19、43、23、37、76、67、86和92条,以NR数据库注释比率最高,达95.83%。
差异表达基因的GO功能注释 GO功能注释的43条DEGs共分为3大类,分别为生物过程、细胞组分和分子功能,又划分为27个功能小类(表3),生物过程的DEGs以代谢过程、细胞过程和单一生物过程3个功能小类的数量最多;细胞组分的DEGs以细胞、细胞成分、细胞器和膜结构4个功能小类的数量最多;分子功能的DEGs以催化活性和结合活性2个功能小类的数量最多。
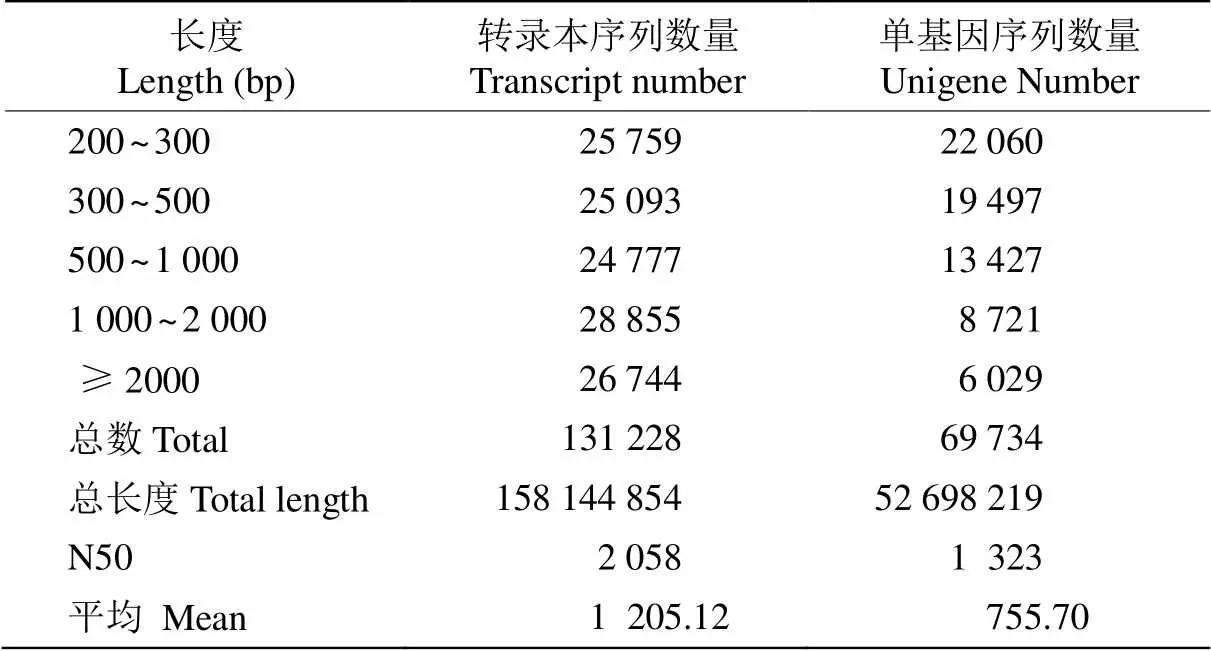
表1 组装结果统计

表2 单基因序列的功能注释

表3 差异表达基因GO功能注释
差异表达基因的COG功能注释 COG数据库注释的19条DEGs进行直系同源分类,并获得11个功能分类(表4),主要集中在E (氨基酸的转运和代谢)、G (碳水化合物转运与代谢)、Q (次生代谢产物生物合成、运输和分解代谢)和R (一般性功能预测)。植物生长发育过程有大量基因表达,在盐胁迫下,部分涉及到了氨基酸、碳水化合物的转运和代谢,以及次生代谢产物生物合成、运输和分解代谢等过程,说明这些生物学过程对于冰菜生命活动特别是耐盐性的重要性。
差异表达基因的KEGG功能注释 有23个DEGs得到注释(表5),分别富集在10条代谢通路,包括植物激素信号转导(4个)、类胡萝卜素生物合成(2个)、半乳糖代谢(1个)、酪氨酸代谢(1个)、玉米素生物合成(1个)、异喹啉生物碱生物合成(1个)、氧化磷酸化(1个)、-亚麻酸代谢(1个)、淀粉和蔗糖代谢(1个)和角质、次分泌和蜡生物合成(1个)。这说明盐胁迫下冰菜叶片的各种代谢途径都发生了变化。
2.4 差异基因分析
通过对盐胁迫下冰菜叶片转录组测序结果进行功能注释、功能分类及代谢途径分析表明(表6),生长素响应蛋白、细胞分裂素合酶、棉子糖合成酶和H+-ATPase基因的表达分别上调了351.4%、500.0%、877.9%和459.6%倍,吲哚-3-乙酰酸酰胺合成酶、脱落酸8ʹ-羟基化酶、茉莉酮酸酯ZIM结构域蛋白和脱水蛋白基因的表达则分别下调了100.0%、80.7%、85.4%和100.0%。可见,这些差异表达基因参与了植物激素、糖等的代谢途径,影响着冰菜的生长发育。
3 讨论
虽然目前已经利用生理生化手段揭示和阐明了冰菜许多的耐盐机制,但从分子水平上冰菜仍有很多抗性策略和机制有待深入研究和分析。转录组主要指所有编码蛋白质的mRNA总和[16],能够从整体水平探索基因的结构和功能。冰菜为非模式生物,需要通过与同源物种的测序信息进行比对分析,从而研究各种特定的生物学过程和分子机理。在本研究中,利用Illumina HiSeq TM2500高通量测序技术构建了冰菜在400 mmolL–1盐胁迫下的叶片转录组数据库,借助其他物种数据库已有信息,对表达差异的基因进行比对分析,表明盐胁迫后冰菜的植物激素代谢、糖代谢和其它相关功能蛋白基因的表达发生了改变。

表4 差异表达基因COG功能注释

表5 差异表达基因的KEGG功能注释

表6 盐胁迫下冰菜叶片差异基因的表达丰度
植物激素几乎参与了所有的生命活动,其对盐胁迫的响应也不例外。植物中的激素没有明显的专一性,一种激素可以有多种生理效应,而多种激素又具有调节同一生理过程的作用[17]。盐胁迫下,冰菜叶片中脱落酸8ʹ-羟基化酶、吲哚-3-乙酰酸酰胺合成酶和茉莉酮酸酯ZIM结构域蛋白基因均下调表达,而生长素响应蛋白、细胞分裂素合酶基因上调表达,均有利于合成脱落酸(ABA)、生长素(AUXIN)、茉莉酸(JA)和细胞分裂素(CTK),促进细胞分裂,加速细胞伸长生长,提高冰菜的盐胁迫耐受性。
盐胁迫下,植物通过细胞积累大量可溶性物质来提高渗透势,增强对渗透胁迫的抗性[18],在表达差异基因中,冰菜叶片中的棉子糖合成酶基因上调表达,有利于合成可溶性糖棉子糖,以提高细胞内的渗透势,从而提高冰菜的盐胁迫耐受性。
质膜H+-ATPase是细胞质膜上的一种重要功能蛋白,在植物的生命活动过程中起着重要作用。在转录水平上,质膜H+-ATPase参与了植物生长发育过程中的多种胁迫反应[19]。盐胁迫对植物造成离子毒害、渗透胁迫和营养不平衡,使植物的生长发育受到抑制,这可能与盐胁迫下植物细胞的跨质膜物质转运发生改变有关。有研究表明,物质通过植物细胞的跨膜转运依赖于质膜H+-ATPase转运质子产生的驱动力[20–21]。在盐胁迫下,冰菜叶片中的H+-ATPase基因上调表达,可以增强H+泵的质子动力势,驱动Na+/H+逆向转运蛋白,提高Na+外排的能力,减少K+离子外流,维持了K+/Na+平衡,从而提高冰菜的盐胁迫耐受性。脱水蛋白是在干旱胁迫下产生的最具有代表性的逆境响应蛋白,脱水蛋白基因下调表达,反映了盐胁迫下冰菜叶片自身降低脱水的生理现象。
植物耐盐性是由一系列基因相互协调而共同发挥作用来表现的[22]。冰菜经过400 mmolL–1盐胁迫后,在一系列基因共同作用下进行自我调节,从而正常生长发育。从分子水平上,可更详细地研究其对应的耐盐基因,为后期筛选冰菜耐盐关键基因和功能验证提供重要的分子基础,同时为改善土壤盐渍化具有重要意义。
[1] LU L M, WU F M, ZHANG Q, et al. Growth and physiological indexes of‘Gillettese How’ under Nacl stress [J]. J Trop Subtrop Bot, 2015, 23(3): 262–267. doi: 10.11926/j.issn.1005-3395. 2015.03.005.陆銮眉, 吴福妹, 张琼, 等. NaCl胁迫对大黄龙船花生长及生理生化的影响 [J]. 热带亚热带植物学报, 2015, 23(3): 262–267. doi: 10. 11926/j.issn.1005-3395.2015.03.005.
[2] LI G L, HU Z H, LENG P S. The physiological responses ofL. to NaCl stress [J]. J Beijing Univ Agric, 2015, 30(1): 64–70. doi: 10.13473/j.cnki.issn.1002-3186.2015.0004.李广鲁, 胡增辉, 冷平生. 冰叶日中花对NaCl胁迫的生理响应[J]. 北京农学院学报, 2015, 30(1): 64–70. doi: 10.13473/j.cnki.issn.1002- 3186.2015.0004.
[3] XU W F, QIN H Y, LIU J, et al. Changes of oxidative stress and anti- oxidant enzyme activity ofLinnaeus in response to different concentrations of seawater [J]. Jiangsu J Agric Sci, 2017, 33(4): 775–781. doi: 10.3969/j.issn.1000-4440.2017.04.008.徐微风, 覃和业, 刘姣, 等. 冰菜在不同浓度海水胁迫下的氧化胁迫和抗氧化酶活性变化[J]. 江苏农业学报, 2017, 33(4): 775–781. doi: 10.3969/j.issn.1000-4440.2017.04.008.
[4] CHEN H Y. The biological characteristics and cultivation techniques ofL. [J]. Vegetables, 2016, (8): 42–45. doi: 10.3969/j.issn.1001-8336.2016.08.015. 陈宏毅. 冰菜的生物学特性与栽培技术[J]. 蔬菜, 2016, (8): 42–45. doi: 10.3969/j.issn.1001-8336.2016.08.015.
[5] ŚLesak I, Libik M, Miszalski Z. The foliar concentration of hydrogen peroxide during salt-induced C3-CAM transition inL. [J]. Plant Sci, 2008, 174(2): 221–226. doi: 10.1016/j.plantsci.2007.11.007.
[6] OH D H, BARKLA B J, VERAESTRELLA R, et al. Cell type-specific responses to salinity-the epidermal bladder cell transcriptome of[J]. New Phytol, 2015, 207(3): 627– 644. doi: 10.1111/nph.13414.
[7] COSENTINO C, FISCHER-SCHLIEBS E, BERTL A, et al. Na+/H+antiporters are differentially regulated in response to NaCl stress in leaves and roots of[J]. New Phytol, 2010, 186(3): 669–680. doi: 10.1111/j.1469-8137.2010.03208.x.
[8] HIZ M C, CANHER B, NIRON H, et al. Transcriptome analysis of salt tolerant common bean (L.) under saline conditions [J]. PLoS One, 2014, 9(3): e92598. doi: 10.1371/journal.pone.0092598.
[9] LIU J T, ZHOU Y L, LUO C X, et al.transcriptome sequencing of desert herbaceous(Achnatherum)seedlings and identification of salt tolerance genes [J/OL]. Genes, 2016, 7(4): 12. doi: 10.3390/genes7040012.
[10] SONG B, HU A H, HALIL kurban. Transcriptome analysis of differentially expressed genes inL. under salt stress [J]. J Xinjiang Agric Univ, 2017, 40(2): 92–98. doi: 10.3969/j. issn.1007-8614.2017.02.003.宋彬, 胡安鸿, 海利力·库尔班. 沙棘NaCl胁迫下差异表达基因的转录组分析[J]. 新疆农业大学学报, 2017, 40(2): 92–98. doi: 10. 3969/j.issn.1007-8614.2017.02.003.
[11] JI F S, LI Y Y, TANG L, et al. Analysis of banana leaves responses salt stress of transcriptome [J]. Mol Plant Breed, 2017, 15(3): 875–882. 吉福桑, 李元元, 唐露, 等. 香蕉叶片响应盐胁迫转录组分析[J]. 分子植物育种, 2017, 15(3): 875–882.
[12] FU C, SUN Y G, FU G R. Advances of salt tolerance mechanism in hylophyate plants [J]. Biotechnol Bull, 2013(1): 1–7.
付畅, 孙玉刚, 傅桂荣. 盐生植物耐盐分子机制的研究进展[J]. 生物技术通报, 2013(1): 1–7.
[13] DEYHOLOS M K. Making the most of drought and salinity trans- criptomics [J]. Plant Cell Environ, 2010, 33(4): 648–654. doi: 10.1111/ j.1365-3040.2009.02092.x.
[14] SAHU B B, SHAW B P. Isolation, identification and expression analysis of salt-induced genes in, a natural halophyte, using PCR-based suppression subtractive hybridization [J]. BMC Plant Biol, 2009, 9: 69. doi: 10.1186/1471-2229-9-69.
[15] Epstein E, Bloom A J. Mineral Nutrition of Plants: Principles and Perspectives [M]. 2nd ed. Sunderland (Massachusetts): Sinauer Associates, 2005: 1–400.
[16] LIU H L, ZHENG L M, LIU Q Q, et al. Studies on the transcriptomes of non-model organisms [J]. Hereditas, 2013, 35(8): 955–970. doi: 10. 3724/SP.J.1005.2013.00955.刘红亮, 郑丽明, 刘青青, 等. 非模式生物转录组研究[J]. 遗传, 2013, 35(8): 955–970. doi: 10.3724/SP.J.1005.2013.00955.
[17] MUNNS R, TESTER M. Mechanisms of salinity tolerance [J]. Annu Rev Plant Biol, 2008, 59: 651–681. doi: 10.1146/annurev.arplant.59. 032607.092911.
[18] TÜRKAN I, DEMIRAL T. Recent developments in understanding salinity tolerance [J]. Environ Exp Bot, 2009, 67(1): 2–9. doi: 10. 1016/j.envexpbot.2009.05.008.
[19] LIU N G, WANG Z Y, MO B B, et al. Plasma membrane H+-ATPase and environmental stress [J]. J Trop Subtrop Bot, 2006, 14(3): 263–268. doi: 10.3969/j.issn.1005-3395.2006.03.017.刘尼歌, 王占义, 莫丙波, 等. 质膜H+-ATPase与环境胁迫[J]. 热带亚热带植物学报, 2006, 14(3): 263–268. doi: 10.3969/j.issn.1005- 3395.2006.03.017.
[20] MICHELET B, BOUTRY M. The plasma membrane H+-ATPase: A highly regulated enzyme with multiple physiological functions [J]. Plant Physiol, 1995, 108(1): 1–6. doi: 10.1104/pp.108.1.1.
[21] PALMGREN M G. Regulation of plant plasma membrane H+-ATPase activity [J]. Physiol Plant, 1991, 83(2): 314–323. doi: 10.1111/j.1399- 3054.1991.tb02159.x.
[22] ZHOU C F, LIN P, YAO X H, et al. Selection of reference genes for quantitative real-time PCR in six oil-tea camellia based on RNA-seq [J]. Mol Biol, 2013, 47(6): 836–851. doi: 10.1134/S0026893313060198.
Transcriptome Analysis ofunder Salt Stress
LIAN Dong-mei, LAI Zheng-feng, YAO Yun-fa, LIN Bi-zhen, HONG Jian-ji*
(Subtropical Agriculture Research Institute, Fujian Academy of Agricultural Sciences, Zhangzhou 363005, Fujian, China)
In order to understand the salt related genomics in leaves of, the expression of transcriptome genes was studied under 400 mmol L–1NaCl stress by using Illumina Hiseq TM2500 high-throughput sequencing technology. The results showed that there were 13.01 Gb Clean Data from the leaves treated with 400 mmol L–1NaCl and untreated, the Q30 base was more than 90.08%. Among them, there were 123 differential expression genes (DEGs), including 73 up-regulated genes and 50 down-regulated genes, in which 96 genes were annotated. The eight salt-resistance DEGs of were screened out based on GO, COG and KEGG annotation. The plant hormone metabolism-related genes, including abscisic acid 8ʹ-hydroxylase gene, indole-3-acetamide synthase gene, jasmonate ZIM structure domain protein gene were down-regulated, auxin responsive protein and cytokinin synthase genes were up-regulated. The sugar metabolism-related raffinose synthase genes was up-regulated, and plasma membrane H+-ATPase and dehydrated protein genes were up and down-regulated, respectively. These would provide a foundation for studing on salt tolerance genomics and molecular biology of.
;Salt stress; Leaf;Transcriptome;DEGs
10.11926/jtsb.3972
2018–07–16
2018–12–03
福建省公益类科研院所专项(2018R1101025-2)资助
This work was supported by the Special Project for Public Welfare Research Institutes in Fujian (Grant No. 2018R1101025-2).
练冬梅,女,助理研究员,主要从事特色蔬菜研究。E-mail: woshildm1987@163.com
通信作者 Corresponding author.E-mail: 76227508@qq.com
