利用VIGS技术初步验证 WSR1基因功能的研究
2019-04-12吕亮杰宿振起孙丽静王莉梅
吕亮杰,宿振起,孙丽静,王莉梅,李 辉
(河北省农林科学院粮油作物研究所/河北省作物遗传育种实验室,河北石家庄 050035)
小麦(TriticumaestivumL.)是我国仅次于玉米和水稻的第三大粮食作物,其播种面积占全国耕地面积的20%~30%[1]。河北是我国重要的商品粮基地和小麦主产省,其小麦总产量和种植面积位居全国第三,发展河北优质小麦对保障全国商品小麦的市场需求具有重要作用[2]。淀粉是小麦籽粒的主要成分,约占小麦籽粒重量的65%[3],总淀粉含量和直、支链淀粉含量对小麦品质有重要影响[4]。近年来,国外对淀粉品质的研究越来越重视,特别是在面条加工和食用品质方面;国内关于小麦淀粉性状的遗传研究起步较晚,主要集中于淀粉含量[5-6]及淀粉糊化特性的遗传规律[7-8]、淀粉含量的影响因素[9]及淀粉合成相关基因的定位[10-11]等方面。淀粉的合成途径及参与合成的酶对小麦的淀粉品质有较大影响,其中淀粉合成过程中的酶直接影响淀粉的合成和直、支链淀粉的含量,进而影响淀粉粒的结构、淀粉理化特性,改变淀粉品质。一般认为ADP-葡萄糖焦磷酸化酶(AGPase)、可溶性淀粉合成酶(SSS)、淀粉粒束缚淀粉合成酶(GBSS)和淀粉分支酶(SBE)对淀粉合成非常关键[12-13],同时环境对淀粉合成也有较大的影响[14-15]。近年来的研究主要集中在与淀粉合成途径相关的转录因子上。Zhu等[16]研究发现,EREBP(乙烯反应元件结合蛋白)可以提高Waxy(GBSS颗粒结合淀粉合成酶)基因的转录水平。Rook等[17]发现apetala2-type蛋白能提高AGPL3(葡萄糖焦磷酸化酶大亚基)的转录水平。Fu等[18]通过基因探针共表达分析,发现一个与15个水稻淀粉合成相关基因表达负向调控有关的淀粉合成转录因子(Rice Starch Regulatory,RSR1)。然而,目前有关调控小麦籽粒淀粉合成的调控转录因子(wheat starch regulatory,WSR)的研究较少。
VIGS 是基于转录后基因沉默(post-transcriptionalgene silencing,PTGS)引起内源mRNA 序列特异性降解发展起来的快速分析基因功能的新技术[19-20]。它通过整合部分靶基因表达序列的重组病毒侵染植物,引起植物内同源基因沉默,诱发表型变异进而实现基因的功能分析[20]。大麦条纹花叶病毒(barleystripemosaicvirus,BSMV)由于具有广泛的寄主范围而成为单子叶植物良好的VIGS沉默载体,为小麦和水稻等的基因功能研究提供了有效手段[21]。Scofield等[22]用BSMV-VIGS技术证明在抗叶锈病基因Lr21介导的抗性途径中要有RAR1、SGT1和HSP90基因的参与。赵 丹等[23]利用该技术沉默小麦的TNBL1基因,沉默的YW642植株对黄矮病敏感,显现感病症状。张立荣等[24]利用该技术证明小麦TaRAR1是抗叶锈病基因Lr24行使抗病作用所需要的。目前已证实BSMV-VIGS方法在小麦基因研究中的作用,但在小麦抽穗期对淀粉调控基因进行VIGS体系优化的研究还有待进一步深入。
本研究以国审小麦品种冀麦325为材料,克隆小麦WSR1基因,并以小麦PDS(八氢番茄红素脱氢酶)基因为指示基因,构建WSR1基因的BSMV-VIGS重组表达载体,进行小麦叶片和幼穗接种,再通过实时荧光定量PCR(qRT-PCR)技术,检测WSR1基因在小麦籽粒发育期间的表达,以期初步阐明小麦WSR1基因对籽粒淀粉合成的影响,并为通过转基因或分子标记辅助选择来改善小麦品质、提高粒重和产量提供理论依据。
1 材料与方法
1.1 试验材料
供试材料为普通小麦品种冀麦325,该品种由河北省农林科学院粮油作物研究所培育。BSMV重组病毒载体α、β、γ和γ-PDS由中国农业科学院张增艳研究员惠赠。
1.2 试验方法
1.2.1WSR1基因的克隆
以水稻RSR1基因(GenBank登录号:JN192938)为探针,在NCBI-BLASTn(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&BLAST_SPEC=&LINK_LOC=blasttab&LAST_PAGE=tblastn)进行同源比对,获得的同源片段为靶序列,设计引物W1-F:5′-TGAAGTTGCT GCTGAAGG-3′和W1-R:5′-CGATGACGAC GAGAAGAG-3′进行PCR扩增和基因克隆,以获得的比对得分最高的片段为靶序列,按SMARTTM RACE cDNA Amplification Kit(Clontech公司,美国)说明书的要求,应用Primer Premier 6.0软件,在其5′端设计反向特异引物WSR1-5:5′-GCCAGATTGGTGGATGCTCG GTCAGA-3′,在其3′端设计正向特异引物WSR1-3:5′-AGATGGTCGCAATCCCTCATC GATACCC-3′进行PCR扩增和基因克隆;将WSR1基因5′RACE和3′RACE的测序结果进行拼接,根据拼接的全长序列,在其开放阅读框两端设计全长引物WSR1-F:5′-TCTTCTTGAA TCGTGAGGTT-3′和WSR1-R:5′-GCTGAGG TTAGTATCTGACA-3′进行PCR扩增和基因克隆。本研究涉及到的引物均由生工生物工程(上海)股份有限公司合成。
1.2.2WSR1基因的生物信息学分析
用DNAMAN软件对测序结果进行分析,用NCBI-CDD(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)和NCBI-ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)查找基因的保守结构域和开放阅读框(ORF)。用GSDS(http://gsds.cbi. pku. edu. cn/)明确小麦WSR1基因的外显子/内含子结构。用NCBI-BLASTP(https://blast.ncbi.nlm.nih.gov/Blast.cgi)对所克隆基因进行相似性比对和同源性分析,将比对结果输入MAGE 7.0,采用邻接法(neighbor-joining,NJ)构建系统进化树,其中, bootstrap值设置为1 000,其余均为默认参数。
1.2.3 VIGS载体构建
根据已克隆的WSR1基因的全长cDNA序列,选取2个不同位置,利用Primer Premier 6.0软件设计带有NheⅠ酶切位点和保护碱基的引物,扩增WSR1基因的沉默片段,其对应的扩增片段分别命名为V1和V2,V1为WSR1基因的保守区段,V2为WSR1基因的特异性区段。引物序列为V-WSR1-1F:5′-TACGCTAGCGTGT ACTTGGGTGGATTTGAC-3′,V- WSR1-1R:5′-TACGCTAGCTGACGCCTCTGTACTTGG AG-3′;V- WSR1-2F:5′-TACGCTAGCGGATA AGAGCAGCCTTGG-3′,V- WSR1-2R:5′-TAC GCTAGCCCTCCATGCCCAGGTTGGAA C-3′。用MluⅠ酶切α、γ、γ-PDS、γ-V1和γ-V2,SpeⅠ酶切β载体,酶切完成后,用TaKaRa MiniBEST DNA Fragment Purification Kit Ver.4.0(TaKaRa,日本)进行纯化,将酶切完全的γ载体和沉默基因的片段进行连接;提取α、β、γ、γ-PDS、γ-V1和γ-V2的质粒并对其进行线性化,用RiboMAX Large Scale RNA Production Syetems-T7(Promega,美国) 和Ribo m7G Cap Analog(Promega,美国)对线性化产物进行体外转录。
1.2.4WSR1功能验证
在小麦开花12 d后,选取生长健壮的小麦植株。将体外转录好的大麦条纹花叶病毒α、β和γ转录产物,各取10 μL混合后,加入2倍体积(60 μL)的DEPC水,再加入120 μL的GKP Buffer(50 mM甘氨酸,30 mM K2HPO4,pH 9.2,1%膨润土,1%硅藻土),制得总体积为210 μL的接种病毒混合液。接种时佩戴刚开封橡胶手套,用DEPC水处理过的枪头取10 μL接种液放在食指上,一只手固定麦穗,另一手用大拇指和食指轻轻来回摩擦麦穗及叶片。接种20 min后向接种部位喷施DEPC水,保湿24 h。接种后每天观察、记录变化,并定期取材。
根据WSR1基因cDNA序列设计qRT-PCR(实时荧光定量PCR)引物WSR1RT-F:5′-TGAAGTTGCTGCTGAAGG-3′和WSR1RT-R:5′-CGATGACGACGAGAAGAG-3′,PDS-F:5′- TCGAAGGGTTCTATCTGG -3′和PDS-R:5′-CTACAACAATGTGGCAAT-3′;分别对田间对照冀麦325和接种后5、10、15、20和25 d的冀麦325取样, -80 ℃保存。在Icycler iQ5 Real-time PCR仪(Bio-Rad,美国)上进行qRT-PCR分析,反应体系与程序参考SYBR Premix ExTaq Ⅱ(TaKaRa,日本)试剂盒说明书,试验设置3个生物学重复和3个技术重复,采用2-△△Ct法计算相对表达量。
采用双波长法测定籽粒直、支链淀粉含量,二者相加得出总淀粉含量。具体测定方法参照何照花[25]和戴 双等[26]的方法并做适当调整。抗性淀粉测定方法参照 Megazyme RS assay kit(Megazyme International,爱尔兰)试剂盒说明书并做适当调整,每个样品测定三次求平均作为样本值。
2 结果与分析
2.1 WSR1基因克隆结果
根据水稻RSR1基因的同源片段设计引物,以冀麦325的cDNA为模板进行PCR扩增。PCR产物用1.0%的琼脂糖凝胶电泳检测,发现在小于500 bp处有一条带(图1),该目的条带经测序大小为459 bp。为获得WSR1基因的全长序列,以459 bp片段为靶序列,分别设计3′ RACE和5′ RACE引物,扩增后经琼脂糖凝胶电泳检测,发现3′ RACE的扩增产物约为700 bp,5′ RACE扩增产物约为1 600 bp。将扩增片段回收克隆测序,3′RACE和5′RACE产物分别为710 bp和1 664 bp(图1),与靶序列的3′端和5′端均有重叠区,将3′RACE和5′RACE 序列进行拼接,得到长2 284 bp的拼接序列。
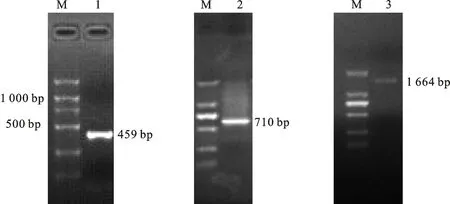
M:DL2000 marker;1:电子克隆PCR产物;2:3′ RACE 的PCR产物;3:5′ RACE的PCR产物。
2.2 WSR1基因在cDNA和DNA中的验证
在拼接序列的3′末端和5′末端设计特异性引物WSR1-F和WSR1-R,以冀麦325的cDNA和DNA为模板,进行PCR扩增,扩增产物用1.0%的琼脂糖凝胶电泳检测,发现在cDNA和DNA中分别扩增出一条2 000 bp和5 000 bp左右的条带。经测序发现WSR1的cDNA长2 003 bp,DNA长4 316 bp(图2),测序结果和拼接序列基本一致。
2.3 WSR1基因的生物信息学分析
将所获得的cDNA序列和DNA序列进行查找比较,结果表明,WSR1基因含8个外显子和7个内含子(图3)。用NCBI-ORF Finder查找拼接序列的ORF,结果显示,该基因包含一个1 584 bp 的ORF,编码527个氨基酸以及469 bp的5′非翻译区(non translated region,UTR)和231 bp的3′UTR。SMART软件分析结果表明,WSR1基因编码蛋白的181~239和273~317 位的氨基酸是保守的AP2核苷酸结合域,属于AP2/ERF家族基因(图3)。
2.4 WSR1基因的系统发育分析
经BLAST分析发现,小麦WSR1基因的编码蛋白与粗山羊草(Aegilopstauschii)的一致性为96%,与大麦(Hordeumvulgare)的一致性为94%,与二穗短柄草(Brachypodiumdistachyon)的一致性为76%,与麻竹(Dendrocalamuslatiflorus)、黍属(Setaria)和栗(Castaneamollissima)的一致性为70%,与高粱(Sorghumbicolor)的一致性为66%。利用MAGE 7.0对WSR1蛋白进行系统发育分析(图4),发现WSR1基因与大麦、山羊草、二穗短柄草的亲缘关系比较近,与水稻、麻竹、高粱的亲缘关系比较远。

M:DL2000;C:cDNA的 PCR产物;D:DNA 的PCR产物。
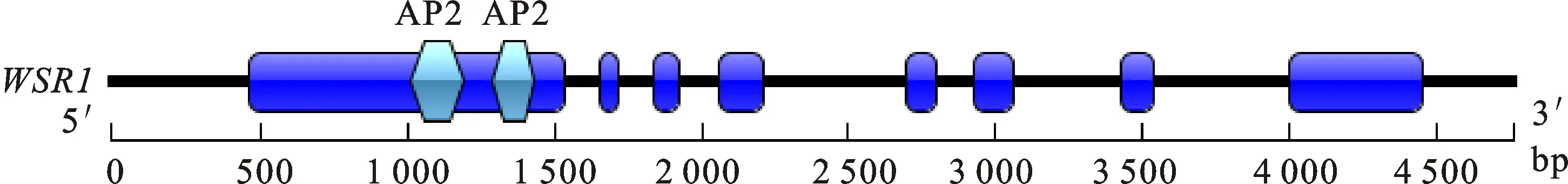
图3 WSR1基因的结构域和基因结构图
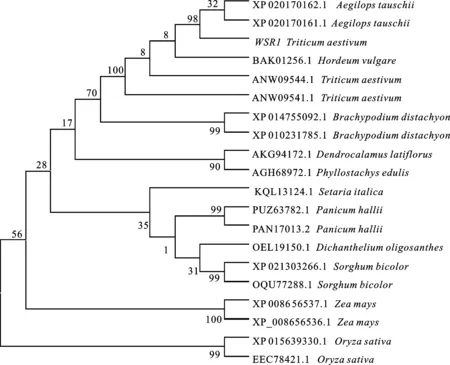
图4 WSR1基因及其编码蛋白的系统进化树
2.5 WSR1基因的功能分析
以加入NheⅠ酶切位点和保护碱基的引物V-WSR1-1F和V-WSR1-1R、V-WSR1-2F和V-WSR1-2R分别扩增WSR1基因上的目的片段(图5A),产物测序后验证,已经成功引入酶切位点和保护性碱基,片段大小依次为220 bp和247 bp,并且序列没有发生突变,与原始序列一致,2个片段分别命名为V1和V2。提取α、β、γ、γ-PDS质粒及γ-V1和γ-V2重组质粒并对其进行线性化,线性化后的质粒大约是6 kb的单一条带(图5B),表明线性化成功。
在小麦开花12 d后,选取生长健壮的冀麦325,在穗部和叶片摩擦接种BSMV:00、BSMV:PDS、BSMV:V1和BSMV:V2,各接种20株。以BSMV:00为对照,以BSMV:PDS确保能将小麦的基因沉默。接种后每天观察、记录变化,并定期取材。发现接种BSMV:PDS的小麦在第5天开始出现光漂白现象,在第10天时,光漂白现象比较明显,在第20 天时光漂白达到最大程度(图6),而接种BSMV:00的对照组却没有明显变化,接种率达90%。
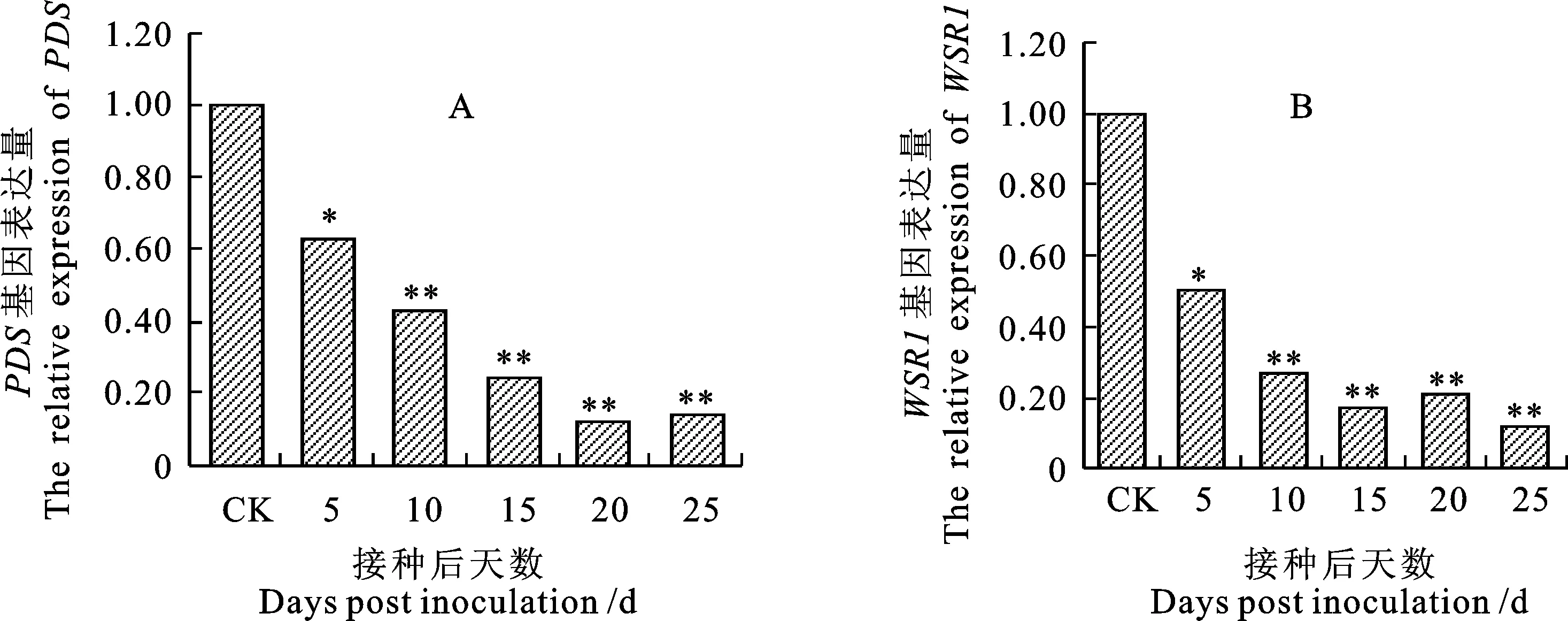
为确保接种BSMV:PDS后可引起PDS基因沉默,造成光漂白现象,提取接种小麦的RNA,反转录得到cDNA,对接种PDS基因的相对表达量进行分析。结果如图7A,在接种BSMV:PDS后,小麦PDS基因的相对表达量显著降低,接种后第5天下降了37%,第15天下降了75%,持续到第25天PDS基因的相对表达量仍保持低水平状态,而接种BSMV:00的对照组却没有变化。证明在该条件下利用BSMV-VIGS技术可以引起小麦穗部基因的沉默。qRT-PCR检测接种麦穗中WSR1基因的表达情况,如图8所示,在接种了BSMV:V1和BSMV:V2后,WSR1基因的表达量,第5天下降了50%,第25天下降到了12%,说明沉默WSR1基因成功引起了基因相对表达量的下降。
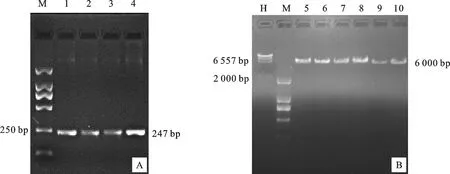
H:λDNA/HindⅢ marker;M:Marker 2000; 1和2:扩增片段V1;3和4:扩增片段V2;5~10:α、β、γ、γ-PDS、γ-V1(S1)和γ-V2(S2)。

从左至右分别是:接种BSMV:00第15天的叶片,接种BSMV:PDS第5天、10天、20天的叶片。
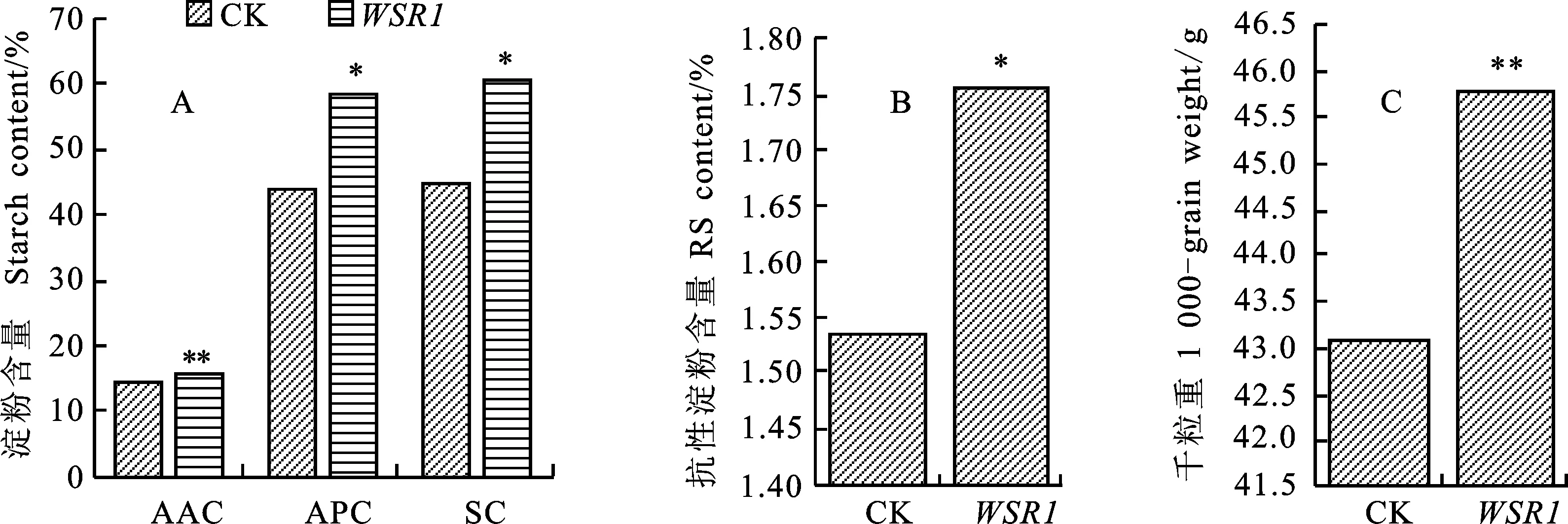
淀粉含量测定结果如图8所示,在接种了BSMV:WSR1之后,接种材料的直链淀粉含量由14.47% 增加到15.60%,千粒重由43.1 g增加到45.8 g。t测验结果表明,直链淀粉含量、千粒重与对照的差异均达到极显著水平。支链淀粉含量由43.95%增加到44.80%,总淀粉含量由58.42%增加到60.40%,抗性淀粉含量由1.53%增加到 1.76%。t测验结果表明,支链淀粉含量、总淀粉含量、抗性淀粉含量与对照的差异均达到显著水平。说明沉默WSR1基因后促进了淀粉合成,淀粉含量、千粒重增加。
3 讨 论
植物AP2/ERF是一个庞大的转录因子基因家族,参与多种生物学过程,包括种子发育、植物生长、病菌防御、果实发育、高盐、干旱等[27]。本研究利用同源克隆技术和RACE技术获得了小麦WSR1基因的全长cDNA,生物信息学分析表明,该基因含有AP2保守结构域。与已克隆的WSR1基因所编码的蛋白进行比较,发现WSR1基因所编码的蛋白序列与大麦、山羊草、二穗短柄草的亲缘关系比较近,与水稻、麻竹、高粱、拟南芥、大豆和苜蓿的亲缘关系比较远。因此推断WSR1基因为AP2/ERF家族基因内调控种子中淀粉含量发育的转录因子。
*和**分别表示与对照的差异显著(P<0.05)和极显著(P<0.01)。
AAC:直链淀粉含量; APC:支链淀粉含量;SC:总淀粉含量;RS:抗性淀粉。
BSMV-VIGS 技术的作用机制是 RNA 干扰,与研究基因功能的传统方法相比具有高效、高通量、避免突变、克服基因功能重复等优点;但它也存在诱导的靶基因未100%沉默、靶基因沉默不同步、沉默后被解除等局限性[28]。马 猛[29]用BSMV-VIGS技术沉默小麦的PSD基因,以麦穗和籽粒作为接种的受体组织,将BSMV-VIGS技术应用到了小麦品质基因的研究上。李淼淼[30]也应用 BSMV-VIGS 技术诱导沉默了淀粉合成相关基因SBEⅡa和SSⅡa,抑制了淀粉的合成。利用摩擦接种时,由于局部摩擦不均或浓度不均匀及病毒扩展时效问题,可能导致病毒在穗部的发展不均,基因表达不尽相同。因此要排除穗部的部分区域感染病毒不均引起的实验干扰。本研究探索了BSMV-VIGS技术在小麦穗部进行WSR1基因沉默的相对最优条件,成功构建了WSR1基因的 BSMV 重组载体,并用实时荧光定量 PCR 从分子水平对实验结果进行了验证,同时还证明 BSMV-VIGS技术诱导淀粉合成转录因子沉默的方法是可行的。
小麦籽粒淀粉合成的主要场所是造粉体,光合产物以蔗糖的形式运输到合成器官的胚乳细胞中,然后水解形成葡萄糖-1-磷酸(G-1-P),在造粉体内腺苷二磷酸葡萄糖焦磷酸化酶(ADP-Glc pyrophosphorylase,AGPase)催化其 ATP 反应生成腺苷二磷酸葡萄糖(ADP-glucose,ADPG)参与淀粉的合成。在造粉体内淀粉合成的最后阶段还有 3 个关键调控酶:淀粉合成酶(starch synthase,SS)、淀粉分支酶(starch branching enzyme,SBE)、淀粉脱分支酶(starch debranching enzyme,DBE)[31]。转录因子是能与目的基因5′端上游序列结合,调控目的基因时空表达功能的蛋白质,分为特异型和非特异性转录因子。它们既可以有选择性的调控目的基因的表达;也可以对基因的转录表达调控没有选择性,如拟南芥的CBF1转录因子。本研究通过VIGS技术,在WSR1基因中选取2个不同部位的片段进行沉默,沉默的2个片段均影响了小麦淀粉的合成;在接种了BSMV:WSR1之后,直链淀粉含量、支链淀粉含量、总淀粉含量、抗性淀粉含量及千粒重均有显著提高,表明冀麦325在接种WSR1基因后从表型上和内部组织中均表现出淀粉含量增高的现象,说明WSR1基因参与了小麦淀粉合成的过程,是抑制淀粉合成的转录因子。
