DNA条形码技术在野生哺乳动物物种鉴定中的可行性
2019-02-26阿迪力艾合麦提阿依努尔阿卜杜艾尼布威海丽且姆阿巴拜科日2伊拉木江托合塔洪塔依尔江买买提马合木提哈力克
阿迪力·艾合麦提 阿依努尔·阿卜杜艾尼 布威海丽且姆·阿巴拜科日,2伊拉木江·托合塔洪 塔依尔江·买买提 马合木提·哈力克*
(1.新疆大学生命科学与技术学院,乌鲁木齐,830046;2.新疆维吾尔医学专科学校,和田,848000)
DNA条形码技术(DNA Barcoding technology)是通过对一个标准目的基因的DNA序列进行分析从而对物种快速鉴定的技术[1-2]。基于线粒体DNA(mtDNA)细胞色素C氧化酶亚单位基因I(cytochromeCoxidase I,COI)序列的 DNA 条形码技术具有操作简便、准确、不受鉴定经验和标本生态特征的限制等优势,在鱼类、鸟类、昆虫类的分类鉴定中广泛应用[3-7]。
新疆是我国面积最大的一个省区,约占我国国土总面积的1/6[8]。新疆的地域辽阔,远离海洋,具有典型的大陆性气候特点,复杂的地形形成“三山夹两盆”的地貌轮廓。气候、植被类型多样等生态环境为野生动物提供了良好的栖息繁衍条件[9]。虽然新疆野生动物资源很丰富并且国家相关机构对野生动物资源的保护提出了一系列对策,但是由于很多野生动物长期被用于饮食、医药、观赏和传统需求并且有关监管部门在鉴定方面存在困难,而野生动物非法盗猎、贸易现象时有发生,导致野生保护动物资源严重的损害[10-16],所以在规范管理和保护执法的过程中,野生动物物种鉴定起着重要的作用。本研究采用DNA条形码技术对新疆野生哺乳动物mtDNA COI基因进行分析,找出鉴别新疆野生哺乳动物的最好方法,给海关、森林公安、工商管理等有关执法部门的物种鉴定提供科学依据和数据库的建立提供相关资料。
1 材料与方法
1.1 实验材料
本研究以已知的新疆野生哺乳动物作为研究对象。实验样本少数的在实验室保存好的以外,其他的来自乌鲁木齐林业局。取样时为避免个体重复,取样时保证每一份标本来自不同的个体,共包括3目6科12种52只野生哺乳动物,所有标本在95%酒精保存备用,标本信息见表1。
1.2 方法
1.2.1 试剂及仪器
肌肉、粪便、血液标本基因组DNA提取的试剂分别参考赵建军、日沙来提、萨姆布鲁克等人的方法配制[17-19]。COI基因通用引物LCO1490,序列为5′-GGTCAACAAATCATAAAGATATTGG-3′,HCO2198序列为5′-TAAACTTCAGGGTGACCAAAAAATCA-3′,引物由上海生工生物工程有限公司合成。PCR仪(德国,型号:T personal);电泳仪(北京市六一仪器厂,型号:DYY-10C);凝胶成像仪(美国伯乐公司,型号:Gel Doc XR+)。
1.2.2 DNA提取
模板DNA提取:参照赵建军、日沙来提、萨姆布鲁克[17-19]等人的方法从肌肉、粪便、血液中提取基因组DNA并纯化,测OD260、OD280定量、溶解后在4℃保存备用。
1.2.3 PCR反应
反应体系为10×缓冲液6.0 μL,2.5 mM dNTP 6.0 μL,3.7 nmol引物各 2.0 μL,TaqDNA聚合酶 1.0 μL,模板DNA 4.0 μL,ddH2O 29.0 μL。PCR反应条件:95℃变性1 min,50℃退火1 min,72℃延伸90 s,35个循环;72℃终延伸7 min。扩增产物经1%琼脂糖凝胶电泳检测,并送上海生工生物工程有限公司进行双向测序。
1.2.4 DNA序列分析
采用Chrosma 2.3软件读取基因序列并峰图质量进行判断。若峰图质量不理想不能准确地判断碱基的,则重新进行PCR扩增和测序。用MEGA7软件进行比对,同时将成功测定的序列在美国国家生物技术信息中心(NCBI)网站上进行BLAST同源性比较,以确定测出来的序列是否正确。使用DnaSP 6.1 软件分析52条序列的遗传多样性参数。运用MEGA7软件分析碱基组成,基于Kimura-2-parameter(K2P)[20]模型的不同替换类型计算各物种的种内和种间遗传距离。同时采用邻居法(Neighbor jointing,NJ)和最小进化法(Maximum evolution,ME)构建全部COI基因序列的NJ和ME分子进化树,对进化树进行内部分支检验与1 000次Bootstrap检验分析,从而确定个支系的置信度。
表1 实验样本信息

Tab.1 Experimental sample information
2 结果
2.1 基因组DNA浓度测定结果
基因组DNA浓度测定结果:CTAB裂解法提取的野生动物哺乳总DNA样本用紫外分光光度计测量260 mn、280 mn处的OD值,依据公式计算DNA浓度(μg/mL)=稀释OD260×50×稀释倍数。DNA纯度=OD260/OD280,所得结果为1.69。
2.2 COI基因扩增结果
共检测片段长度为642~729 bp的52份样本,实验采用的所有标本都成功扩增出来,这个结果与预期的目标片段COI基因的长度相符合,取每种野生哺乳动物PCR产物进行电泳,未出现引物二聚体和非特异杂带,结果见图1。

图1 11种野生哺乳动物PCR扩增结果Fig.1 PCR amplification product of 11 wild mammals 注:M:DL2000 DNA Marker;1.盘羊Ovis ammon;2.鹅喉羚Gazella subgutturosa;3.藏羚Pantholops hodgsoni;4.岩羊Pseudois nayaur;5.北山羊Capra sibirica;6.藏原羚Procapra picticaudata;7.蒙古野驴Equus hemionus;8.雪兔Lepus timidus;9.雪豹Uncia uncia;10.马鹿Cervus elaphus;11.狍Capreolus pygargus
2.3 遗传多样性和碱基组成分析
该研究所得的全部COI基因序列用DnaSP 6.1软件分析,结果表示:52条COI基因序列分到29个单倍型,共发现256个多态位点,391个突变位点。单倍型多样性Hd:0.965,核苷酸多样性Pi:0.1396。52条COI基因序列碱基分析结果所示:全部COI基因序列当中A、T、C和G的平均含量分别为26.3%、30.3%、26.0%和17.4%,A+T含量(56.6%)高于C+G含量(43.4%)。最高的狍A+T含量61.1%,G+C含量38.9%。最低的蒙古野驴A+T含量53.5%,G+C含量46.5%。
2.4 COI基因序列同源性比对
对新疆3目6科12属12种52只野生哺乳动物mtDNA COI基因序列进行同源性比较分析。结果COI基因序列同源性达到98%~100%,相似度达到90%~99%,结果见表2。
2.5 样本分子进化树
基于Kimura-2-parameter(K2P)模型的不同替换类型计算种内和种间遗传距离,发现替换类型Syn-Nonsynonomous、替换种类Nonsynonymous only时的结果最好,各野生哺乳动物种内遗传距离0%~2%,种间遗传距离为15%~42%(北山羊和雪豹各有1个标本,无法计算种内遗传距离)。基于COI基因序列构建6科12种野生哺乳动物系统发育树见图2。
表2 新疆12种野生哺乳动物样品测序比对结果
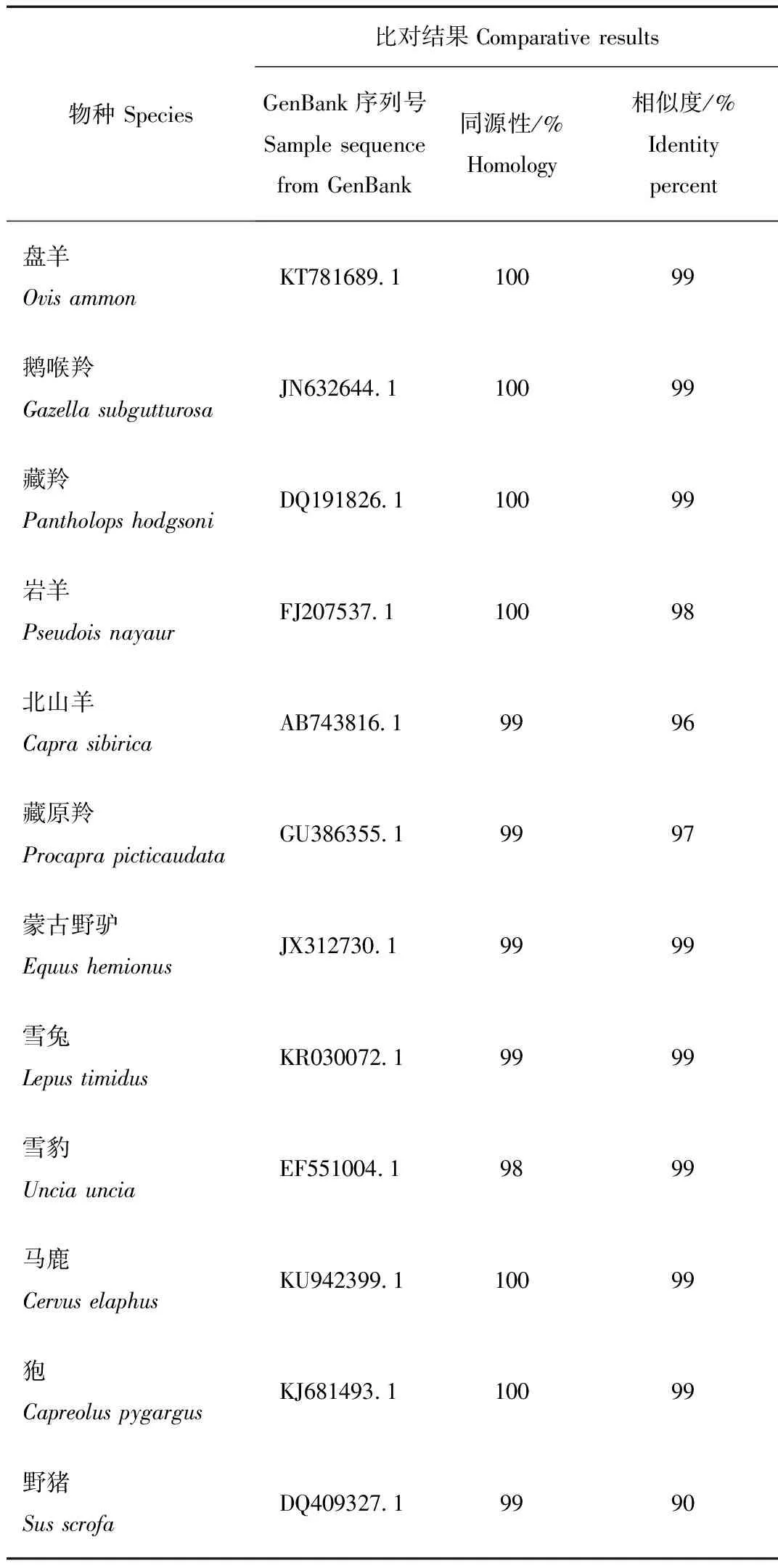
Tab.2 Comparative results of 12 species of wild mammals in Xinjiang
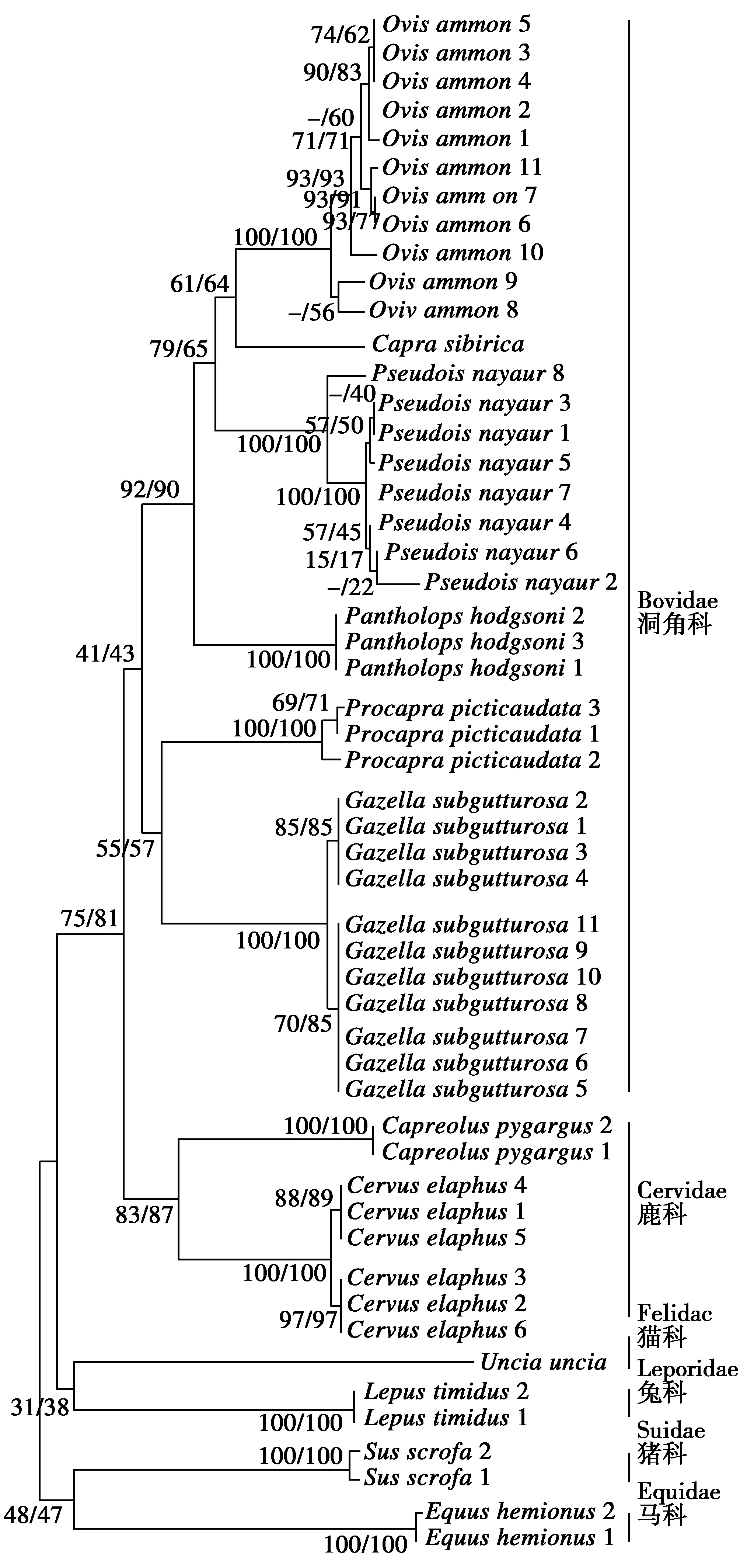
图2 基于COI基因序列构建的12种野生哺乳动物系统发育树Fig.2 12 Wild mammal phylogenetic trees constructed based on COI gene sequences 分支上的数字分别表示NJ树和ME树的置信度(分支有差异的不显示),每个标本用已知的学名来记标本号 The numbers on the branches indicate the confidence of the NJ tree and the ME tree(the difference on the branch does not display),each specimen number was record by the known scientific name of the sample
3 讨论
野生动物资源是一种可更新的,有着巨大生产力的自然资源。一直以来,野生动物资源为人们做出了很大的贡献,只要科学地保护和管理野生动物资源,就可以对它们持续利用而造福于人类[21]。目前新疆已被记录的野生脊椎动物有726种。就哺乳动物而言,新疆有154种野生兽类,占中国哺乳动物种类的37.4%,其有很多新疆野生珍稀特有物种[22-23]。采用DNA条形码技术能进一步提高海关、工商管理、森林公安等有关执法部门的物种鉴定水平并弥补其他物种鉴定方法,控制野生保护动物的贸易、偷猎、饮食等非法行为,从而为野生动物的科学保护和管理起重要作用。
本研究对采集于新疆3目6科12属12种52只野生哺乳动物mtDNA COI基因进行研究。本实验研究对象包括:录入国务院1988年7月批准的中国《国家重点保护野生动物名录》的国家级重点保护的动物有10种,其中Ⅰ级保护动物有4种(雪豹、蒙古野驴、藏羚和北山羊),Ⅱ级保护动物有6种(马鹿、盘羊、岩羊、藏原羚、鹅喉羚和塔里木兔);录入中国《国家保护的有益的或者有重要经济、科学研究价值的陆生野生动物名录》的有2种(狍和野猪)[24]。从研究结果发现:经典酚-氯仿法、改良的CTAB裂解法适用于取提取新疆野生动物基因组DNA,本研究所得的DNA纯度1.69,纯度比值在1.6~1.8范围内说明所得的DNA纯度较高[25]。物种多样性、遗传多样性分析结果虽然比较高,但人均资源量还是很少,加上我国经济快速发展时期,因此,野生动物资源的保护仍然面临着严峻的形势。mtDNA COI基因碱基组成分析结果有明显的碱基偏倚性,这个结果符合线粒体DNA的碱基组成特点。已知的52种野生哺乳动物mtDNA COI 基因序列同源性比较,发现已知的52只野生哺乳动物和mtDNA COI基因序列同源性鉴定结果高度一致,说明测出来的序列是正确的。从系统进化树可见,基于Kimura-2-parameter(K2P)模型计算各野生哺乳动物的种内和种间遗传距离,替换类型为Syn-Nonsynonomous、替换种类为Nonsynonymous only时种内遗传距离0%~2%,种间遗传距离为15%~42%,二者的分离程度比其他类型较远,发生误判的概率就很低。两种方法构建的系统进化树拓扑结构大致相同,同一个科、属和种均能够被明确地分入相应的分支,但是置信度较低,仍存在不足。此外,各个种间遗传距离不同,而现有的DNA条形码COI基因数据的物种判断阈值可能对物种鉴定造成一定误判。
综上所述,DNA条形码COI基因能够用于新疆野生哺乳动物的分类鉴定,该技术不仅弥补传统物种鉴定中的模式标本的依赖性、专业经验方的限制,而且在动物个体不完整的情况下进行种类鉴定拥有绝对优势。它将对野生动物资源的保护,控制野生保护动物的贸易、偷猎、饮食等违法行为等方面起到积极作用,为海关、森林公安、工商管理等有关执法部门提供有效、快速、精确的物种鉴定平台,保护野生动物资源中有重要意义。
