生物医学期刊开放代码政策调研
2019-02-21冯昌扬陈雨雪
■冯昌扬 陈雨雪
1)武汉大学信息管理学院,湖北省武汉市武昌区八一路299号 4300722)福建船政交通职业学院,福建省福州市仓山区首山路80号 350007
在学术交流活动中,越来越多的学术期刊意识到开放代码的好处,开始鼓励作者共享代码,并要求他们在每篇论文中包含一份关于代码可用性的声明。开放代码指的是可供免费分发和重复使用的计算机代码(软件),其源代码不受限制[1]。结合Easterbrook的观点[2],本研究将“开放代码”定义为在某些平台上自由发布代码、模型和算法的过程,因此,其他学科的研究人员可以对这些代码进行分析,并可以重新运行代码来验证结果。例如,为了解决日益复杂的数据和分析问题,《科学》(Science)扩展了数据访问要求,包括涉及数据创建或分析的代码[3];《自然》(Nature)、《自然方法》(NatureMethods)、《自然生物技术》(NatureBiotechnology)和《自然神经科学》(NatureNeuroscience)等期刊鼓励作者提供源代码、安装指南和样本数据集,以供审稿人检查[4];《生物统计学》(Biostatistics)创建了“再现性副主编”(Associate Editor for Reproducibility)这一职位,致力于根据收到的数据和代码重复论文;《生物信息学》(Bioinformatics)要求作者在提交论文时描述如何访问其软件,并在文章的标题页中指明能够访问源代码的统一资源定位符(Uniform Resource Locator, URL)[5];《内科医学年鉴》(AnnalsofInternalMedicine)则要求作者说明他们是否愿意在论文出版后分享他们在研究中开发和使用的源代码、数据和协议[6]。
鉴于期刊对科学传播的重要作用和对研究人员具有较大的影响,国外一些学者已经展开期刊开放代码政策的相关研究,如Stodden等[7-8]通过评估样本期刊的数据共享政策、代码共享政策、补充材料政策和开放获取状态,建立了期刊采用开放数据和代码政策的预测模型;通过向作者请求数据和代码并尝试复现已发表的结果来评估开放代码政策的有效性,研究发现,作者的数据和代码发布情况比没有推出开放代码政策时有所改进,但目前还不足以复现实验结果。此外,Rowhani-Farid等[9]发现《生物统计学》采用再现性政策奖励带有数据和代码共享徽章的文章,他们通过样本期刊文章的提交日期绘制代码共享概率,并进行贝叶斯逻辑回归建模,发现生物统计学期刊的徽章并没有影响代码共享。如前所述,生物医学领域的研究人员更多地关注代码可用性及可重复性,然而笔者通过文献调研发现我国生物医学领域期刊鲜有对开放代码提出要求,目前也鲜有关于期刊开放代码政策的研究,因此,本研究试图采用内容分析法调研生物医学期刊开放代码政策的相关特征。
1 数据来源与概况
本研究使用的数据集来自2017年《期刊引证报告》(JournalCitationReports,JCR)中的生物医学研究期刊,这些期刊广泛分布于Web of Science(WoS)数据库的生物化学和分子生物学、生物学、细胞生物学、晶体学、发育生物学、生物医学工程、免疫学、医学信息学、微生物学、显微术、多学科科学和神经科学等分类中。由于生物医学期刊数量较多,本研究将研究样本限制为Q1区的期刊。具体检索式为:Select Categories:Biochemistry & Molecular Biology,Biology,Cell Biology,Crystallography,Developmental Biology, Engineering、Biomedical,Immunology,Medical Informatics,Microbiology,Microscopy,Multidisciplinary Sciences,Neurosciences; Select JCR Year:2017; Select Edition:SCIE; Category Scheme:WoS; JIF Quartile:Q1。
初始数据集包括299种期刊。经过人工审核,排除非英语语种期刊、简短报告和评论期刊、基础医学或临床研究期刊,最终确定的研究样本包括152种期刊,占JCR该领域Q1区期刊总量的50.84%。从JCR报告中获取的数据,包括期刊标题(Full Journal Title)、期刊的总被引频次(Total Cites)、影响因子(Impact Factor)和特征因子分值(Eigenfactor Score)。2017年各影响因子区间的期刊数量和相应的占比如表1所示。提取这些期刊的期刊政策(Information for Authors和Editorial Policies)作为编码文本。

表1 期刊影响因子分布
2 研究方法
采用内容分析法研究国外期刊开放代码政策。内容分析法是从文本语料库中发现定量模式的有效方法。在内容分析法中,编码是数据收集和数据解释之间的关键联系,它可以为研究人员提供一套系统的指导方针(即编码方案)来解释数据。
编码的第一步是确定研究目标并创建一个明确的编码方案。本研究结合Stodden的编码方案[7],创建一个编码草案。为了补充在拟定草案时未注意到的元素,笔者采用基础理论方法将草案应用于数据集的一个子集,以帮助进一步改进编码方案,得到最终版编码方案(表2)。
两位具有编程背景,并具有文本编码经验的信息科学专业的研究生作为编码人员,对50个随机抽取的期刊样本进行编码。本研究使用Cohen′s kappa系数来测量编码人员之间的可信度(Interrater Reliability,IRR),其IRR为0.8,这为一位编码人员提供了足够的可靠性来编码所有样本。

表2 最终版编码方案
3 研究结果
3.1 开放代码政策概况
在152种样本期刊中,40种(26.32%)期刊将开放代码作为发表条件, 57种(37.5%)期刊明确鼓励开放代码,但并未强制要求,55种(36.18%)期刊未提及任何有关开放代码的内容(表3)。

表3 样本期刊开放代码政策的概况
3.2 期刊对公开代码的处理方式
65种(67.01%)期刊没有明确提及公开代码的处理方式,2种(2.06%)期刊愿意托管作者提交的代码,30种(30.92%)期刊会对代码进行审核。与仅鼓励作者开放代码的期刊相比,将开放代码作为发表必要条件的期刊审核代码的概率更大(表4)。

表4 样本期刊对公开代码的处理方式
3.3 期刊推荐的代码公开方式
75种(49.34%)期刊建议作者通过公共在线存储库开放代码,2种(1.32%)期刊推荐通过期刊托管方法,2种(1.32%)期刊建议读者请求作者共享,73种(48.03%)期刊没有指定代码公开方式(表5)。
在要求开放代码(编码为A-3)的40种期刊中,36种期刊建议通过公共存储库开放代码,4种期刊没有指定代码公开方式。
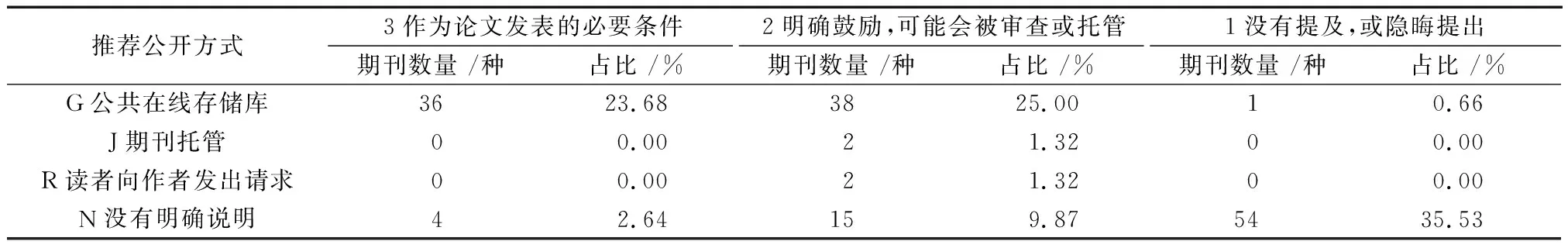
表5 样本期刊推荐的代码公开方式
3.4 开放获取期刊的开放代码概况
对开放获取期刊的开放代码概况进行Fisher精确检验(Fisher′s Exact Test),结果得出P值为0.007,表明开放代码政策强度与期刊访问模式之间差异有统计学意义。笔者进一步将编码A分为两大类,期刊政策将开放代码列为必需条件(编码为A-3的期刊)和期刊政策认为开放代码是非必需的(编码为A-2或A-1的期刊),并使用卡方检验(Chi-Square Test)来测试此关联,发现两类期刊开放代码政策的差异有统计学意义(P=0.009)。两个结果都表明,开放获取期刊更有可能要求开放代码。
3.5 不同影响因子期刊的开放代码要求

图1 不同影响因子期刊的开放代码要求
图1所示为2017年每个开放代码级别期刊的影响因子中位数。2017年具有最强开放代码政策(编码为A-3)的期刊,其期刊影响因子中位数为6.772;然而,没有提及开放代码(编码为A-1)的期刊的影响因子中位数为5.186。
影响因子与编码A的差异具有统计学意义(Kruskal-Wallis检验,P<0.0001)。通过检查编码A中各类别之间的成对差异,笔者发现将开放代码政策作为论文发表必要条件(编码为A-3)的期刊具有显著高于期刊政策认为开放代码是非必需(编码为A-2或A-1)的期刊影响因子(Wilcoxon检验,均为P<0.0001)。笔者进一步将期刊政策分为两类,即期刊政策将开放代码列为必需条件(编码为A-3的期刊)和期刊政策认为开放代码是非必需的(编码为A-2和A-1的期刊),发现需要开放代码期刊的影响因子显著增加(Wilcoxon检验,P<0.0001)。
3.6 期刊开放代码版权声明概况
只有60种(61.86%)期刊明确提到版权或许可,即使是要求开放代码(编码为A-3)的期刊,也只有30种(30.93%)期刊提到版权或许可(表6)。

表6 期刊开放代码版权声明
4 讨论、结论与展望
4.1 讨论
从整体来看,生物医学期刊对开放代码有不同程度的要求,但大多数期刊没有明确提及公开代码的处理方式,这或许与这项工作背后需投入的巨大人力有关。但从另一方面考虑,期刊审核无异于一种作者代码监督机制,可以在一定程度上减少粗糙的代码。如若可行,期刊还可不定期跟踪作者共享代码的后续,如研究人员是否对代码进行更新和维护、代码的影响力如何等。
从代码公开方式来看,大多数期刊推荐作者使用GitHub等公共在线存储库分享代码,这与实际情况相吻合。在实践中,GitHub、FigShare、Zenodo和Bitbucket已成为预选的学术交流工具,尤以GitHub为甚[10]。GitHub成立于2008年,广泛用于存储、分享、更新数据集和软件代码。截至2018年6月13日,谷歌学术(Google Scholar)中有超过22.3万篇学术论文引用GitHub存储的代码[11]。GitHub在2018年7月的美国Alexa网站排名中位居32位[12]。
从期刊获取方式来看,开放获取期刊更倾向于出台开放代码政策,这也是开放科学的题中之意。尽管开放代码与开放获取、开放数据的目的不尽相同,开放代码更多地是为了让读者重复,甚至更新模型、算法、实验步骤,但开放获取、开放数据和开放代码之间存在着紧密联系,开放科学、数据共享、软件共享都是未来的发展趋势[13]。
从影响因子来看,影响因子与开放代码政策显著相关,高影响因子的期刊如《自然》《科学》等更有可能要求作者开放代码。但反过来,影响因子计算方法中的总被引频次是根据WoS所收录的SCI期刊论文对该期刊两年内发表论文的引用情况计算出来的,那么开放代码是否与期刊被引频次存在相关关系,并对期刊影响因子产生影响,则是笔者在下一个研究中讨论的问题。
从版权声明来看,明确提及版权声明的期刊所占比例不高,知识产权是否是作者不愿分享代码的原因还有待考量。但从既往研究来看,Stodden等[14]从用户角度对机器学习社区进行调查,并指出了开放代码没有得到广泛实践的原因,其中权属不清晰占44%,其次是专利问题(40%);Barnes[15]也发现公开代码与机构知识产权相悖,这是科学家没有公布其代码的原因之一 。可见,知识产权问题确是影响作者共享代码的因素之一,至于如何拟定版权声明,包括代码公开范围、使用范围、程度等需要声明的条款,则可以成为后续研究考虑的问题。
4.2 结论
普遍和大规模的计算正在改变人们对科学方法的实践。如果没有代码,就会导致所提供的信息不足,影响他人再现已发布的计算结果。在这项研究中,笔者试图了解期刊在出版结果的代码公开可用性方面发挥的作用。
本研究通过对2017年JCR中Q1区的152种生物医学期刊开放代码政策的相关特征进行分析,发现开放获取期刊比传统订阅制期刊的开放代码政策更普遍;公共在线存储库如GitHub等是大多数期刊推荐的公开代码存储平台;高影响因子期刊比低影响因子期刊更有可能出台开放代码政策。
4.3 局限与展望
本研究的局限性主要体现在样本的选择上:(1)由于选取的是JCR中生物医学Q1区的期刊,鉴于样本期刊已具有较高的影响力,它们选择通过开放获取来扩大影响力的可能性相对较低,因此本研究的样本期刊选取具有一定的局限性;(2)部分期刊如《自然》影响因子显著高于Q1区期刊影响因子的平均水平,这可能对显著性检验产生了一定影响;(3)笔者假设年轻期刊更有可能出台开放代码政策,并意图研究期刊创刊年份对开放代码政策的影响,但由于JCR Q1区期刊多为老牌期刊,不适合开展此研究。
后期研究,笔者将集中在以下3个方面:(1)笔者在对期刊政策进行编码时发现,一些出版社旗下期刊存在共用该出版社同一套期刊政策的现象,出版社对开放代码政策的影响可以进行回归分析和预测分析;(2)期刊开放代码政策的有效性,即验证作者的执行情况,甚至可以对比期刊论文开放代码出台政策前后有何不同,以检验开放代码政策的影响力;(3)研究期刊创刊年份对开放代码政策的影响,是否年轻期刊出台开放代码政策的可能性更大。
作者贡献声明:
冯昌扬:确定选题,提出研究框架,撰写论文初稿;
陈雨雪:文献检索,图表制作,修改论文。
