PCR快速检测淋病奈瑟菌NspA基因方法的建立及分析
2019-02-10王彦王琨刘小林刘霞王西珍
王彦 王琨 刘小林 刘霞 王西珍

【摘要】目的 构建PCR检测淋病奈瑟菌NspA基因的方法,为寻找快速检测淋病奈瑟菌的方法提供理论依据。方法 根据NspA基因序列设计一对引物,从10例已成功培养出淋病奈瑟菌的患者尿道分泌物中用PCR方法扩增NspA基因,并测序鉴定。结果 1.0%琼脂糖凝胶电泳显示成功获得一个约528 bp的基因片段,基因测序鉴定与本实验设计相符。结论 淋球菌常规培养检测和鉴定需2~3天的时间,而PCR快速检测淋病奈瑟菌NspA基因,只需2~3小时便能获得数据,缩短了检测时间,给临床诊治赢得宝贵时间。
【关键词】PCR;淋病奈瑟菌;NspA基因
【中图分类号】R392 【文献标识码】A 【文章编号】ISSN.2095.6681.2019.29..02
淋病病原体是淋病奈瑟菌(Neisseria gonorrhoeae,NG)俗称淋球菌,它的唯一天然宿主是人类。淋病奈瑟菌NspA基因是淋球菌表面蛋白A的表达基因,有研究发现NspA抗原不但在细菌表面持续表达,而且高度保守,淋球菌NspA氨基酸序列在淋球菌菌株之间的同源性高达98%。本研究旨在寻找一种快速检测淋球菌的方法,为临床诊治赢得宝贵时间。
1 材料与方法
1.1 标本采集
收集自2016年9月~2018年3月我院皮肤性病科、男性科、泌尿外科门诊疑似淋病患者,其中男性取尿道分泌物,女性取距宫颈口1~1.5 cm处分泌物,装入无菌试管,-80℃冷冻备用。从中选取10例经VITEK-2 COMPACT全自动微生物分析仪鉴定为淋病奈瑟菌的患者分泌物用PCR方法扩增NspA基因。
1.2 方法
1.2.1 设计和合成引物
以淋球菌DNA为模板,根据GeneBank报道的NspA (gi:26324161) 核甘酸序列,利用Primer Premier 5.0软件自行设计引物和探针,由invitrogen公司合成。引物如下:
上游引物 P1: 5′-GCATGAAAAAAGCACTTGCC-3′
下游引物P2: 5′-CCGTCAGAATTTGACGCGCAC-3′
用PCR来验证所设计引物的特异性,并使用淋球菌标准菌株CMCC 29403作为阳性对照;用可能成为尿道感染致病菌的标准菌株大肠埃希菌ATCC25922,作为阴性对照(由江苏省临检中心提供)。
1.2.2 DNA的提取和扩增
1.2.2.1 临床样本的DNA提取
把10例临床样本的棉拭子在磷酸盐缓冲液(PBS)中(2 mL)挤压洗1~2 min,并用Votex漩涡振荡器振荡1min,弃去棉拭子使分泌物尽量溶于生理盐水中,悬浮液放入离心机3000 r/min离心5 min(离心半径16 cm),弃上清液,用1mL无菌生理盐水悬浮细胞,放入离心机,3000 r/min离心5 min(离心半径16 cm),弃上清液,细胞重新溶于1×TE缓冲液中(100 μL),100℃加热煮沸10 min后放入离心机12000 r/min离心10 min(离心半径16 cm),上清液含DNA模板,用于PCR扩增。
1.2.2.2 阳性对照、阴性对照
按照《全国临床检验操作规程》要求,取淋球菌标准菌株CMCC 29403增菌后接种于巧克力培养基中培养,用苯酚-氯仿法提取淋球菌基因组DNA,PCR扩增NspA基因,作为阳性对照。以同样方法取标准菌株大肠埃希菌ATCC25922作为阴性对照。
1.2.2.3 NspA基因的PCR扩增
取已准备好的10例临床样本DNA,用设计的P1和P2为引物,PCR扩增NspA基因,25 μL反应体系如下:10×PCR Buffer 2.5 μL;10 μmol/L上游引物1 μl;10 μmol/L下游引物1 μL;dNTP 1.5 μL;Taq 酶0.5 μL;模板DNA 1μL;去离子水17.5 μL。反应条件为:94℃ 5 min;94℃ 60 S;58℃ 45 S;72℃ 1 min;共35个循环;最后72℃ 10 min。
1.2.2.4 NspA基因核苷酸序列鉴定
按PCR胶回收纯化试剂盒说明书纯化PCR产物,将经柱式胶回收纯化临床PCR产物送往INVITROGEN公司进行测序鉴定。
2 结 果
2.1 PCR扩增NspA基因
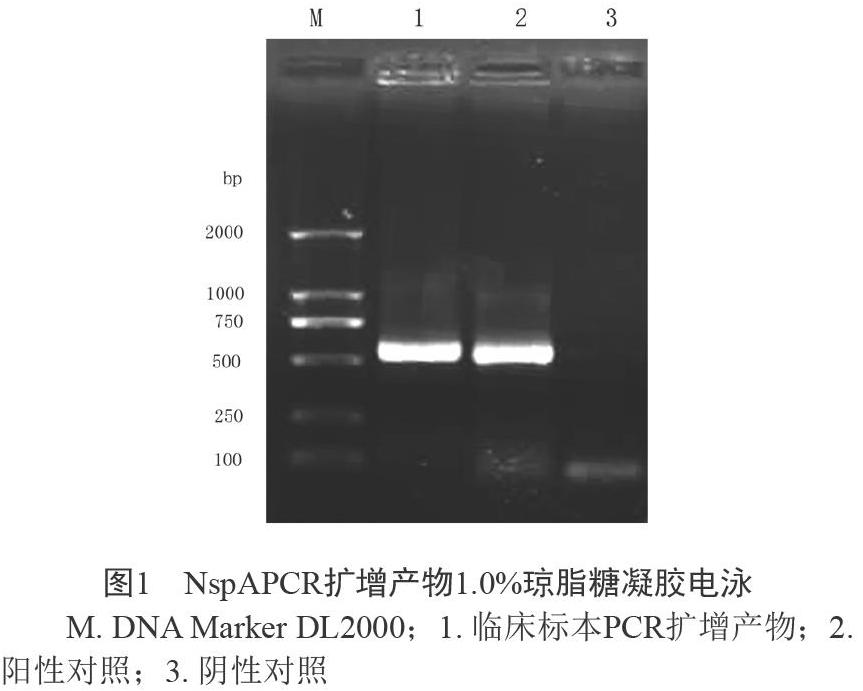
采用P1和P2为引物,对已准备好的10例临床样本DNA模板进行PCR扩增,在1.0%琼脂糖凝胶中电泳,得到一条与NspA预期相符大小约为528 bp的清晰条带,与本实验设计相符,见图1。
2.2 序列测定
本研究PCR扩增的10例临床NspA基因全长528 bp(含起始密码和终止密码),编码175个氨基酸。对测序结果作Blastn分析,结果发现PCR扩增的临床NspA基因序列与GeneBank报道的淋球菌(CMCC 29403)NspA(gi:215260656)核苷酸序列完全一致,见图2。
3 讨 论
淋球菌能够引起泌尿生殖系统的化脓性感染,临床表现为有典型的尿痛及尿道脓性分泌物,人类是它唯一的天然宿主。淋病好发于尿道引起淋病性尿道炎,如治疗不及时,还可侵入泌尿生殖器的其他部位,引起前列腺炎、附睾炎、盆腔炎、输卵管炎等,也可侵犯眼睛、咽部、直肠,甚至经血液播散到全身形成淋球菌性关节炎、心内膜炎、腱鞘炎等合并症。
目前一种用来放大扩增特定的DNA片段的分子生物学技术是聚合酶链式反应(PCR),其一次性地将反应液加好后进行变性-退火-延伸反应。扩增反应一般在2~4小时内完成,扩增产物一般用电泳分析,无放射性污染、易推广。
细菌培养法是世界卫生组织推荐诊断淋球菌的“金标准”,然而报告时间较长、操作繁琐。不少患者在就诊前都接受过非正规的抗生素治疗,再加上淋球菌生长条件苛刻,对外界抵抗力较低,增加细菌培养难度,影响检出率。而PCR检测法由于有较高的特异性和敏感性,因而广泛应用于医学领域,不管患者分泌物中的活菌或死菌都可检出[1]。一般细菌培养及VITEK.MS质谱鉴定,需要较长时间才能最后确定到菌种[2],应用VITEK.MS质谱技术来进行细菌鉴定,虽然有较完整的微生物图谱库进行结果比对[3],但仪器设备价格昂贵,中小型医院难以普及。淋球菌常规培养检测和鉴定也是需2~3天的时间,常常延误诊断和治疗。而PCR检测淋球菌保守的NspA基因只需2~3小时便能获得数据,具有较高的特异性及敏感性,缩短了检测时间,快速做出诊断,早发现、早治疗,给临床诊治赢得宝贵时间。
参考文献
[1] 赵晓兰,冯 琳.四种方法检测淋病奈瑟球菌的临床应用对比研究[J].中国性科学,2015(7):45-47.
[2] Lim SH,Mix S,Xu Z,et a1.Colorimetric sensor array allows fast detection and simultaneous identification of sepsis-causing bacteria in spiked blood culture[J].J Clin Microbiol,2014,52(2):592-598.
[3] Altun O,Botero-Kleiven S,Carlsson S,et a1.Rapid identification of bacteria from positive blood culture bottles by MALDI-TOF MS following short—term incubation on solid media[J].J Med Microbiol, 2015,64(1 1):1346-1352.
本文編辑:刘欣悦
