Trichoderma Reesei菌生物降解酶CIP 1和CIP 2的生物信息学分析*
2018-12-15魏雪莹陈加蓓陈思佳贺建武
张 红,魏雪莹,陈加蓓,陈思佳,贺建武,2
(1.吉首大学生物资源与环境科学学院,湖南 吉首 416000;2.吉首大学杜仲综合利用技术国家地方联合工程实验室,湖南 吉首 416000)
随着世界人口不断增长,环境污染问题日益严峻,化石能源面临枯竭,能源的供需矛盾进一步升级.生物质能源(Biomass Energy)是地球上最丰富的可再生物资源,有替代化石燃料的广阔发展潜力.在生物质能源中,纤维素和半纤维素类物质是最主要的部分.[1]农业生产中的玉米芯、玉米秸秆、小麦秸秆和水稻秸秆等废弃物,产量大、可再生,利用其木质纤维素类生物质可用来生产液体燃料和高附加值化合物,变废为宝,可望成为保障人类社会可持续发展的重要替代能源.
相对于物理降解和化学降解而言,微生物降解木质纤维素具有成本低、反应条件温和、对环境安全友好等优点,有报道[2]称来自真菌的木质纤维素降解酶通常可将木质纤维素材料水解为葡萄糖等碳水化合物.里氏木霉Trichodermareesei是多细胞的丝状真核微生物,是红褐肉座菌Hypocreajecorina的无性型酶,隶属于丛梗孢目Moniliales木霉属Penicillium[3],具有高效的纤维酶水解活性,是重要的纤维素降解模式菌株和工业菌株.虽然T.reesei基因组中仅包含200多种负责编码糖苷水解酶的基因,远少于其他丝状真菌,但其外分泌纤维素酶的能力较其他丝状真菌最高[4].2003年,Foreman等[5]发现有2个蛋白可以伴随着纤维素酶一起被诱导,并且协助纤维素酶对木质纤维素进行有效降解.这2个蛋白酶基因被分别命名为CIP 1和CIP 2.近期发现CIP 1与裂解酶在结构上有相似性,CIP 2属于CE15家族的葡萄醛酸酯酶[6],但其更详细的生物学信息没有得到阐述.笔者选取里氏木霉生物降解酶CIP 1和CIP 2,利用生物信息学方法,分析蛋白CIP 1和CIP 2的理化性质、二级结构、信号肽、定位、跨膜结构和磷酸化位点等信息,进行结构和功能的预测,了解蛋白CIP 1和CIP 2功能和它们与纤维素酶协同作用,以期实现纤维素酶更高效率的降解,降低工业上酶用量,减少生产成本.
1 材料与方法
1.1 蛋白基因序列获取
根据Forman[5]在GneBank(https:∥www.ncbi.nlm.nih.gov/)中提交的TrichodermareeseiQM6a菌株生物降解酶基因序列CIP 1(Genbank Accession Number:AY281370.1)和CIP 2(Genbank Accession Number:AY281368.1)获取相应的酶基因序列,以FASTA格式保存为计算机本地文件.
1.2 氨基酸分析
NCBI数据库应用在线程序ORFfinder(http:∥www.ncbi.nlm.nih.gov/gorf/orfig.cgi)读取开放阅读框,并且获取相应的氨基酸序列.利用ProtParam程序(http:∥web.expasy.org/protparam)分别对酶CIP 1和CIP 2进行氨基酸残基、分子量、理论等电点、脂溶指数、不稳定指数和氨基酸的亲水性和疏水性等进行分析.
1.3 二级结构分析
利用SOPMA在线程序(https:∥npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)进行分析,系统预测其α螺旋、延伸链、β折叠和无规则卷曲等次级结构.
1.4 蛋白信号肽预测与分析
通过SignalP 4.1 server(http:∥www.cbs.dtu.dk/services/SignalP)利用人工神经网络和HMM(Hidden Markov Models)的原理对氨基酸序列的信号肽存在情况和信号肽切割位点进行预测分析.
1.5 蛋白跨膜结构预测
使用TMHMM(http:∥www.cbs.dtu.dk/services/TMHMM-2.0)进行酶CIP 1和CIP 2二级跨膜域分析.
1.6 蛋白卷曲螺旋预测
利用EMBnet的COILS卷曲螺旋预测GO工具(http:∥www.ch.embnet.org/software/COILS_form.html)分别预测酶CIP 1和CIP 2的卷曲螺旋结构.
1.7 亚细胞定位分析
利用TargetP 1.1(http:∥www.cbs.dtu.dk/services/TargetP/)对酶CIP 1和CIP 2分别进行亚细胞定位分析.
1.8 三级结构预测
利用SWISS-MODEI(http:∥swissmodel.expasy.org/)对酶CIP 1和CIP 2氨基酸序列进行三级结构预测.
1.9 内含子和外显子检测
利用NCBI提供的Graphics工具,在NCBI搜索AY281370.1和AY281368.1之后,用NCBI提供的Graphics工具来查看,得到相应基因的内含子和外显子的序列,此序列同时也是一个基因结构图.
1.10 磷酸化位点预测
利用KinasePhos(http:∥kinasephos.mbc.nctu.edu.tw/)进行蛋白磷酸化位点预测分析.
2 结果与分析
2.1 基因序列与氨基酸序列
用相应的登录号在GneBank查找得到里氏木霉生物降解酶CIP 1和CIP 2的基因序列和编码的氨基酸序列,其中CIP 1基因序列长度为951 bp,CIP 2长度为1 383 bp,CIP 1编码氨基酸数为316,CIP 2为460.
2.2 氨基酸分析
利用ProtParam程序[7]分析酶CIP 1和CIP 2的理化特性,得到相应的氨基酸理化特性分析数据(表1)、氨基酸组成(表2).结果表明,编码CIP 1和CIP 2的氨基酸一级结构残基、分子量、脂溶指数等都存在差异,其中CIP 1与CIP 2的理论等电点分别为4.93和7,CIP 1为酸性蛋白.根据ProtParam的算法[8],不稳定指数小于40时,预测的蛋白在试验中比较稳定,反之则较差.CIP 1与CIP 2的不稳定指数分别为26.53和37.67,则CIP 1和CIP 2属于稳定蛋白.CIP 1的平均疏水性预测结果为负值,CIP 2的平均疏水性预测结果为正值,则很有可能CIP 1为亲水性蛋白,CIP 2为疏水性蛋白.从表2可以看出,酶CIP 1和CIP 2都由20种氨基酸组成,其中Gly,Ser,Thr和Ala组成比例相对较大,且都不含Pyl和Sec.
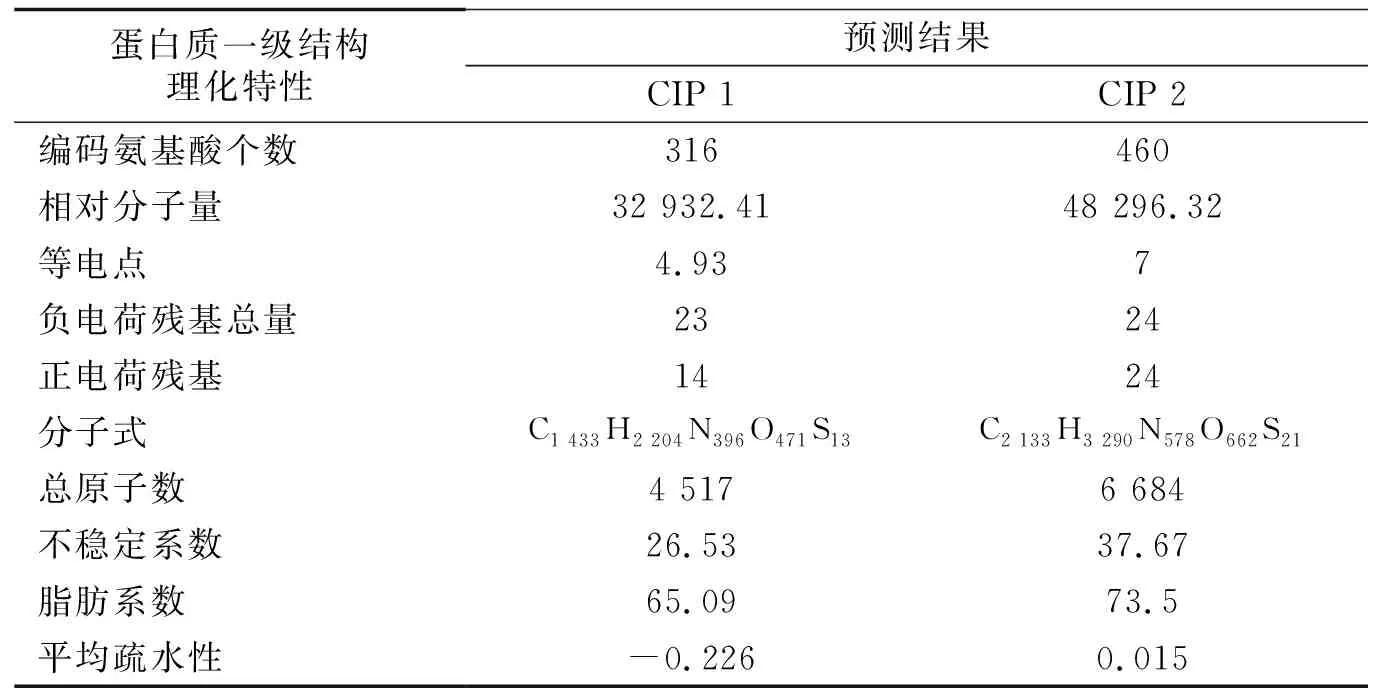
表1 降解酶CIP 1和CIP 2的一级结构预测分析

表2 里氏木霉生物降解酶CIP 1和CIP 2的氨基酸组成
2.3 蛋白质二级结构
利用SOPMA在线程序得到酶CIP 1和CIP 2二级空间结构预测特征(表3).从表3可知,酶CIP 1和CIP 2均含有大量无规则卷曲,分别是56.01%和44.13%,其次是延伸链;CIP 2的α螺旋比例高于CIP 1,分别是22.83%和11.71%,两者的β折叠比例都相对较少;且酶CIP 1和CIP2 二级结构中的α螺旋和无规则卷曲分布较集中,而β折叠结构分布相对分散.

表3 里氏木霉生物降解酶CIP 1和CIP 2的二级结构预测分析
2.4 蛋白信号肽预测与分析
通过SignalP 4.1 server对酶CIP 1和CIP 2氨基酸序列利用神经网络模型进行信号肽分析,得到3种C,Y和S值计算结果(图1).对于一个典型的信号肽,C值和Y值趋向于+1,S值在剪切位点之前高,而在剪切位点之后变低[9].由图1可知:CIP 1的第20位氨基酸残基具有最高的原始剪切位点分值0.456,第3位氨基酸有最高的信号肽分值0.918,第1~19 位氨基酸残基的信号肽分值为0.896,第20位氨基酸残基具有最高的综合剪切位点分值0.629,信号肽与成熟肽链间的剪切位点很可能位于第19~20位氨基酸之间;CIP 2的第18位氨基酸残基具有最高的原始剪切位点分值0.786,第10位氨基酸有最高的信号肽分值0.974,第1~17位氨基酸残基的信号肽分值为0.951,第18位氨基酸残基具有最高的综合剪切位点分值0.862,信号肽与成熟肽链间的剪切位点很可能位于第17~18位氨基酸之间.

图1 降解酶CIP 1和CIP 2信号肽截图Fig. 1 Signal Peptide of Biodegrading Enzyme CIP 1 and CIP 2
2.5 蛋白跨膜结构预测
利用TMHMM得到的蛋白CIP 1和CIP 2二级跨膜域分析结果表明,氨基酸的位置在膜外的可能性评估分值均大于1,位于跨膜区和内部的可能性极小,可忽略不计,说明酶CIP 1和CIP 2无跨膜区域,且都位于膜外.
2.6 蛋白卷曲螺旋预测
利用EMBnet的COILS卷曲螺旋预测GO工具分别预测蛋白CIP 1和CIP 2的卷曲螺旋结构,结果显示蛋白CIP 1和CIP 2几乎不含有卷曲螺旋结构,表明该蛋白属于膜蛋白、结构蛋白和转录因子的可能性很小[10].
2.7 亚细胞定位分析
利用TargetP 1.1对酶CIP 1和CIP 2分别进行亚细胞定位分析(图2),结果表明CIP 1和CIP 2定位在分泌途径信号肽(SP)的可能性分别为82.6%和97.9%,定位于线粒体等其他部位的可能性较小,且定位结果可靠性较高,说明酶CIP 1和CIP 2为定位于胞外细胞间隙的分泌蛋白.
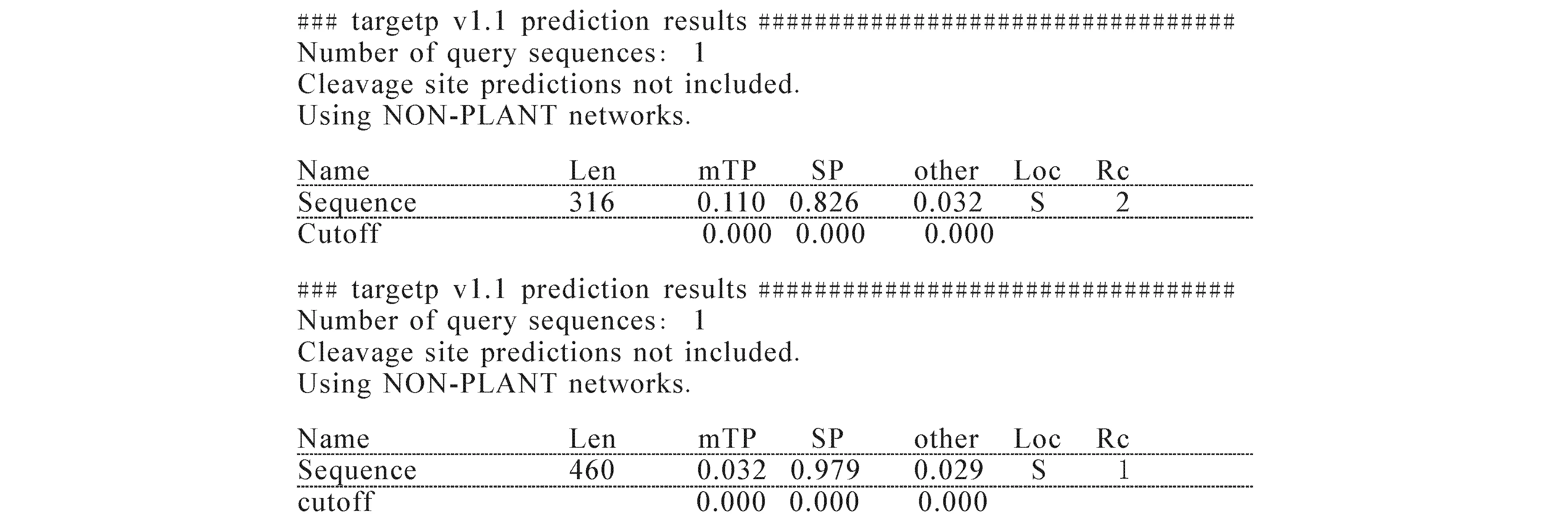
图2 里氏木霉生物降解酶CIP 1和CIP 2亚细胞定位截图Fig. 2 Sub-Cellular Localization of Biodegrading Enzyme CIP 1 and CIP 2
2.8 三级结构预测
应用Swiss-Model在线软件,根据同源建模理论预测里氏木霉生物降解酶CIP 1和CIP 2的三级结构(图3),结果表明CIP 1蛋白和CIP 2蛋白的三级结构主要由无规卷曲构成,延伸链的数目很多,α螺旋和β折叠比例相对较少,与二级结构预测结果一致.

图3 里氏木霉生物降解酶CIP 1和CIP 2的三级结构预测Fig. 3 Tertiary Structure of Biodegrading Enzyme CIP 1 and CIP 2
2.9 内含子和外显子检测
利用NCBI提供的Graphics工具在NCBI搜索AY281370.1和AY281368.1,结果显示CIP 1的FCBD是849~948这一段基因序列,CIP 2的CBM-1是64~147这一段基因序列,即CIP 1基因序列中的1~848,948~951属于内含子,849~948属于外显子,CIP 2基因序列中的1~63,148~1 383属于内含子,64~147属于外显子.
2.10 磷酸化位点预测
根据磷酸化位点分析结果,CIP 1共有14个磷酸位点,分别为9个Ser、3个Thr、2个Tyr;CIP 2共有10个磷酸位点,分别为7个Ser、3个Thr、0个Tyr.
3 讨论
基因功能最终通过其表达产物——蛋白质来实现,因此,要了解相关基因功能,最终也必须回到蛋白质上.CIP 1和CIP 2都存在一段信号肽,信号肽剪切位点分别在19~20,17~18位的氨基酸之间.蛋白质分子中的信号肽是引导新合成肽链转移到内质网上的一段多肽,位于新合成肽链的N端,由于信号肽又是引导肽链进入内质网腔的一段序列,又称为开始转移序列(start transfer sequence).在信号肽位置对其进行修饰,可以提高编码蛋白在体内特定位置的表达效率,为进一步表达该蛋白提供依据.信号肽的功能,不仅决定一个蛋白质是否为分泌蛋白,而且和蛋白质或其新生肽链在细胞内的全方位的定位有关.[11]综合亚细胞定位分析和蛋白跨膜结构预测结果,CIP 1和CIP 2都定位在分泌途径信号肽(SP),可能性分别为82.6%和97.9%,而定位于线粒体等其他部位的可能性很小.定位结果可靠性较高,且CIP 1和CIP 2都无跨膜结构域,在膜外,CIP 1和CIP 2都属于分泌蛋白.
酶CIP 1和CIP 2几乎不含有卷曲螺旋结构(coiled-coil,CC).构成CC的链都是α-螺旋,CIP 1与CIP 2的α-螺旋比例分别是22.83%和11.71%,含量不高.自然界中,CC是一个介导蛋白质相互作用或形成蛋白质骨架的通用结构域,CC常见于蛋白质的折叠结构中.多种跨膜蛋白的跨膜部分含有CC结构,在基因组数据库中,有20%~30%的产物被预测为跨膜蛋白[12].蛋白卷曲螺旋预测结果表明CIP 1与CIP 2属于膜蛋白、结构蛋白和转录因子的可能性很小,与跨膜结构预测结果能相互印证.
根据磷酸化位点分析结果,CIP 1含有9个Ser、3个Thr、2个Tyr,CIP 2含有7个Ser、3个Thr、0个Tyr,这些可能是酶的磷酸化位点.蛋白质磷酸化是蛋白质转录后修饰的一个重要可逆机制,它影响许多重要的细胞过程.一条蛋白质链的磷酸化一般只发生在丝氨酸(serine,S)、苏氨酸(threonine,T)、酪氨酸(tyrosine,Y)这3个残基上,在生命现象的许多关键调节机制中,蛋白质的磷酸化是重要的翻译后修饰,它与信号传导、细胞周期、生长发育以及癌症机理等诸多生物学问题有密切关系.[13]
4 结论
里氏木霉生物降解酶CIP 1和CIP 2均属于稳定蛋白,CIP 1是亲水性蛋白,CIP 2是疏水性蛋白;含有大量无规则卷曲,几乎不含有螺旋卷曲结构,都有一段信号肽.无跨膜结构域,并定位在分泌途径信号肽(SP)上,含有少量的磷酸化位点.为保证预测准确性,本研究对里氏木霉生物降解蛋白CIP 1和CIP 2的各种结构性质进行分析预测,均选用了多种不同软件,其所使用的原理和算法各有不同,但结果基本一致,能相互印证.因此,本研究结果具有较强的可信度.但由于生物信息学是根据已知的信息预测结果,所以还有很大的局限性,进一步确定蛋白质的功能和性质还需要实验的验证.
