Gateway技术构建菊芋块茎全长cDNA文库及EST测序分析
2018-12-06杨少丽
杨少丽,秦 松
(中国科学院烟台海岸带研究所,中国山东烟台264003)
菊芋(Helianthus tuberosusL.)俗名洋姜、鬼子姜,是一种兼具产业化开发及生态应用价值的植物,被联合国粮农组织称为“21世纪人畜共用作物”,开发前景十分广阔。菊芋病虫害少,产量高,适应性极强,能耐旱、耐贫瘠、耐盐碱。菊芋全身都可利用,是工业(燃料乙醇)、农业(饲料)、食品、保健品等行业的重要原料[1,2]。菊芋块茎含有丰富的菊糖,具有特殊的保健作用,既可以食用,又可进行深加工,制成菊粉、低聚果糖、食品添加剂、水溶性膳食纤维等功能食品[3,4]。另外,菊芋能在滩涂、盐碱地、边际地种植,不与地争粮,同时是优良的固沙植物,具有重要的生态应用价值,目前已被用于开发中国废弃盐碱地等[5~7]。
目前虽然有部分报道对菊芋Na+/K+转运蛋白基因、金属硫蛋白基因、果糖基转移酶基因等功能基因进行了克隆分析、初步转化验证等方面的研究[8~10],但功能基因的鉴定与研究工作仍然相对滞后,可利用的基因组信息较少,这在一定程度上阻碍了菊芋遗传资源的有效开发和利用。构建cDNA文库能够全面发掘组织、器官的新基因,是系统研究功能基因的重要方法。目前cDNA文库被广泛应用于大规模功能及比较基因组学、转录组学等研究,并成为生物、医学等领域克隆相关性状基因的重要方法和途径[11~13]。许多生物如欧洲油菜(Brassica napus)、水稻(Oryza sativa)、玉米(Zea mays)、葡萄(Vitis vinifera)等的cDNA文库已被构建,积累的表达序列标签(expressed sequence tag,EST)数据变得越来越庞大,极大地促进了相关物种功能基因组学的研究[14~16]。然而,菊芋EST报道尚少,在NCBI的GenBank数据库中公布的菊芋EST仅有4万余条,数量远低于玉米、水稻、葡萄等其他作物。因此,菊芋EST的获得,尤其是重要成熟期阶段EST,显得非常必要和迫切。
传统的cDNA文库构建涉及酶切和连接酶的连接步骤,而这一过程会使cDNA被酶切损失,也容易l成低拷贝数基因克隆的丢失。此外,建库过程需要PCR,这会使文库cDNA产生偏向性。利用Gateway技术构建文库,通过BP反应就可将cDNA片段重组至目的载体,整个过程无需酶切、连接,克隆效率非常高,并且能够较大程度地避免低拷贝数基因克隆的丢失,可使材料中mRNA的原始丰度在文库中得以真实的反映[15,17]。此外,应用Gateway技术成功构建入门载体后,通过LR反应即可将目的基因迅速且定向地重组到各种表达系统如细菌、酵母或昆虫中进行基因的表达分析[18,19]。
块茎是菊芋的可食部分,其中菊糖含量占干物质的80%以上,具有多种保健功能,是菊粉产业的主要原料[2~4]。而且,菊芋块茎中菊粉的聚合度(DP值)一般较低,其中DP值大于10的寡聚糖仅占20%左右,较易实现微生物转化,是燃料乙醇的理想底物[1,2]。对菊芋块茎组织基因的转录表达特征进行全面考察将有助于了解其特定的功能体系。本研究首次通过Gateway技术构建菊芋块茎全长cDNA文库,并进一步进行EST序列测定分析,以期丰富菊芋的遗传信息库,为菊芋的深入研究及开发利用提供分子信息。
1 材料与方法
1.1 总RNA的提取
新鲜菊芋块茎取自山东莱州盐碱地。将菊芋块茎用生理盐水冲洗干净并切成小块,放入研钵中加上液氮充分研磨后,加入Trizol提取液(Invitrogen公司,美国)按照说明书提取总RNA。使用分光光度计测量RNA浓度。RNA产量计算如下:RNA 产量(μg/μL)=A260×0.04 μg/μL RNA×稀释因子。用溴化乙锭染色的1%琼脂糖-甲醛凝胶电泳检测RNA质量。
1.2 mRNA的分离
使用FastTrack 2.0 mRNA分离试剂盒(Invitrogen公司,美国),按照说明书对符合要求的RNA进行mRNA分离。用1%琼脂糖凝胶电泳确认mRNA的质量,用紫外分光光度法测定mRNA浓度。
1.3 cDNA文库构建
对菊芋块茎mRNA纯化和质检后,使用CloneMiner域cDNA文库构建试剂盒(Invitrogen公司,美国)构建cDNA文库。合成双链cDNA后,加入T4DNA聚合酶使双链cDNA平末端化。向双链 cDNA 产物中加入 10 μL 5× Adapter Buffer、10 μL 1 μg/μL attB1 接头、7 μL 0.1 mol/L DTT,在T4DNA连接酶的作用下16℃孵育16~24 h,使得在双链cDNA的3′末端连接上attB1序列。将上述产物上样于cDNA Size Fractionation Columns,对cDNA片段进行分离,收集大小主要在500~4 000 bp之间的双链cDNA。将收集的双链cDNA与250 ng pDONR222质粒混合,通过BP重组反应连接载体。在冰上将2 μL重组产物和50 μLDH10B感受态细胞(北京博迈德基因有限公司)混合,电击转化后加入SOC培养基(北京鼎国昌盛生物技术有限责任公司),37℃、225~250 r/min摇床培养1 h,获得原始入门文库。测定文库容量后,剩余培养物加入甘油至终浓度20%存于-80℃。
1.4 cDNA文库质量鉴定
取转化后的细菌原液10 μL,分别稀释10、100、1 000倍后,各取50 μL涂布SOC琼脂平板,37℃过夜孵育,次日统计菌落数目。文库滴度(CFU/mL)=培养皿中单菌落数×稀释倍数/涂布菌液体积,原始库容量(CFU)=文库滴度(CFU/mL)×文库体积(mL)。随机挑取24个单克隆,以M13FM13R为引物(宝生物工程大连有限公司),通过PCR的方法检测插入片段大小并估计文库重组率。
1.5 EST测序及分析
随机挑取2 800个阳性克隆送交华大基因进行测序。获得EST片段后利用Phrap软件对EST片段进行组装。样本基因序列分别与NCBI的蛋白质库NR进行比对,取相似度>30%且E<1E-5的注释。基于比对Uniprot的结果对基因进行分子功能、细胞成分以及生物过程方面的GO功能分类注释。
2 结果与分析
2.1 总RNA的提取及mRNA分离纯化
用Trizol试剂提取菊芋块茎总RNA,取2 μL RNA进行1%琼脂糖凝胶电泳。电泳结果如图1A所示,28S与18S rRNA条带清晰,亮度比大约为2∶1,且无其他S带,说明提取的RNA未发生降解,质量良好。经分光光度法测定RNA的总产量约为980 μg,A260/A280=2.02,比值接近2,说明提取的总RNA基本无蛋白质污染,纯度较高,可以用来分离mRNA。使用FastTrack 2.0 mRNA分离试剂盒从总RNA中分离和纯化mRNA,电泳结果如图1B所示,mRNA呈弥散状,且无S带污染,表明分离的mRNA质量较高,为建立高质量菊芋cDNA文库奠定了基础。
2.2 菊芋cDNA文库的构建及质量鉴定
按照CloneMiner域cDNA文库构建试剂盒提供的方法构建菊芋cDNA文库。原始文库稀释1 000倍后,取3个平板进行涂布,每个平板涂布50 μL,获得每个平板的克隆平均数为96个,经计算得文库滴度为1.92×106CFU/mL,库容量为5.76×106CFU,说明cDNA文库的丰富度较好。采用菌落PCR的方法检测插入片段大小并估计文库重组率,结果如图2所示,插入片段的长度在500~3 000 bp之间,其中80%的长度为1 000~3 000 bp,插入片段较长,达到了构建高质量文库的标准。此外,PCR鉴定表明扩增文库的重组效率为100%(24/24),亦达到了构建高质量文库的标准。

图1 总RNA提取及mRNA分离纯化的琼脂糖凝胶电泳图(A)菊芋块茎总RNA电泳图。1,2:总RNA,M:DNA相对分子质量标准;(B)mRNA电泳图。1:纯化后的mRNA。Fig.1 Agarose gel electrophoresis of total RNA and mRNA(A)Electrophoresis of total RNA from Jerusalem artichoke tuber.1,2:Total RNA;M:DNA marker.(B)Result of mRNA electrophoresis.1:Purified mRNA.
2.3 cDNA文库测序分析
从文库中随机挑选2 800个阳性克隆进行EST测序,去除载体序列和短片段(<100 bp),拼接后得到2 639条高质量EST,序列的平均长度为507.76 bp。将这些有效序列进行组装,共得到了337个含一条以上EST的重叠群(contig)和1 558个单一序列(singlet),即代表了1 895条非重复的唯一表达序列(unigene)。其中,含EST数量最多的一个重叠群由18条EST拼接而成。文库的冗余度为28.19%,表明构建的文库具有较好的代表性。
2.4 基因注释
NR数据库同源比对分析表明,共有1 533条(80.9%)与已知基因有显著的同源性;362条(19.1%)无显著的同源性,是新序列。在NR注释上的基因中,注释数目最多的前10个物种注释了71%的unigene,其中17%比对上了葡萄,12%比对上了番茄,8%比对上了可可,7%比对上了蓖麻,7%比对上了毛果杨,6%比对上了碧桃,4%比对上了草莓属,4%比对上了大豆,3%比对上了黄瓜,3%比对上了向日葵。
2.5 基因的GO功能分类分析
对1 895条unigene进行GO功能分类预测,有1 220个unigene被注释上GO分类。结果如图3所示,样本基因的功能在生物学过程(biological process,BP)分类中主要聚集于细胞过程和代谢过程;在细胞成分(cellular component,CC)主要聚集于细胞和细胞部分;在分子功能(molecular function,MF)分类中结合和催化活性占有最高比例。

图2 cDNA文库插入片段大小的PCR检测1~24:随机挑取的24个克隆;M:DNA相对分子质量标准。Fig.2 PCR analysis of the insert size in the cDNA library1~24:PCR products for clones selected randomly;M:DNA marker.
3 讨论
cDNA文库筛选和EST测序分析是一种高效率发现新基因的技术,特别是对非模式生物,通过cDNA文库进行基因表达分析及鉴定研究更具优势[11~13]。库容量是衡量cDNA文库质量高低的一个重要指标,一个好的cDNA文库一般至少含有5×106个克隆[17]。我们构建的文库滴度为1.92×106CFU/mL,库容量为5.76×106个克隆,符合高质量文库的库容要求。随机挑取24个菌落,经PCR检测显示插入片段长度在500~3 000 bp,其中80%的长度为1 000~3 000 bp,文库中长片段所占比例较大,表明插入片段的完整性较好,适合以后的测序及基因筛选。此外,PCR鉴定表明扩增文库的重组效率为100%(24/24),亦达到了构建高质量文库的标准。
本研究利用Gateway技术构建菊芋块茎cD-NA文库。cDNA合成采用SuperScriptⅢ逆转录酶,降低Rnase H活性,提高低丰度基因合成率。文库构建全程无酶切,保证cDNA完整性,避免普通酶切连接方式建库中cDNA出现酶切受损的情况。此外,在文库构建过程中第2链合成采用的是置换合成法,而没有采用PCR扩增,能够保持材料中mRNA的原始丰度,这使一些较低拷贝数基因的克隆具有优势,这也为今后基因分离的效率提供了有力保障。此文库所用的克隆载体pDONR222在其多克隆位点的双侧翼含有Gateway位点特异性的重组位点(attB1和attB2),可以迅速方便而且定向地重组到任何Gateway化的表达载体上,在后期的研究中便于形成各种表达文库用以进行基因的表达分析。
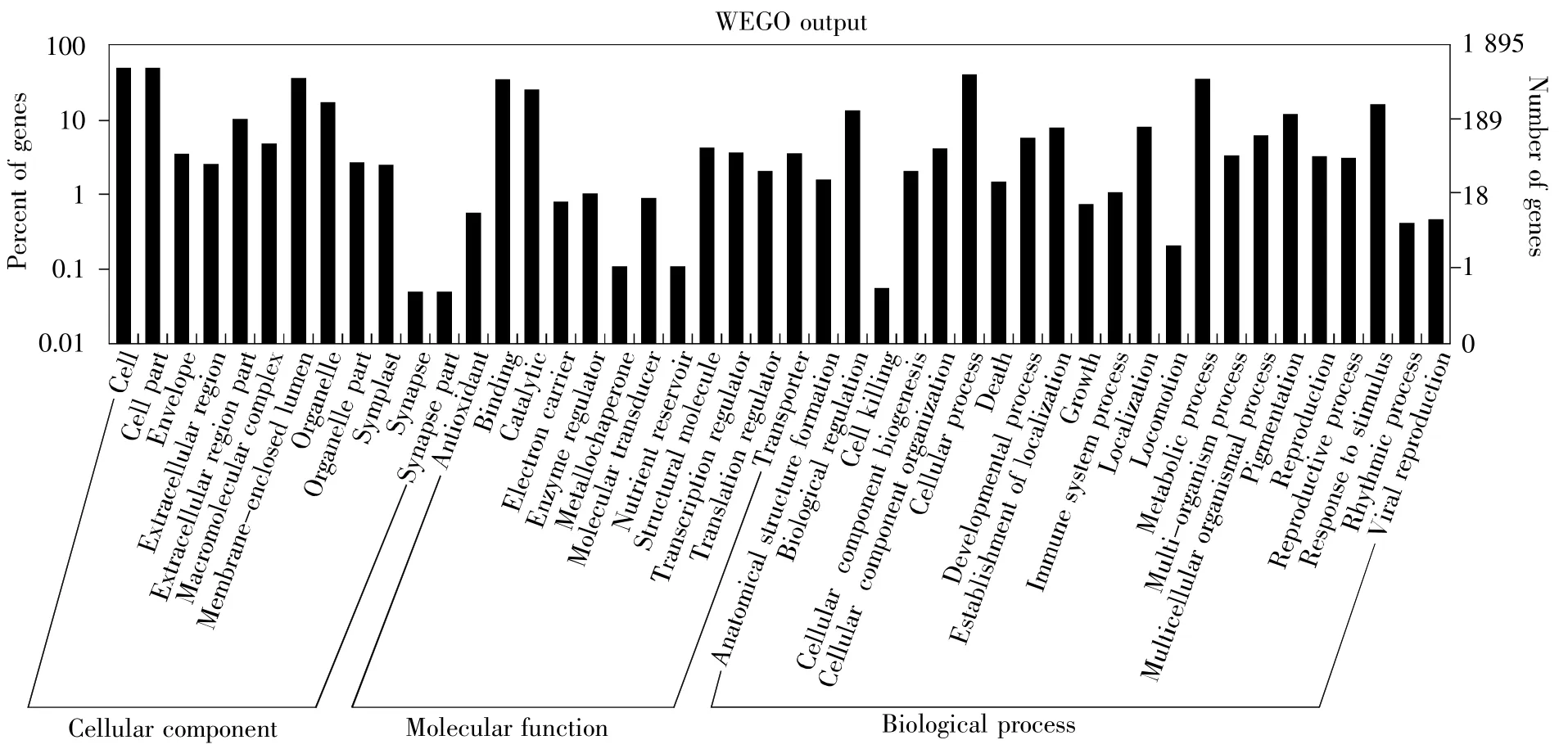
图3 Level 2水平GO注释上的基因分布Fig.3 Gene distribution of level 2 GO annotation
EST序列初步分析结果显示,共有1 533条unigene(80.9%)与NR数据库已知基因有显著的同源性,但其中预测蛋白质和未知蛋白质占比较多,许多基因的功能信息没有明确的注释。在NR注释中,注释数目最多的前10个物种注释了71%的unigene,包括葡萄(17%)、向日葵(3%)等,仅有约1%的unigene比对上了菊芋。测序获得的EST序列在数量上并不与菊芋序列同源性最多,这是因为没有数量足够大的菊芋EST与基因数据库可供参考,而菊芋块茎组织基因的鉴定与研究工作也相对更为滞后,使得测得的菊芋块茎cDNA文库中大部分EST没有对应的菊芋所表达基因序列相匹配。通过Blastx比对,我们发现了很多与营养及信号转导相关的基因,这可能包含了一些菊芋组织特异表达的基因序列,对这些基因进行深入研究,必将有助于推动菊芋功能基因组学的发展。
Gene Ontology 数据库(http://www.geneontology.org)是将全世界所有与基因有关的研究结果进行分类汇总的综合数据库,通过GO分类图可以大致了解基因产物的分类情况,有助于理解基因背后所代表的生物学意义[20]。GO分析结果表明:菊芋块茎表达基因主要包括细胞成分、分子功能和生物学过程3个类群。在细胞成分类群中,细胞和细胞部分所占比例最高;在分子功能类群中,结合和催化活性所占比例最高;而在生物学过程类群中,以细胞过程和代谢过程最突出。同时分析结果显示,有30%~40%的序列无分子功能、生物学过程和细胞成分,有待进一步挖掘。
本研究构建的菊芋块茎cDNA文库符合高质量文库的标准,可能包含大量新基因,为菊芋新功能基因发掘、功能基因组分析、基因芯片制作等研究奠定了基础。此外,菊芋EST报道尚少,本研究为充实菊芋EST数据库提供了基础数据。
