西瓜枯萎病菌遗传多样性的相关序列扩增多态性分析
2018-12-05刘丽芳肖姬玲张屹朱菲莹魏林梁志怀
刘丽芳 肖姬玲 张屹 朱菲莹 魏林 梁志怀

摘要
對30个西瓜枯萎病菌Fusarium oxysporum f.sp. niveum菌株基因组DNA进行相关序列扩增多态性(SRAP)分子标记分析,以探究其遗传多样性与地理来源的关系。采用尖孢镰刀菌西瓜专化型Fusarium oxysporum f.sp. niveum 0、1、2号生理小种的基因组DNA为模板,对225对SRAP引物进行筛选,筛选出20对多态性、重复性较好且条带清晰的引物,对30个菌株进行PCR扩增,共扩增出386条带,其中多态性条带有371条,多态性比率为96.11%,平均每对引物扩增出19.3个位点和18.55个多态性位点。UPGMA法聚类分析结果显示,供试菌株两两之间的遗传相似系数范围为0.69~0.90,平均为0.79,说明尖孢镰刀菌西瓜专化型的遗传多样性较为丰富。基于SRAP标记聚类分析表明,30个菌株在遗传相似系数为0.70处被划分为3个类群,I类群包含24个菌株,其中18个来自湖南省,Ⅱ类群只包含1个来自黑龙江省哈尔滨市的菌株,它和另一个来自黑龙江地区的菌株被划分到不同的类群,且遗传距离相对较远;Ⅲ类群包含了5个菌株,其中3个来自海南三亚,其余两个来自湖南省。根据菌株的分布情况来看,菌株的聚集与地理来源没有明显的相关性。
关键词
西瓜枯萎病菌; SRAP分析; 遗传多样性
中图分类号:
S 436.5
文献标识码: A
DOI: 10.16688/j.zwbh.2017435
Genetic diversity analysis of Fusarium oxysporum f.sp. niveum by
sequence-related amplified polymorphism
LIU Lifang1,2, XIAO Jiling ZHANG Yi ZHU Feiying WEI Lin3, LIANG Zhihuai1,2
(1. Longping Branch, Graduate School of Hunan University, Changsha 410125, China;
2. Hunan Agricultural Biotechnology Research Institute, Changsha 410125, China;
3. Hunan Plant Protection Institute, Changsha 410125, China)
Abstract
In order to explore the relationship between genetic diversity and geographical origin, genomic DNA of 30 strains of Fusarium oxysporum f.sp. niveum isolates were analyzed by sequence-related amplified polymorphism (SRAP) molecular markers. With genomic DNA of F. oxysporum f.sp. niveum race 0, race 1 and race 2 as templates, 20 pairs of primers which had rich product and high polymorphism and repeatability were screened from total 225 pairs of SRAP primers. The 20 pairs of SRAP primers generated a total of 386 reproducible bands by PCR with the genomic DNA from 30 tested materials as templates. 371 bands were polymorphic, with polymorphism ratio of 96.11%. On average, the bands amplified by each primer combination were 19.3 including 18.55 polymorphism bands. The UPGMA cluster analysis revealed that the genetic similarity coefficient of all the stains was ranged from 0.69 to 0.90, averaged 0.79, indicating the genetic diversity of F. oxysporum f. sp. niveum was abundant. Based on SRAP marker clustering analysis results, 30 isolates were classified into 3 groups with genetic similarity of 0.70. Group I contains 24 isolates, with 18 isolates from Hunan Province. Group Ⅱ only contains one strain from Harbin City, Heilongjiang Province. The two strains from Harbin City were divided into different groups and the genetic distance was relatively far. Group Ⅲ contains 5 isolates, 3 from Sanya, Hainan Province and two from Hunan Province. As far as the strains distribution was concerned, there was no significant correlation between the aggregation of the strains and the geographical origin.
Key words
Fusarium oxysporum f.sp. niveum; SRAP analysis; genetic diversity
我国是西瓜Citrullus lanatus生产与消费第一大国。西瓜枯萎病是由尖孢镰刀菌西瓜专化型Fusarium oxysporum f.sp. niveum (FON)引起的一种世界性真菌土传性病害。西瓜产业中,西瓜枯萎病已成为导致产量、品质下降最为严重的病害之一,利用抗病品种和嫁接换根是防治该病最经济、有效的措施[1]。国际上公认的西瓜枯萎病菌有4个生理小种,即生理小种0号、1号、2号和3号。生理小种3号由Zhou[2]于2010年鉴定分离,能导致兼抗生理小种0、1和2号的野生种质PI296431-FR感病,2017年在美国的佛罗里达州也被分离鉴定[3],但在中国尚未见报道。
相关序列扩增多态性(sequence-related amplified polymorphism,SRAP)是一种基于PCR的显性标记系统的DNA分子标记技术,由美国加州大学的Li[4]于2001年提出。该技术利用独特的引物对开放阅读框(open reading frames, ORFs)进行扩增,上游引物对外显子进行特异扩增,下游引物对内含子区域、启动子区域进行特异扩增,因个体不同以及物种的内含子、启动子与间隔长度不等而产生多态性,具有简便、稳定、产率高和便于得到选择条带序列等特点。目前,SRAP技术已广泛用于植物遗传多样性的分析[5-7]。
本研究以尖孢镰刀菌西瓜专化型0号、1号和2号生理小种为标准试验材料,对其进行差异分析和遗传多样性分析,并筛选得到了20对多态性好的SRAP引物组合,利用筛选的20对SRAP引物对收集到的湖南省内大部分地区及省外部分西瓜主产区枯萎病菌进行多态性分析,为监测与防控西瓜枯萎病的流行提供基础。
1 材料与方法
1.1 材料
1.1.1 试验材料
尖孢镰刀菌西瓜专化型Fusarium oxysporum f.sp. niveum (FON) 0、1、2號生理小种由北京市农林科学院惠赠;各地区收集分离的菌株详情见表1。
1.1.2 试剂及仪器
SRAP引物由生工生物工程(上海)股份有限公司合成,Trans Tap DNA Polymerase High Fidelity(HiFi)、dNTPs、DNA分子量标准Trans DNA Marker I,均购于全式金生物技术有限公司;T100 PCR仪,购于美国Bio-Rad公司;电泳仪,北京六一仪器厂生产。
表1 供试菌株信息
Table 1 Information of tested isolates
1.2 研究方法
1.2.1 DNA提取
供试菌株基因组DNA的提取参考曾东方等[8]的研究方法。
1.2.2 SRAP扩增反应
SRAP的反应体系参考Li[4]的方法,并加以改进。反应体系为12 μL,其中ddH2O 7.6 μL,HiFi buffer Ι 1.2 μL,2.5 mol/L dNTPs 1.0 μL,HiFi酶0.2 μL,DNA模板1.0 μL,上下游引物各0.5 μL。PCR反应程序为:94℃预变性5 min;94℃变性1 min,35℃复性1 min,72℃延伸2 min,5个循环;94℃变性1 min,55℃复性1 min,72℃延伸2 min,34个循环;最后72℃延伸10 min。
1.2.3 SRAP引物的筛选及PCR产物的检测
采用0、1、2号生理小种的基因组DNA为模板,用合成的225对SRAP随机组合引物对其进行PCR扩增,产物通过8%聚丙烯酰胺变性胶进行电泳[9],140 v恒电压电泳1.5 h,电泳结束后采用银染法[10]进行染色并拍照记录,对扩增结果进行比较分析。从中选择多态性和重复性较好的引物对30株供试菌株的基因组DNA进行PCR扩增,并进行电泳图谱分析。
1.2.4 电泳谱带的记录与数据分析[7,9]
根据电泳结果,电泳图谱中每条扩增条带均代表了引物与模板DNA互补的一个结合位点,可记为一个分子标记,在电泳图谱中相同迁移位置上有扩增带的记为1,无扩增带的记为0,如有数据缺失的情况则用其他数字代替,然后将PCR扩增的DNA条带数据统计成01矩阵。并用NTSYS软件计算两两菌株间的Dice相似系数,利用UPGMA法进行聚类分析建立聚类树状图。
2 结果与分析
2.1 SRAP引物筛选
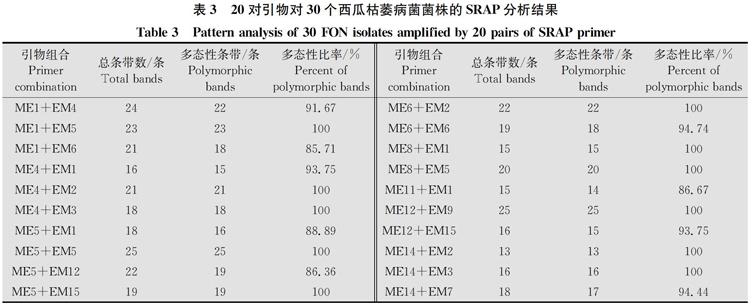
根据SRAP引物筛选的扩增结果,从225对引物中筛选出20对扩增条带清晰、多态性明显、扩增稳定的引物对:ME1+EM4、ME1+EM5、ME1+EM6、ME4+EM1、ME4+EM2、ME4+EM3、ME5+EM1、ME5+EM5、ME5+EM12、ME5+EM15、ME6+EM2、ME6+EM6、ME8+EM1、ME8+EM5、ME11+EM1、ME12+EM9、ME12+EM15、ME14+EM2、ME14+EM3、ME14+EM7,具体引物序列见表2。
表2 筛选的引物序列
Table 2 Sequences of the screened primers
2.2 尖孢镰刀菌SRAP引物扩增的多态性
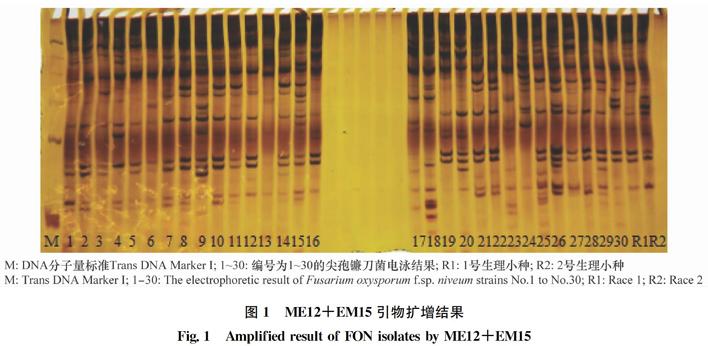
采用筛选出的20对引物,对30株的供试尖孢镰刀菌菌株进行了SRAP-PCR扩增。共扩增出386条带,其中多态性条带有371条,平均每对引物扩增出19.3个位点和18.55个多态性位点,多态性比率为96.11%(引物组合ME12+EM15的扩增结果见图1)。说明尖孢镰刀菌西瓜专化型的遗传多样性丰富且遗传差异性较大。在这20对SRAP引物中,不同的引物组合扩增出来的条带数存在一定的差异,其中引物组合ME5+EM5和ME12+EM9扩增出来的条带数最多,达到了25条;引物组合ME14+EM2扩增出来的条带数最少,只有13条,同时这几个引物组合的多态性比率均为100%(表3)。
图1 ME12+EM15引物扩增结果
Fig.1 Amplified result of FON isolates by ME12+EM15
2.3 SRAP聚类分析
用NTSYS软件计算两两菌株间的相似系数并利用UPGMA法进行聚类分析建立聚类树状图(图2),结果表明,供试尖孢镰刀菌西瓜专化型菌株两两之间的遗传相似系数范围为0.69~0.90,平均为0.79。说明这30个西瓜枯萎病菌遗传分化较大,群体遗传多样性比较丰富。在遗传相似系数为070处,可以把供试菌株划分为3个类群:Ⅰ类群包含24个菌株,占总数的80%,其中18个来自湖南省,占总数的75%,6个分别来自广东(3号)、安徽(12号)、黑龙江(14号)、海南(25/26号)和河南(27号)省,占总数的25%。在遗传相似系数为0.75处,该类群又可进一步被划分为多个亚群,其中来自湖南省株洲市天元区的6号菌株和来自海南海口的26号菌株各自独立成为一个亚群,与其他来自同一地区的菌株的遗传距离相对较远,其余菌株组成的亚群遗传距离较近,由图还可得知来自湖南省以外的6个菌株分布在不同的亚群;Ⅱ类群只包含1个菌株,是来自黑龙江省哈尔滨市的15号菌株,该菌株和来至同一地区的14号菌株分属于不同的类群,且遗传距离相对较远;Ⅲ类群包含5个菌株,其中17、18、23号菌株关系较近,均来自海南三亚,其余两个分别来自湖南省的怀化辰溪县(13号)和衡阳南岳区(24号),且来自湖南省衡阳南岳区24号菌株与来自海南三亚的3个菌株关系更近。进一步分析发现来自同一地区的菌株被划分在不同的类群中,而同一类群中包括了不同地区的菌株。所以基于SRAP多态性而划分的西瓜枯萎病菌菌株类群与菌株地理来源没有明显的相关性。
表3 20对引物对30个西瓜枯萎病菌菌株的SRAP分析结果
Table 3 Pattern analysis of 30 FON isolates amplified by 20 pairs of SRAP primer
图2 30个西瓜枯萎病菌菌株的SRAP聚类树状图
Fig.2 Dendrogram of 30 FON isolates based on
SRAP marker clustering analysis
3 讨论
利用SRAP进行的遗传多样性分析结果表明,这30个西瓜枯萎病菌的遗传距离变化在0.69~0.90之间,平均为0.79,从分子水平上说明群体遗传多样性比较丰富。可能的原因是西瓜枯萎病的病菌主要依靠种子进行传播,而种子的来源途径广泛,同一地区的种子可能来源不一致,不同地区的种子也可能来自同一地区[11]。
本研究筛选出了20对SRAP引物组合,共扩增出386条带,平均每对引物扩增出19.3个位点,多态性比率为96.11%,高于苏家等[12]利用ISSR-PCR分子标记技术得到的多态性比率(90.82%),也高于段会军等[13]利用AFLP分子标记技术得到的多态性比率(33.6%),表明尖孢镰刀菌基因组DNA在SRAP分子标记区域的分子遗传多态率高,且在种内存在丰富的遗传多样性。但低于李伟[11]等利用RAPD-PCR分子标记技术得到的多态性比率(97.18%)。而段会军等[14]利用RAPD、ISSR和AFLP三种分子标记进行对比发现RAPD不能将绝大部分菌株分开,说明不同的分子标记技术因为所揭示的遗传位点不同,所以在分类结果上会存在差异。
基于SRAP聚类分析结果将各个地区30个西瓜枯萎病菌划分为3个类群,SRAP类群的划分与地域之间没有直接的关系。段会军等[14]用AFLP分子标记技术分析表明,尖孢镰刀菌西瓜专化型不同菌株间的亲缘关系与地理来源关系不明显;延涵等[15]利用SRAP分子标记技术分析得出,44株供试甜瓜枯萎病菌菌株之间的亲缘关系与地理来源没有明显相关性。而田叶韩等[16]用SRAP分子标记技术分析认为尖孢镰刀菌苦瓜专化型不同菌株间的亲缘关系与地理来源有一定的相关性;张述义等[17]用ISSR分子标记分析了33株尖孢镰刀菌遗传多样性,表明寄主來源相同的供试菌株间遗传相似性与其地理来源也有一定的相关性。由于本研究采集到的样本有限,若需更全面地了解还需更深更全面的研究分析。
参考文献
[1] NAVEENKUMAR R, MUTHUKUMAR A, MOHANAPRIYA R. Occurrence, virulence and evaluation of essential oils against Fusarium oxysporum f.sp. niveum causing wilt of watermelon [J/OL]. International Journal of Plant Research, 2017, 30:4.doi:10.5958/229-4473.2017.00181.1.
[2] ZHOU X G, EVERTS K L, BRUTON B D. Race 3, a new and highly virulent race of Fusarium oxysporum f.sp. niveum causing Fusarium wilt in watermelon [J].Plant Disease, 2010, 94(1):92-98.
[3] AMARADASA S, SANCHEZ T, BECKHAM K A, et al. First Report of Fusarium oxysporum f.sp. niveum race 3 causing wilt of watermelon in Florida,USA [J].Plant Disease, 2018,102(5):1029.
[4] LI G, QUIROS C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 2001, 103:455-461.
[5] ANEJA B, YADAV N R, CHAWLA V, et al. Sequence-related amplified polymorphism (SRAP) molecular marker system and its applications in crop improvement[J]. Molecular Breeding, 2012, 30(4):1635-1648.
[6] GUENNI K, AOUADI M, CHATTI K, et al. Analysis of genetic diversity of Tunisian pistachio (Pistacia vera L.) using sequence-related amplified polymorphism (SRAP) markers.[J]. Genetics and Molecular Research, 2016, 15(4):gmr15048760.
[7] 白玲玲,劉行风,陈政瑜,等.基于SRAP标记的黄瓜霜霉菌遗传多样性分析[J].植物保护,2017,43(4):145-150.
[8] 曾东方,陈玢,何一正,等.冻融法快速提取真菌微量培养物基因组DNA[J].生物技术,2010,20(6):49-51.
[9] 马洪平,潘明洪,彭丹丹,等.四川白三叶根瘤菌SRAP遗传多样性分析[J].中国草地学报,2016,38(6):36-44.
[10] 伊六喜,斯钦巴特尔,张辉,等.胡麻种质资源遗传多样性及亲缘关系的SRAP分析[J].西北植物学报,2017,37(10):1941-1950.
[11] 李伟,孙文秀.西瓜枯萎病菌致病型的遗传多样性分析[J].河南农业科学,2008,37(2):70-74.
[12] 苏家,高增贵,姚远,等.利用ISSR-PCR分子标记技术分析瓜类枯萎病病原菌遗传多样性[J].北方园艺,2014(17):97-101.
[13] 段会军,张彩英,李喜焕,等.河北省西瓜枯萎病菌生理小种鉴定与AFLP分析[J].中国农业科学,2007,40(5):925-931.
[14] 段会军,张彩英,李喜焕,等.基于RAPD、ISSR和AFLP对西瓜枯萎病菌遗传多样性的评价[J].菌物学报,2008,27(2):267-276.
[15] 延涵,杨瑞秀,姚远,等.东北地区甜瓜枯萎病菌遗传多样性分析[J].北方园艺,2017(11):130-134.
[16] 田叶韩,侯圆圆,彭彩云,等.瓜类枯萎病菌遗传多样性和亲缘关系的SRAP分析[J].应用生态学报,2017,28(3):947-956.
[17] 张述义,李新凤,韦晓艳,等.33株尖孢镰刀菌遗传多样性的ISSR分析[J].生态学杂志,2013,32(5):1195-1202.
(责任编辑: 杨明丽)
