哈族食管鳞癌中PLCE1基因启动子甲基化水平的实验研究
2018-10-26王彬彭昊陈云昭
王彬,彭昊,陈云昭
哈族食管鳞癌中PLCE1基因启动子甲基化水平的实验研究
王彬,彭昊,陈云昭
832000 新疆,石河子大学医学院病理科新疆地方与民族高发病重点实验室
探讨 PLCE1基因启动子甲基化与食管鳞癌发生发展的关系。
分别用免疫组化(IHC)和甲基化特异 PCR(MSP)方法检测 51 例新疆哈萨克族食管鳞癌及相应癌旁正常组织中 PLCE1 蛋白表达水平及其启动子甲基化水平,分析其与病理资料相关性;分别用 Western blot 及 MSP 检测食管鳞癌细胞在 5-aza-dC 作用前后 PLCE1 蛋白表达及启动子甲基化变化情况。
新疆哈萨克族食管鳞癌组织中 PLCE1 蛋白表达高于癌旁正常组织,而其基因甲基化水平则较低(< 0.001),且蛋白高表达的癌组织中其甲基化水平低于蛋白低表达的癌组织(= 0.028),PLCE1 甲基化水平与其蛋白表达水平具有明显的相关性(2= 4.791,= 0.028),并与患者的淋巴结及远处转移(2= 7.242,= 0.027)和 TNM 分期(2= 7.883,= 0.019)明显相关;在食管鳞癌细胞系中 PLCE1 甲基化程度同样与蛋白表达水平成反比,5-aza-dC 处理可抑制 TE-1 和 Kyse150 的 PLCE1 甲基化,提高其蛋白表达。
食管鳞癌组织中 PLCE1 甲基化低与其蛋白表达高、淋巴结和远处转移及 TNM 分期相关,5-aza-dC 处理可抑制 PLCE1 甲基化程度,增高其蛋白表达。
甲基化; 食管鳞癌; 磷脂酶Cε1; 甲基化特异 PCR
食管癌在我国的发病率及病死率都高居前列[1],且有明显的地域分布,在冀、豫、苏、晋、陕、闽等省的食管鳞癌发病率最高[2]。磷脂酶 Cε1(phospholipase C epsilon-1,PLCE1)是最近发现的 PLC 最复杂的同工酶,它独有的 RA 结构域,可以结合Ras 和 Rap1。Ras 介导的 PLCE1 活化并诱导酶转运到质膜,而 Rap1 诱导的 PLCE1 活化使酶在核周区域聚集[3]。此外,PLCε的 CDC25 结构域有鸟嘌呤核苷酸交换因子(GEF)属性,参与 RAS/MAPK 和细胞外信号调节激酶(ERK)的激活。因此,PLCE1 通过参与调节一系列细胞内的信号传导通路,而调节细胞的生长和凋亡,促进细胞增殖、分化,甚至恶变。近年来,很多报道采用全基因组关联分析(GWAS)技术对食管鳞癌患者和正常对照人群进行研究,发现 PLCE1 是中国食管鳞癌重要的易感基因[4-5]。研究发现 PLCE1 在新疆哈萨克族食管鳞癌中高表达,且与肿瘤的转移与侵袭密切相关[6]。作为最常见的表观遗传改变,食管鳞癌组织中 PLCE1 蛋白表达增高,其原因是否与基因甲基化异常相关鲜有研究。
1 材料与方法
1.1 材料
1.1.1 病历资料 本实验收集伊犁哈萨克自治州友谊医院等医院病理科 2000 – 2013 年间石蜡包埋的术前未经任何治疗的新疆哈萨克族食管鳞癌患者癌组织及相应癌旁正常组织各 51 例,同时收集患者临床病理资料。
1.1.2 材料与试剂 人食管鳞癌细胞株 Eca109、EC9706、TE-1、Kyse150 均购自上海复祥生物科技有限公司;山羊抗人 PLCE1(sc-28404)抗体购自 Santa Cruz 公司;小鼠抗人 β-actin(TA-09)抗体购自北京中杉金桥生物技术有限公司;QIAamp DNA FFPE Tissue Kit、EpiTect Fast DNA Bisulfite Kit 购自德国 Qiagen 公司;甲基转移酶抑制剂 5-aza-dC 购自美国 Sigma 公司。
1.2 方法
1.2.1 细胞培养及实验分组 食管癌细胞 Eca109、EC9706、TE-1、Kyse150 用含 10% 胎牛血清及青、链霉素(均为 100 U/ml)的 DMEM 培养基重悬,以 1 × 104个/ml 的浓度接种 24 孔板,置于 37 ℃、5% CO2细胞培养箱中孵育。5-aza-dC 处理组:细胞培养 24 h 后用含 5-aza-dC(1.0 × 10-3mol/L)的培养基换液;对照组:细胞同期培养,只换液,不加入 5-aza-dC。
1.2.2 免疫组织化学观察 选择患者病理组织蜡块,制作切片,按梯度浓度用二甲苯对切片脱蜡,用乙醇进行水化,用 EDTA 进行抗原修复,用 H2O2孵育,切片用牛血清封闭后添加一抗在 4 ℃冰箱孵育过夜,PBS 洗 3 次,再添加二抗 37 ℃温箱孵育 30 min,PBS 洗 3 次,DAB 显色,清水冲洗后苏木素复染、盐酸分化、自来水蓝化、乙醇脱水、二甲苯中透明切片、中性树胶封片。免疫组化评分标准:0 ~ 1 分为阴性,2 ~ 4 分为低表达组,5 ~ 12 分为高表达组。
1.2.3 甲基化特异性 PCR 提取组织及细胞 DNA,对 DNA 进行亚硫酸氢盐转化并提纯,引物序列见表 1,配制体系后进行扩增,反应条件为94 ℃变性 3 min;94 ℃ 30 s、50 ℃ 30 s、72 ℃30 s,重复 35 个循环;72 ℃延伸 5 min,4 ℃时结束。2% 琼脂糖凝胶电泳分离 PCR 产物后拍照。结果判定以 DNA MarkerI 为指示并参考甲基化和非甲基化阳性对照条带。

表 1 引物名称与序列
1.2.4 Western blot 提取细胞蛋白后,按不同分子量大小配制不同浓度的 SDS-PAGE 凝胶,按分子量大小选择电泳及转膜的条件,胎牛血清封闭2 h,一抗 4 ℃孵育过夜,洗膜,二抗室温孵育2 h,洗膜,曝光,成像。以β-actin 为内参。
1.3 统计学处理
采用 SPSS17.0 软件进行统计学分析,用 Mann-whitney U 检验评价免疫组化评分在癌和正常组织中的差异,采用2检验和 Fisher’s 确切概率法分析 PLCE1 启动子区甲基化表达水平与临床病理资料之间的关系。< 0.05 时,差异具有统计学意义。
2 结果
2.1 食管鳞癌及癌旁正常组织中 PLCE1 蛋白的表达
在 51 例新疆哈萨克族食管鳞癌样本中,有 34 例(34/51,66.67%)PLCE1 蛋白呈高表达,而癌旁正常组织中只有 4 例(4/51,7.84%)呈高表达(图 1)。癌组织中的 PLCE1 蛋白表达明显高于癌旁正常组织(7.27 ± 2.89 vs. 3.12 ± 2.35,< 0.001)。
2.2 食管鳞癌与相应癌旁正常组织中 PLCE1 基因启动子甲基化状态
MSP 结果(图 2)显示,食管鳞癌组织中完全甲基化、部分甲基化、非甲基化分别有 11、17、23 例,而在癌旁正常组织中分别为 28、19 和4 例。癌组织中 PLCE1 甲基化程度远远低于癌旁正常组织(2= 20.892,< 0.001)。
2.3 食管鳞癌中 PLCE1 基因启动子甲基化与蛋白表达的关系
在 28 例 PLCE1 基因启动子出现甲基化现象的食管鳞癌组织中,有 13 例的 PLCE1 蛋白呈现低表达,15 例高表达;而在 23 例未出现甲基化现象的食管鳞癌组织中,仅有 4 例 PLCE1 蛋白呈现低表达,其余 19 例均为高表达。两者之间比较差异有统计学意义(2= 4.791,= 0.028)(表 2)。

图 1 A、B 为食管鳞癌组织 PLCE1 表达,C、D 为正常食管组织 PLCE1 表达(A、C:×100 倍;B、D:× 400 倍)
Figure 1 Pictures A and B are cancer tissue, C and D are normal tissue (A, C: ×100 magnification; B, D: × 400 magnification)
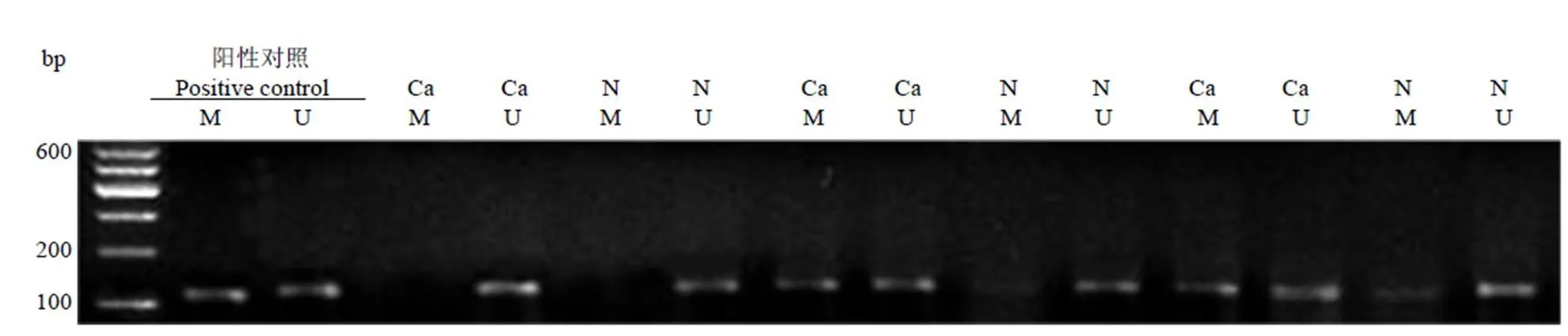
Ca:癌组织;N:正常组织;M:甲基化引物扩增产物;U:非甲基化引物扩增产物
Figure 2 Expression of methylation of PLCE1 promoter in ESCC cancer tissue and normal tissue

表 2 食管鳞癌中 PLCE1 基因启动子甲基化与蛋白表达的关系

表 3 不同病理特征的食管鳞癌组织中 PLCE1 基因启动子甲基化的比较
2.4 不同病理特征的食管鳞癌组织中 PLCE1 基因启动子的甲基化状态
分析 PLCE1 启动子甲基化情况与病理参数的关系发现,食管鳞癌组织中 PLCE1 甲基化程度越低,其发生淋巴结及远处转移的可能性就越大(2= 7.248,= 0.027),且其 TNM 分期越高(2= 7.883,= 0.019)(表 3)。
2.5 甲基转移酶抑制剂 5-aza-dC 对食管鳞癌细胞 PLCE1 启动子甲基化的影响
在 Eca109 和 EC9706 两株 PLCE1 启动子低甲基化细胞株中,5-aza-dC 处理未见明显影响PLCE1 的低甲基化状态;而在 TE-1 和 Kyse150 两株 PLCE1 启动子高甲基化细胞株中,5-aza-dC 处理后,PLCE1 非甲基化条带在粗细和亮度上明显超过甲基化条带,可见 5-aza-dC 有效地抑制了 TE-1 和 Kyse150 的 PLCE1 甲基化程度(图 3)。
2.6 甲基转移酶抑制剂 5-aza-dC 对食管鳞癌细胞 PLCE1 蛋白表达的影响
PLCE1 蛋白在 Eca109、EC9706 中表达较高,而在 TE-1、Kyse150 中表达量很低,用甲基转移酶抑制剂 5-aza-dC 处理后,Eca109、EC9706 的 PLCE1 蛋白表达并无明显变化,而 TE-1 和Kyse150 中的 PLCE1 蛋白表达增高(图 4),这与其基因启动子甲基化水平变化相对应:Eca109 和 EC9706 细胞系在经过 5-aza-dC 处理后,PLCE1 基因启动子甲基化水平几乎未发生改变,因此其蛋白表达也几乎无变化;TE-1 和 Kyse150 在经过 5-aza-dC 处理后,PLCE1 基因启动子甲基化水平由部分甲基化变化为接近非甲基化,其蛋白表达也显示提高。
可见 5-aza-dC 抑制了 PLCE1 甲基化程度后,其蛋白表达升高,即 PLCE1 的 DNA 甲基化反向影响着其蛋白表达。
3 讨论
食管鳞癌的发生,是环境因素与遗传因素长久以来相互作用的结果。众所周知,吸烟和大量酒精的摄入是食管鳞癌发生的危险因素[7]。不同地区的一些特殊生活习惯也与食管鳞癌的发生有着不可推卸的关系。例如在东南亚,嚼槟榔增加了患食管鳞癌的可能性[8]。新疆哈萨克族人的一些生活习惯,例如喜食熏马肠、各种奶制品、喝热奶茶以及新鲜水果蔬菜摄入较少,使得哈族人群成为了食管鳞癌的一个高发人群。此外,遗传因素对于食管鳞癌的发生也是必不可少的一个重要因素。
我们前期研究发现,新疆哈萨克族食管鳞癌组织中 PLCE1 蛋白表达远高于癌旁正常组织[6],而且,PLCE1 蛋白表达的增加伴随着癌变的进展,在正常上皮组织、低级别上皮内瘤变组织、高级别上皮内瘤变组织及食管鳞癌组织中,PLCE1 蛋白表达量依次增高。本次实验检测的 51 例食管鳞癌病例再次印证了之前的发现,癌组织中有 34 例 PLCE1 蛋白呈现高表达而在癌旁正常组织中只有 4 例(7.27 ± 2.89 vs. 3.12 ± 2.35,= 0.000)。
表观遗传学是基于当基因的核苷酸序列未改变时,基因表达却呈现可遗传的变化,即基因型未改变而表型发生变化,且这种变化在发育和细胞增殖中能稳定传代[9-11]。其中 DNA 甲基化是最早发现也是目前最为常见的表观修饰途径[12]。现如今人们已认识到,DNA 甲基化能引起染色质结构、DNA 构象、DNA 稳定性及 DNA 与蛋白质相互作用方式的改变,从而控制基因表达[13]。DNA 的异常甲基化与食管癌发生息息相关。一些分子的 DNA 甲基化率在正常食管上皮、Barrett’s 食管、Barrett’s 食管伴随不典型增生、食管腺癌和食管鳞癌中是逐渐增加的[14]。
越来越多研究表明,PLCE1 与肿瘤发生发展关系密切,有证据显示 PLCE1 的单核苷酸多态性rs2274223 SNP 与胃癌发生相关[15-16],在食管癌中 PLCE1 的表达还受到 micro-RNA 调控[17],在结肠癌中,PLCE1 启动子高甲基化导致其蛋白低表达[18],但并无 PLCE1 在食管癌中的甲基化研究。本研究中我们首次发现,新疆哈萨克族食管鳞癌的癌旁正常组织的 PLCE1 基因启动子甲基化水平明显高于癌组织(2= 20.892,= 0.000),这与蛋白表达水平呈负相关(2= 4.791,= 0.028),说明在新疆哈萨克族食管鳞癌中,PLCE1 基因启动子的甲基化水平降低,是促使其蛋白表达增高的原因之一。进一步探究它与临床病理资料及预后的相关性,发现其与病人的性别、年龄及肿瘤大小没有相关性,但是 PLCE1 甲基化水平越低,越容易发生淋巴结及远处转移(2= 7.248,= 0.027),且 TNM 分期越高(2= 7.883,= 0.019)。

M:甲基化引物扩增产物;U:非甲基化引物扩增产物
Figure 3 Effect of 5-aza-dC on the methylation status of PLCE1 in ESCC cells

图 4 5-aza-dC 对食管鳞癌细胞 PLCE1 蛋白表达的影响
Figure 4 Effect of 5-aza-dC on the protein status of PLCE1 in ESCC cells
通过检测 4 株食管鳞癌细胞系中 PLCE1 的蛋白表达和基因启动子甲基化水平,我们发现2 株 PLCE1 蛋白表达高的细胞系 Eca109 和 EC9706 其启动子区甲基化水平要低于蛋白表达低的细胞系 TE-1 和 Kyse150,这印证了组织中 PLCE1 的蛋白表达与基因甲基化状态的关系,提示食管鳞癌中 PLCE1 表达增高可能与其启动子区甲基化水平降低有关。随后,当我们用甲基转移酶抑制剂 5-aza-dC 作用于这 4 株细胞系后,PLCE1 基因启动子处于未甲基化状态的 2 株细胞 Eca109、EC9706 的 PLCE1 甲基化水平和蛋白表达水平几乎未发生改变,而 TE-1 和 Kyse150 的 PLCE1 甲基化水平被减弱,其蛋白表达水平大大提高,进一步提示了食管鳞癌中 PLCE1 表达增高与其启动子区甲基化水平降低有关,且甲基转移酶在这一过程中起着促进作用。
本研究阐明,在新疆哈萨克族食管鳞癌中 PLCE1 启动子低甲基化导致其蛋白高表达,且与肿瘤的淋巴结和远处转移及 TNM 分期相关,为干预食管鳞癌的发生发展提供了一个潜在的作用靶点,并为阐明 PLCE1 在食管鳞癌发生发展中的作用机制奠定了基础。
[1] Chen WQ, Zheng RS, Zeng HM, et al. Incidence and mortality of malignant tumors in China, 2011. China Cancer, 2015, 24(1):1-10. (in Chinese)
陈万青, 郑荣寿, 曾红梅, 等. 2011年中国恶性肿瘤发病和死亡分析. 中国肿瘤, 2015, 24(1):1-10.
[2] Wang M, Hao CQ, Zhao DL, et al. Distribution of esophageal squamous cell cancer and precursor lesions in high-risk areas, Linzhou in Henan province and Feicheng in Shandong province of China, 2005-2009. Chin J Prev Med, 2015, 49(8):677-682. (in Chinese)
王孟, 郝长青, 赵德利, 等. 2005-2009年中国食管癌高发区河南省林州市、山东省肥城市食管癌及其癌前病变人群分布研究. 中华预防医学杂志, 2015, 49(8):677-682.
[3] Song C, Satoh T, Edamatsu H, et al. Differential roles of Ras and Rap1 in growth factor-dependent activation of phospholipase C epsilon. Oncogene, 2002, 21(53):8105-8113.
[4] Wang LD, Zhou FY, Li XM, et al. Genome-wide association study of esophageal squamous cell carcinoma in Chinese subjects identifies susceptibility loci at PLCE1 and C20orf54. Nat Genet, 2010, 42(9): 759-763.
[5] Wu C, Hu Z, He Z, et al. Genome-wide association study identifies three new susceptibility loci for esophageal squamous-cell carcinoma in Chinese populations. Nat Genet, 2011, 43(7):679-684.
[6] Chen YZ, Cui XB, Hu JM, et al. Overexpression of PLCE1 in Kazakh esophageal squamous cell carcinoma: implications in cancer metastasis and aggressiveness. APMIS, 2013, 121(10):908-918.
[7] Chung CS, Lee YC, Wang CP, et al. Secondary prevention of esophageal squamous cell carcinoma in areas where smoking, alcohol, and betel quid chewing are prevalent. J Formos Med Assoc, 2010, 109(6):408-421.
[8] Akhtar S. Areca nut chewing and esophageal squamous-cell carcinoma risk in Asians: a meta-analysis of case-control studies. Cancer Causes Control, 2013, 24(2):257-265.
[9] Esteller M. The necessity of a human epigenome project. Carcinogenesis, 2006, 27(6):1121-1125.
[10] Mulero-Navarro S, Esteller M. Epigenetic biomarkers for human cancer: the time is now. Crit Rev Oncol Hematol, 2008, 68(1):1-11.
[11] Kelly AD, Issa JJ. The promise of epigenetic therapy: reprogramming the cancer epigenome. Curr Opin Genet Dev, 2017, 42:68-77.
[12] Paska AV, Hudler P. Aberrant methylation patterns in cancer: a clinical view. Biochem Med (Zagreb), 2015, 25(2):161-176.
[13] Hoffman RM. Is DNA methylation the new guardian of the genome? Mol Cytogenet, 2017, 10:11.
[14] Kaz AM, Grady WM. Epigenetic biomarkers in esophageal cancer. Cancer Lett, 2014, 342(2):193-199.
[15] Wadhwa R, Song S, Lee JS, et al. Gastric cancer-molecular and clinical dimensions. Nat Rev Clin Oncol, 2013, 10(11):643-655.
[16] Mocellin S, Verdi D, Pooley KA, et al. Genetic variation and gastric cancer risk: a field synopsis and meta-analysis. Gut, 2015, 64(8):1209- 1219.
[17] Cui XB, Li S, Li TT, et al. Targeting oncogenic PLCE1 by miR-145 impairs tumor proliferation and metastasis of esophageal squamous cell carcinoma. Oncotarget, 2016, 7(2):1777-1795.
[18] Wang XL. Screening and identification of candidate tumor suppressor gene PLCE1 in colorectal cancer. Shanghai: Shanghai Jiao Tong University, 2009. (in Chinese)
王晓亮. 结直肠癌候选抑癌基因PLCE1的筛选及鉴定研究. 上海: 上海交通大学, 2009.
Analysis of the status of PLCE1 promoter methylation in Kazakh esophageal squamous cell carcinoma
WANG Bin, PENG Hao, CHEN Yun-zhao
Department of Pathology, School of Medicine, Shihezi University, Xinjiang 832000, China
To explore the methylation status of PLCE1 promoter in ESCC.
51 cases of ESCC from Kazakh nationality were collected and examined by IHC and MSP. Western blot was used to detect the cellular protein.
The expression of PLCE1 protein in ESCC from Kazakh nationality tissue was higher and the methylation status was lower (< 0.001) than those in the normal tissues around. In the cancer tissues with high PLCE1 protein expression, the methylation level was lower than those with the low PLCE1 group (= 0.028). PLCE1 methylation was associated with lymph nodes metastasis (2= 7.242,= 0.027) and TNM stage (2= 7.883,= 0.019). The status of PLCE1 promoter methylation and protein expression were similarly inverse in sophageal squamous cell lines. Inhibition of PLCE1 methylation in TE-1 and Kyse150 cell lines by 5-aza-dC increased the protein expression of PLCE1.
The methylation status of PLCE1 promoter in ESCC is assiocated with the PLEC1 protein expression and lymph nodes metastasis and TNM stage.
Methylation; Esophageal squamous cell carcinoma; Phospholipase C epsilon 1; Methylated specific PCR
WANG Bin, Email: 814235427@qq.com
王彬,Email:814235427@qq.com
2018-07-23
10.3969/j.issn.1673-713X.2018.05.010