广西凡纳滨对虾源副溶血弧菌耐药性和耐药基因的检测
2018-10-19黄伟德肖双燕黎姗梅黄德生张振豪钟昌艳陈福彩梁静真
黄伟德,肖双燕,黎姗梅,黄德生,张振豪,钟昌艳,陈福彩,梁 毅,梁静真*,黄 钧*
(1. 广西水生动物病害诊断实验室, 广西 南宁 530005;2. 钦州市水产技术推广站, 广西 钦州 535000;3. 广西大学 动物科学技术学院, 广西 南宁 530005;4. 广西水产技术推广总站, 广西 南宁 530022;5. 北海市合浦县水产技术推广站, 广西 北海 536100;6. 防城港市港口区渔业技术推广站, 广西 防城港 538002;7. 钦州市钦南区水产技术推广站,广西 钦州 535000;8. 东兴市渔业技术推广站,广西 东兴 538100)
【研究意义】凡纳滨对虾(Litopenaeusvannamei)又称南美白对虾,具有出肉率高、营养需求低、生长迅速、适应性强等优点。从1996年引进广西后,一直是广西沿海重要的对虾养殖品种。而近年来细菌性疾病常给广大养殖户造成巨大的损失,已成为制约广西凡纳滨对虾养殖业可持续健康发展的重要因素。副溶血弧菌(Vibrioparahaemolyticus)为革兰氏阴性菌,主要分布于淡咸水交界处、沿海以及鱼虾贝类等多种水产动物中,能引起对虾的烂鳃病、红体病、早期死亡综合征等多种疾病,是限制我国海水对虾养殖业发展的主要病原菌之一[1-3]。目前对弧菌病主要以抗生素进行治疗。但随着抗生素的大量使用,副溶血弧菌耐药问题日渐突出,必需引起养殖及疾病防治工作者的高度重视。此外,副溶血弧菌还是夏秋季节沿海地区食物中毒和急性腹泻的主要病原菌[4]。可见副溶血弧菌病的流行不仅严重影响凡纳滨对虾养殖业的健康发展,还对人类健康与食品安全造成很大威胁。因此,深入探讨副溶血弧菌的耐药性及其机制,无论是对凡纳滨对虾弧菌病防控提供指导还是对人类的公共健康都显得非常迫切。【前人研究进展】有关副溶血弧菌的耐药性研究目前已有不少报道,如Oh等[5]和Yutaka等[6]分别对韩国鱼源和泰国虾源副溶血弧菌的耐药性进行了研究;国内众多学者也先后对浙江[7]、江苏[8]、福建和海南[9]等地水产品源副溶血弧菌的耐药表型进行了研究,并发现部分菌株在一定程度上产生了多重耐药性。Ceccarelli等[10]认为,副溶血弧菌多重耐药的形成与耐药基因随可移动遗传元件(如整合子、转座子、质粒等)在菌株间的水平传播密切相关。目前已知,磺胺类耐药基因Sul1位于I型整合子的3'末端,而整合子通常存在于可移动的质粒或转座子上;ant(3")-I位于质粒或整合子的基因盒上介导氨基糖苷类的耐药[11];TEM是革兰氏阴性菌最常见的β-内酰胺类耐药基因,由质粒编码[12];tet(A)为革兰氏阴性菌最主要的四环素类耐药基因,通常位于转座子或质粒上[13]。关于水产动物源副溶血弧菌的耐药基因,Yutaka等[6]检测了泰国虾源副溶血弧菌β-内酰胺类和四环素类耐药基因的携带情况,娄阳等[14]分析了上海水产品源副溶血弧菌β-内酰胺类、四环素类、氯霉素类、氨基糖苷类耐药基因的分布。【本研究切入点】副溶血弧菌的耐药性和耐药机制日益受到人们的重视。但目前副溶血弧菌耐药性研究主要集中于耐药表型,有关病原菌耐药表型与耐药基因型相关性以及广西对虾源副溶血弧菌耐药基因的研究报道仍属鲜见。【拟解决的关键问题】对分离自广西沿海养殖凡纳滨对虾的30株副溶血弧菌进行药敏试验,并检测4种不同类别耐药基因的携带情况,分析对虾源副溶血弧菌耐药表型与耐药基因之间的相关性,为凡纳滨对虾弧菌病防控提供科学指导,为副溶血弧菌耐药机制的深入研究提供理论基础。
1 材料与方法
1.1 菌株来源
试验菌株为2014-2016年广西水生动物病害诊断实验室于广西钦州市、防城港市和北海市从凡纳滨对虾肝胰腺分离并鉴定保存的30株副溶血弧菌。其中,来自钦州市14株,防城港市9株,北海市7株(表1)。
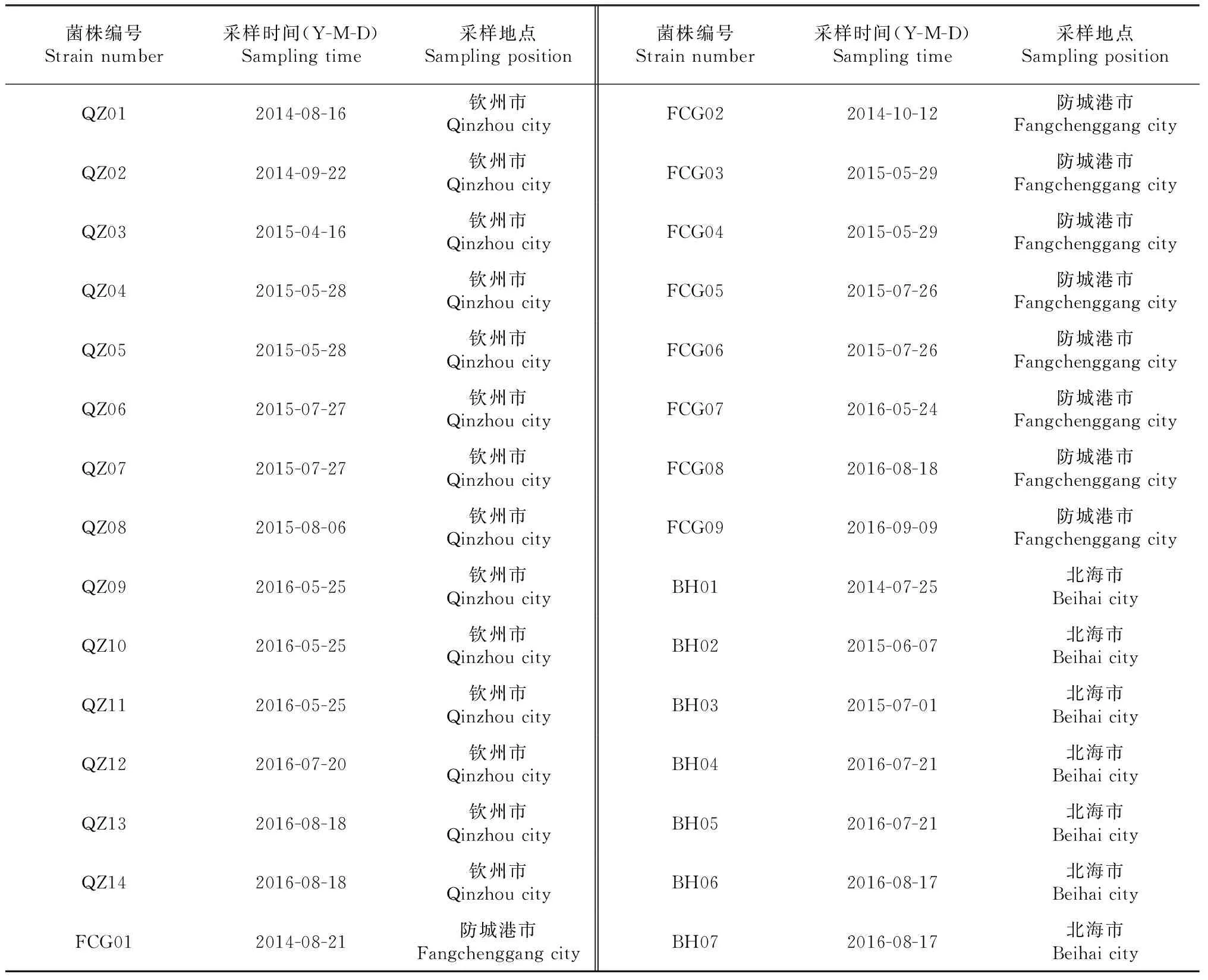
表1 株来源信息
1.2 主要试剂
PCR扩增引物由生工生物工程技术服务(上海)有限公司合成。PCR缓冲液、dNTPs、Taq聚合酶、MgCl2、ddH2O均购于宝生物工程(大连)有限公司。琼脂糖购于美国Invitrogen公司。细菌基因组DNA提取试剂盒购自北京天根生化科技有限公司。引物设计参考Bryan等[15],序列及产物长度见表2。
12种药敏片均购自杭州天和微生物试剂有限公司,包括β-内酰胺类:青霉素G(PG,10 μg/片)、氨苄青霉素(AMP,10 μg/片);四环素类:多西环素(DOX,30 μg/片);氯霉素类:氟苯尼考(FFC,30 μg/片);氨基糖苷类:新霉素(NEO,30 μg/片)、链霉素(STR,10 μg/片)、庆大霉素(GEN,10 μg/片);喹诺酮类:恩诺沙星(ENR,30 μg/片)、环丙沙星(CLX,5 μg/片);磺胺类:复方磺胺嘧啶(SDM,25 μg/片)、磺胺二甲嘧啶(SMD,25 μg/片);大环内酯类:红霉素(ERY,15 μg/片)。所有试验用药敏片均保存于4 ℃冰箱备用。
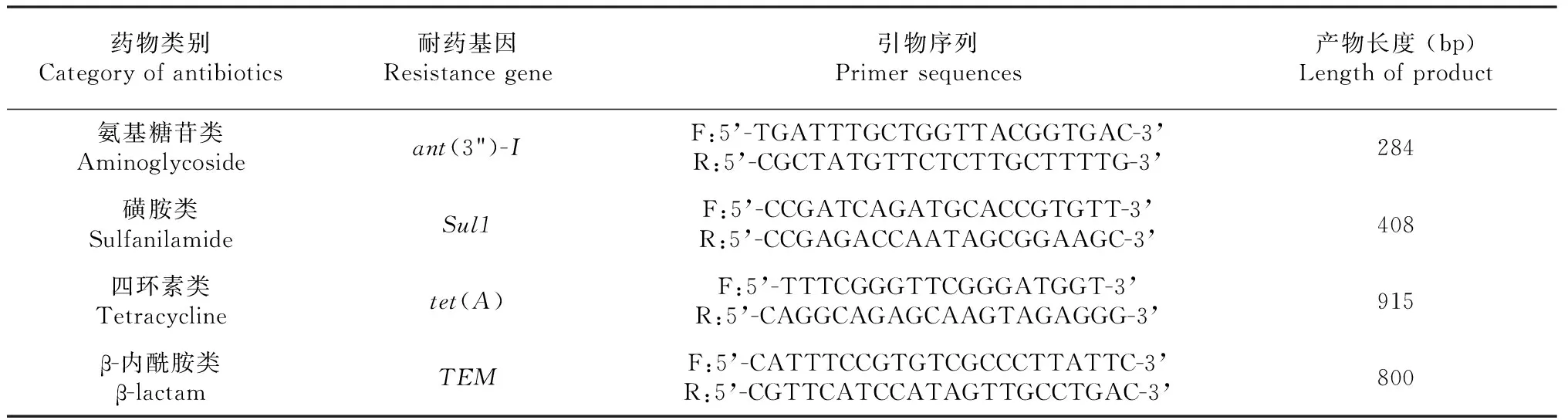
表2 4种耐药基因PCR扩增引物序列

表3 4种耐药基因的PCR反应条件
1.3 药敏试验
采用Kirby-Bauer(K-B)纸片扩散法进行药物敏感性试验。参照美国临床和实验室标准协会(CLSI)抗生素敏感试验标准,将每种抗生素的药敏结果用S(敏感)、I(中介)、R(耐药)进行记录。
1.4 耐药基因的PCR检测
按试剂盒说明书提取细菌基因组DNA,-20 ℃保存备用。4种耐药基因的PCR反应条件见表3。反应体系(25 μl):PCR缓冲液2.5 μl,dNTPs 0.5 μl,MgC122.5 μl,上下游引物各1 μl,Taq聚合酶0.3 μl,DNA模板3 μl,ddH2O补足余量。扩增条件[15]见表3。PCR反应结束后以1.0 %琼脂糖凝胶在100 V电压下电泳35 min,凝胶成像分析系统(北京六一,WD-9413B)观察电泳结果。PCR产物由生工生物工程技术服务(上海)有限公司测序。采用ClustalX及DNAstar软件进行核苷酸序列一致性分析以及系统发育树构建。
对未出现预期条带的菌株,退火温度以上述各温度为中心温度,±5 ℃再做梯度PCR并调试其他条件,反复试验仍无预期条带出现者视为所检测基因检测结果阴性。
1.5 统计分析
耐药表型与耐药基因的符合率计算公式:
符合率(%)=携带耐药基因并具有相应耐药表型菌株数÷具有相应耐药表型菌株总数。
使用SPSS 18.0中Fisher’s exact test进行菌株耐药表型和耐药基因的相关性分析。
2 结果与分析
2.1 副溶血弧菌药物敏感性
检测的副溶血弧菌对12种抗生素存在不同程度的多重耐药现象(表4),其中,6重耐药1株(3.3 %),5重耐药3株(10.0 %),4重耐药10株(33.3 %),3重耐药13株(43.3 %)。30株副溶血弧菌的优势耐药谱是SDM/SMD/PG/AMP和SMD/PG/AMP,所含菌株数分别占被检菌株数的23.3 %和20.0 %,其余依次为DOX/SDM/SMD/PG/AMP、SDM/SMD/PG、DOX/SMD/PG、SMD/PG(各占6.7 %),PG/AMP/ERY、SMD、STR/SMD/PG、STR/SMD/PG/AMP、SMD/PG/CLX、GEM/SMD/PG/AMP、GEN/SDM/SMD/PG/AMP、GEN/DOX/SDM/SMD/PG/AMP、DOX/SMD/PG/AMP(各占3.3 %)。
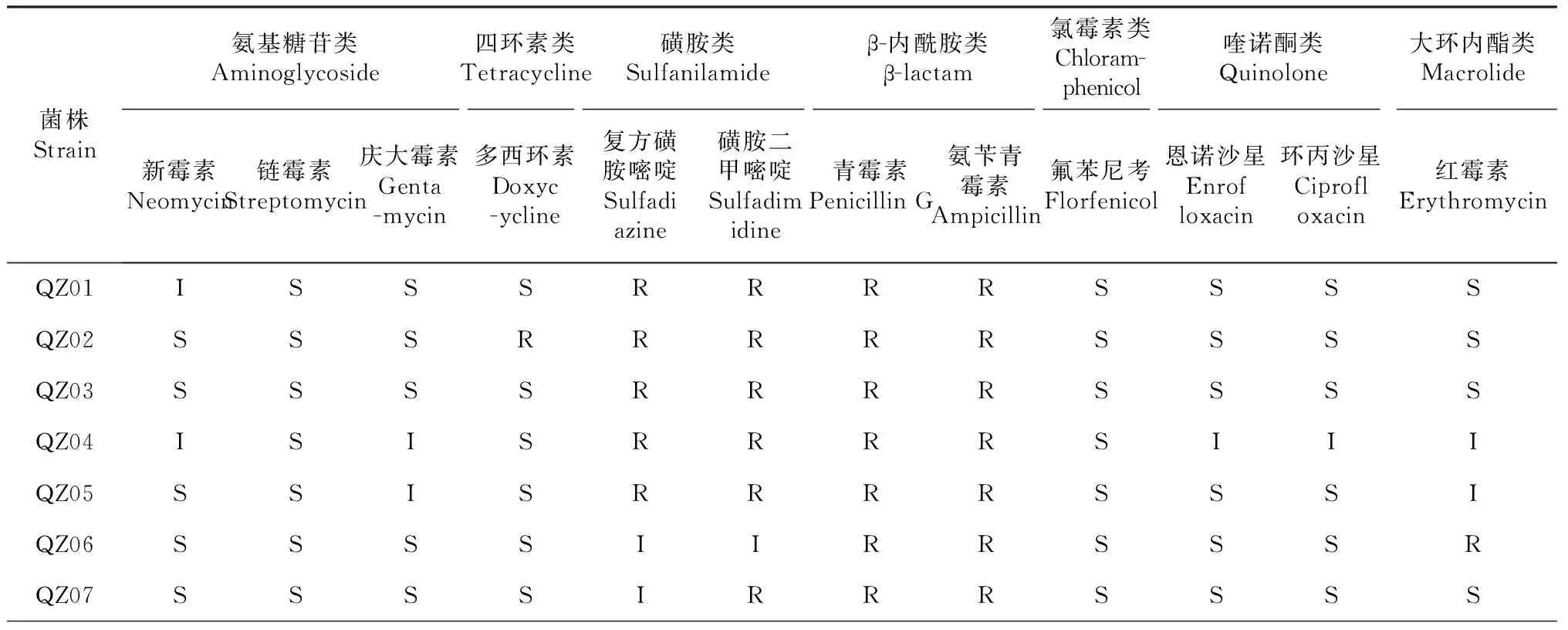
表4 30株副溶血弧菌菌株的药物敏感性
续表4 Continued table 4
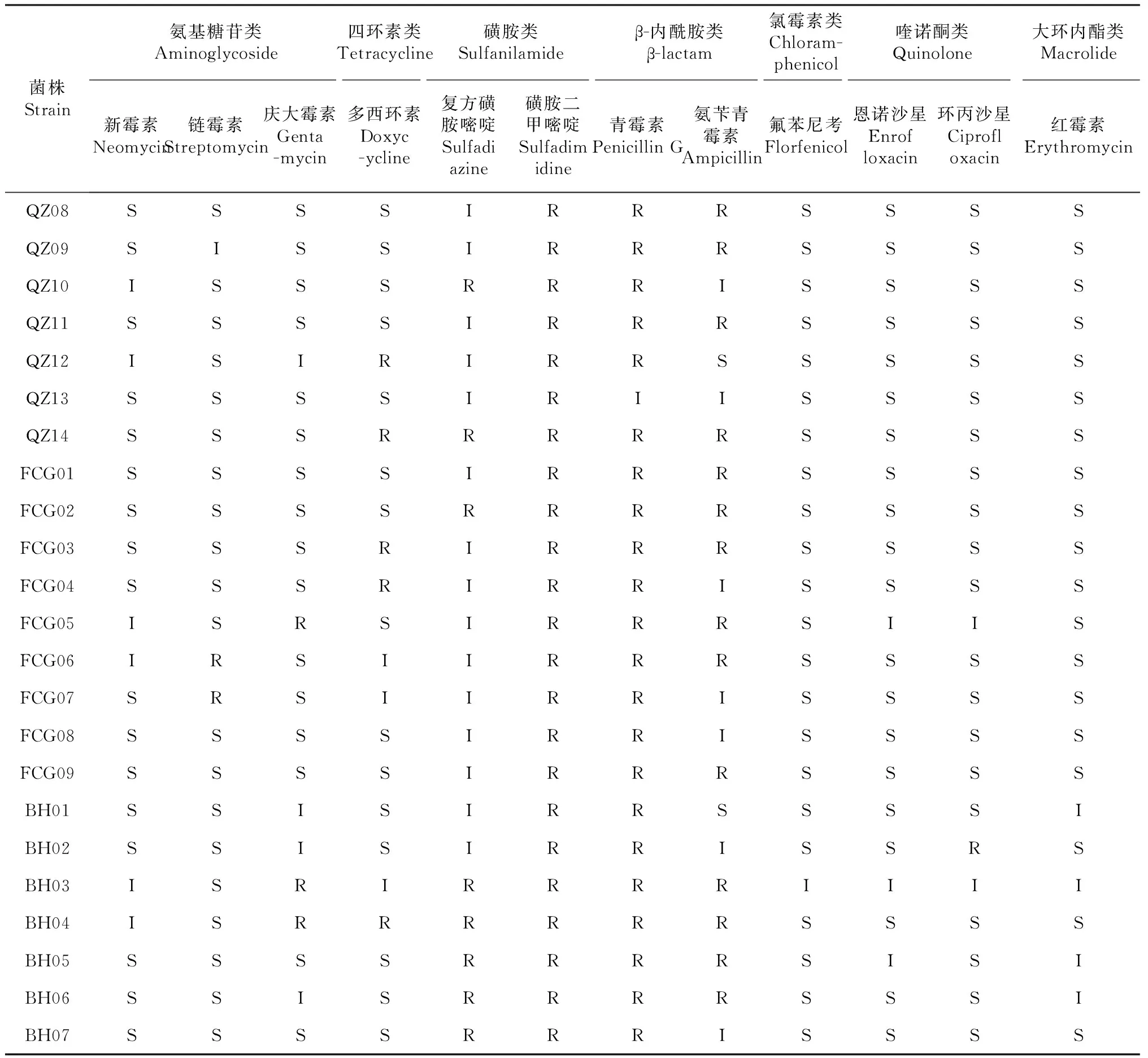
菌株Strain氨基糖苷类Aminoglycoside四环素类Tetracycline磺胺类Sulfanilamideβ-内酰胺类β-lactam氯霉素类Chloram-phenicol喹诺酮类Quinolone大环内酯类Macrolide新霉素Neomycin链霉素Streptomycin庆大霉素Genta-mycin多西环素Doxyc-ycline复方磺胺嘧啶Sulfadiazine磺胺二甲嘧啶Sulfadimidine青霉素Penicillin G氨苄青霉素Ampicillin氟苯尼考Florfenicol恩诺沙星Enrofloxacin环丙沙星Ciprofloxacin红霉素ErythromycinQZ08SSSSIRRRSSSSQZ09SISSIRRRSSSSQZ10ISSSRRRISSSSQZ11SSSSIRRRSSSSQZ12ISIRIRRSSSSSQZ13SSSSIRIISSSSQZ14SSSRRRRRSSSSFCG01SSSSIRRRSSSSFCG02SSSSRRRRSSSSFCG03SSSRIRRRSSSSFCG04SSSRIRRISSSSFCG05ISRSIRRRSIISFCG06IRSIIRRRSSSSFCG07SRSIIRRISSSSFCG08SSSSIRRISSSSFCG09SSSSIRRRSSSSBH01SSISIRRSSSSIBH02SSISIRRISSRSBH03ISRIRRRRIIIIBH04ISRRRRRRSSSSBH05SSSSRRRRSISIBH06SSISRRRRSSSIBH07SSSSRRRISSSS
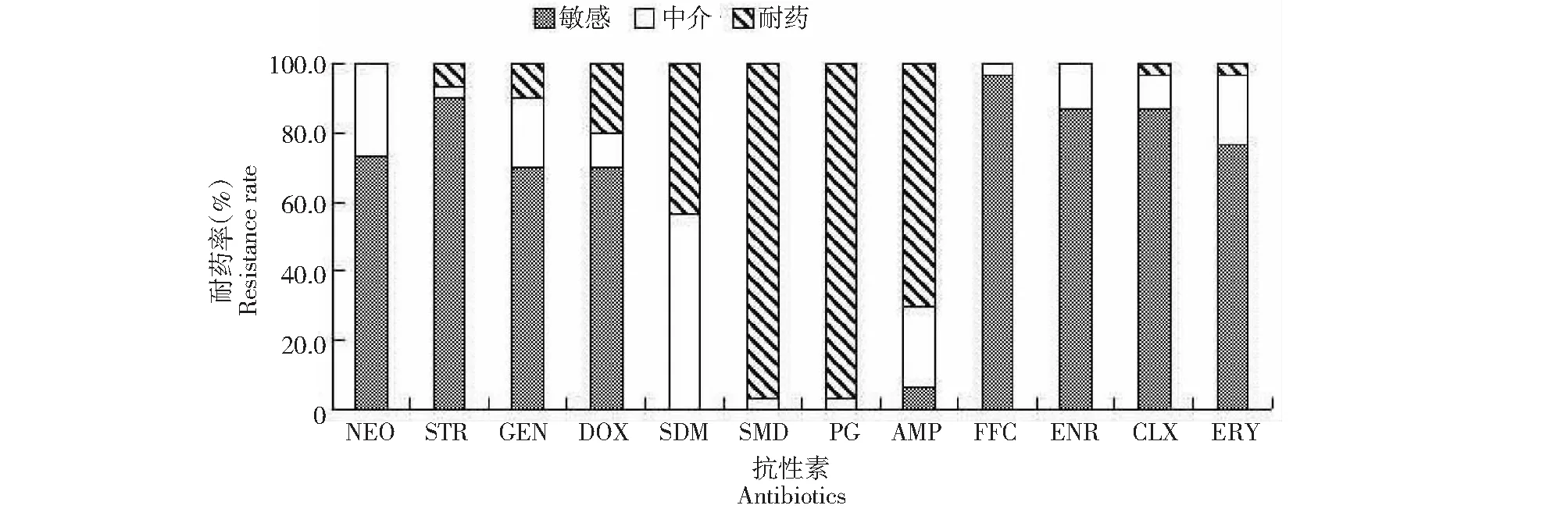
PG:青霉素;G;AMP:氨苄青霉素;SDM:复方磺胺嘧啶;SMD:磺胺二甲嘧啶;DOX:多西环素;ERY:红霉素;FFC:氟苯尼考;NEO:新霉素;STR:链霉素;GEN:庆大霉素;ENR:恩诺沙星;CLX:环丙沙星PG:Penicillin G; AMP:Ampicillin; SDM:Sulfadiazine; SMD:Sulfadimidine; DOX:Doxycycline; ERY:Erythromycin; FFC:Florfenicol; NEO:Neomycin; STR:Streptomycin; GEN:Gentamycin; ENR:Enrofloxacin; CLX:Ciprofloxacin图1 副溶血弧菌菌株对抗生素的耐药情况比较Fig.1 Comparison of resistant situation of Vibrio parahaemolyticus strains against antibiotics

M为100 bp DNA ladder,N为阴性对照;1~9为被检测的部分菌株M indicates the 100 bp DNA ladder, N indicates the negative control; Lane 1-9 indicate some strains detected图2 部分菌株4种耐药基因的PCR扩增结果Fig.2 PCR amplification results of four kinds of antibiotics resistance genes in some strains
30株副溶血弧菌对12种抗生素的耐药程度也不同(图1),对β-内酰胺类和磺胺类抗生素的耐药性相对较高,对青霉素G、氨苄青霉素的耐药率分别为96.7 %和70.0 %,对磺胺二甲嘧啶、复方磺胺嘧啶的耐药率分别为96.7 %和43.3 %,远超过其他类抗生素的耐药率。所有菌株对新霉素、氟苯尼考、恩诺沙星均为敏感或中介;其中,对氟苯尼考的敏感率最高,达96.7 %,对新霉素、链霉素、庆大霉素、多西环素、恩诺沙星、环丙沙星和红霉素的敏感率为70.0 %~90.0 %。
2.2 副溶血弧菌的耐药基因检测结果
以30株副溶血弧菌的总DNA为模板,采用PCR方法扩增4种耐药基因。4种耐药基因的PCR扩增结果见图2。部分受试菌株能扩增出大小约为284 bp的ant(3")-I基因、408 bp的Sul1基因和800 bp的TEM基因片段,与预期产物大小相符。将阳性PCR扩增产物进行测序,并将所得序列与NCBI数据库中的相应基因序列进行比对,结果显示,PCR阳性样品测序片段与NCBI数据库中的对应基因序列相似性高。经反复试验,均未扩增出tet(A)目的条带,表明所有供试菌株均不携带tet(A)基因。
根据检测结果(表5),30株受试菌株的ant(3")-I、Sul1、tet(A)、TEM基因的检出率分别为6.7 %(2/30)、43.3 %(13/30)、0.0 %(0/30)和13.3 %(4/30)。共包含5种耐药基因型,即ant(3")-I-Sul1+tet(A)-TEM-、ant(3")-I-Sul1-tet(A)-TEM-、ant(3")-I-Sul1-tet(A)-TEM+、ant(3")-I+Sul1-tet(A)-TEM-和ant(3")-I-Sul1+tet(A)-TEM+,分布率依次为40.0 %(12/30)、40.0 %(12/30)、10.0 %(3/30)、6.7 %(2/30)、3.3 %(1/30)。
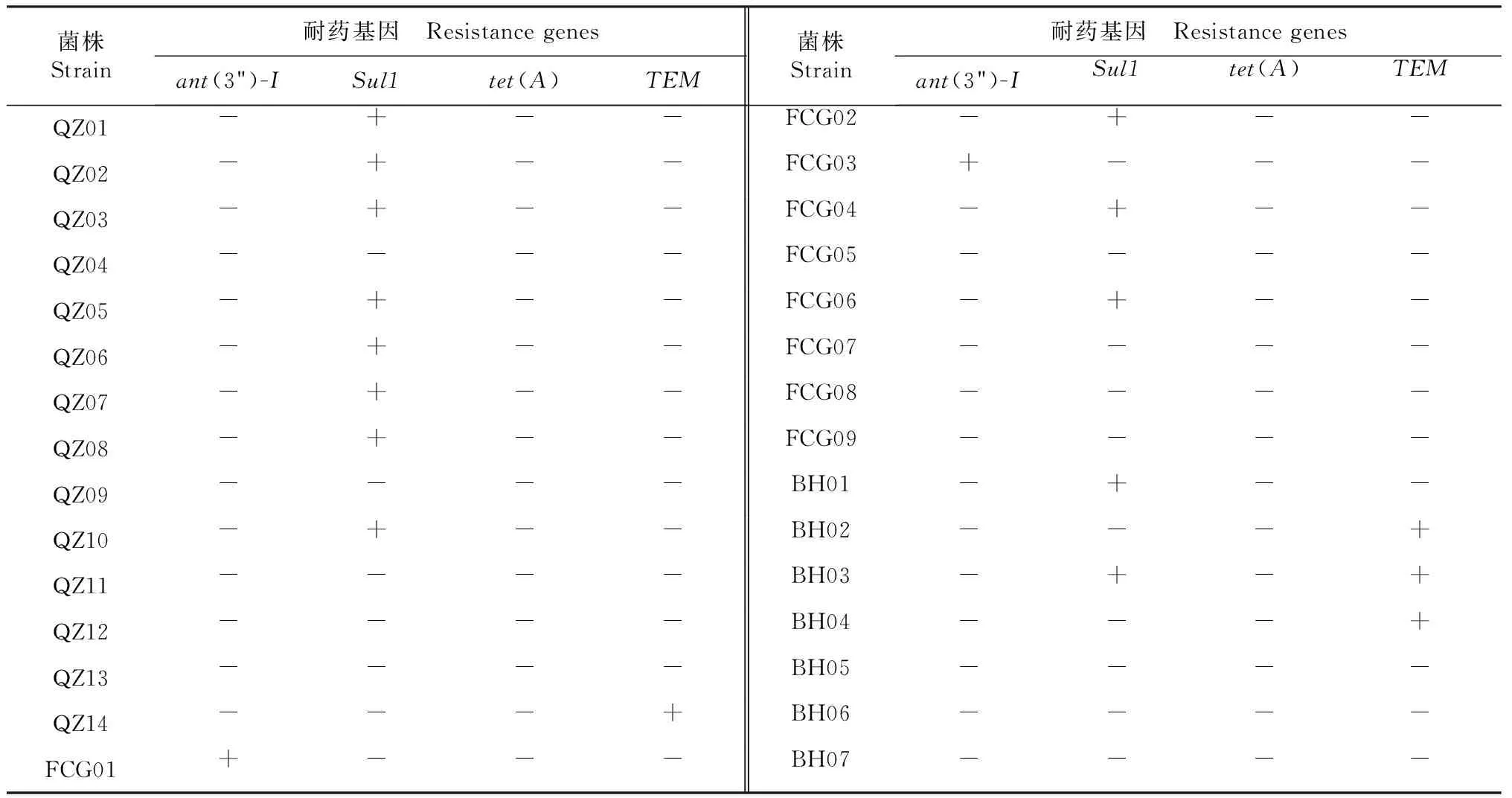
表5 副溶血弧菌菌株耐药基因的检测结果
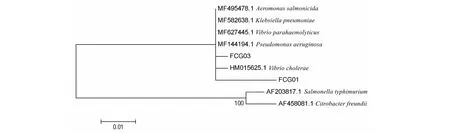
图3 基于ant(3")-I部分序列的系统发育树Fig.3 Phylogenetic tree of partial sequences of ant(3")-I
在30株受试菌株中,只有FCG01和FCG03菌株携带ant(3")-I基因,GenBank登录号分别为MG870346和MG870347。基于284 bp的ant(3")-I部分序列构建系统发育树(图3),可见菌株FCG01和FCG03的ant(3")-I部分序列与副溶血弧菌(V.parahaemolyticus,MF627445.1)、霍乱弧菌(V.cholerae,HM015625.1)、杀鲑气单胞菌(Aeromonassalmonicida,MF495478.1)、肺炎克雷伯氏菌(Klebsiellapneumoniae,MF582638.1)、铜绿色假单胞菌(Pseudomonasaeruginosa,MF144194.1)聚为一支,在进化关系上比较近;与弗氏柠檬酸杆菌(Citrobacterfreundii,AF458081.1)、鼠伤寒沙门氏杆菌(Salmonellatyphimurium,AF203817.1)在进化关系上距离较远。基于ant(3")-I部分序列,与副溶血弧菌、杀鲑气单胞菌、肺炎克雷伯氏菌、铜绿色假单胞菌、霍乱弧菌、鼠伤寒沙门氏杆菌、弗氏柠檬酸杆菌的核苷酸一致性:FCG01分别为98.2 %、98.2 %、98.2 %、98.2 %、97.9 %、88.8 %和88.4 %;FCG03分别为99.6 %、99.6 %、99.6 %、99.6 %、99.3 %、89.9 %和89.5 %。
有4株菌株(QZ14、BH02、BH03和BH04)携带TEM基因,GenBank登录号依次为MG870348、MG931979、MG931980和MG931981。基于TEM部分序列的系统发育树见图4。根据图4,4株菌株的TEM部分序列与副溶血弧菌(V.parahaemolyticus,JF327791.1)、嗜水气单胞菌(Aeromonashydrophila,FJ767900.1)、荧光假单胞菌(Pseudomonasfluorescens,GU733416.1)聚为一支,在进化关系上比较近;与摩氏摩根氏菌(Morganellamorganii,GU011973.1)、肺炎克雷伯氏菌(Klebsiellapneumoniae,LNIM01000067.1)、粘质沙雷菌(Serratiamarcescens,AY538699.1)、大肠杆菌(Escherichiacoli,KU664682)在进化关系上距离较远。基于TEM部分序列,与副溶血弧菌、嗜水气单胞菌、荧光假单胞菌、摩氏摩根氏菌、肺炎克雷伯氏菌、粘质沙雷菌、大肠杆菌的核苷酸一致性:QZ14分别为100 %、100 %、100 %、99.7 %、99.6 %、99.3 %和99.3 %;BH02分别为99.7 %、99.9 %、99.9 %、99.6 %、99.6 %、99.4 %、99.1 %和99.1 %;BH03分别为100 %、100 %、99.9 %、99.7 %、99.6 %、99.3 %和99.3 %;BH04分别为99.9 %、99.9 %、99.9 %、99.6 %、99.6 %、99.1 %和99.1 %。
检测到Sul1基因的菌株有13株,分别为QZ01、QZ02、QZ03、QZ05、QZ06、QZ07、QZ08、QZ10、FCG02、FCG04、FCG06、BH01和BH03,GenBank登录号依次为MG870352~MG870362、MG931977、MG931978。根据基于Sul1部分序列的系统发育树(图5)可知,上述菌株的Sul1部分序列与副溶血弧菌(V.parahaemolyticus,MF627445.1)、霍乱弧菌(V.cholerae,DQ227350.1)、大肠杆菌(Escherichiacoli,U04278.1)聚为一支,进化关系较近;与鼠伤寒沙门氏杆菌(Salmonellatyphimurium,DQ205474.1)序列在进化关系上相对较远。DNAStar软件分析结果显示,本试验被测菌株的Sul1部分序列与副溶血弧菌、霍乱弧菌、大肠杆菌核苷酸一致性均为100 %,与鼠伤寒沙门氏杆菌核苷酸一致性为97.7 %。

图4 基于TEM部分序列的系统发育树Fig.4 Phylogenetic tree of partial sequences of TEM

图5 基于Sul1部分序列的系统发育树Fig.5 Phylogenetic tree of partial sequences of Sul1
2.3 副溶血弧菌对4类抗生素耐药表型与耐药基因的相关性
在30株副溶血弧菌中,链霉素和庆大霉素耐药表型与ant(3")-I基因之间符合率均为0 %,相关性不显著(P>0.05,下同)。由于未检测到对新霉素有抗性的菌株,因此新霉素耐药表型与ant(3")-I基因之间的相关性无法计算。复方磺胺嘧啶耐药表型与Sul1基因之间符合率为53.8 %,相关性不显著。磺胺二甲嘧啶耐药表型与Sul1基因之间符合率为41.4 %,相关性不显著。由于未检测到tet(A)基因,因此多西环素耐药表型与tet(A)基因之间的相关性也无法计算。青霉素G的耐药表型与TEM基因之间符合率为13.8 %,相关性不显著。氨苄青霉素的耐药表型与TEM基因之间符合率为14.3 %,相关性不显著(表6)。
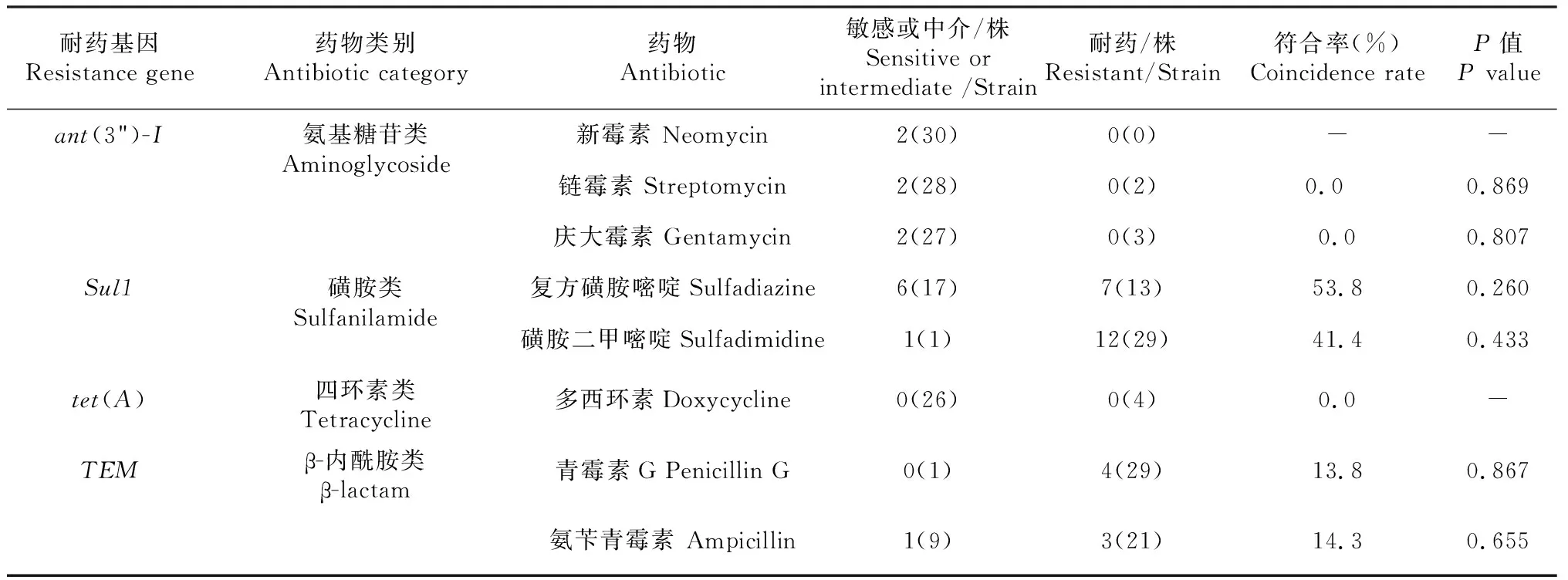
表6 副溶血弧菌对4类药物耐药性和耐药基因的相关性
注:“-”表示无法计算。括号外的数字为耐药基因为阳性的菌株数量,括号里的数字为对应耐药表型的菌株总数。
Note:‘-’ indicates that the value could not be calculated. The number outside the bracket indicated the quantity of strains which carry the resistance gene, and the number inside the bracket indicated the quantity of strains possessing the relevant antibiotic resistance.
3 讨 论
3.1 广西凡纳滨对虾源副溶血弧菌的耐药性
水生养殖动物源副溶血弧菌的耐药现象普遍存在,在本试验中,广西沿海凡纳滨对虾源副溶血弧菌对青霉素G和氨苄青霉素耐药程度较高,与在浙江海产品[7]、江苏凡纳滨对虾[8]源弧菌中的研究结果类似,这可能是因为青霉素G主要对革兰氏阳性菌有效;而氨苄青霉素虽具有广谱抗菌活性,但弧菌可能容易对其发生耐药[6]。本试验中,试验菌株对复方磺胺嘧啶和磺胺二甲嘧啶耐药率较高,与对浙江海产品来源副溶血弧菌的研究结果[7]相似;试验菌株也具有一定的多重耐药性。有学者认为,由于磺胺类滥用现象严重,目前在我国环境中磺胺类的检出率与浓度远高于其他国家[16],因而推测副溶血弧菌对磺胺类的耐药程度可能与环境中的磺胺类残留量较高有关,多种抗生素可在虾池中长期存在,并诱导水体细菌产生耐药性,同时造成耐药基因的广泛传播[16]。但2013-2015年,本研究团队对某公司设在沿海各地的5个渔药销售店进行初步调查结果,抗生素类药物在广西沿海的销售量很少,所调查的5个渔药店基本上不销售抗生素类药物;另对广西沿海近6500 hm2虾塘的调查结果也证明,在对虾养殖过程中极少使用磺胺类等抗生素类药物,虾病都是通过改水、控料等方法进行防控,一旦发生虾病,几乎都是即卖或排塘。表明导致本试验的试验菌株对复方磺胺嘧啶和磺胺二甲嘧啶耐药率较高和多重耐药性形成的原因较为复杂,除与抗生素的滥用有关外,可能还与其他因素有关。总之,在对虾等水生动物养殖过程中,建议养殖户在选择用药时应以药敏试验结果为依据,做到科学用药。根据本试验结果,虽然氟苯尼考的敏感率达96.7 %,可作为广西沿海凡纳滨对虾副溶血弧菌病防控的首选药物,但也不可乱用和滥用,以最大限度减少耐药菌株的产生。
3.2 4类耐药基因检测以及与耐药表型的相关性
在耐药基因携带情况与耐药表型相关性的研究结果中,靳晓敏等[15]认为,ant(3")-I为弧菌主要的氨基糖苷类修饰酶,能使游离羟基核苷化从而导致细菌产生耐药性[15]。但在本试验中,携带ant(3")-I的2株副溶血弧菌均对3种氨基糖苷类药物敏感。在对单增李斯特菌(Listeriamonocytogenes)的研究中也有类似结果,这一现象被认为是ant(3")-I基因需在一定条件下才能表达功能蛋白[17]。Sul1基因编码的二氢叶酸合成酶能保护细菌不被磺胺类所抑制[15]。而本试验携带Sul1基因的13株副溶血弧菌中,有6株对复方磺胺嘧啶中介、1株对磺胺二甲嘧啶中介,可见Sul1和磺胺类耐药表型间并非完全对应,这是否与菌株生理条件或环境导致Sul1控制的性状未能得到有效表达有关尚待进一步深入研究。tet(A)为革兰氏阴性菌最主要的四环素类耐药基因,其编码的tet(A)蛋白可将抗生素外排至细胞外导致细菌耐药[13],本试验在30株试验菌株中均未检测到tet(A)基因,与对大菱鲆源鳗弧菌的检测结果[15]一致。尽管未检测到tet(A),仍有13.3 %的试验菌株对多西环素耐药。已知四环素类耐药机制除tet(A)介导的外排泵机制外,还有核糖体保护和四环素类钝化机制等[18],据此推测,本试验的副溶血弧菌对四环素类抗生素可能存在其他耐药机制。TEM是革兰氏阴性菌最常见的β-内酰胺类耐药基因,其编码的β-内酰胺酶可水解各类青霉素类[12]。本试验TEM的检出率(13.3 %)低于宁波(91.47 %)[19]而高于泰国(0 %)[6]。造成TEM检出率不同的原因可能与细菌宿主及所处环境、地理位置、检测条件等不一致有关。本试验中青霉素G、氨苄青霉素的耐药表型和TEM相关性不显著(P>0.05),有学者认为菌株对青霉素G、氨苄青霉素的耐药性可能还受环境的抗生素含量、耐药基因拷贝数等影响[20]。
据本试验结果,未发现4类抗生素耐药表型和耐药基因之间存在显著相关性,综合前人的研究成果,认为原因可能有:①菌外膜蛋白有可能参与调节抗生素经细菌外膜的通透性[21]。如副溶血弧菌具有外膜蛋白复合物VmeA-VmeB-vpoC,该复合物可将抗生素泵出细菌胞外以达到耐受抗生素的目的[22]。②不同菌株其基因拷贝数不一,导致部分菌株耐药基因未被检出[20]。③一些耐药基因在染色体、质粒及其他可移动遗传元件上的分布情况复杂,其有效表达受到各种因素影响[10]。I型整合子耐药基因(如ant(3")-I和Sul1)与整合酶基因intI的表达呈此消彼长的状况[23]。位于转座子上的tet(A)基因其转录和翻译受启动子、茎环结构的影响[18]。④不同菌株其耐药基因启动子强弱不一[20],如I型整合子中耐药基因ant(3")-I和Sul1的表达与启动子的种类、强弱以及与启动子的距离密切相关[23]。TEM基因4种启动子强弱顺序为P4>Pa/Pb>P3[24]。⑤菌株可能存在其他耐药基因介导的耐药机制等。例如当外部环境条件不适宜细菌生存时,细菌可通过整合外源耐药基因及调节整合子中下游基因盒的表达以适应外部变化[23]。⑥细菌耐药形成与抗生素使用方式有关。如嗜水气单胞菌(Aeromonashydrophila)等病原菌在亚抑菌浓度诱导下可以获得对氟苯尼考等抗生素的高耐药性[25-26]。可见,除了耐药基因,副溶血弧菌对4类抗生素的耐药性可能还受上述6个方面情况的影响,导致本试验4类抗生素耐药表型和耐药基因之间的相关性不显著。但由于许多耐药基因可以随可移动遗传组件在菌株之间水平转移[10],耐药基因的存在给凡纳滨对虾弧菌病防控带来的风险仍需引起高度重视。总之,建议加强细菌耐药监测和耐药基因的研究,以提高凡纳滨对虾弧菌病防控效率,同时为进一步弄清弧菌耐药机制提供依据。
4 结 论
广西沿海30株凡纳滨对虾源副溶血弧菌对磺胺二甲嘧啶和青霉素G耐药性最强,对氟苯尼考敏感性最高。30株弧菌的优势耐药基因型是ant(3")-I-Sul1+tet(A)-TEM-和ant(3")-I-Sul1-tet(A)-TEM-。4类抗生素耐药基因与耐药表型之间均未发现显著相关性。
