最大团问题的三链DNA计算模型
2018-09-07陈海芳殷志祥
陈海芳,殷志祥
(安徽理工大学数学与大数据学院,安徽淮南232001)
最大团是著名的NP(Non-deterministic Polynomial)完全问题,也是图论中一个重要的研究方向,无论是在理论研究还是在实际应用中都具有重要意义,诸如市场分析、方案选择、信号传输、计算机视觉、故障诊断等领域都有直接的应用。从1957年Hararv和Ross首次给出最大团的一个确定性算法以来,人们就在不停地寻求更好的方案。但是随着图规模的增大,确定性算法无法有效解决此类NP问题,人们又开始寻找新的突破。随着分子生物学的发展以及DNA分子具有高并行性和信息储存量的优势,研究者们开始着手使用DNA计算来求解NP问题。1994年,Adleman[1]利用DNA编码成功地解决了一个具有7个顶点的有向Hamilton路径问题,开创了DNA计算的先河;1997年,Ouyang等[2]又利用DNA计算成功求解了6个顶点的图的最大团问题;文献[3]利用纳米材料属性,提出Tile自组装高效模型,减少了解的空间规模,提高了运算的并行性;文献[4]基于二维DNA分子tiler自组装,构建了一个三维DNA图结构来建立模型求解;文献[5]使用DNA三维自组装的算法来解决最大团问题,使得计算的时间复杂度是线性的,瓦片结构的独特类型也使得所需的DNA链数是恒定的;文献[6-7]分别运用圆形DNA分子模型和粘贴模型求解了最大团问题。在上述DNA计算中,都是利用双链DNA来求解问题,为了更好地解决最大团问题,本文引入三链DNA的概念。
1 三链DNA的形成
三链DNA是在双螺旋结构的基础上,以Hoogsteen和反式Hoogsteen型氢键与新加入的第三条链相连接形成三螺旋结构。2004年,Shigemori等发现在RecA蛋白及ATPrS存在的情况下,寡聚脱氧核苷酸与线性双螺旋DNA可形成稳定的三链结构[8]。三链DNA的具体形成过程如图1所示。三链DNA在参与反应时,由于反应前参与反应的数据池中皆为双链DNA,使得形成的三链结构很稳定,这样避免了DNA链在参与反应时碱基的错配,也不会出现由于单链过长而出现发夹结构的现象。使用三链结构可以减少错误率,同时也可以降低编码的复杂度。三链模型已经成功解决了许多图论中的问题[9-17]。
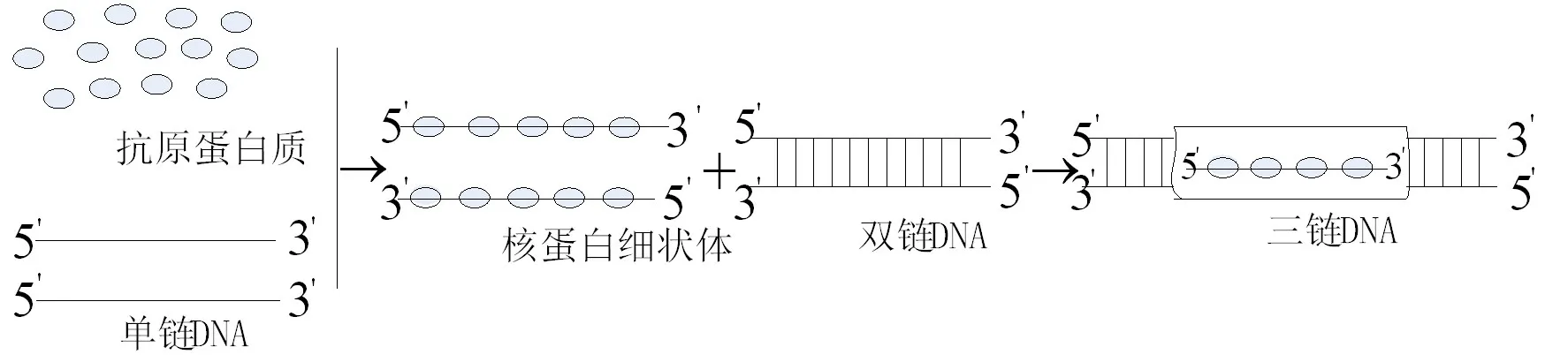
图1 三链DNA的形成
2 最大团问题
设G是一个图,V(G)和E(G)分别表示图的顶点集和边集,S是G的一个顶点子集,若S中任意两点都与E(G)中的边相连,则称S是图G的一个团。若对G的任意其他团S′,都有 ||S′≤ ||S,则称S是G的最大团。图2是一个具有6个顶点的简单图。
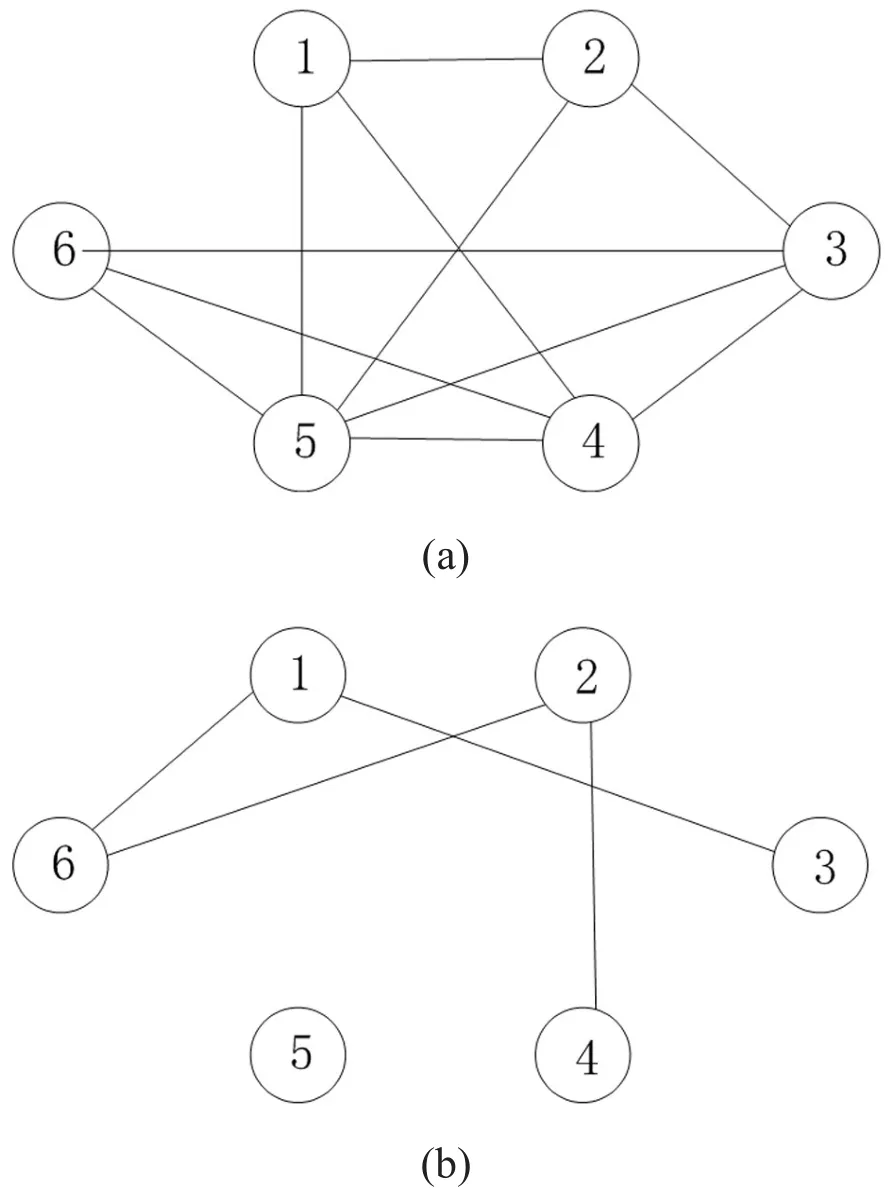
图2 6个顶点的简单图
3 最大团问题的DNA算法
3.1 基本算法
步骤1:DNA单链根据Waston-Click原理生成数据池;
步骤2:删除不满足条件的解;
步骤3:输出结果。
3.2 生物算法
对于n个顶点的简单图的团,可以用一个n位的二进制的数字串来表示。具体表示方式:当图的某个顶点在团中时,编码为1;当该顶点不在团中时,编码为0。如图2中,顶点1、2、5组成的一个团可以用110 010来表示。因此可以用0、1组成的n位数字串来表示所有可能的情况,称所有二进制数的集合为数据池。为了满足最大团的定义,需要对数据池进行筛选,只保留正确解,具体的操作:
(i)对图的各个顶点进行编码,构造出代表不同顶点的DNA单链,然后将这些单链与连接酶一起加入到溶液中进行生化反应得到双链DNA,即生成初始数据池T0。
(ii)为了满足团定义的要求,需要对初始数据池中顶点的组合进行筛选。结合图G的补图,因为图中的团在其图的补图中对应的是空图,所以在补图中相连的两顶点在原图中是断开的,因此,他们不可能是同一团中的顶点。因此对应于编码来说,在补图中相连的两个顶点不可能同时为1。据此,借助三链DNA进行检测,配制代表补图中相连的两个顶点的DNA片段的补链,将其与抗原蛋白质相结合,制作成探针,加入到数据池中,形成三链DNA。没有形成三链的就是满足在补图中未被连接的顶点,则该顶点都包含在团中。利用生物操作技术将数据池中的三链DNA去除,剩下的双链DNA就是满足团定义的点所对应的DNA链。最后根据1的个数(即团中所包含顶点的个数)来读出最大团的规模。
(iii)通过凝胶电泳来对数据池中的最大团方案进行分离,读出最大团。
4 算法实例
下面以图2为例,求解图的最大团问题。
(1)对图2中简单图的各个顶点进行编码,顶点的编码设计如图3所示:中间是表示顶点位置的Vi,两边是粘性末端Si,并且第i个顶点右边的黏性末端的补链与第i+m个顶点左边的黏性末端的补链可以通过连接酶链接构成i→i+m或。将Vi、和放入缓冲液中,在一定的温度下,根据Watson-Crick原则,随机生成DNA双链,这些双链里包含了顶点组合的所有可能,即生成了初始的数据池T0。又因为每个顶点可能在团中,也可能不在团中,所以每个顶点有两种不同的编码。图2中有6个顶点,共有12段不同的编码。为了对顶点进行区分,给它设计为不同的编码与长度:定义每一个黏性末端的长度为5 bp,顶点的长度为10 bp。对应于编码:若Vi的值在团中,由定义知Vi=1,则Vi肯定不在其补图中,故在补图中的长度为0 bp;若Vi的值不在团中,由定义知Vi=0,则Vi在其补图中,长度为10 bp。我们可以知道最长的DNA链长为120 bp(包含6个顶点的长度和12个黏性末端的长度),对应于数字串(000 000),最短的DNA链长为60 bp(没有顶点,只有12个黏性末端),对应于数字串(111 111)。由于检测时是借助补图来进行的,因此要寻找代表最大团的双链DNA,就是寻找满足团的定义的补图中所含顶点最少的双链DNA,即最短的双链DNA,并且为了防止反应过程中错配现象的发生,不同序列具有相同碱基的长度不超过4 bp,具体的编码如表1所示。
(2)从图G的补图出发,检查补图中相连的点。在图2(b)中,顶点1、3相连接,当数据池中顶点1、3同时存在时(即编码分别为11、31),先将代表初始数据池的试管T0分为T1和T2,然后将11、31的补链分别与抗原蛋白质混合,制作成探针P1,P2。将探针P1、P2分别放入到试管T1、T2中,探针P1在试管T1中与11进行杂交反应,形成三链DNA;同样,探针P2在试管T2中与31进行杂交反应,形成三链DNA。利用生物操作将试管T1、T2中的三链DNA去除,再将T1和T2合并为T0,此时的试管T0中不再含有顶点1、3同时存在的数字串了。同理,补图中顶点1、6有边连接,即顶点1、6同时存在时(编码分别为11、61)制作11、61的补链,操作方法同上,此时得到的T0里面没有顶点1、6同时存在的数字串。依次对补图中相连的顶点进行该操作,最终得到的T0里面都是满足团定义的顶点,根据DNA双链的长度可以读出团的规模。

图3 DNA链的设计

表1 顶点及颜色的编码
(3)为了读出最大团及每个顶点,我们需要借助凝胶电泳来完成。由于检测是借助补图来操作的,所以在所有团中寻找最大团,就是寻找补图中含有顶点数最少的团。含有顶点数较少的双链分子量小,在凝胶电泳中的迁移速度较快。利用凝胶电泳技术找到迁移速度最快的DNA链,也就是最短的DNA片段。利用PCR扩增和纯化后,提取出这些链,采用非放射性标记DNA测序的方法进行测序,可以得到最短的DNA链的排序,也就得到了图的最大团。
5 结束语
本文通过三链DNA来求解图的最大团问题,并给出了具体的实例验证该方法的可行性。在三链DNA模型中,在反应前参与反应的都是双链DNA,稳定性好,避免了单链DNA在反应过程中发生错配现象,形成发夹结构,降低了错误率,并且操作较简单,无需对双链DNA进行解链,直接将探针放入试管即可,避免了在加热、解链、退火过程中产生伪解,提高了效率。在本文中,仅讨论了顶点数较少的图,对于顶点数较多的图,也可用此方法来求解,但实际操作中可能会存在一些不足。例如随着顶点数的增加,所需要的DNA序列也相应增加,呈线性增长ο(2n);在筛选的过程中未能实现自动化操作,需要逐一进行检验;在最后解的读取过程中,也需要依靠相关生物手段的进步才能准确地读出解。总体而言,该模型较好地处理了图的最大团问题,并且可以使用该模型解决一定规模的最大团问题。
