替加环素不敏感鲍曼不动杆菌的分子流行病学及耐药机制
2018-08-28何秀娟李荷楠王启赵春江李曙光王晓娟张雅薇王若冰尹玉瑶靳龙阳张菲菲王辉
何秀娟,李荷楠,王启,赵春江,李曙光,王晓娟,张雅薇,王若冰,尹玉瑶,靳龙阳,张菲菲,王辉
王辉 北京大学人民医院检验科主任、教授、博导,北京大学医学部检验学系主任,国家自然科学基金杰出青年获得者,中国医促会临床微生物与感染分会主委、中华医学会微生物学和免疫学分会常委兼临床微生物学组组长、美国临床和实验室标准化协会 (CLSI) 药敏委员会折点工作组委员。共发表SCI论文90余篇;主持及参与制定专家共识17项、临床指南3项、行业标准5项;主编“十二五”精品书籍《临床微生物学检验》,主译专业经典书籍美国《临床微生物学手册》 (第11版)。

替加环素不敏感鲍曼不动杆菌的分子流行病学及耐药机制
何秀娟,李荷楠,王启,赵春江,李曙光,王晓娟,张雅薇,王若冰,尹玉瑶,靳龙阳,张菲菲,王辉
北京大学人民医院 检验科,北京 100044

何秀娟, 李荷楠, 王启, 等. 替加环素不敏感鲍曼不动杆菌的分子流行病学及耐药机制. 生物工程学报, 2018, 34(8): 1218–1225.He XJ, Li HN, Wang Q, et al. Molecular epidemiology and resistance mechanisms of tigecycline-non-susceptible Acinetobacter baumannii. Chin J Biotech, 2018, 34(8): 1218–1225.
为探讨替加环素不敏感鲍曼不动杆菌的耐药机制,为院内感染控制及临床合理用药提供理论依据,采用琼脂稀释法和微量肉汤稀释法检测全国多中心12个城市20家医院临床分离的94 株非重复的替加环素不敏感鲍曼不动杆菌的最低抑菌浓度 (Minimum inhibitory concentration,MIC),应用多位点序列分型 (Multilocus sequence typing,MLST) 技术进行分子流行病学研究,应用eBURST软件对MLST结果进行分析;用PCR和测序技术分析常见耐药基因 (OXA-40-like、OXA-58-like、OXA-23-like、OXA-51-like、bla),与替加环素耐药相关的外排泵调控基因和的突变位点、的突变位点。经检测94株鲍曼不动杆菌除对多粘菌素B 100%敏感、对米诺环素敏感率25.5%外,其他抗菌药物的敏感率均低于3.5%,亚胺培南和美罗培南敏感率均只有1.1%。MLST分型得到12种ST分型,以ST195 (45株,47.9%)、ST208 (19株,20.2%) 和ST457 (10株,10.6%) 为主,eBURST分析发现其中8个ST型均属于克隆复合体92 (Clonal Complex 92,CC92);99%菌株OXA-23-like型碳青霉烯酶基因阳性;均未扩增出bla基因;外排泵调控基因和的检出率分别是73.4%和91.5%,Asp26Asn和Ala97Glu分别为和的高频突变位点;在12株鲍曼不动杆菌中检测到了基因的IS,以北部地区为主;基因均在第240位核苷酸发生缺失突变。综上所述,替加环素不敏感鲍曼不动杆菌对除多粘菌素B外的大多数抗菌药物具有很高的耐药性,AdeABC外排泵上游的双组分调控系统和的缺失和突变,缺失突变是导致鲍曼不动杆菌对替加环素敏感性降低的主要原因。
鲍曼不动杆菌,替加环素,多位点序列分型,
鲍曼不动杆菌是一种非发酵、严格需氧革兰阴性杆菌,广泛分布于自然界和医院环境,是我国医院内感染重要的病原菌之一[1],主要引起肺炎、脑膜炎、尿路感染、皮肤软组织感染及菌血症等。随着抗生素的广泛使用,鲍曼不动杆菌引起的医院感染不断增加,尤其是多重耐药鲍曼不动杆菌。中国细菌耐药监测研究2015–2016年革兰阴性菌监测报告显示,鲍曼不动杆菌对多数被测药物耐药率>70%以上,耐药率低于10%的药物依次为多粘菌素E、替加环素和磷霉素,但磷霉素的中介率达84%,替加环素对亚胺培南不敏感鲍曼不动杆菌的敏感率为82.9%[2]。替加环素是米诺环素的衍生物,一种新型甘胺酰环类抗生素,具有广谱抗菌活性,对革兰阳性菌、革兰阴性菌、需氧菌、厌氧菌均具有很好的抗菌活性[3-4],它是治疗多重耐药鲍曼不动杆菌的少数可选药物之一。然而,随着替加环素在临床抗感染治疗中的频繁使用,自2007年第一例替加环素耐药病例报告以来,替加环素耐药菌株不断被分离[5-8],给临床治疗带来了极大困难。目前替加环素耐药机制主要有主动外排泵介导、修饰酶介导、药物作用靶点介导及细胞膜渗透性介导[9]。本研究主要对替加环素不敏感鲍曼不动杆菌进行分子流行病学分析,并探讨其耐药机制,从而指导抗菌药物的合理应用,以避免替加环素耐药鲍曼不动杆菌的暴发流行。
1 材料与方法
1.1 菌株来源
收集全国12个城市20家医院在2013年中国医院内感染的抗菌药物耐药监测项目(Chinese Antimicrobial Resistance Surveillance of Nosocomial Infections of 2013,2013CARES) 和2014年、2016年中国美罗培南监测研究项目(The Chinese Meropenem Surveillance Study of 2014 and 2016,2014 CMSS、2016 CMSS) 非重复性替加环素不敏感鲍曼不动杆菌94株,其中2013年CARES项目30株 (31.9%)、2014年CMSS项目45株(47.9%) 和2016年CMSS项目19株(20.2%)。所有菌株经MALDI-TOF质谱和OXA-51-like测序鉴定,确定为鲍曼不动杆菌。药物敏感性试验的质控菌株为大肠埃希菌ATCC25922。
1.2 方法
1.2.1 药敏试验
采用微量肉汤稀释法测定替加环素对鲍曼不动杆菌的最低抑菌浓度 (Minimum inhibitory concentration,MIC)。MIC结果参照美国食品药品管理局 (Food and Drug Administration,FDA) 判读标准判读 (敏感:MIC<4 μg/mL,中介:MIC=4 μg/mL,耐药:MIC>4 μg/mL)。采用琼脂稀释法测定菌株对其他药物头孢哌酮/舒巴坦(1∶1)、哌拉西林/他唑巴坦、头孢他啶、头孢吡肟、亚胺培南、美罗培南、阿米卡星、环丙沙星、左氧氟沙星、多粘菌素B、米诺环素的MIC,并根据美国临床和实验室标准化协会 (CLSI) 2016判断标准进行药物敏感性判断[10],其中头孢哌酮/舒巴坦按照头孢哌酮对肠杆菌科折点判定 (敏感≤16 μg/mL;耐药≥64 μg/mL)。采用Whonet 5.6进行耐药率的分析。
1.2.2 提取DNA
采用煮沸法提取菌株DNA。无菌管内加入200 μL无菌水,从血平板上刮取适量新鲜培养的菌落进行稀释,100 ℃水浴10 min,然后12 000 r/min离心10 min,取上清液于另一干净无菌的管中,即为所需DNA,–20 ℃保存。
1.2.3 多位点序列分型 (Multilocussequence typing,MLST)
PCR扩增鲍曼不动杆菌MLST的7个管家基因和[11]。将PCR产物测序结果在鲍曼不动杆菌数据库(https://pubmlst.org/abaumannii/) 进行比对。使用软件 (http://eburst.mlst.net/) 对获得的ST型数据进行进化关系分析,构建进化关系图谱。
1.2.4 碳青霉烯酶与金属酶基因检测
根据文献中报道的OXA碳青霉烯酶和NDM的引物序列[12],利用PCR扩增鲍曼不动杆菌碳青霉烯酶OXA-40-like、OXA-58-like、OXA-23-like、OXA-51-like与金属酶基因NDM-1。
1.2.5 替加环素耐药相关基因检测
根据文献中报道的、和的引物序列[13-14],利用PCR扩增替加环素耐药相关基因,并对PCR产物进行测序。IS-的上、下游引物序列分别为:5′-ATGAAAAGTAAGTTA GGAATTAGTAAG-3′和5′-CTCTGTACACGACA AATTTC-3′。
2 结果与分析
2.1 菌株基本特征
94株非重复替加环素不敏感鲍曼不动杆菌来源于2013、2014和2016年,不同年份替加环素不敏感鲍曼不动杆菌 (TNSAba) 地区分布见表1。
94株鲍曼不动杆菌的来源的标本类型以血液 (43株,45.7%) 和痰液 (30株,31.9%) 为主,还包括腹水 (4株,4.3%)、导管 (3株,3.2%) 和脑脊液 (3株,3.2%),其他标本类型 (组织、尿液、胸水等) 共11株,占11.7%。分离的菌株大部分来源于老年男性患者。男性59株,占62.8%;女性35株,占37.2%。按年龄,≥66岁有46株,占49%;18–65岁有46株,占49%;≤17岁只有2株,占2%。
2.2 药敏试验结果
94株鲍曼不动杆菌除对多粘菌素B均敏感、对米诺环素25.5%敏感率外,对其他检测药物的敏感率均低于3.5%。94株鲍曼不动杆菌不同时间药物敏感性结果见表2。
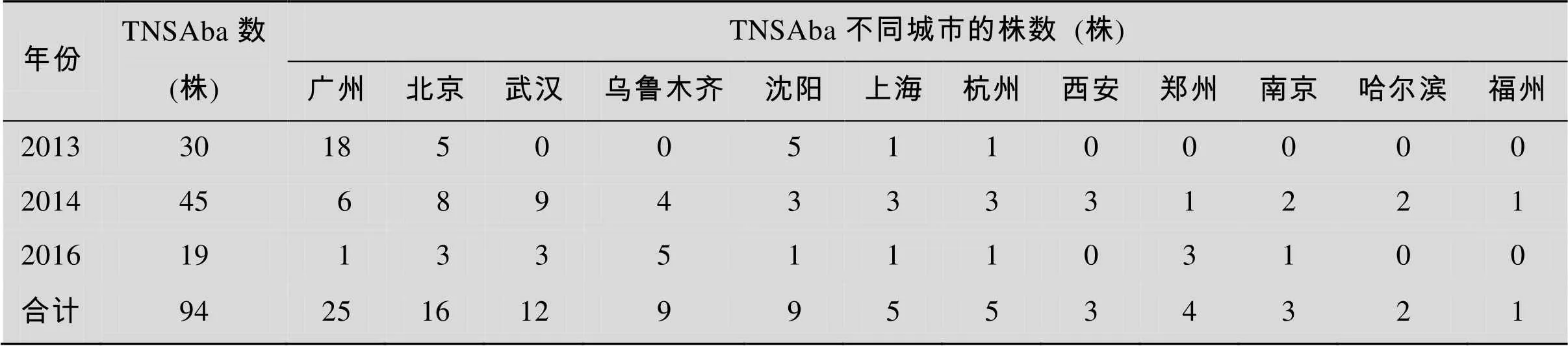
表1 94株替加环素不敏感鲍曼不动杆菌地区分布
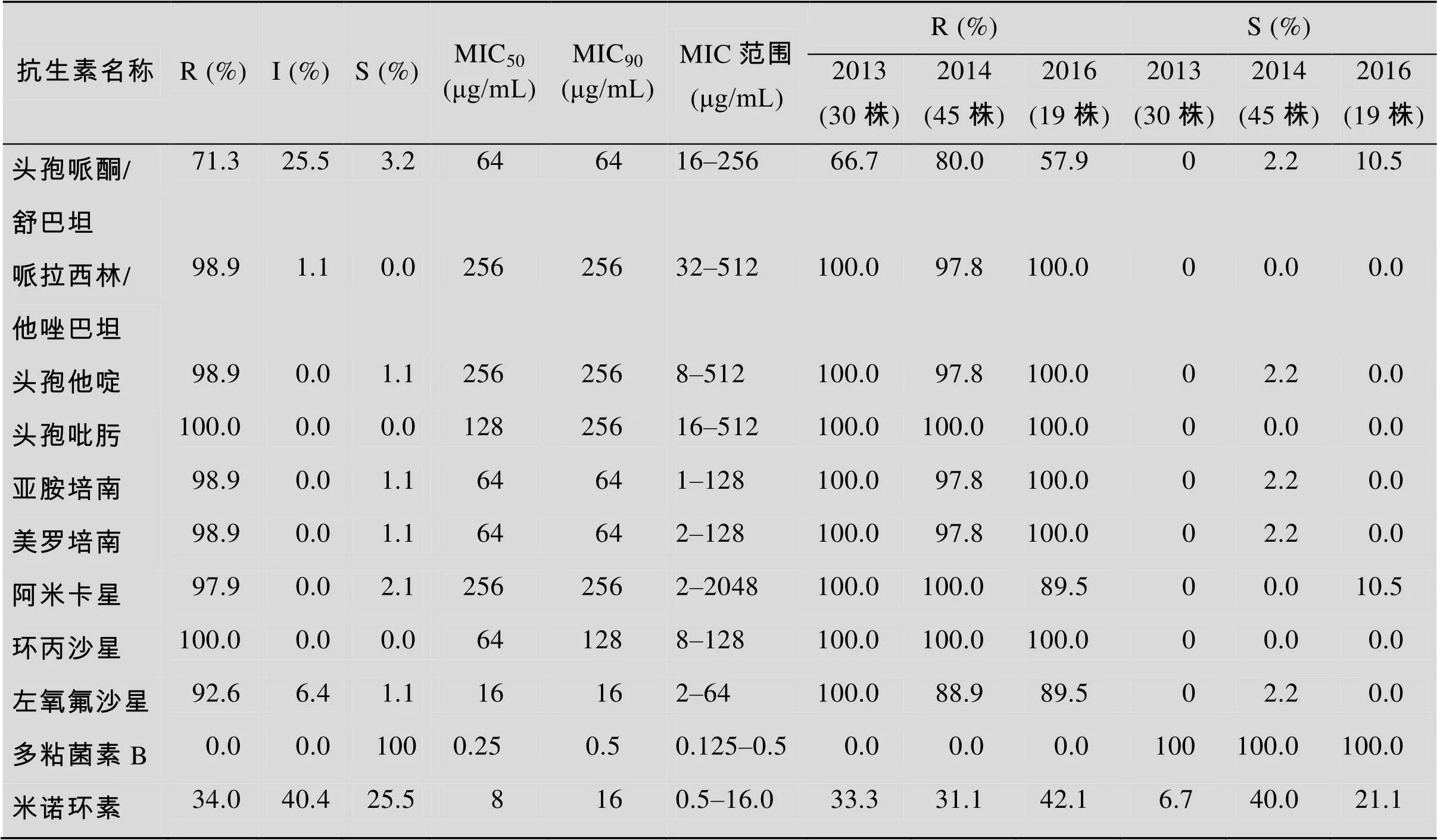
表2 94株替加环素不敏感鲍曼不动杆菌药物敏感性
注:头孢哌酮/舒巴坦按照头孢哌酮对肠杆菌科折点判定 (敏感:≤16 μg/mL;耐药;64 ≥μg/mL);R:表示耐药,S:表示敏感,I表示中介。
2.3 MLST分型结果
MLST分析得到12种ST分型,以ST195和ST208为主,分别占47.9%和20.2%,为我国替加环素不敏感鲍曼不动杆菌的流行克隆,并且分布范围最广,在所有地域均有分布。其次为ST457 (3个地区,10.6%)、ST540 (2个地区,5.3%)、ST369 (3个地区,4.3%) 和ST191 (2个地区,3.1%),见表3。eBURST分析发现其中8个ST型均属于克隆复合体92 (Clonal Complex 92,CC92),这些ST型之间仅相差一个管家基因(),具有较近的亲缘关系。
2.4 碳青霉烯酶与金属酶基因检测结果
94株替加环素不敏感鲍曼不动杆菌中,94株检出OXA-51-like基因 (100%),93株检出OXA-23-like基因 (99%),未检出NDM-1、OXA-40-like和OXA-58-like基因。仅1株AB90未携带OXA-23-like基因,MLST为ST368型,对美罗培南和亚胺培南的MIC分别是1、2 μg/mL。
2.5 替加环素耐药相关基因结果
2.5.1 AdeABC外排泵上游的双组分调控系统和
检出有69株,占73.4%;有86株,占91.5%。同时检出和的菌株有69株。与敏感鲍曼不动杆菌克隆株BM4454比对后,AdeR的氨基酸突变位点有Asp26Asn (5株)、Met197Thr (1株)和His210Tyr (1株),AdeS的氨基酸突变位点有Thr66Ser (1株)、Ala97Glu (10株)、Ile100Asn (2株)、Ala107Ser (2株)、Arg108Ile (1株)、Ser136Phe (1株) 和Phe177Ile (1株) (表4)。
2.5.2 Is检测
双组分调节系统中上游存在Is有12株,占12.8%,分别来自沈阳 (6株)、北京 (3株)、乌鲁木齐 (1株)、南京 (1株) 和广州 (1株)。这12株鲍曼不动杆菌对替加环素的MIC均为4 μg/mL,MLST分型 ST208占11株,1株ST369。

表3 94株替加环素不敏感鲍曼不动杆菌ST型及地区分布
注:华北 (北京)、华东 (上海、杭州、南京)、东北 (沈阳、哈尔滨)、西北 (西安、乌鲁木齐)、华南 (广州、福州)、华中 (武汉、郑州)

表4 AdeR和AdeS的氨基酸突变位点
2.5.3基因检测
所有菌株均存在缺失突变,都在第240位核苷酸缺失导致该基因编码蛋白提前终止,未发现其他突变。
3 讨论
非发酵革兰阴性菌多重耐药已成为世界关注的问题,2017年2月27日世界卫生组织 (World Health Organization,WHO) 发表了首份抗生素耐药“重点病原体”清单,其中碳青霉烯耐药鲍曼不动杆菌 (Carbapenem-resistant,CRAB) 被列为“极为重要”的新型抗生素研发重点病原体。它可引起呼吸机相关性肺炎及创口、血流、泌尿系、中枢神经系统等感染,在长期收住于ICU的免疫力较低及持续使用抗生素的患者中感染率尤其高[15]。由于鲍曼不动杆菌有多种内在和获得性耐药机制,易导致其多重耐药、广泛耐药甚至泛耐药表型的发展,给临床治疗带来巨大困难[16-17]。替加环素具有抗菌谱广、抗菌活性强等特点,曾被认为是治疗多重耐药鲍曼不动杆菌的最后选择。然而,由鲍曼不动杆菌引起的感染在接受替加环素治疗过程中由敏感变为耐药在不断地被报道[18-19]。
本研究分析了2013CARES、2014CMSS和2016CMSS项目中12个城市20家医院分离的非重复替加环素不敏感鲍曼不动杆菌。94株替加环素不敏感鲍曼不动杆菌对临床常用的第三代、第四代头孢菌素、氨基糖苷类抗生素、氟喹诺酮类和碳青霉烯类等抗生素均具有很高的耐药率。MLST分型得到12种ST分型,以ST195和ST208为主,分别占47.9%和20.2%。经eBRUST软件计算ST195是Founder,ST208 (1-3-3-2-2-97-3)、ST191 (1-3-3-2-2-94-3)、ST368 (1-3-3-2-2-140-3)、ST540 (1-3-3-2-2-160-3)、ST784 (1-3-3-2-2-107-3)、ST136 (1-3-3-2-2-16-3) 和ST369 (1-3-3-2-2-106-3) 是ST195的单个位点变异型(Single-locus variant,SLV),仅管家基因与ST195不同;S445 (1-81-3-2-2-16-3)、ST457 (1-15-3-2-2-157-3)、ST469 (1-12-3-2-2-103-3) 是ST195的双位点变异型(Double-locus variant,DLU),在管家基因和上存在不同。ST195和ST208都属于CC92克隆复合体,CC92是多重耐药鲍曼不动杆菌最主要的克隆复合体,在全球广泛流行,最早在荷兰发现,此后在韩国、泰国、日本、意大利等超过50个国家分离到,在我国多个地区和不同医院已分离到[20-23]。
鲍曼不动杆菌的耐药机制较复杂,与替加环素耐药相关的耐药结节细胞分化(Resistance-nodulatian- cell division family,RND) 系统外排泵主要包括AdeABC、AdeFGH和AdeIJK[24]。目前研究认为鲍曼不动杆菌对替加环素产生耐药性与外排泵AdeABC表达活性过度增加有关[18,25-26]。作为一个双组分调控系统,操纵子位于基因上游,包含基因和,分别编码感应激酶AdeS和反应调节蛋白AdeR[27]。本研究数据同时存在和有69株,占73.4%,有86株,占91.5%,和与替加环素耐药密切相关。值得一提的是,在12株鲍曼不动杆菌中检测到了基因的IS,以北部地区为主,上游中的IS引入了新的启动子,可能增强下游AdeABC持续高表达,导致替加环素耐药。后续我们将对IS的作用和机制进行深入研究。
替加环素耐药相关的甲基化转移酶基因(Tigecycline-related-methyltransferase gene,)编码S-腺苷酸依赖的甲基化转移酶。在细菌中,甲基化转移酶主要参与保护基因组不受外来DNA破坏,同时在抗生素耐药中也发挥重要作用[28]。本研究所有替加环素不敏感鲍曼不动杆菌的均存在缺失突变,与Trebosc等[29]报道结果一致。陈琼等利用基因回补功能验证试验证实,基因突变导致替加环素敏感性降低,野生型则能回复替加环素的敏感性,基因作为一种新的耐药机制在鲍曼不动杆菌的替加环素耐药过程中发挥重要作用[14]。
综上所述,替加环素不敏感的鲍曼不动杆菌有很高的亲源性且对大多数抗生素的耐药率极高,RND外排泵在替加环素耐药过程中发挥重要作用,IS在替加环素耐药中的机制有待进一步研究。
[1] Wang H, Ren JK, Wang MG. Clinical Microbiological Examination. Beijing: People’s Medical Publishing House, 2015: 417 (in Chinese). 王辉, 任健康, 王明贵. 临床微生物学检验. 北京: 人民卫生出版社, 2015: 417.
[2] Li Y, Lv Y, Deng B, et al. Antimicrobial susceptibility of Gram-negative organisms: results from China antimicrobial resistance surveillance trial program, 2015–2016. Chin J Clini Pharmacol, 2017, 33(23): 2521–2542 (in Chinese). 李耘, 吕媛, 郑波, 等. 中国细菌耐药监测研究2015–2016革兰氏阴性菌监测报告. 中国临床药理学杂志, 2017, 33(23): 2521–2542.
[3] Gordon NC, Wareham DW. A review of clinical and microbiological outcomes following treatment of infections involving multidrug-resistantwith tigecycline. J Antimicrob Chemother, 2009, 63(4): 775–780.
[4] Stein GE, Babinchak T. Tigecycline: an update. Diagn Microbiol Infect Dis, 2013, 75(4): 331–336.
[5] Keeney D, Ruzin A, McAleese F, et al. MarA-mediated overexpression of the AcrAB efflux pump results in decreased susceptibility to tigecycline in. J Antimicrob Chemother, 2008, 61(1): 46–53.
[6] Sun Y, Cai Y, Liu X, et al. The emergence of clinical resistance to tigecycline. Int J Antimicrob Agents, 2013, 41(2): 110–116.
[7] Chen YH, Lu PL, Huang CH, et al. Trends in the susceptibility of clinically important resistant bacteria to tigecycline: results from the tigecyclinesurveillance in Taiwan study, 2006 to 2010. Antimicrob Agents Chemother, 2012, 56(3): 1452–1457.
[8] Hawser SP, Bouchillon SK, Hackel M, et al. Trending 7 years ofactivity of tigecycline and comparators against Gram-positive and Gram-negative pathogens from the Asia-Pacific region: Tigecycline Evaluation Surveillance Trial (TEST) 2004–2010. Int J Antimicrob Agents, 2012, 39(6): 490–495.
[9] Chen HJ, Li CR. Research updates on the mechanisms of tigecycline resistance in. Chin JInfecChemother, 2017, 17(3): 336–340 (in Chinese). 陈浩俊, 李从荣. 鲍曼不动杆菌对替加环素耐药机制的研究进展. 中国感染与化疗杂志, 2017, 17(3): 336–340.
[10] Clinical and Laboratory Standards Institute. M100S26 Performace standards for antimicrobial susceptibility testing: twenty-sixth edition. Wayne. PA: CLSI. 2016.
[11] Lee HY, HuangCW, Chen CL, et al. Emergence in Taiwan of novel imipenem-resistantST455 causing bloodstream infection in critical patients. J Microbiol, Immunol Infect, 2015, 48(6): 588–596.
[12] Woodford N, Ellington MJ, Coelho JM, et al. Multiplex PCR for genes encoding prevalent OXA carbapenemases inspp. Int J Antimicrob Agents, 2006, 27(4): 351–353.
[13] Peleg A, Adams J, Paterson DL. Tigecycline efflux as a mechanism for nonsusceptibility in. Antimicrob Agents Chemother, 2007, 51(6): 2065–2069.
[14] Chen Q, Li X, Zhou H, et al. Decreased susceptibility to tigecycline inmediated by a mutation inencoding SAM-dependent methyltransferase. J Antimicrob Chemother, 2014, 69(1): 72–76.
[15] Consales G, Gramigni E, Zamidei L, et al. A multidrug-resistantoutbreak in intensive care unit: antimicrobial and organizational strategies. J Crit Care, 2011, 26(5): 453–459.
[16] Visca P, Seifert H, Towner KJ.infection–an emerging threat to human health. IUBMB Life, 2011, 63(12): 1048–1054.
[17] Paterson DL, Harris PNA. Editorial Commentary: The newequation: hypervirulence plus antibiotic resistance equals big trouble. Clin Infect Dis, 2015, 61(2): 155–156.
[18] Hornsey M, Ellington MJ, Doumith M, et al. AdeABC-mediated efflux and tigecycline MICs for epidemic clones of. J Antimicrob Chemother, 2010, 65(8): 1589–1593.
[19] Dhabaan GN, AbuBakar S, Shorman MA, et al.activity of tigecycline againstisolates from a teaching hospital in Malaysia. J Chemother, 2012, 24(2): 87–92.
[20] Fu YQ. The study of multilocus sequence typing and the carbapenem resistance mechanism offrom partial regions of China[D]. Hangzhou: Zhejiang University, 2012 (in Chinese).符一琪. 中国部分地区鲍曼不动杆菌多位点序列分型及碳青霉烯类抗生素耐药机制研究[D]. 杭州: 浙江大学, 2012.
[21] Zhong Q, Xu WD, Wu YJ, et al. Clonal spread of carbapenem non-susceptiblein an intensive care unit in a teaching hospital in China. Ann Lab Med, 2012, 32(6): 413–419.
[22] Huang L, Sun LY, Yan Y. Clonal spread of carbapenem resistantST92 in a Chinese hospital during a 6-year period. J Microbiol, 2013, 51(1): 113–117.
[23] Zhou JJ. Molecular epidemiology and drug resistance mechanism of multidrug-resistant[D]. Shanghai: Fudan University, 2014 (in Chinese).周娇娇. 多重耐药鲍曼不动杆菌分子流行病学和耐药机制研究[D]. 上海: 复旦大学, 2014.
[24] Li HN, Wang XJ, Zhang YW, et al. The role of RND effux pump and global regulators in tigecycline resistance in clinicalisolates. Fut Microbiol, 2015, 10(3): 337–346.
[25] Deng M, Zhu MH, Li JJ, et al. Molecular epidemiology and mechanisms of tigecycline resistance in clinical isolates offrom a Chinese university hospital. Antimicrob Agents Chemother, 2014, 58(1): 297–303.
[26] Krishnamoorthy S, Shah BP, Lee HH, et al. Microbicides alter the expression and function of RND-type efflux pump AdeABC in biofilm-associated cells ofclinical isolates. Antimicrob Agents Chemother, 2015, 60(1): 57–63.
[27] Yoon EJ, Courvalin P, Grillot-Courvalin C. RND-type efflux pumps in multidrug-resistant clinical isolates of: major role for AdeABC overexpression and AdeRS mutations. Antimicrob Agents Chemother, 2013, 57(7): 2989–2995.
[28] Kumar R, Rao DN. Role of DNA methyltransferases in epigenetic regulation in bacteria//Kundu T, Eds. Epigenetics: Development and Disease. Subcellular Biochemistry, vol 61.Dordrecht: Springer 2013, 61: 81–102.
[29] Trebosc V, Gartenmann S, Royet K, et al. A novel genome-editing platform for drug-resistantreveals an AdeR-unrelated tigecycline resistance mechanism. Antimicrob Agents Chemother, 2016, 60(12): 7263–7271.
(本文责编 郝丽芳)
Molecular epidemiology and resistance mechanisms of tigecycline-non-susceptible
Xiujuan He, Henan Li, Qi Wang, Chunjiang Zhao, Shuguang Li, Xiaojuan Wang, Yawei Zhang, Ruobing Wang, Yuyao Yin, Longyang Jin, Feifei Zhang, and Hui Wang
Department of Clinical Laboratory,Peking University People’s Hospital,Beijing 100044, China
To investigate the resistance mechanisms of tigecycline-non-susceptibleand for providing the evidence of the control of nosocomial infection and rational use of antibiotics. The minimum inhibitory concentrations (MICs) of 94 non repetitive tigecycline-non-susceptible.from 20 hospitals in 12 cities of China were determined by agar dilution method and broth microdilution method. The molecular epidemiology was studied by Multilocus sequence typing (MLST) and eBURST software. PCR and sequencing techniques were used to analyze the resistance genes (OXA-40-like,OXA-58-like,OXA-23-like,OXA-51-like,NDM-1), IS, and the mutation sites of,, and. The activity of polymyxin B and minocyclinem against tigecycline-non-susceptible.were 100% and 25.5%, respectively. The sensitivities of other antibiotics were less than 3.5%, and the sensitivities of imipenem and meropenem totigecycline-non- susceptible.were only 1.1%. A total of 12 ST types were identified, including ST195 (45, 47.9%), ST208 (19, 20.2%) and ST457 (10, 10.6%). EBURST analysis found that 8 of the ST types belonged to the clone complex 92 (Clonal Complex 92, CC92). TheOXA-23-liketype carbapenem gene was identiefied in 93 strains (99% positive); and none of the strains contained theNDM-1gene. The detection rates ofandwere 73.4% and 91.5% respectively and high frequency mutation sites were located in(Asp26Asn) and(Ala97Glu); The ISlocated upstream of thegene was detected in 12 strains of., mainly from the northern region of China. The 240 nucleotide deletion of thegene caused a frameshift leading to a premature stop. So the tigecycline-non-susceptible.showed high resistance against most antibiotics except polymyxin B. The deletion and mutation of,andwere the main resistant mechanisms in tigecycline-non-susceptible.in China.
, tigecycline, multilocus sequence typing,
May 9, 2018;
July 10, 2018
National Natural Science Foundation of China (No. 31170125), Natural Science Foundation of Beijing, China (No. 7153177).
Hui Wang. Tel/Fax: +86-10-88326300; E-mail: whuibj@163.com
国家自然科学基金 (No. 31170125),北京市自然科学基金 (No. 7153177)资助。
2018-08-15
http://kns.cnki.net/kcms/detail/11.1998.q.20180814.1328.002.html
10.13345/j.cjb.180196
