应用多重实时荧光PCR方法对8种动物毛纤维种类鉴别的研究
2018-08-07刘艳艳范阳阳东野升金谭晴晴张全芳
刘艳艳,范阳阳,步 迅,胡 悦,东野升金,谭晴晴,张全芳
(山东省农业科学院生物技术研究中心/山东省农业科学院应用生命科学实验室,济南 250100)
动物毛绒纤维是畜牧业的重要畜产品和纺织工业的高档纺织原料,具有弹性好,吸湿性好,保暖性好,光泽柔和等优点[1]。由于各种纤维纺织性能、市场价格差异较大,市场上许多动物毛绒制品出现了掺假现象[2],动物毛纤维快速鉴别标准的建立已成为消费者和执法者关注的焦点。经调查,动物毛绒纤维掺假:向狐狸、貉子等高端昂贵的动物毛纤维中掺入大量羊毛、兔毛等价格较低的动物纤维,或者是利用合成的纤维来冒充[3]。由于在原料和产品的监管上缺乏统一的检测标准,消费者难辨真伪,对大众造成很大的经济损失,也严重影响了生产厂家的信誉,扰乱了市场秩序。因此如何快速、准确地鉴定貂、狐狸等各种高端动物毛纤维真伪,对于维护市场的健康稳定发展、维护消费者的合法权益及监督管理技术保障等方面都具有重要的意义。
对于动物毛纤维源性成分的鉴定主要是通过感官和扫描电镜法等传统手段和观察微观结构来鉴别,如李维红和吴建平[4]利用扫描电镜检测野生杂皮动物毛皮种类,郭海涛等[5]利用红外光谱对毛发的分析。从1988年开始,德国和英国的生物学专家就证实了可以从动物毛纤维中提取DNA[6-7]。随后渐渐出现了从动物毛纤维中提取DNA,并进行PCR扩增来鉴定动物毛的方法[8-9],已有的动物毛纤维检测鉴定标准有羊毛(羊绒)源性成分鉴定[10-11],Tang等[12]采用Real-time PCR技术,设计2套针对羊绒和羊毛的探针和引物对。2004年,印度科学家Subramanian等[13]利用PCR-RFLP技术和绵羊、山羊线粒体核酸序列的差异成功建立了羊毛与羊绒的鉴别方法。近年的检测应用基于荧光定量PCR的兔毛[14-15]、狐狸毛[15]纤维定性检测方法。
感官和扫描电镜法的主观性较强,要求有较多的从业经验,而且出错率高,已不能满足目前面临的鱼龙混杂的原料检测需求[16]。从DNA层面的检测方法多样但物种单一,而对于水貂、狐狸、羊、骆驼、牛、兔等动物毛纤维的源性成分鉴定还没有统一的检测方法。本研究采用改进的动物毛纤维DNA提取方法,根据水貂、狐狸、骆驼、兔、山羊、绵羊、鸭和鹅8种动物的特异核苷酸序列设计通用PCR引物和特异荧光探针,并建立多重实时荧光PCR检测体系,以鉴定动物毛纤维的源性成分,为市场上区分动物毛纤维的真伪提供有效的鉴别依据。
1 材料与方法
1.1 试剂与仪器
动物组织提取试剂盒(D3396)为OMEGA产品。2×Taq Man Master Mix为DBI Bioscience产品。20 mg·mL-1蛋白酶K溶液、RNA酶、二硫苏糖醇(DTT)和Chelex-100树脂购自Sigma公司,引物与探针由生工生物工程(上海)有限公司合成。超高效硅珠纯化DNA提取试剂盒购自天津诺维莱博科技有限公司。
实时荧光PCR仪(ABI-7500),电子天平(余姚记铭称重校验设备有限公司,JM-A10002),高压灭菌锅(申安,LDZX-50KBS),台式离心机(Eppendorf5810),振荡仪(杭州瑞城,TS100)。
1.2 材料
水貂皮、狐狸皮、兔皮、山羊绒、绵羊毛、骆驼绒、鹅毛和鸭毛采自山东某些毛皮动物及畜禽养殖场。
1.3 方法
1.3.1 毛纤维DNA提取方法优化 毛干部位的DNA提取:在进行动物毛纤维DNA裂解之前,首先取30 mg动物毛纤维用70%乙醇清洗1次,再用蒸馏水清洗2次,采用液氮研磨法将毛干研磨成粉末。然后称取3 mg置于2 mL的离心管中,添加400 μL的DNA细胞裂解液,10 μL蛋白酶K,10 μL二硫苏糖醇(DTT),充分混匀,放置恒温振荡仪56 ℃ 800 r·min-1裂解3~5 h。
1.3.1.1 方法1:裂解完成后,采用酚氯仿抽提法对DNA进行抽提和纯化,具体操作参考杨芳芳等[17]方法。
1.3.1.2 方法2:裂解完成后,加入100 μL 5% Chelex-100树脂,煮沸8 min,8 000 r·min-1离心3 min,取上清置于1.5 mL EP管中。采用硅珠吸附法DNA纯化试剂盒进行纯化DNA。
1.3.1.3 方法3:采用OMEGA生物公司动物组织提取试剂盒(D3396)。
1.3.1.4 毛囊部位的DNA提取:采用OMEGA生物公司动物组织提取试剂盒操作说明进行提取。
1.3.2 DNA浓度及纯度测定 将水貂、狐狸、兔、山羊、绵羊、骆驼、鹅和鸭共8种动物毛纤维的基因组DNA 用紫外分光光度法检测提取DNA浓度和纯度。测定并记录其在OD260 nm和OD280 nm吸光值,OD260 nm/OD280 nm比值为1.7~1.9,试验用DNA浓度为0.1~50.0 ng·μL-1。用于PCR反应的DNA溶液A260 nm/A280 nm比值>1.5,如<1.5则重新提取。
1.3.3 设计PCR引物序列和特异性探针序列 根据GenBank数据库中已公布的水貂、狐狸、骆驼、兔、山羊、绵羊、鸭和鹅的线粒体16SrRNA基因组序列(每个物种不同品种的线粒体16SrRNA基因序列都下载),使用Primer 5.0软件在保守区设计通用引物,使用Primer 3.0软件在可变区设计相应的特异性探针,设计的探针必须满足种间特异种内保守原则。应用TaqMan探针的荧光定量PCR反应,退火温度为60 ℃,探针的退火温度比引物的退火温度高约10 ℃。通过比较确定用于8种动物源性DNA的定性检测的通用引物组合为UNF/UNR,扩增产物长度为228~241 bp。以16SrRNA基因作为内参靶基因,设计可同时扩增哺乳类动物及禽类的通用引物及通用探针,作为PCR内参质控体系,可有效验证提取的基因组DNA质量,避免出现假阴性,引物序列及探针序列见表1。
1.3.4 优化确定PCR反应体系和条件
1.3.4.1 单重实时荧光PCR检测方法建立:建立单重PCR体系:以8种供试动物毛纤维基因组DNA为研究对象,反应体系采用20 μL,2×qPCR Master Mix 10 μL,对引物的浓度、探针浓度及退火温度进行筛选,以确定最佳的浓度和退火温度。首先将探针终浓度设定为0.20 μmol·L-1,引物设置4个加样梯度,分别为0.25、0.50、0.75、1.00 μmol·L-1;然后将引物设置为0.25 μmol·L-1,探针设置4个梯度的加样量分别为0.10、0.20、0.40、0.80 μmol·L-1,每个梯度设置3个平行样。针对每条探针进行温度梯度试验,退火温度以2 ℃的梯度递增,分别设置为58、60、62和64 ℃。每次试验采取等量的模板,根据Ct值、荧光信号和平台期等因素作为参考。
1.3.4.2 多重实时荧光PCR检测方法建立:以提取并用紫外分光光度计检测好的DNA为模板,根据上述单重荧光PCR优化好的引物、探针浓度和退火温度进行组合,按照优化好的反应条件进行多重荧光PCR反应。采用PCR体系A(包括水貂、狐狸和兔的混合探针)、PCR体系B(包括骆驼、山羊和绵羊的混合探针)和PCR体系C(包括鸭和鹅的混合探针)同时进行特异检测。实时荧光PCR反应条件随仪器不同略有改变,如使用ABI-7500荧光定量PCR仪程序:95 ℃ 3 min; 95 ℃ 10 s, 62 ℃ 35 s(收集荧光信号),40个循环。
1.3.5 方法验证
1.3.5.1 DNA特异性试验:用建立好的多重PCR体系对水貂、狐狸、兔、骆驼、山羊、绵羊、牛、猪、马、貉子、狗、驴、鸡、鸭、鹅、鱼和玉米共17种物种的基因组DNA进行特异性验证。用NanoDrop2000紫外分光光度计检测浓度,统一稀释到10 ng·μL-1左右,每个样本做3次平行样。
1.3.5.2 DNA灵敏度试验:将水貂、狐狸、兔、山羊、绵羊、骆驼、鹅、鸭共8种动物毛纤维的基因组DNA模板浓度定为0.200 0、0.020 0、0.002 0和0.000 2 ng·μL-1,设立3个平行样,每个反应取5 μL模板量,即DNA用量分别为1.000、0.100、0.010、0.001 ng,进行多重PCR检测、评价DNA检测灵敏度。
1.3.5.3 方法检出限的确立:根据各国的标识制度确定实时荧光PCR检测方法的检出限。根据阈值范围设定5.0%、1.0%、0.5%和0.1% 4个浓度(质量比)作为实时荧光PCR法的检出限的评估标准。以鸡或鱼的DNA样本为背景基质对目标样本进行掺杂,分别制备5.0%、1.0%、0.5%和0.1% 4个浓度(质量分数)的各标准样品,每种标准品重复测试3次。为了进一步确认实时荧光PCR法的DNA检测灵敏度,在0.10%和0.01%的评估标准下,每个样品重复10次。
表18种动物毛纤维源性实时荧光PCR定性检测所用引物和探针序列
Table1Primerandprobesequencesforqualitativedetectionof8animalhairfiber-derivedreal-timefluorescencePCR

引物和探针名称Primer and probe name缩写Abbreviation序列(5'→3')Sequence扩增片段/bpAmplified fragment上游通用引物Universal primer-forwardUNFAAGACGAGAAGACCCTTGGACTTTA228~241下游通用引物Universal primer-reverseUNRGATTGCGCTGTTATCCCTAGGGTA上游内参引物 Internal reference primer-forward16SFCCTAGCGATAACAGGGCAATC125下游内参引物Internal reference primer-reverse16SRTTAAGCCTTGAACAAACGAACC内参探针Internal reference probe16SP5'CY5-ACGACCTCGATGTTGGATCAGGAC-3' BHQ2水貂探针Mink probeSD-P5'ROX-TCTAACACAACATTATTACTGGGT-3'BHQ2狐狸探针Fox probeHL-P5'FAM-CAAACCCCTCCGGGAATAACTTACT-3'DAB兔探针Rabbit probeTU-P5'JOE-TACAATGAGCCTAACCAAGGAAATCCC-3'BHQ2骆驼探针Camel probeLT-P5'FAM-ATAACCGCCAAGGGATAATAATCTTCTACC-3' DAB山羊探针Goat probeSY-P5'ROX-TATGGACTAGCAGTTTTGGTTGGGG-3'DAB绵羊探针Sheep probeMY-P5'JOE-CCACCAAGGGATAACAACACTCTGGTGG-3' BHQ2鸭探针Duck probeYA-P5'JOE-CGGGGCTACAGACATCGCAGAG-3' BHQ2鹅探针Goose probeE-P5'FAM-ACTGGGGCCACTACCATCGC-3' DAB
1.3.6 模拟混合样本检测 狐狸毛纤维中分别掺入0.5%、1.0%、5.0%、10.0%、20.0%、50.0%兔毛纤维,提取混合样本基因组DNA,进行多重实时荧光PCR检测;骆驼毛纤维中分别掺入0.5%、1.0%、5.0%、10.0%、20.0%、50.0%绵羊毛纤维,提取混合样本基因组DNA,进行多重实时荧光PCR检测;鸭毛纤维中分别掺入0.5%、1.0%、5.0%、10.0%、20.0%、50.0%鹅毛纤维,提取混合样本基因组DNA,进行多重实时荧光PCR检测。
2 结 果
2.1 DNA提取方法比较
将3种方法提取的8种动物毛纤维基因组DNA利用紫外分光光度计检测其核酸的浓度和纯度(表2),结果表明:方法1对于只有毛干的骆驼毛提取效果较差,检测不到浓度。方法3虽然能将8个 动物毛纤维DNA提取出来,但是浓度高低差别较大。优化的方法2提取的DNA浓度在10~20 ng·μL-1,纯度优于方法1和3。另外,以水貂源性和兔源性为例,通过实时荧光PCR扩增比较其扩增曲线发现,本实验室优化的方法2(用硅珠纯化提取动物毛纤维DNA)效果最佳,见图1和图2。
2.2 实时荧光定量PCR引物的选择
分别以提取水貂、狐狸、骆驼、兔、山羊、绵羊、鸭和鹅的基因组DNA为模板,以表1中的通用引物,用染料法进行PCR扩增,其结果扩增曲线见图3A。8种物种的扩增曲线均正常,说明通用引物扩增良好;8种物种的熔解曲线均为单一峰值(图3B),说明通用引物特异性强。同时还利用DNA测序进行验证,其所得序列在NCBI数据库中比对均与目标物种一致。
表28种动物基因组DNA纯度及浓度
Table2Purityandconcentrationof8animalsgenomicDNA

样品Sample方法1 Method 1方法2 Method 2方法3 Method 3A260 nm/A280 nm浓度/(ng·μL-1)ConcentrationA260 nm/A280 nm浓度/(ng·μL-1)ConcentrationA260 nm/A280 nm浓度/(ng·μL-1)Concentration水貂Mink1.199.181.9616.491.225.71狐狸Fox2.261.661.9416.551.439.94兔Rabbit1.951.501.9517.211.636.63骆驼Camel0.000.001.8211.671.168.85山羊Goat1.534.361.8125.751.1312.99绵羊Sheep1.238.031.8015.871.367.70鸭Duck3.503.292.0520.441.337.93鹅Goose3.002.862.0514.421.2219.79

FAM.狐狸源性探针;JOE.兔源性探针;ROX.水貂源性探针;CY5.内参探针。下同FAM.The fox-derived probe;JOE. The rabbit-derived probe;ROX. Mink-derived probe;CY5. Internal reference probe.The same as below图1 不同方法提取的水貂源性DNA的实时荧光PCR扩增曲线Fig.1 Real-time fluorescence PCR amplification curve of mink-derived DNA extracted by different methods

图2 不同方法提取的兔源性DNA的实时荧光PCR扩增曲线Fig.2 Real-time fluorescence PCR amplification curve of rabbit-derived DNA extracted by different methods
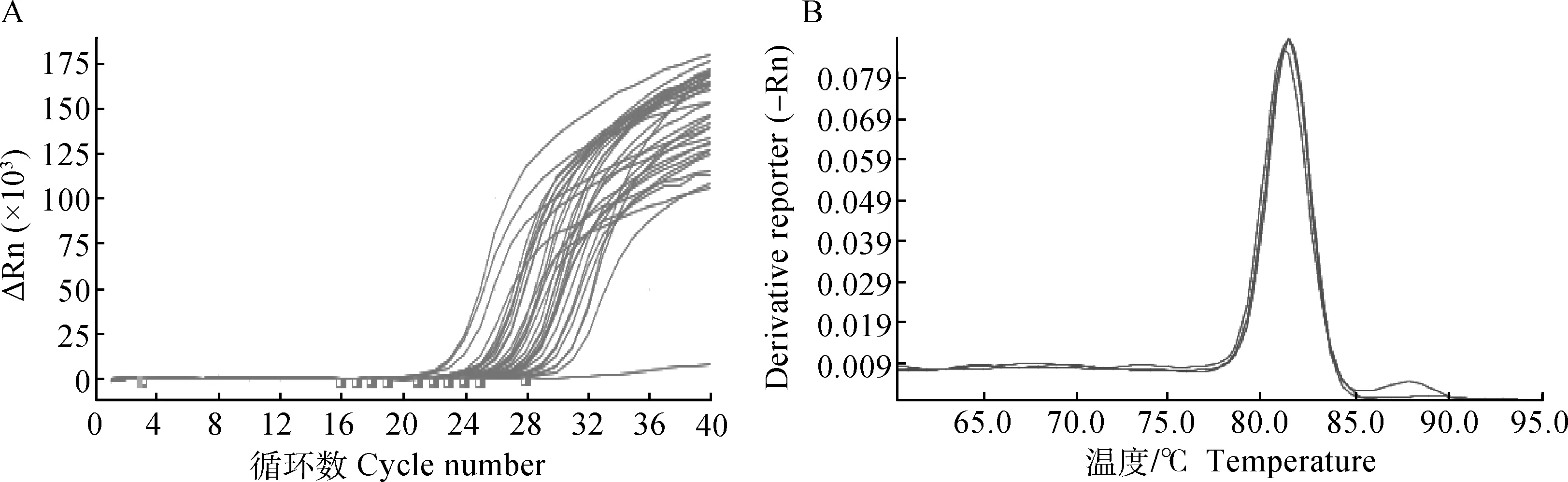
图3 8种物种的扩增曲线(A)和熔解曲线(B)Fig.3 The amplification curves (A) and melting curves (B) of 8 species
2.3 优化确定PCR反应体系和条件
引物为0.75、1.00 μmol·L-1时扩增曲线信号明显高于0.25、0.50 μmol·L-1,探针扩增曲线信号随着浓度的增加逐渐增强,同时引物及探针浓度会影响扩增效率,初步确定引物浓度0.50 μmol·L-1,根据探针信号强度差异优化浓度,最终构建3组多重PCR体系鉴定8种毛纤维动物源性,20 μL PCR反应体系:2×TaqManMaster Mix 10 μL,10× Primer Mix 2 μL,10× Probe Mix 2 μL,DNA模板 2 μL(浓度为0.2~100.0 ng·μL-1),ddH2O 4 μL,3组多重PCR体系探针及引物终浓度为0.1~0.5 μmol·L-1。最佳反应条件:95 ℃ 3 min; 95 ℃ 10 s, 62 ℃ 35 s(收集荧光信号),40个循环。
2.4 多重PCR体系方法验证
2.4.1 特异性验证 用建立好的多重PCR体系对水貂、狐狸、兔、骆驼、山羊、绵羊、牛、猪、马、貉子、
狗、驴、鸡、鸭、鹅、鱼和玉米共17种物种的基因组DNA进行特异性验证。
2.4.1.1 多重PCR体系组合A(包括水貂、狐狸和兔混合探针),只对应的对水貂、狐狸和兔DNA为模板时才有荧光信号,且Ct值<35,其他样品均没有荧光信号,表明体系A具有良好的扩增特异性(图4A)。
2.4.1.2 多重PCR体系组合B(包括骆驼、山羊和绵羊混合探针),只对应的对骆驼、山羊和绵羊DNA为模板时才有荧光信号且Ct值<35,其他样品均没有荧光信号,表明体系B具有良好的扩增特异性(图4B)。
2.4.1.3 多重PCR体系组合C(包括鸭、鹅混合探针),只对应的对鸭、鹅DNA为模板时才有荧光信号,且Ct值<35,其他样品均没有荧光信号,表明体系C具有良好的扩增特异性(图4C)。

图4 3组(A、B和C)多重PCR体系的扩增曲线Fig.4 Amplification curves of multiplex PCR system in 3 groups (A, B and C)
2.4.2 灵敏度检测 将水貂、狐狸、兔、山羊、绵羊、骆驼、鹅和鸭共8种动物毛纤维的基因组DNA模板浓度定为0.200 0、0.020 0、0.002 0和0.000 2 ng·μL-1,设立3个平行样,每个反应取5 μL模板量,即DNA用量分别为1.000、0.100、0.010、0.001 ng。qPCR反应结果见图5,当基因组DNA模板含量≥0.010 ng时均有扩增曲线、Ct值<35,当基因组DNA模板含量为0.001 ng时无扩增曲线,所以DNA灵敏度为0.010 ng。
2.4.3 模拟混合样本检测 模拟混合样本的检测结果见表3~5,表3表明,狐狸毛纤维中分别掺入1.0%、5.0%、10.0%、20.0%、50.0%的毛纤维时兔毛均能被检出,掺入0.5%未被检出;从表4可看出骆驼毛纤维中掺入1.0%、5.0%、10.0%、20.0%、50.0%的绵羊毛纤维时均能被检出,掺入0.5% 未被检出;从表5可看出鹅毛纤维中掺入1.0%、5.0%、10.0%、20.0%、50.0%的鸭毛纤维时均能被检出,掺入0.5%未被检出。说明本方法检出限为1.0%。

A~H. 水貂、狐狸、兔、骆驼、山羊、绵羊、鹅和鸭A-H. Mink, fox, rabbit, camel, goat, sheep, goose and duck图5 8种动物毛纤维DNA灵敏度扩增曲线Fig.5 DNA sensitivity amplification curves of 8 animal hair fibers
3 讨 论
研究和开发动物毛纤维的DNA检测技术,关键技术难点在于动物毛纤维DNA的提取[18],动物毛发由毛干和毛根构成,虽然毛根带有毛囊DNA含量丰富,但是经过染色等初加工处理的动物纤维样本多缺失毛囊,而毛干主要由蛋白质组成,DNA含量极低,导致DNA提取与纯化困难,从而使得毛干这种极易取得的生物样品在分子生物学研究领域得不到有效利用[19]。根据文献报道[20-22],毛干DNA提取的主要方法包括经典的酚-氯仿抽提法、Chelex-100法、试剂盒法以及多种组合方法。本研究对3种动物毛囊及毛干基因组DNA提取方法进行了比较。发现优化后的方法2效果最好,该方法使用SDS-二硫苏糖醇DTT-蛋白酶K结合Chelex-100法提取DNA,并利用硅珠纯化DNA[23]。其DNA提取过程中使用的SDS碱裂解液中加入的二硫苏糖醇DTT及蛋白酶K能有效的将毛干全部裂解,Chelex-100是一种由苯乙烯、二乙烯苯共聚体组成的化学螯合树脂[24],可螯合多价离子,特别是对高价金属离子有很高的亲和力和螯合作用。在低离子强度、碱性及煮沸的条件下,使蛋白质变性,通过离心除去Chelex颗粒,使其结合的物质与DNA分离。本方法采用硅珠吸附法来纯化DNA,提取的核酸纯度高而且省时,更重要的是避免了酚类物质对操作人员的毒性伤害。
表3狐狸毛混合兔毛检测结果
Table3Thetestresultsoffoxfurmixedrabbithair
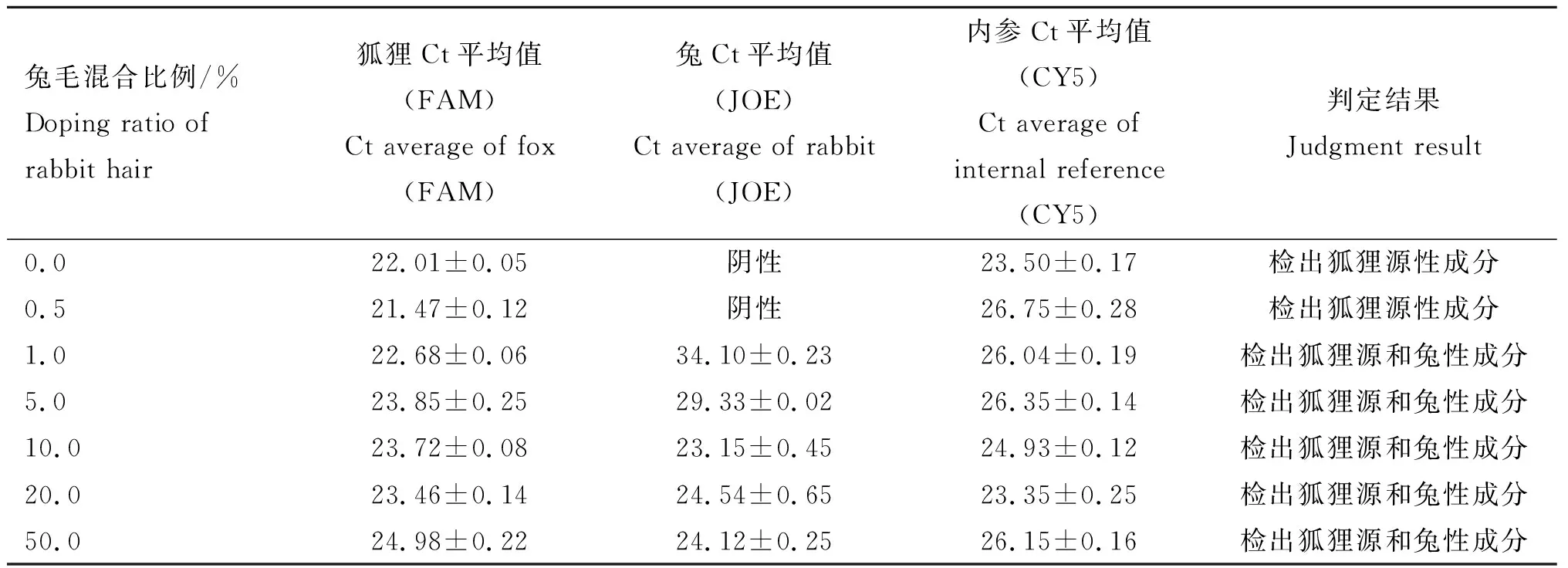
兔毛混合比例/%Doping ratio of rabbit hair狐狸Ct平均值(FAM)Ct average of fox(FAM)兔Ct平均值(JOE)Ct average of rabbit(JOE)内参Ct平均值(CY5)Ct average of internal reference(CY5)判定结果Judgment result0.022.01±0.05阴性23.50±0.17检出狐狸源性成分0.521.47±0.12阴性26.75±0.28检出狐狸源性成分1.022.68±0.0634.10±0.2326.04±0.19检出狐狸源和兔性成分5.023.85±0.2529.33±0.0226.35±0.14检出狐狸源和兔性成分10.023.72±0.0823.15±0.4524.93±0.12检出狐狸源和兔性成分20.023.46±0.1424.54±0.6523.35±0.25检出狐狸源和兔性成分50.024.98±0.2224.12±0.2526.15±0.16检出狐狸源和兔性成分
表4骆驼毛混合绵羊毛检测结果
Table4Thetestresultsofcamelwoolmixedwool
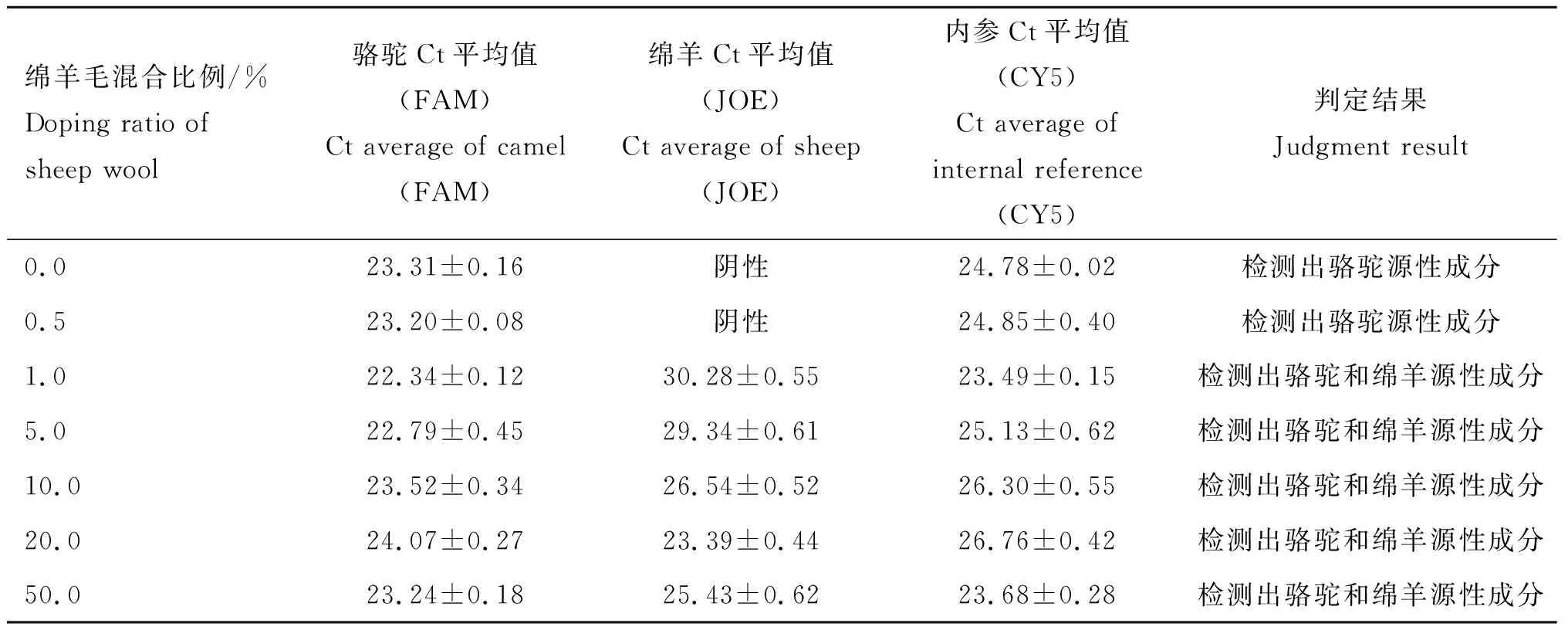
绵羊毛混合比例/%Doping ratio of sheep wool骆驼Ct平均值(FAM)Ct average of camel (FAM)绵羊Ct平均值(JOE)Ct average of sheep(JOE)内参Ct平均值(CY5)Ct average of internal reference(CY5)判定结果Judgment result0.023.31±0.16阴性24.78±0.02检测出骆驼源性成分0.523.20±0.08阴性24.85±0.40检测出骆驼源性成分1.022.34±0.1230.28±0.5523.49±0.15检测出骆驼和绵羊源性成分5.022.79±0.4529.34±0.6125.13±0.62检测出骆驼和绵羊源性成分10.023.52±0.3426.54±0.5226.30±0.55检测出骆驼和绵羊源性成分20.024.07±0.2723.39±0.4426.76±0.42检测出骆驼和绵羊源性成分50.023.24±0.1825.43±0.6223.68±0.28检测出骆驼和绵羊源性成分
表5鸭毛混合鹅毛检测结果
Table5Thetestresultsduckfeathersmixedgoosehair

鹅毛混合比例/%Doping ratio of goose feathers鸭Ct平均值(JOE)Ct average of duck(JOE)鹅Ct平均值(FAM)Ct average of goose(FAM)内参Ct平均值(CY5)Ct average of internal reference (CY5)判定结果Judgment result0.031.03±0.22阴性26.12±0.11检出鸭源性成分0.531.24±0.63阴性26.33±0.18检出鸭源性成分1.028.78±0.6633.83±0.3725.73±0.22检出鸭和鹅源性成分5.029.21±0.4231.41±0.4126.25±0.42检出鸭和鹅源性成分10.030.57±0.5632.35±0.8027.93±0.21检出鸭和鹅源性成分20.029.11±0.3230.36±0.0224.88±0.56检出鸭和鹅源性成分50.030.86±0.4430.84±0.0926.60±0.43检出鸭和鹅源性成分
动物毛发中的DNA本身含量较低,但在动物毛发中线粒体DNA比核DNA拷贝数要高很多,大量研究报道线粒体DNA D_loop区、COI基因、Cytb基因以及16SrDNA基因在物种间变异大,具有很高物种特异性,种内相对保守,适合于遗传多样性、种属鉴定和亲缘关系的研究[25-27]。本研究以线粒体16SrRNA基因为靶基因,设计8种动物的特异性探针及通用引物,显著提高了DNA扩增效率及物种特异性。通过优化PCR反应体系和条件,建立了同时对水貂、狐狸、兔、骆驼、山羊、绵羊、鸭和鹅8种动物毛纤维源性成分的特异性鉴定体系。通过特异性方法验证表明除以上8种动物能被检出外,其他物种源性均未被检出。通过梯度稀释DNA检测发现,本方法建立的8种源性动物多重实时荧光PCR检测体系扩增效率为90%~120%,未受到多重探针之间相互干扰的影响,而且DNA灵敏度检测为0.01 ng。通过常见的贵重毛纤维掺杂廉价的动物毛纤维以不同比例进行混合,模拟混合样本检测,结果证明本方法最低检出限为1.0%,说明本研究所建立的多重PCR检测方法具有很高的灵敏度。本方法针对动物毛纤维进行物种鉴别,可以避免因采血或取活检组织而对动物造成应激反应等生理伤害,尤其濒临灭绝的国家保护动物,为拓展动物毛纤维物种识别、分子进化和分子标记辅助育种、遗传资源分子等领域中的应用奠定了研究基础。
4 结 论
通过3种DNA提取方法的比较,采用硅珠吸附法可成功的提取动物毛干及毛囊中的DNA,省时、操作简便且提取纯度高,解决了传统检测方式因原材料DNA含量低导致检测繁琐的困难。另外,本研究所建立的8种动物毛纤维源性成分鉴定的多重实时荧光PCR体系特异性强,DNA检测灵敏度可达到0.010 ng·μL-1。因此,本方法可以快速、准确地鉴定貂、狐狸、山羊等各种高端动物毛纤维真伪,同时克服了感官检测和显微电镜检测的主观性误差,降低了工作经验的要求,为市场上毛皮的鉴定提供了有力技术支持。
